蜻蜓凤梨 AfERF113基因的克隆与表达特性
2017-01-04李志英王加宾
雷 明,李志英,王加宾,乔 飞,徐 立
(中国热带农业科学院 热带作物品种资源研究所,农业部华南作物基因资源与种质创新重点实验室,海南儋州 571737)
蜻蜓凤梨 AfERF113基因的克隆与表达特性
雷 明,李志英,王加宾,乔 飞,徐 立
(中国热带农业科学院 热带作物品种资源研究所,农业部华南作物基因资源与种质创新重点实验室,海南儋州 571737)
观赏凤梨是一类重要的热带花卉,但有关其生长发育调控分子机理的研究相对匮乏,这在一定程度上限制了新品种的开发和利用。以热带观赏花卉蜻蜓凤梨(Aechemiafasciata)为材料,在分析蜻蜓凤梨转录组数据的基础上,结合cDNA末端快速扩增(rapid amplification of cDNA ends,RACE)技术克隆了APETALA2/Ethylene Response Element Binding Protein(AP2/EREBP)家族的1个转录因子编码序列,并将其命名为AfERF113。生物信息学分析表明,AfERF113cDNA全长1 093 bp,有1个864 bp的开放阅读框(open reading frame,ORF),编码287个氨基酸。蛋白质理化性质分析表明,该蛋白分子质量为29.768 5 ku,理论等电点为6.06,为一类不稳定的酸性亲水蛋白。亚细胞定位软件预测表明该蛋白定位于细胞核。结构域分析表明,该蛋白有1个保守的AP2结构域,分属ERF亚族的B-4类,与拟南芥中所有ERF蛋白的ERF113亲源关系最近。实时荧光定量PCR(quantitative real time PCR,qRT-PCR)结果表明,AfERF113转录本在成熟株的根中表达量最高,且外源乙烯诱导后,AfERF113的表达量无论是在幼株还是在成株的各组织中均显著上调。研究结果证实,AfERF113响应了外源乙烯调控,为进一步研究AfERF113基因的功能及通过基因工程手段调控凤梨科植物生长发育提供了理论依据。
蜻蜓凤梨;AP2/EREBP家族;AfERF113;乙烯;表达分析
凤梨科(Bromeliaceae)植物原产于南美,包括58个属3 248个种,是一类广泛分布于热带亚热带地区形态最为多样的种属之一[1-2]。凤梨科植物的一些品种如食用凤梨(菠萝Ananascomosus)在6 000多年前就已经被驯化[3-4]。除了菠萝外,人工栽培的凤梨科植物还包括一类重要的热带花卉,即观赏凤梨。观赏凤梨进入人们的生活不过百余年历史,传入中国则在20世纪90年代中期,但因其形态多样、花期持久,市场发展突飞猛进,现已成为仅次于兰花和红掌的第3大热带花卉,效益超过了菠萝[5]。
作为一种重要的植物激素,乙烯参与种子萌发、幼苗生长、开花诱导、果实成熟、器官衰老和病原菌响应等植物生长发育的诸多过程,是植物体应答很多生物和非生物胁迫的关键因子[6-7]。拟南芥中,目前已知响应乙烯信号的受体蛋白有5个,分别为ETHYLENE RESPONSE1(ETR1)、ETR2、ETHYLENE RESPONSE SENSOR1(ERS1)、ERS2和ETHYLENEINSENSITIVE4(EIN4)[8-9]。乙烯受体蛋白结合乙烯后抑制了Raf-like Ser/Thr 蛋白酶 CONSTITUTIVE TRIPLE RESPONSE1(CTR1)的磷酸化能力,使得膜蛋白ETHYLENE INSENSITIVE2(EIN2)被激活[10-11]。活化的EIN2稳定了转录因子EIN3在核内的表达,EIN3蛋白则通过结合到ERFs基因(如ERF1)的启动子区,调控后者的表达,最终启动了植物对各种信号的应答响应[12]。
ERFs属于APETALA2/Ethylene Response Element Binding Protein(AP2/EREBP)家族。依据所含AP2结构域以及序列同源性的差异,AP2/EREBP家族又可以分为5个亚族:AP2亚族、干旱响应元件结合蛋白亚族(Dehydration Responsive Element Binding Protein,DREB)、乙烯响应元件结合蛋白亚族ERF、Related to ABSCISIC ACID INSENSITIVE3 (ABI3)/VIVIPAROUS1 (VP1)(RAV)亚族以及其他蛋白类[13]。其中,ERF亚族含有1个AP2结构域。根据进化树及同源性分析结果,ERF亚族蛋白可以分为B-1到B-6共6类,这6类大部分都是转录激活因子,但有些含有ERF相关双亲抑制(ERF-associated amphiphilic repression ,EAR)基序的B-1类蛋白则是转录抑制因子[14-19]。ERFs转录因子主要通过结合到靶标基因启动子区的核心AGCCGCC(GCC box)顺式元件上调控靶标基因的表达,应答细菌、真菌或病毒病原体等造成的生物胁迫[20-22],也有一些ERFs可以通过识别干旱响应元件(dehydration-responsive elements,DREs/CRT)应答非生物胁迫[23]。另外,一些ERFs转录因子则可以同时识别GCC box和DREs/CRT元件调控靶标基因的表达[24]。
目前,国内外有关凤梨科植物AP2/EREBP转录因子的研究很少。Lei等[25]报道了蜻蜓凤梨(Aechemiafasciata)中的1个AP2亚族的基因AfAP2-1的转录本表达响应了外源乙烯处理,且在拟南芥中过表达显著延迟了开花。前期研究中,通过乙烯处理和未处理的蜻蜓凤梨转录组数据比较分析发现,1个AP2家族的基因在乙烯处理24 h后的蜻蜓凤梨成株心叶中表达量显著上升(未发表),基于此,通过RACE技术克隆该基因的全长,并将其命名为AfERF113。此外,利用生物信息学方法对其编码的蛋白进行结构和功能分析,并通过转录水平的表达分析解析其组织表达特异性,同时发现其的确受外源乙烯处理的调控。研究为进一步解析AfERF113基因响应外源乙烯或其他胁迫因素的分子机制及通过基因工程手段调控凤梨科植物生长发育提供了理论依据。
1 材料与方法
1.1 材料与处理
蜻蜓凤梨取自中国热带农业科学院热带作物品种资源研究所离体保存与繁育研究室大棚,环境温度为30~32 ℃。所用材料均为移栽成活的试管苗,幼株为成活后株龄6个月植株;成株为成活后株龄11~14个月的植株。外源乙烯利处理时,取10 mL体积分数为400 μL·L-1的乙烯利灌心,分别处理1、6、24 h,以清水灌心的材料为对照。不同组织材料在处理后迅速于液氮中冷冻,之后置于-80 ℃保存,备用。
1.2 总RNA提取及cDNA第1条链的合成
蜻蜓凤梨总RNA的提取采用改良的CTAB法[26]。cDNA第1条链的合成采用TranScript-Uni One-Step gDNA Removal and cDNA Synthesis SuperMix试剂盒(Transgene),具体过程按照试剂盒说明书进行。
1.3 5′和3′ RACE
前期转录组数据获得了414 bp的基因片段。为了获得该基因的全长,首先参照SMARTerTM RACE cDNA Amplification Kit(Clontech)的说明书合成了5′和3′ RACE模板。根据已有的414 bp的序列,分别设计了5′和3′特异引物进行后续扩增。其中,5′的外侧和内侧引物分别为5′ RACE GSP51和5′ RACE GSP52,3′的外侧和内侧引物分别为3′ RACE GSP31和3′ RACE GSP32。引物序列见表1。PCR扩增得到的特异性条带采用OMEGA(Omega)的胶回收试剂盒进行。纯化后的特异条带分别连接到pEASY-blunt克隆载体上,测序。测序后的条带与转录组序列拼接后再做进一步验证分析。
1.4 生物信息学分析
利用NCBI网站的ORF Finder在线软件(http://www.ncbi.nlm.nih.gov/projects/gorf/)进行开放阅读框的预测分析;利用NCBI网站的BLAST在线软件(http://blast.ncbi.nlm.nih.gov/Blast.cgi)进行核苷酸和氨基酸的序列比对与分析;利用DNAMAN 6.0软件进行氨基酸序列的同源性比对;利用MEGA 6.0软件和邻接法进行进化树构建;利用ProtParam在线软件(http://web.expasy.org/protparam/)分析蛋白质分子质量、等电点、疏水性及稳定性;利用TMHMM在线软件(http://www.cbs.dtu.dk/services/TMHMM/)对蛋白质的跨膜区进行预测;利用SignalP 4.1在线软件(http://www.cbs.dtu.dk/services/SignalP/)对蛋白质的信号肽进行预测;利用Cell-PLoc package(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc/)、WOLF Psort(http://www.genscript.com/wolf-psort.html)和ProtComp(http://www.softberry.com/berry.phtml?topic=protcomppl& group=programs&subgroup=proloc)在线软件预测蛋白的亚细胞定位;利用Phyre2软件(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)预测蛋白质三维结构。
1.5 实时荧光定量PCR(qRT-PCR)
运用Primer premier 5.0 设计用于实时荧光定量PCR的各基因特异引物。选用TransStart Tip Green qPCR SuperMix(Transgene)进行PCR反应,在Therma PikoReal 96TM荧光定量PCR仪(Thermo Fisher Scientific)上进行。反应体系(10 μL):qRT-PCR混合反应液SuperMix 5 μL,模板1 μL,正反向引物各0.3 μL,灭菌的去离子水补齐至10 μL;反应程序:95 ℃预变性7 min,然后在95 ℃ 5 s、60 ℃ 30 s的条件下扩增40个循环,循环结束后65 ℃ 30 s,20 ℃停止。每个样品2个生物学重复,3次技术重复。数据分析采用双delta法,选取蜻蜓凤梨的β-actin(AfACTB)为内参。通过制作标准曲线获得目的基因和内参基因的扩增效率。所有引物序列见表1。
2 结果与分析
2.1AfERF113cDNA全长的克隆与序列分析
以蜻蜓凤梨心叶cDNA为模板,利用RACE及PCR技术获得1条长度为1 093 bp的AP2/EREBP家族基因的cDNA序列。分析后发现,其5′非编码区(5′ untranscriptional region,5′-UTR)长度为114 bp,3′非编码区(3′ untranscriptional region,3′-UTR)长度为115 bp(包括25 bp的polyA),开放阅读框(open reading frame,ORF)长度为864 bp,编码287个氨基酸残基。将其在NCBI网站上进行BLAST比对,发现其与很多植物中AP2/EREBP家族ERF亚族的ERF113或ERF113-like基因具有很高的相似性,故将其命名为AfERF113(Genbank登录号:KX371269)。
2.2 AfERF113蛋白的同源性和进化树分析
序列比对分析表明,AfERF113与油棕(Elaeisguineensis)的ERF113、海枣(Phoenixdactylifera)的ERF113、荷花(Nelumbonucifera)的ERF113和小果野芭蕉(Musaacuminatasubsp.malaccensis)的ERF114分别具有51.92%、48.43%、40.46%和39.57%的同源性(图1)。结构域分析发现,AfERF113具有1个典型的AP2结构域,包含保守的YPG和RAYD元件(图1)。此外,该保守的AP2结构域的第14位和第19位氨基酸残基分别为丙氨酸(Ala,A)和天冬氨酸(Asp,D)(图1),因此,AfERF113属于AP2/EREBP家族的ERF亚族。
为了进一步分析AfERF113在ERF亚族中的类别,将其与拟南芥(Arabidopsisthaliana)ERF亚族B1~B6类别中的部分蛋白序列进行进化树分析,结果发现AfERF113与拟南芥的ERF112、ERF113、ERF114、ERF115等进化关系最近,同属于B4类(图2)。
2.3AfERF113的理化性质和空间结构
利用ProtoParam在线软件分析AfERF113后发现,该蛋白质分子质量为29.768 5 ku,理论等电点为6.06,总平均亲水性(GRAVY)为-0.460,不稳定系数为60.51,因此推测AfERF113是一类不稳定的酸性亲水蛋白。利用TMHMM在线软件对AfERF113进行分析后发现,该蛋白位于跨膜螺旋中的氨基酸残基数为0.003 62,远小于18;此外,N端60个氨基酸残基中位于跨膜螺旋中的氨基酸残基数为0.000 82,远小于10,推测该蛋白不含跨膜区(图3-A),这与ProtoParam预测其为亲水性蛋白的结果一致。进一步使用SignalP 4.1在线软件对AfERF113进行了信号肽预测,结果表明该蛋白的N端70个氨基酸残基中并不含信号肽(图3-B),推测该蛋白不属于分泌蛋白。为了验证该蛋白的亚细胞定位,分别使用Cell-PLoc package、WOLF Psort和ProtComp软件分析对其进行分析。3个不同的软件分析结果都预测该蛋白定位于细胞核,且ProtComp软件预测其为转录因子。

表1 本研究所用的引物

AfERF113. 蜻蜓凤梨(Aechemia fasciata) ERF113;EgERF113.油棕(Elaeis guineensis)ERF113,XP_010929690.1;NnER113.荷花(Nelumbo nucifera)ERF113,XP_010249458.1;MaERF114. 小果野芭蕉(Musa acuminata subsp.)ERF114,XP_009405112.1;PdERF113.海枣(Phoenix dactylifera)ERF113,XP_008791015.1
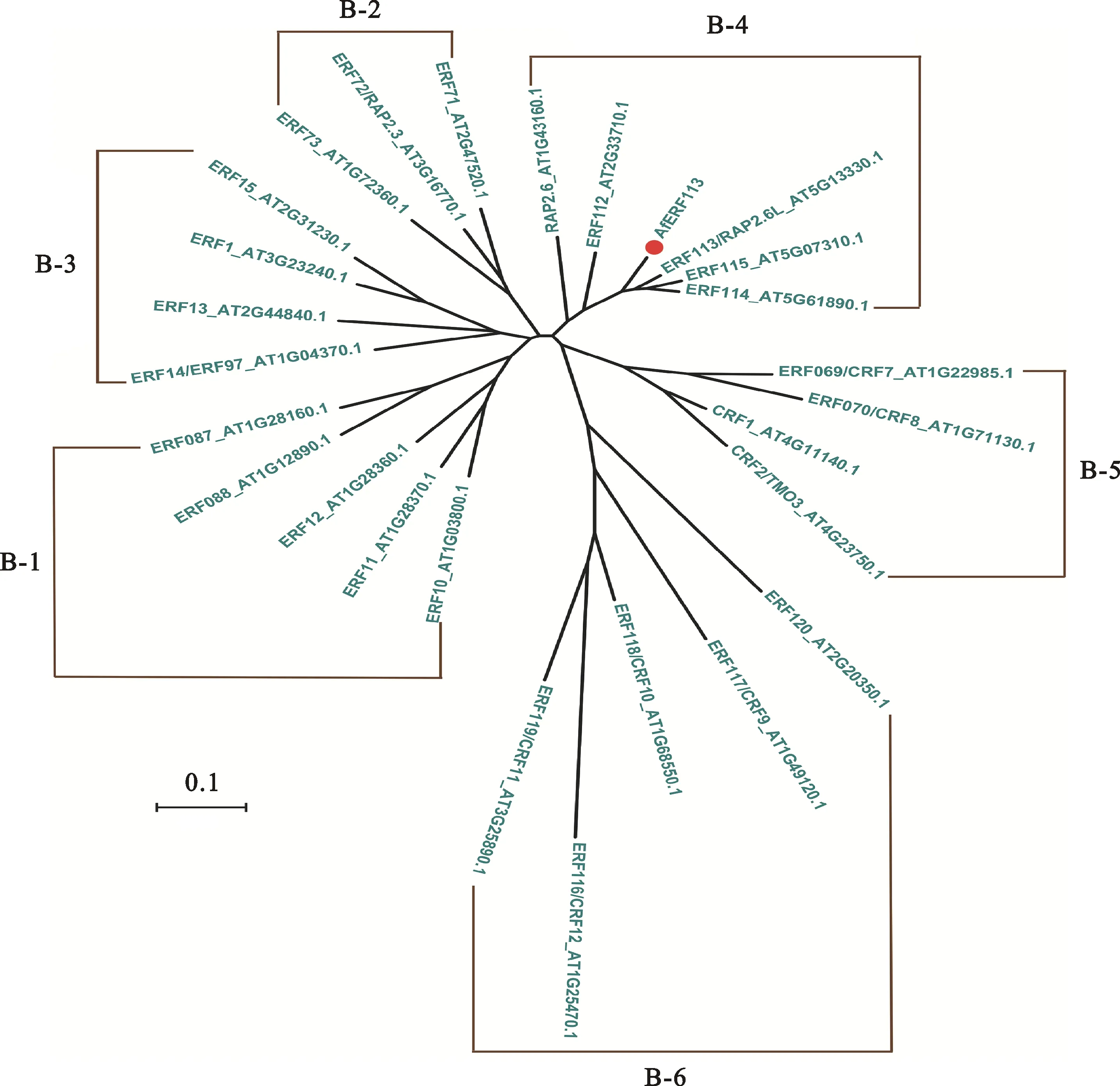
图2 AfERF113的分子进化树构建

A.AfERF113的信号肽分析 The analysis of signal peptide of AfERF113;B.AfERF113的跨膜区分析 The analysis of transmembrane region of AfERF113
为了更进一步明晰AfERF113蛋白的结构特点,利用Phyre2软件对其进行三维建模。以拟南芥ERF1蛋白(PDB ID:1gcc.1.C)为模板,获得AfERF113蛋白保守结构域91-152 氨基酸肽段区域的三维结构模型(图4-A,4-B)。从该模型中可以看到, AfERF113的该保守结构域主要含有3个β折叠和1个α螺旋,其中2个β折叠由一级结构中的YPG元件构成,另一个β折叠和α螺旋主要由RAYD元件构成,这与很多ERF蛋白核心区域的构象类似,表明该模拟构象合理,空间结构稳定。
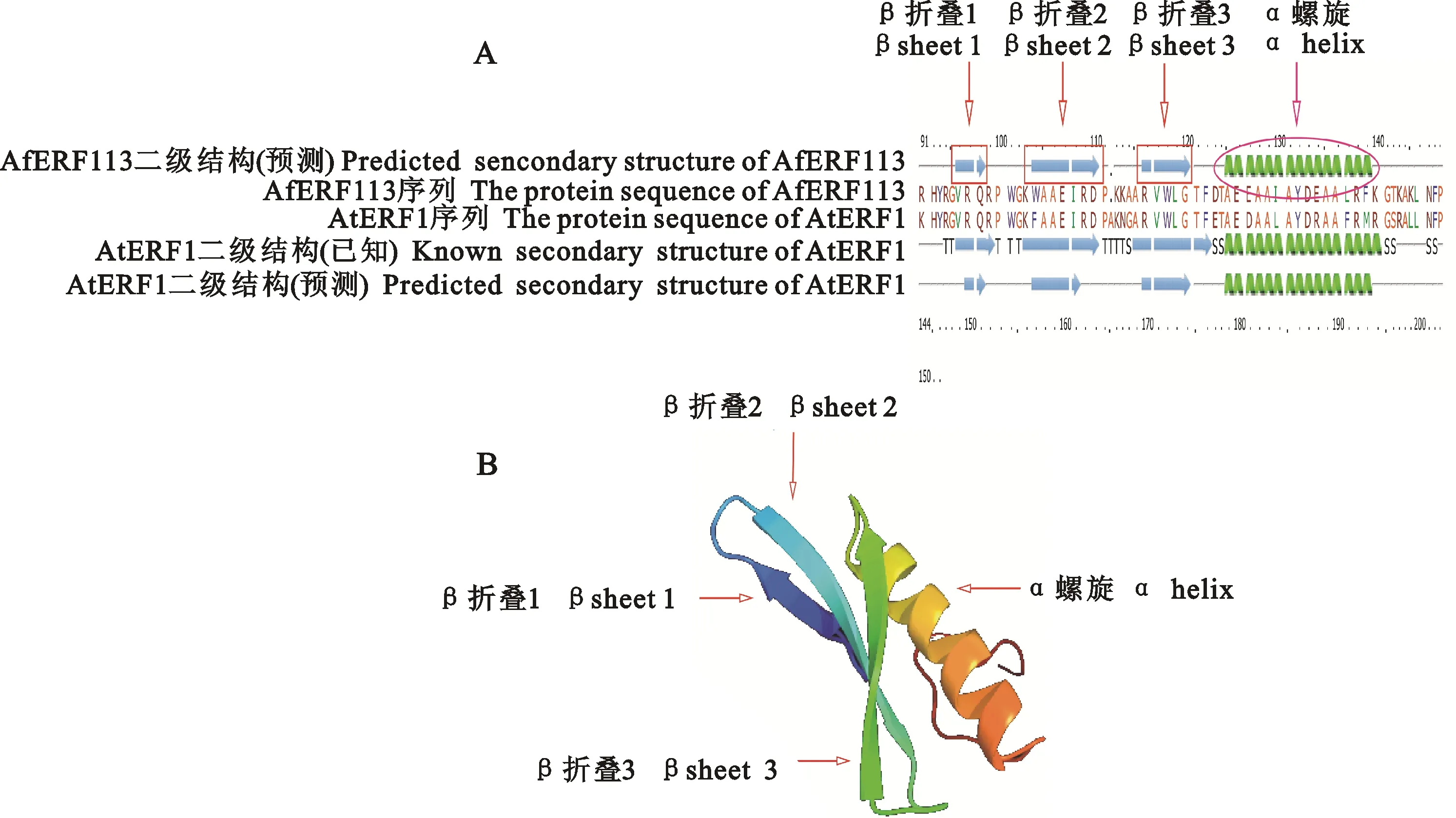
A.二级结构 The second structure;B.三维模型 The three-dimension structure
2.4 AfERF113的组织表达特性
为了研究AfERF113在蜻蜓凤梨各组织中的表达特性,分别选取幼株、成株和抽薹39 d后成株的外叶、心叶、茎和根,提取RNA,反转录成cDNA,以此为模板,利用qRT-PCR技术对其进行验证。结果显示,AfERF113转录本在幼株各组织以及未开花的成熟株和抽薹39 d后的成熟株的外叶、心叶和茎中的表达量相对较低,但在未开花的成熟株和抽薹39 d后的成熟株根中表达量则显著升高(图5-A)。此外,AfERF113转录本在花器官中的表达量均很低(图5-B)。
2.5AfERF113对外源乙烯处理的响应特性
为了验证AfERF113对外源乙烯处理的响应情况,分别用10 mL体积分数为400 μL·L-1的乙烯利对蜻蜓凤梨幼株和成株的整株进行灌心处理,然后于处理后的1、6、24 h分别对幼株和成株的外叶、心叶、茎和根中AfERF113转录本的表达情况进行检测。结果显示,在幼株中,乙烯处理1 h后AfERF113在各检测组织中的表达量显著上调(图6-A);在成株中,乙烯处理1 h后AfERF113在外叶和心叶中的表达量上调显著,在茎中的表达量则随着外源乙烯处理时间的延长稳步上升,而在根中的表达量则在乙烯处理后呈现先上升后下降的趋势(图6-B)。这些结果表明,在蜻蜓凤梨的幼株和成株中,AfERF113在不同的组织中可能具有不尽相同的功能。
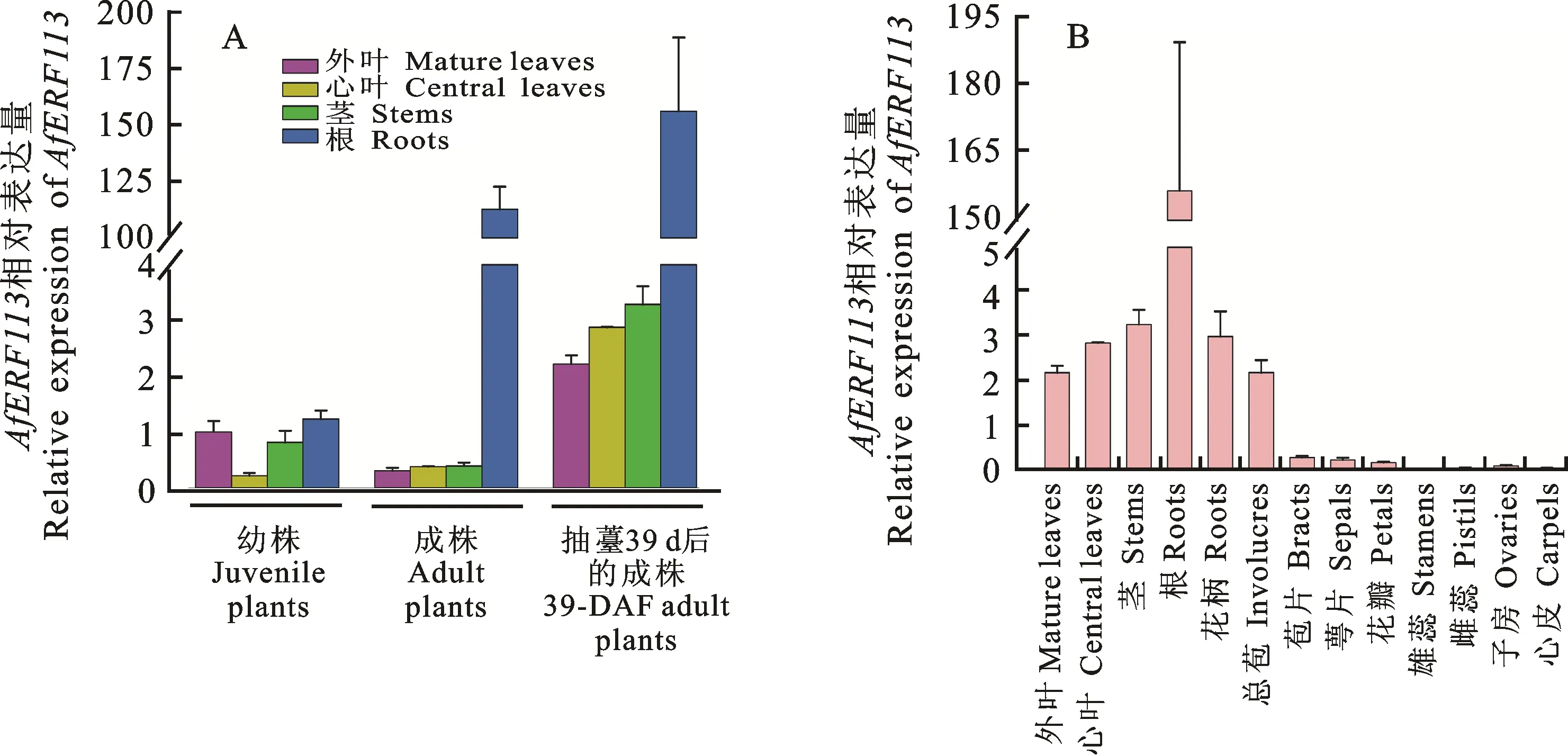
A. AfERF113在蜻蜓凤梨幼株、成株和抽薹39 d后的成株各组织中的相对表达量 Relative expression of AfERF113 in variable tissues of juvenile plants, adult plants before flowering, and 39-day after flowering (DAF) adult plants;B. AfERF113在抽薹39 d后的成株营养器官和生殖器官中的相对表达量 Relative expression of AfERF113 in vegetative and reproductive organs of 39-DAF adult plants
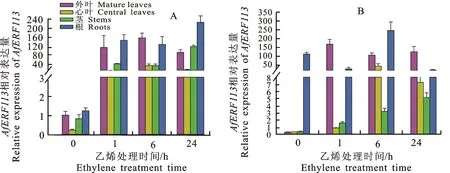
A. AfERF113在乙烯处理后幼株各组织中的相对表达量 Relative expression of AfERF113 transcripts in variable tissues of juvenile plants treated with ethylene;B. AfERF113在乙烯处理后成株各组织中的相对表达量 Relative expression of AfERF113 transcripts in variable tissues of adult plants treated with ethylene

A. AfERF113在乙烯处理后幼株各组织中的相对表达量 Relative expression of AfERF113 transcripts in variable tissues of juvenile plants treated with ethylene;B. AfERF113在乙烯处理后成株各组织中的相对表达量 Relative expression of AfERF113 transcripts in variable tissues of adult plants treated with ethylene
3 讨 论
自从AP2转录因子和ERFBP转录因子分别首次于1994年和1995年从拟南芥和烟草中被分离鉴定出以来[20, 27],现已发现AP2/EREBP转录因子广泛存在于植物、原生生物、蓝藻和真菌中[28-29]。本研究首次从重要的热带花卉蜻蜓凤梨中克隆到AP2/EREBP转录因子ERF亚族的一个基因,根据BLAST同源比对结果,将其命名为AfERF113。
AfERF113具有一个非常典型的AP2结构域,该结构域在一级结构上含有2个特别保守的氨基酸序列(图1-A, 1-B)。其中,YPG元件在二级结构上可以形成2个β折叠,RAYD元件可以形成1个β折叠和1个α螺旋(图4)。在模拟的三级结构上, AfERF113的AP2结构域与拟南芥的ERF1(PDB ID:1gcc.1.C)非常相似(图4)。已知这3个反向平行的β折叠在帮助AP2结构域识别各种顺式作用元件中可能起重要作用[30],而两亲性的α螺旋则参与了DNA或其他转录因子之间的相互作用[31]。此外, AfERF113的AP2结构域中第14位和第19位分别是保守的丙氨酸(Ala,A)和天冬氨酸(Asp,D),区别于大部分DREB家族的缬氨酸(Val14,V14)和谷氨酸(Glu19,E19)[13]。 AfERF113在蛋白结构上的保守性暗示其在功能上也可能具有一些保守性。
3.1 AfERF113的转录本具有组织表达特异性
基因表达模式的分析是进一步研究生物学功能的基础[32]。研究结果显示,ERF113同源基因在不同物种中的组织特异性表达既有共性,又有差别。拟南芥的ERF113(RAP2.6L)在成熟干燥的种子、花(尤其是雄蕊)、子叶下胚轴、成熟的根以及发育中的木质部均高表达[33-34];杨树(Populus)的一个RAP2.6L同源基因则在雌雄花、木质部和根中有强表达[35];黑胡桃(JuglansnigraL.)的一个RAP2.6L同源基因JnRAP2-like则在雌花中表达量最高,而在根中其次,在雄花中则检测不到其表达[36]。本研究结果显示,在蜻蜓凤梨幼株的外叶、心叶、茎和根中,AfERF113的表达量均较低(图5-A)。但是随着植株年龄的增长,AfERF113在成株根中的表达量显著上升,为幼株根中表达量的126倍(图5-B),这暗示AfERF113可能调控蜻蜓凤梨根的生长发育。另外,AfERF113在蜻蜓凤梨花器官中的表达量均维持在相对较低的水平,与拟南芥、杨树和黑胡桃的RAP2.6L同源基因的表达模式不尽相同,说明蜻蜓凤梨中的AfERF113可能进化出新的功能。
3.2AfERF113转录本的表达响应了外源乙烯处理
植物生长发育同时受外源环境信号和内源信号的调控,其中,重要的内源信号则包括植物激素,比如乙烯[37]。乙烯进化为植物激素已有4.5亿年的历史[38]。乙烯调控了种子发育、幼苗生长、叶子和花瓣的脱落、果实成熟、器官衰老和病虫害响应等多种植物生长发育的过程[7]。研究发现,很多物种的ERF113及其同源基因响应了乙烯处理。拟南芥RAP2.6L转录本在乙烯处理6~24 h逐渐上升,过表达RAP2.6L的拟南芥植株提高了对乙烯处理的抗性[39]。柑橘(Citrus)的一个RAP2.6L同源基因在乙烯调控的叶片衰老中上调[40]。黑胡桃的JnRAP2.6-like在拟南芥中过表达后参与了木质部等组织中乙烯和茉莉酸协同调控路径[36]。本研究发现,外源乙烯利处理1 h后,蜻蜓凤梨幼株各组织中AfERF113的转录本的表达量均显著上升(图6-A);成株中,外叶、心叶和茎中AfERF113的转录本达到最大表达量所用的时间分别为1、6、24 h,而在根中的表达量则在处理24 h后下调了(图6-B)。AfERF113在幼株和成株各组织中受外源乙烯处理诱导表达的差异性表明:首先,蜻蜓凤梨的株龄影响了乙烯对AfERF113的调控;其次,组织特异性造成AfERF113的转录本表达量受乙烯诱导后达到最大值时出现了时间差。已知ERF家族中拟南芥的ERF1在转录水平上直接受乙烯信号路径的EIN3调控[12],因此,通过克隆AfERF113的基因组和启动子序列,找到并验证是否存在乙烯响应元件,同时通过体内外方法寻找不同组织中与AfERF113DNA或蛋白发生相互作用的因子,有望为更好地解析AfERF113在不同株龄及不同组织中对乙烯诱导响应差异的机制。
已知拟南芥的RAP2.6L参与了从根外植体再生出芽的调控[41]、病害防疫[42]及髓细胞分化和器官再生[43]。未来通过转基因结合高低温胁迫、盐胁迫及其他激素胁迫处理的方法进一步探究AfERF113基因在蜻蜓凤梨生长发育调控中的基本功能,无疑可以为通过基因工程手段调控蜻蜓凤梨乃至凤梨科植物生长发育提供理论依据。
Reference:
[1] LUTHER H E.An Alphabetical List of Bromeliad Binomials[M].Florida:The Bromeliad Society International,2010:4.
[2] VERSIEUX L M,BARBARA T,WANDERLEY M,etal.Molecular phylogenetics of the Brazilian giant bromeliads(Alcantarea,Bromeliaceae):implications for morphological evolution and biogeography [J].MolecularPhylogeneticEvolution,2012,64(1):177-189.
[3] CLEMENT C R,DE CRISTO-ARAJO M,dEECKENBRUGGE G C,etal.Origin and domestication of native amazonian crops[J].Diversity,2010,2(1):72-106.
[4] MING R,VAN BUREN R,WAI C M,etal.The pineapple genome and the evolution of CAM photosynthesis [J].NatureGenetics,2015,47(12):1435-1442.
[5] 李志英.凤梨科植物组织培养技术[M].北京:中国农业出版社,2014:1.
LI ZH Y.Tissue Culture Technology of Bromeliads [M].Beijing:Chinese Agricultural Press,2014:1(in Chinese).
[6] MLLER M,MUNN-BOSCH S.Ethylene response factors:a key regulatory hub in hormone and stress signaling [J].PlantPhysiology,2015,169:32-41.
[7] SHAKEEL S N,GAO Z,AMIR M,etal.Ethylene regulates levels of ethylene receptor-CTR1 signaling complexes inArabidopsisthaliana[J].JournalofBiologicalChemistry,2015,290(19):12415-12424.
[8] BLEECKER A B.Ethylene perception and signaling:an evolutionary perspective [J].TrendsinPlantScience,1999,4(7):269-274.
[9] CHANG C,STADLER R.Ethylene hormone receptor action inArabidopsis[J].BioEssays:NewsandReviewsinMolecular,CellularandDevelopmentalBiology,2001,23(7):619-627.
[10] KKEBER J J,ROTHENBERG M,ROMAN G,etal.CTR1,a negative regulator of the ethylene response pathway inArabidopsis,encodes a member of the raf family of protein kinases [J].Cell,1993,72(3):427-441.
[11] ALONSO J M,HIRAYAMA T,ROMAN G,etal.EIN2,a bifunctional transducer of ethylene and stress responses inArabidopsis[J].Science,1999,284(5423):2148-2152.
[12] SOLANO R,STEPANOVA A,Chao Q,etal.Nuclear events in ethylene signaling:a transcriptional cascade mediated by ETHYLENE-INSENSITIVE3 and ETHYLENE-RESPONSE-FACTOR1 [J].Genes&Development,1998,12(23):3703-3714.
[13] SAKUMA Y,LIU Q,DUBOUZET J G,etal.DNA-binding specificity of the ERF/AP2 domain ofArabidopsisDREBs,transcription factors involved in dehydration- and cold-inducible gene expression [J].BiochemicalandBiophysicalResearchCommunications,2002,290(3):998-1009.
[14] FUJIMOTO S Y,OHTA M,USUI A,etal.Arabidopsisethylene-responsive element binding factors act as transcriptional activators or repressors of GCC box-mediated gene expression [J].ThePlantCell,2000,12:393-404.
[15] OHTA M,MATSUI K,HIRATSU K,etal.Repression domains of class II ERF transcriptional repressors share an essential motif for active repression [J].ThePlantCell,2001,13(8):1959-1968.
[16] SONG C P,AGARWAL M,OHTA M,etal.Role of anArabidopsisAP2/EREBP-type transcriptional repressor in abscisic acid and drought stress responses [J].ThePlantCell,2005,17(8):2384-2396.
[17] ZHANG G,CHEN M,CHEN X,etal.Isolation and characterization of a novel EAR-motif-containing gene GmERF4 from soybean(GlycinemaxL.) [J].MolecularBiologyReports,2009,37(2):809-818.
[18] ZHANG G,CHEN M,LI L,etal.Overexpression of the soybeanGmERF3gene,an AP2/ERF type transcription factor for increased tolerances to salt,drought,and diseases in transgenic tobacco [J].JournalofExperimentalBotany,2009,60(13):3781-3796.
[19] ZHAI Y,LI J W,LI X W,etal.Isolation and characterization of a novel transcriptional repressor GmERF6 from soybean [J].BiologiaPlantarum,2012,57(1):26-32.
[20] OHME-TAKAGI M,SHINSHI H.Ethylene-inducible DNA binding proteins that interact with an ethylene-responsive element [J].ThePlantCell,1995(7):173-182.
[21] YI S Y,KIM J H,JOUNG Y H,etal.The pepper transcription factor CaPF1 confers pathogen and freezing tolerance inArabidopsis[J].PlantPhysiology,2004,136(1):2862-2874.
[22] ZUO K J,QIN J,ZHAO J Y,LING H,etal.Over-expression GbERF2 transcription factor in tobacco enhances brown spots disease resistance by activating expression of downstream genes [J].Gene,2007,391(1/2):80-90.
[23] MCGRATH K C.Repressor and activator-type ethylene response factors functioning in jasmonate signaling and disease resistance identified via a genome-wide screen ofArabidopsistranscription factor gene expression [J].PlantPhysiology,2005,139(2):949-959.
[24] CHENG M C,LIAO P M,KUO W W,etal.TheArabidopsisETHYLENE RESPONSE FACTOR1 regulates abiotic stress-responsive gene expression by binding to different cis-acting elements in response to different stress signals [J].PlantPhysiology,2013,162(3):1566-1582.
[25] LEI M,LI Z Y,WANG J B,etal.AfAP2-1,an age-dependent gene ofAechmeafasciata,responds to exogenous ethylene treatment [J].InternationalJournalofMolecularSciences,2016,17(3):303.
[26] 丛汉卿,信彩云,张银东,等.‘阿蒂晴天’凤梨谷胱甘肽-S-转移酶基因的克隆与乙烯诱导表达特性的初步分析[J].分子植物育种,2013,11(3):365-370.
CONG H Q,XIN C Y,ZHANG Y D,etal.Cloning of glutathione-S-transferase gene and primary expression analysis inGuzmaniawittmackii‘Attila’ induced by ethylene [J].MolecularPlantBreeding,2013,11(3):365-370(in Chinese with English abstract).
[27] JOFUKU K D,DEN BOER B G,VAN MONTAGU M,etal.Control ofArabidopsisflower and seed development by the homeotic geneAPETALA2[J].ThePlantCell,1994,6(9):1211-1225.
[28] WESSLER S R.Homing into the origin of the AP2 DNA binding domain [J].TrendsinPlantScience,2005,10(2):54-56.
[29] LICAUSI F,OHME-TAKAGI M,PERATA P.APETALA2/Ethylene responsive factor(AP2/ERF) transcription factors:mediators of stress responses and developmental programs [J].TheNewPhytologist,2013,199(3):639-649.
[30] LIU Y,ZHAO T J,LIU J M,etal.The conserved Ala37 in the ERF/AP2 domain is essential for binding with the DRE element and the GCC box [J].FEBSLetters,2006,580(5):1303-1308.
[31] OKAMURO J K,CASTER B,VILLARROEL,R,etal.The AP2 domain of APETALA2 defines a large new family of DNA binding proteins inArabidopsis[J].ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica,1997,94(13):7076-7081.
[32] NAKANO T,SUZUKI K,OHTSUKI N,etal.Identification of genes of the plant-specific transcription-factor families cooperatively regulated by ethylene and jasmonate inArabidopsisthaliana[J].JournalofPlantResearch,2006,119(4):407-413.
[33] WINTER D,VINEGAR B,NAHAL H,etal.An “Electronic Fluorescent Pictograph” browser for exploring and analyzing large-scale biological data sets [J].PLoSONE,2007,2(8):718.
[34] CAI S,LASHBROOK C C.Stamen abscission zone transcriptome profiling reveals new candidates for abscission control:enhanced retention of floral organs in transgenic plants overexpressingArabidopsisZINC FINGER PROTEIN2 [J].PlantPhysiology,2008,146(3):1305-1321.
[35] WILKINS O,NAHAL H,FOONG J,etal.Expansion and diversification of the populus R2R3-MYB family of transcription factors [J].PlantPhysiology,2009,149(2):981-993.
[36] HUANG Z,ZHAO P,MEDINA J,etal.Roles ofJnRAP2.6-likefrom the transition zone of black walnut in hormone signaling [J].PLoSONE,2013,8(11):75857.
[37] ZDARSKA M,DOBISOVA T,GELOVA Z,etal.Illuminating light,cytokinin,and ethylene signalling crosstalk in plant development [J].JournalofExperimentalBotany,2015,66(16):4913-4931.
[38] JU C,VAN DE POEL B,COOPER E D,etal.Conservation of ethylene as a plant hormone over 450 million years of evolution [J].NaturePlants,2015,1(1):14004.
[39] KRISHNASWAMY S,VERMA S,RAHMAN M H,etal.Functional characterization of four APETALA2-family genes(RAP2.6,RAP2.6L,DREB19 and DREB26) inArabidopsis[J].PlantMolecularBiology,2011,75(1/2):107-127.
[40] AGUSTI J,MERELO P,CERCOS M,etal.Comparative transcriptional survey between laser-microdissected cells from laminar abscission zone and petiolar cortical tissue during ethylene-promoted abscission in citrus leaves [J].BMCPlantBiology,2009,9:127.
[41] CHE P,LALL S,NETTLETON D,etal.Gene expression programs during shoot,root,and callus development inArabidopsistissue culture [J].PlantPhysiology,2006,141(2):620-637.
[42] SUN F,LIU P,XU J,etal.Mutation in RAP2.6L,a transactivator of the ERF transcription factor family,enhancesArabidopsisresistance toPseudomonassyringae[J].PhysiologicalandMolecularPlantPathology,2010,74(5/6):295-302.
[43] ASAHINA M,AZUMA K,PITAKSARINGKARN W,etal.Spatially selective hormonal control of RAP2.6L and ANAC071 transcription factors involved in tissue reunion inArabidopsis[J].ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica,2011,108:1128-16132.
(责任编辑:潘学燕 Responsible editor:PAN Xueyan)
Cloning and Expression Analysis ofAfERF113inAechemiafasciata
LEI Ming,LI Zhiying,WANG Jiabin, QIAO Fei and XU Li
(Ministry of Agriculture Key Laboratory of Crop Gene Resources and Germplasm Enhancement in Southern China, Institute of Tropical Crop Genetic Resources, Chinese Academy of Tropical
Agricultural Sciences, Danzhou Hainan 571737, China)
Cultivated bromeliads are important tropical ornamental flowering plants. However, it is little known about its molecular mechanism of growth and development regulation, and which in turn becomes a bottleneck for breeding and exploiting of new varieties. With the ornamental bromeliadsAechemiafasciataas material, based on its transcriptome data, a member of APETALA2/Ethylene Response Element Binding Protein (AP2/EREBP) was isolated by using rapid amplification of cDNA ends (RACE) technology and was named asAfERF113. The length ofAfERF113cDNA was 1 093 bp, with an 864 bp open reading frame (ORF), which encodes a 287-amino acids deduced protein. The protein is acidic hydrophilic and with 29.768 5 ku molecular mass, 6.06 theoretical pI. It is a putative nuclear-localized protein with a conserved AP2 domain. Phylogenetic analysis indicated the domain belong to B-4 group of ERF subfamily and the protein is most close in genetic relationship withArabidopsisERF113. Quantitative real-time PCR (qRT-PCR) results showed that the accumulation ofAfERF113transcripts was highest in roots of adult plants compared with other tested tissues of juvenile. Whereas, after treatment with exogenous ethylene for 1 h, the expression level ofAfERF113transcripts showed significant increase in all tested tissues either of juvenile or adult plants, indicating its rapid response to ethylene. These results provided a theoretical basis of further research ofAfERF113features and manipulating the growth and development of bromeliads using genetic approach in the near future.
Aechemiafasciata; AP2/EREBP family;AfERF113; Ethylene; Expression analysis
LEI Ming, male, Ph.D,assistant professor. Research area: the flowering molecular mechanism of bromeliads.E-mail:leiming_catas@126.com
XU Li, male,research fellow,doctoral supervisor.Research area:the molecular mechanism of the growth and development of bromeliads and bananas.E-mail:xllzy@263.net
常见废弃量名称与标准量名称对照表
2016-04-23
2016-06-15
国家自然基金面上项目(31372106);海南省自然科学基金面上项目(20163127)。
雷 明,男,博士,助理研究员,从事凤梨科植物开花分子机理研究。E-mail:leiming_catas@126.com
徐 立,男,研究员,博士生导师,从事凤梨、香蕉等热带作物生长发育调控研究。E-mail: xllzy@263.net
日期:2016-12-12
网络出版地址:http://www.cnki.net/kcms/detail/61.1220.S.20161212.1117.034.html
S682.39
A
1004-1389(2016)12-1851-10
Received 2016-04-23 Returned 2016-06-15
Foundation item The National Natural Science Foundation of China (No. 31372106); the National Science Foundation of Hainan Province (No. 20163127).
