南海新村湾海草沉积物细菌群落组成及分布
2016-12-23江玉凤董俊德张燕英
江玉凤,凌 娟,董俊德,3,张燕英,3
(1.中国科学院南海海洋研究所 热带海洋生物资源与生态重点实验室,广州 510301;2.中国科学院大学,北京 100049;3.中国科学院海南热带海洋生物重点实验站,三亚 572000)
南海新村湾海草沉积物细菌群落组成及分布
江玉凤1,2,凌 娟1,董俊德1,3,张燕英1,3
(1.中国科学院南海海洋研究所 热带海洋生物资源与生态重点实验室,广州 510301;2.中国科学院大学,北京 100049;3.中国科学院海南热带海洋生物重点实验站,三亚 572000)
采用聚合酶链式反应-变性梯度凝胶电泳(PCR-DGGE)技术及产物序列分析方法,比较研究南海新村湾3个站位海草沉积物中细菌群落组成及分布。结果表明:根据DGGE图谱代表的遗传多样性显示春季二号和春季三号站位样品相似度最高为81.5%,秋季一号和秋季三号站位样品相似度最低为29.3%。春季和冬季细菌多样性指数较高,其Shannon 指数范围分别为3.03~3.07和3.23~3.35。通过对16S rDNA V3 高变区域42条条带克隆测序比对,结果显示:海草沉积物细菌类群主要分布在变形菌门(Proteobactetia)和厚壁菌门(Firmicutes),其中变形菌门中Gammaproteobacteria含量最多,为新村湾海草沉积物中优势菌群,占总数的65.80%。蓝细菌(Cyanobacteria)普遍存在于所有沉积物样品中。细菌群落结构在不同季节、不同站位间具有一定差异。秋季二号站位和三号站位优势类群分别为嗜冷杆菌属和交替假单胞属,其含量分别为31.61%和19.41%。
海草沉积物;细菌群落组成;南海;新村湾
海草床是典型近海岸生态系统,具有较高生物多样性和生产力[1],在近海岸营养物质循环,潮间及潮下带基质沉积和稳定,海洋生物栖息与食物来源等方面具有重要生态价值[2]。海草沉积物中存在大量细菌,其在海洋物质循环、能量流动和海洋生态系统多样性等方面扮演重要角色[3]。同时,细菌群落组成和结构受到沉积物环境、地理位置及季节等因素影响[4-6]。目前,南海新村湾沉积物中细菌多样性分布很少报道。海洋微生物经常处于“活的非可培养状态”(Viable but Nonculturable State),传统纯培养方法难以对海洋微生物多样性进行有效监测[7]。研究表明,纯培养方法获得微生物不足其总数的0.1%[8]。随着分子生物学技术在微生物研究中广泛应用,变性梯度凝胶电泳(Denaturing Gradient Gel Electrophoresis,DGGE)方法逐渐成为一项重要鉴定微生物多样性技术手段[9]。目前,DGGE方法已经广泛应用于土壤和海洋中微生物研究,并且能较好反应微生物菌落组成及分布[9-10]。Bagwell等通过PCR-DGGE分析互花米草(Spartinaalterniflora)中固氮菌类群,结果显示多数固氮菌为厌氧菌[11]。凌娟等通过DGGE方法研究三亚湾表层和底层蓝藻群落结构组成,发现表层和底层群落中普遍存在细菌类群为Synechococcus和Synechococcus-likespecies[12]。Ding 等利用DGGE方法研究苜蓿土壤及其临近灌木丛土壤中细菌群落结构组成时,发现不同土壤中Bacteria,Actinobacteria,Alphaproteobacteria和Betaproteobacteria类群组成差异较大[13]。
因此,本研究通过PCR-DGGE技术,分析南海新村湾不同站位海草沉积物细菌群落组成,并探讨不同站位细菌差异性分布原因。这为进一步认知海草沉积物中细菌群落组成,及其在物质能量循环中的作用提供理论基础。
1 材料与方法
1.1 样品采集
样品采集时间为2012年10月(秋季)及2013年1月(冬季)、4月(春季)、7月(夏季)。采样地点为南海新村湾湾内(110.015°E,18.408°N)、湾中(110.004°E,18.397°N)和湾口(109.984°E,18.404°N),低潮时用无菌1.5 mL eppendorf 小管采集海草周围沉积物,每个样品设5个平行,采集后快速储存于-80℃保存。其中,湾内站位(一号站位)海草较为密集,受到人类活动影响相对较小;湾中站位(二号站位)内侧为网箱养殖区域,水质受到轻微污染;湾口(三号站位)位于新村湾码头附近,旅游业和餐饮业较发达。
1.2 细菌总DNA提取
采用Mobio DNA Isolation Kit提取沉积物中总DNA[14]。
1.3 细菌16S rDNA 片段V3高变序列扩增
以样品总DNA为模板,采用细菌引物GC-338F和518R扩增样品16S rDNA V3高变区序列[10],其中338F为CCT ACG GGA GGC AGC AG,518R为ATT ACC GCG GCT GCT GG,"GC"夹子序列为CGC CCG GGG CGC GCC CCG GGG CGG GGC GGG GGC GCG GGG GGC CTA CGG GAG GCA GCA G。PCR扩增体系(50 μL)为:10× PCR buffer 5 μL;dNTP(2.5 mmol/L)3.2 μL;rTaq(5 U/μL)0.4 μL;GC-338F(20 μmol/L)1 μL;518R(20 μmol/L)1 μL;模板DNA 50 ng;补dd H2O至50 μL。PCR扩增程序为:94℃预变性5 min;94℃变性1 min,55℃复性45 s,72℃延伸1 min,30个循环;最终72℃延伸10 min。
1.4 变性梯度凝胶电泳(DGGE)分析
取10 μL PCR产物进行变形梯度凝胶电泳(DGGE)分析。采用变形梯度为35%~55%,浓度为8%的聚丙烯酰胺凝胶在1×TAE缓冲液中150V、60℃下电泳5 h。
1.5 DGGE图谱中条带回收与测序
用灭菌手术刀切下待回收DGGE条带,采用OMEGA 公司Poly-Gel DNA Extraction Kit回收目的条带。将重新扩增DNA片段切胶回收、纯化后,连接到pMD 18-T载体上,并转化至大肠杆菌DH5α感受态细胞中,筛选阳性克隆,进行序列测定。
1.6 数据分析
DGGE图谱采用Quantity One软件对每个样品的电泳条带数目、条带密度进行数字化分析,Shannon指数(H′)、均衡指数(Evenness,E)和丰度(Richness,S)等指标被用来比较不同样品多样性情况。其算法如下所示:
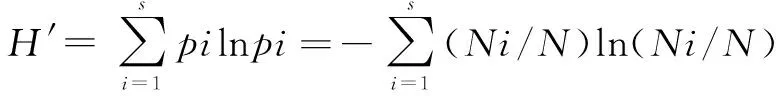
E′=H′/Hmax=H′/lnS
其中,pi为样品中单一条带的强度在该样品所有条带总强度中所占的比率,N为DGGE图谱单一泳道上所有条带的丰度,Ni为第i条带的丰度;S是样品中所有条带数目总和。
2 结果
2.1 细菌16S rDNA V3区 PCR-DGGE图谱分析
PCR-DGGE图谱中条带颜色越深表示细菌种群含量越高,条带位置和数量分别代表细菌种类和丰度。由图1可以看出,海草沉积物样品细菌丰度冬季高于其他季节,不同样品DGGE图谱中条带数目、迁移位置及亮度上均有较大差异,海草沉积物样品间细菌种类及丰度不同。对海草12个沉积物样品分析,共检测到262条条带。每个样品条带数目范围为16~26,平均为21.8;秋季、春季、夏季和冬季样品平均条带数分别为19、22.3、19.3和26.7。根据泳道条带识别匹配结果,由Quantity One软件计算各个样品间戴维斯系数(Dice coefficient)相似矩阵。两两间样品相似性系数平均值为47.56%。春季二号站位和春季三号站位样品间相似度最高为81.5%,秋季一号站位和秋季三号站位样品相似度最低为29.3%(表1)。

图1 沉积物细菌16S rDNA V3 区PCR-DGGE图谱
Fig 1 PCR-DGGE profiles of 16S rDNA V3 region of bacteria in sediment
1Q、2Q、3Q分别代表一号、二号和三号站位秋季样品;1C、2C、3C分别代表一号、二号和三号站位春季样品;1X、2X、3X分别代表一号、二号和三号站位夏季样品;1D、2D、3D分别代表一号、二号和三号站位冬季样品。下同
2.2 沉积物样品细菌多样性指数分析
采用Quantity One软件分析细菌群落多样性表明:春季和冬季细菌多样性指数较高,其Shannon指数范围分别为3.03~3.07和3.23~3.35。秋季和夏季多样性指数相对其他季节较低。冬季和秋季的一号站位细菌多样性指数高于同季节其他站位,春季和夏季细菌多样性指数二号站位最高(表2)。
表1 沉积物样品相似系数(%)
Table 1 Dice coefficients among different sediment samples(%)

UPGMA聚类分析表明:不同站位沉积物细菌群落可以聚为一簇,春季和夏季样品距离较近,秋季样品较为分散。其中,秋季一号站位、二号站位样品和冬季样品在聚类树上比较接近(图2)。
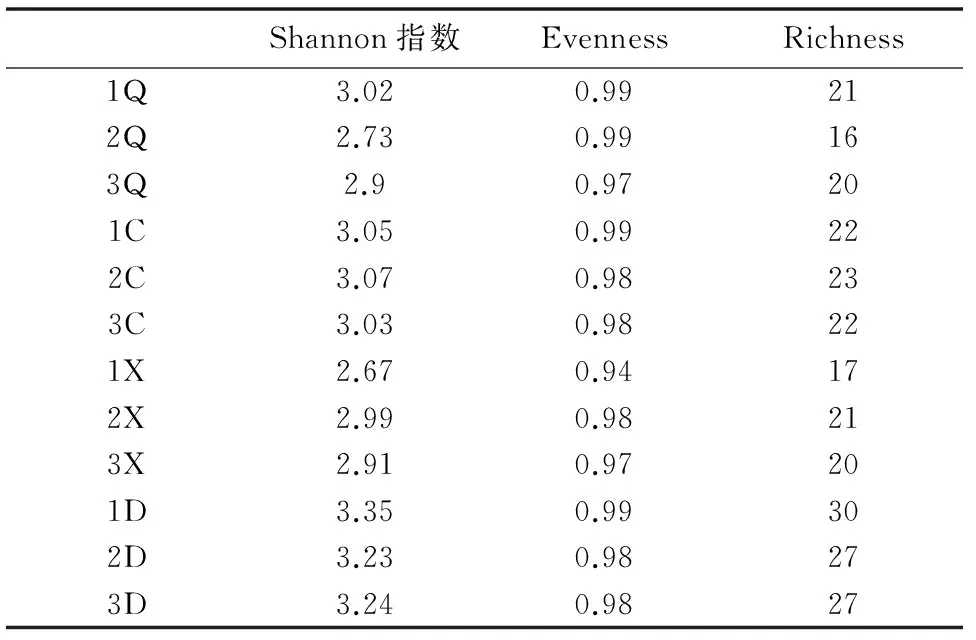
表2 沉积物细菌多样性指数分析
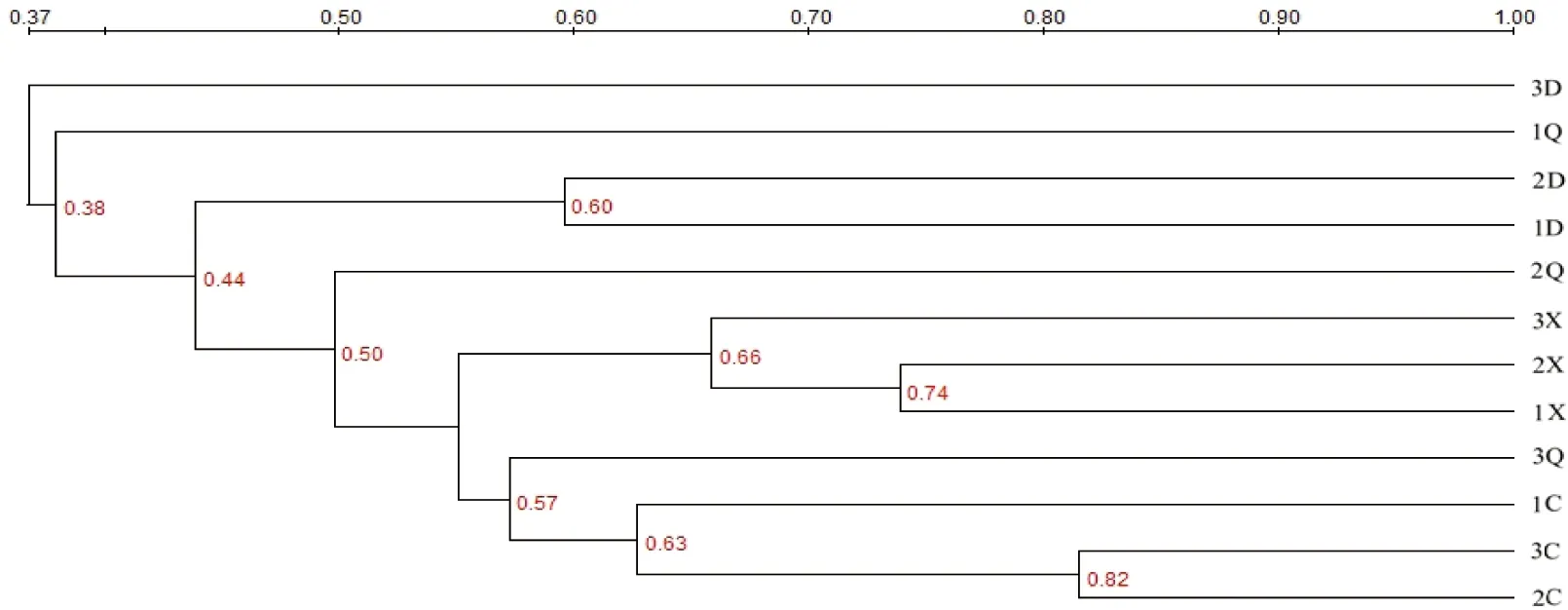
图2 沉积物样品聚类分析图(UPGMA)
Fig 2 Cluster analysis (UPGMA)among different sediment samples
2.3 DGGE切胶条带分析
对DGGE图谱42种迁移位置不同的条带切胶、克隆测序,并与 GenBank数据库中16S rDNA序列比对,所得序列和GenBank中序列相似性在93%~100%之间,对应的同源性最高的细菌主要分布在变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、酸杆菌门(Acidobacteria)和蓝细菌门(Cyanobacteria),见表3,其比例分别为70.87%、10.18%、1.05%和6.64%。其中,变形菌门包含Gammaprobacteria(65.80%)和Epsilonproteobacteria(5.07%)两大类群。不同站位变形菌门平均含量不同,二号站位变形菌门(72.59%)高于一号站位(70.79%)和三号站位(70.65%)。厚壁菌门在不同季节含量不同,秋季厚壁菌门含量最多为11.64%,其次为冬季(9.96%)和春季(9.50%),夏季含量最少(8.57%)。酸杆菌门在冬季一号样品和二号样品及夏季三号样品中出现,其含量分别为3.92%、3.60%和3.11%。另外,所有样品均检测到蓝细菌,其中,一号站位蓝细菌平均含量为7.12%,二号和三号站位平均值为7.04%和6.92%。
沉积物样品中嗜冷杆菌属(Psychrobacter)平均含量约为15.80%,该菌在二号站位含量较高,其中秋季二号站位含量为31.61%。Pseudoalteromonassp.大量存在于秋季三号站位,约为19.41%。Serratianematodiphila在秋季三号站位含量最高为3.5%。Klebsiellasp.在春季三号站位沉积物中含量为0.27%,高于其他样品。Paenibacillus在秋季和冬季一号站位检测到,其含量分别为4.86%和2.96%。Bacillusflexu在新村湾沉积物夏季样品中含量较高。
表3 PCR-DGGE图谱中条带序列分析
Table 3 The band sequence analysis in PCR-DGGE profiles

3 讨论
本研究应用PCR-DGGE技术分析南海新村湾沉积物中细菌群落组成,发现南海新村湾海草沉积物不同站位细菌群落主要包含:嗜冷杆菌属(Psychrobacter),弓形杆菌属(Arcobacter),弧菌属(Vibrio),发光杆菌属(Photobacterium),克雷伯氏菌属(Klebsiella),科贝克氏菌属(Cobetia),假单胞交替菌属(Pseudoalteromonas)和沙雷氏菌属(Serratia)等;其中,变形菌门(Proteobacteria)为主要优势门类,且其含量占细菌总数的70.87%。变形菌门在南海新村湾沉积物中具有很强的适应性,其在海洋生态系统中分布广泛,此结论与Ravenschlag 等研究结果相一致[15]。同时,蓝细菌(Cyanobacteria)在所有沉积物样品中均被检测到,该细菌被当作水体富营养化检测指标,当水体中氮磷含量增加时,会引起蓝细菌数目增加[16]。
不同站位间细菌群落结构具有一定差异。二号站位变形菌门平均含量高于一号和三号站位,一号站位蓝细菌平均含量高于二号和三号站位。不同站位间细菌分布差异可能由于新村湾受潮汐变化及季风气候影响,沉积物再悬浮引起[17];同时,南海新村湾内侧有三条河流入,部分物质如重金属(汞、铅、镉、铬、铜、锌、砷等)、有机物(除草剂、杀虫剂、多烷芳烃、多氯联苯等)和石油烃等进入海岸带,对不同站位细菌分布产生一定影响[18]。另外,随着海草植株衰老脱落,海草组织最终被微生物所分解形成的有机质,也将影响细菌分布[19]。
秋季二号站位沉积物样品中嗜冷杆菌属(Psychrobacter)约为其他站位的2~3倍,含量为31.61%,同时,二号站位秋季、春季和夏季中该菌含量均高于同季节其他站位。该现象可能是由于秋季、春季和夏季二号站位附近网箱养殖区范围较大,水产养殖业造成水域污染,近而对细菌种类分布产生一定影响[20]。另外,研究报道显示,嗜冷杆菌属具有耐盐、需氧等特征[21],主要分布在低温海洋环境和温带海水中[22-24],该菌能降解海水和沉积物所含污染物,对生态环境具修复作用[25]。
秋季三号站位Pseudoalteromonassp.含量较多。研究报道显示,Pseudoalteromonassp.能分泌胞外多糖exopolysaccharide(EPS),EPS可以作为金属离子絮凝剂,结合多种金属离子,如Fe2+、Zn2+、Cu2+和Co2+等[26]。因此,Pseudoalteromonassp.在海草沉积物中可以聚集并结合金属离子,减轻过量金属离子对植物的毒害;并通过沉降悬浮物,改善水质,提高海草周围水质透明度。Serratianematodiphila菌株在秋季三号站位检测到,该菌株具有抗金属性[27]。Klebsiellasp.在新村湾春季三号站位沉积物中分布较多,Lee等发现该菌株可以有效利用葵烷、十六烷及原油,并能够产生生物表面活性剂,具有很强乳化原油的功能[28]。同时,研究表明,海菖蒲沉积物中低浓度多烷芳烃(PAH)可以为微生物提供碳源,能促进PAH及其衍生物降解相关微生物生长[29]。
细菌群落多样性指数结果显示,春季和冬季的多样性指数较高,夏季和秋季多样性减少。该现象可能是由于春季和冬季温度较为适宜,而秋季和夏季温度较高,位于低潮间带海草和细菌受到光照和紫外线胁迫,生长受到抑制[17]。另外,在秋季和冬季一号站位检测到Paenibacillus,该菌能够增强植物抗性,促进植物生长[30]。Bacillusflexu在新村湾沉积物夏季样品中分布高于其他季节,该菌能抵抗多种病原体[31]。细菌在不同季节和不同站位分布有所差异,其与环境及海草相互作用关系有待进一步研究。
[1]凌 娟,董俊德,张燕英,等.一株珊瑚礁-海草床复合生态系统固氮菌的分离与鉴定[J].微生物学通报,2010,37(7): 962-968.
[2]COSTANZA R.The value of ecosystem services[J].Ecological Economics,1998,25(1): 1-2.
[3]FIELD C B,BEHRENFELD M J,RANDERSON J T,et al.Primary production of the biosphere: integrating terrestrial and oceanic components[J].Science,1998,281(5374): 237-240.
[4]NELSON D M,OHENE-ADJEI S,HU F S,et al.Bacterial diversity and distribution in the holocene sediments of a northern temperate lake[J].Microbiology Ecology,2007,54(2): 252-263.
[5]LIU W T,MARSH T L,CHENG H,et al.Characterization of microbial diversity by determining terminal restriction fragment length polymorphisms of genes encoding 16S rRNA[J].Applied and Environment Microbiology,1997,63(11): 4516-4520.
[6]JURGENS G,GLÖCKNER F,AMANN R,et al.Identification of novel Archaea in bacterioplankton of a boreal forest lake by phylogenetic analysis and fluorescent in situ hybridization[J].FEMS Microbiology Ecology,2000,34(1): 45-56.
[7]AYRAPETYAN M,WILLIAMS T C,OLIVER J D.Bridging the gap between viable but non-culturable and antibiotic persistent bacteria[J].Trends in Microbiology,2015,23(1): 7-13.
[8]SHAH S Q,CABELLO F C,L′ABÉE-LUND T M,et al.Antimicrobial resistance and antimicrobial resistance genes in marine bacteria from salmon aquaculture and non-aquaculture sites[J].Environmental Microbiology,2015,16(5): 1310-1320.
[9]XIONG X,HU Y,YAN N,et al.PCR-DGGE analysis of the microbial communities in three different Chinese "Baiyunbian" liquor fermentation starters[J].Journal of Microbiology and Biotechnology,2014,24(8): 1088-1095.
[10]MUYZER G,DE WAAL E C,UITTERLINDEN A G.Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J].Applied and Environmental Microbiology,1993,59 (3): 695-700.
[11]BAGWELL C E,ROCQUE J R,SMITH G W,et al.Molecular diversity of diazotrophs in oligotrophic tropical seagrass bed communities[J].FEMS Microbiology Ecology,2002,39 (2): 113-119.
[12]凌 娟,张燕英,董俊德,等.三亚湾珊瑚礁海域蓝藻群落组成的空间分布特征及其与环境因子的关系[J].科学通报,2013,58(17): 1610-1619.
[13]DING G C,PICENO Y M,HEUER H,et al.Changes of soil bacterial diversity as a consequence of agricultural land use in a semi-arid ecosystem[J].PLOS One,2013,8(3): e59497.
[14]PEDERSEN M W,GINOLHAC A,ORLANDO L,et al.A comparative study of ancient environmental DNA to pollen and macrofossils from lake sediments reveals taxonomic overlap and additional plant taxa[J].Quaternary Science Reviews,2013,75(1): 161-168.
[15]RAVENSCHLAG K,SAHM K,AMANN R.Quantitative molecular analysis of the microbial community in marine Arctic sediments (Svalbard)[J].Applied and Environmental Microbiology,2001,67(1): 387-395.
[16]LEIGH C,BURFORD M A,ROBERTS D T,et al.Predicting the vulnerability of reservoirs to poor water quality and cyanobacterial blooms[J].Water Research,2010,44(15): 4487-4496.
[17]许战洲,黄良民,黄小平,等.海草生物量和初级生产力研究进展[J].生态学报,2007,27(6): 2594-2602.
[18]蔚枝沁,邓 泓,吴可为,等.海南优势海草营养元素的含量及其影响因素研究[J].华东师范大学学报,2012(4):131-141.
[19]李文涛,张秀梅.海草场的生态功能[J].中国海洋大学学报(自然科学版),2009,39(5): 933-939.
[20]郑凤英,邱广龙,范航清,等.中国海草的多样性、分布及保护[J].生物多样性,2013,21(5): 517-526.
[21]JUNI E,HEYM G A.Psychrobacterimmobilisgen.nov.,sp.nov.: geno species composed of Gram-negative,aerobic,oxidase-positive coccobacilli[J].International Journal of Systematic Bacteriology,1986,36(3): 388-391.
[22]ROMANENKO L A,SCHUMANN P,ROHDE M,et al.Psychrobactersubmarinussp.nov.andPsychrobactermarincolasp.nov.,Psychrophilichalophilesfrom marine environments[J].International Journal of Systematic and Evolutionary Microbiology,2002,52(Pt4): 1291-1297.
[23]ROMANENKO L A,LYSENKO A M,ROHDE M,et al.Psychrobactermaritimussp.nov.andPsychrobacterarenosussp.nov.,isolated from coastal sea ice and sediments of the Sea of Japan[J].International Journal of Systematic and Evolutionary Microbiology,2004,54(Pt5): 1741-1745.
[24]YOON J H,LEE C H,YEO S H,et al.Psychrobacteraquimarissp.nov.andPsychrobacternamhaensissp.nov.,isolated from sea water of the South Sea in Korea[J].International Journal of Systematic and Evolutionary Microbiology,2005,55(Pt3): 1007-1013.
[25]BHARATHKUMAR S,PAUL D,NAIR S.Microbial diversity of culturable heterotrophs in the rhizosphere of salt marsh grass,Porteresiacoarctata(Tateoka) in a mangrove ecosystem[J].Journal of Basic Microbiology,2008,48(1): 10-15.
[26]QIN G,ZHU L,CHEN X,et al.Structural characterization and ecological roles of a novel exopolysaccharide from the deep-sea psychrotolerant bacteriumPseudoalteromonassp.SM9913[J].Microbiology,2007,153(5): 1566-1572.
[27]SARMA B,ACHARYA C,JOSHI S R.Characterization of metal tolerantSerratiaspp.isolates from sediments of uranium ore deposit of Domiasiat in Northeast India[J].Proceedings of the National Academy of Sciences,India Section B: Biological Sciences,2013,3078(5):292-300.
[28]LEE S C,LEE S J,KIM S H,et al.Characterization of new biosurfactant produced byKlebsiellasp.Y6-1 isolated from waste soybean oil[J].Bioresource Technology,2008,99(7): 2288-2292.
[29]LING J,ZHANG Y Y,WU M L,et al.Fungal community successions in rhizosphere sediment of seagrassesEnhalusacoroidesunder PAHs stress[J].International Journal of Molecular Sciences,2015,16(6): 14039-14055.
[30]TIMMUSK S,WAGNER E G.The plant-growth-promoting rhizobacteriumPaenibacilluspolymyxainduces changes inArabidopsisthalianagene expression: a possible connection between biotic and abiotic stress responses[J].Molecular Plant Microbe Interactions,1999,12(11): 951-959.
[31]PRIYADARSHINI S,GOPINATH V,MEERA PRIYADHARSSHINI N,et al.Synthesis of anisotropic silver nanoparticles using novel strain,Bacillusflexusand its biomedical application[J].Colloids and Surfaces B: Biointerfaces,2013,102:232-237.
The bacterial communities constitution and distribution insediment of seagrass in Xincun Bay,South China Sea
JIANG Yu-feng1,2,LING Juan1,DONG Jun-de1,3,ZHANG Yan-ying1,3
(1.Key Laboratory of Tropical Marine Bio-Resources and Ecology,South China Sea Institute of Oceanology,CAS,Guangzhou 510301; 2.University of Chinese Academy of Sciences,Beijing 100039; 3.National Experiment Station of Tropical Marine Biology,Sanya 572000,China)
In order to compare the bacterial communities constitution and distribution in three sites of seagrass sediment in Xincun Bay,South China Sea,Polymerase Chain Reaction-Denaturing Gradient Gel Electrophoresis (PCR-DGGE) analysis compounded with cloning and sequencing bands in DGGE profile was used.The results diverse bands of DGGE profile showed the most similarity of bacterial communities was found between the second and the third site in spring with an coefficient of 81.5%,whereas the least was 29.3% between the first and the third site in autumn.Shannon diversity indexes of the bacteria in spring and winter were higher than other seasons,which were of 3.03-3.07 and 3.23-3.25,respectively.Further,based on the 16S rDNA-V3 fragments from 42 individual DGGE bands and clone library sequence,the results showed most of the sequences belonged to Proteobactetia and Firmicutes.The most abundance Proteobactetia was Gammaproteobacteria with a percentage of 65.80%.Cyanobacteriawere widely distributed in all samples.In addition,there were differences in bacterial communities among different seasons and sites.PsychrobacterandPseudoalteromonaswere advantage groups in the second and the third site during autumn,with a percentage of 31.61% and 19.41%,respectively.
seagrass sediment; bacterial communities constitution; South China Sea; Xincun Bay
2016-02-03;
2016-02-24
收稿日期:国家自然科学基金(41276113; 41276114; 41406191; 41676163);湖南省科技兴海专项(XH201415);广东省公益研究与能力建设专项(2015A020216016)
江玉凤,博士研究生,主要从事海洋微生物分子生态学研究,E-mail:jiangyufeng11@mails.ucas.ac.cn
董俊德,研究员,主要从事微生物海洋学、海洋生态学研究,E-mail:dongjunde@vip.163.com
10.3969/j.issn.2095-1736.2016.06.038
Q938
A
2095-1736(2016)06-0038-05
