嘧啶并嘧啶酮类化合物作为L3MBTL1选择性络合剂的设计与合成
2016-12-21周皓,车鑫,彭丽,王娜,柏旭
周 皓, 车 鑫, 彭 丽, 王 娜, 柏 旭
(吉林大学组合化学与创新药物研究中心, 吉林大学药学院, 长春 130021)
嘧啶并嘧啶酮类化合物作为L3MBTL1选择性络合剂的设计与合成
周 皓, 车 鑫, 彭 丽, 王 娜, 柏 旭
(吉林大学组合化学与创新药物研究中心, 吉林大学药学院, 长春 130021)
用嘧啶并嘧啶酮替换Lethal 3 malignant brain tumor 1(L3MBTL1)小分子络合剂UNC669分子中的芳香部分, 合成了一系列嘧啶并嘧啶酮类化合物. 采用同质邻近发光放大法(AlphaScreen®)测试了其活性, 得到IC50值为1.21 μmol/L的化合物8a; 通过对其5位基团进行改造, 最终获得了3个选择性L3MBTL1络合剂8g, 8o与8p, 它们仅对L3MBTL1有活性, 对其同源蛋白L3MBTL3在内的其它甲基化识别蛋白则无活性.
嘧啶并嘧啶酮; L3MBTL1; 选择性络合剂

Fig.1 Structure of UNC669 and UNC926
近十多年来, 表观遗传学领域中的调节蛋白逐渐成为新药研究的热门靶点[1~3]. 信号识别蛋白是调节蛋白中的一大类, 它能够识别染色体中组蛋白上被共价修饰的氨基酸残基, 从而实现调节蛋白之间相互作用的功能[4]. 组蛋白上可被修饰的氨基酸残基主要包括赖氨酸和精氨酸2大类, 而共价修饰这2大类残基的方式主要有甲基化、 乙酰化、 磷酸化和泛素化等[5]. 大约有超过200种蛋白能够识别甲基化修饰的组蛋白赖氨酸残基, 这个庞大的家族主要被分为3大类: 植物同源域(PHD)蛋白, WD40(Tryptophan-aspartic acid 40)重复域蛋白和“国王家族”蛋白. “国王家族”主要由Tudor结构域、 克罗莫结构域(Chromodomain)、 PWWP(Pro-Trp-Trp-Pro)结构域和恶性脑瘤结构域(MBT)蛋白家族组成[6].
Lethal 3 malignant brain tumor 1(L3MBTL1)[7]是MBT家族中研究相对成熟的蛋白之一, 其生物学功能越来越多地被发现. Trojer等[8]证明L3MBTL1的缺失能够促进造血祖细胞分化为红细胞. 通过与染色体结合, L3MBTL1能够抑制E2F-调控基因, 例如与致癌和成长有关的c-myc基因. 非组蛋白p53可由赖氨酸甲基化转移酶Set8/PR-Set7共价修饰得到单甲基化的p53(p53K382me1), 而L3MBTL1能够与p53K382me1结合, 从而抑制p53反式激活其靶基因[9]. 虽然L3MBTL1的生物学作用被逐渐认识, 但是其许多生物学功能还有待探索, 而开发能与其络合的小分子探针不仅可以帮助认识其生物学功能, 还能够证明其靶点成药性.
2011年, Herold等[10,11]发现了第一个L3MBTL1小分子络合剂UNC669(1a), 随后通过构效关系研究得到了化合物UNC926(1b), 结构如图1所示. 化合物1a和1b都含有1个4-吡咯烷基哌啶基团, 该基团能够模仿组蛋白甲基化赖氨酸残基与L3MBTL1的芳香口袋结合, 这种结合力主要是质子化后的吡咯烷与口袋中的天冬氨酸355形成的氢键和盐桥, 此外该基团与芳香残基之间的疏水作用和阳离子π作用也是导致二者结合的因素.
含氮芳杂环是许多活性分子都含有的结构片段[12~14], 本课题组[15~19]一直致力于构建新颖的芳香杂环化合物分子库, 并已报道了多种快速构建嘧啶并环的方法. 本文采用嘧啶并环替换化合物1a和1b中芳香环部分的策略, 开发了L3MBTL1小分子探针, 发现了一系列可作为选择性的L3MBTL1小分子络合剂的以嘧啶并[4,5-d]嘧啶-4(3H)-酮为骨架的化合物.
1 实验部分
1.1 试剂与仪器
二乙苯胺(PhNEt2)、 三氯氧磷(POCl3)、 二(异丙基)氨基锂(LDA)、 四氢呋喃(THF)、 二氯亚砜(SOCl3)、N,N-二甲基甲酰胺(DMF)、 醋酸(HOAc)、 乙腈、 三乙胺(TEA)、 正丁醇(n-BuOH)、 甲醇钠(MeONa)、 苯酚、 碳酸钾、 二氧六环、 双(三环己基膦)二氯化钯[Pd(PCy3)2Cl2]、 锌粉、 间氯过氧苯甲酸(mCPBA)和二氯甲烷(DCM)均为分析纯或化学纯, 除特殊注明外未经进一步处理.
Agilent 1100型液质联机(LC/MS)和Alltech ELSD 2000型蒸发光散射检测仪(美国安捷伦科技有限公司); Varian 300 MHz核磁共振仪(TMS为内标, 美国瓦里安技术中国有限公司); XT5型显微熔点测定仪(微电脑控温型, 未校准, 北京科仪电光仪器厂); 薄层色谱用硅胶G(台州市路桥四甲生化塑料品厂); 柱色谱用硅胶(200~300目, 青岛海洋化工有限公司); Agilent 1290-micrOTOF QⅡ型高分辨液相色谱-质谱联用仪(美国布鲁克·道尔顿公司); Bruker IFS 66V/S 型红外光谱仪(德国布鲁克公司).
1.2 实验过程
嘧啶并嘧啶酮衍生化合物的合成路线如Scheme 1所示.
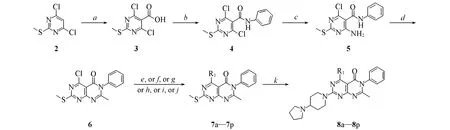
Scheme 1 General syntheses of target compounds
1.2.1 化合物3的合成 参照文献[20]方法合成化合物3, 产率43%, m. p. 164~166 ℃(文献值[21]: 160~163 ℃).
1.2.2 化合物4的合成 参照文献[21]方法合成化合物4.
1.2.3 化合物5的合成 将化合物4(1.87 g, 5.96 mmol)溶于30 mL四氢呋喃中, 加热至回流, 向反应体系中持续通入氨气1 h. 反应完全后, 停止通入氨气并将反应液冷却至室温, 过滤并用乙酸乙酯洗涤滤饼, 即得化合物5.
1.2.4 化合物6的合成 将化合物5(1.0 g, 3.4 mmol)和乙酸(0.39 mL, 6.8 mmol)溶于30 mL乙腈中, 加入三氯氧磷(1.3 mL, 13.6 mmol), 加热至回流反应8 h. 反应完全后, 冷却至室温, 并将反应液缓慢加入碎冰中, 用Na2CO3调节pH值至9, 用乙酸乙酯(60 mL×3)萃取, 合并有机相, 用无水硫酸钠干燥, 过滤蒸干溶剂得到粗品, 经快速柱层析 [V(石油醚)∶V(乙酸乙酯)=2∶1]分离, 即得化合物6.
1.2.5 化合物7a~7p的合成 参照文献[15]方法合成化合物7a~7k, 参照文献[22]方法合成化合物7l, 参照文献[23]方法合成化合物7m. 将化合物6(300 mg, 0.94 mmol)溶于4 mL二氧六环中, 冰浴下分3批加入2-甲氧基乙醇钠(111 mg, 1.13 mmol), 将反应体系升至室温并搅拌2 h. 反应完毕, 用20 mL水稀释, 二氯甲烷(20 mL×3)萃取, 合并有机相并用无水硫酸钠干燥. 将有机相中的溶剂蒸除, 经快速柱层析[V(石油醚)∶V(乙酸乙酯)=1∶1]分离, 得到化合物7n. 将苯酚(177 mg, 1.89 mmol)溶于4 mL二氧六环中, 加入无水碳酸钾(217 mg, 1.58 mmol), 将反应体系升温至80 ℃并搅拌0.5 h; 加入化合物6(200 mg, 0.63 mmol). 反应15 h后, 将反应体系用15 mL水稀释, 二氯甲烷(20 mL×3)萃取. 合并有机相, 用无水硫酸钠干燥, 将有机相中的溶剂蒸除, 经快速柱层析 [V(石油醚)∶V(乙酸乙酯)=1∶1] 分离, 得到化合物7o. 将化合物6(200 mg, 0.64 mmol)和苯硼酸频哪醇酯(252 mg, 1.28 mmol)溶于4 mL二氧六环中, 加入碳酸钾(353 mg, 2.56 mmol), 抽出空气并用氮气置换, 反复操作3次后加入双(三环己基膦)二氯化钯(24 g, 0.032 mmol), 在氮气保护下反应30 h. 反应结束后, 用硅藻土过滤, 滤液用10 mL水稀释, 分出水相后, 用二氯甲烷(20 mL×3)萃取. 合并有机相, 用无水硫酸钠干燥, 将有机相中的溶剂蒸除, 经快速柱层析[V(石油醚)∶V(乙酸乙酯)=1∶1]分离得到化合物7p.
1.2.6 化合物8a~8p的合成 将间氯过氧苯甲酸(114 mg, 0.56 mmol)在超声下溶于4 mL二氯甲烷中, 于0 ℃下滴加溶有化合物7a~7p(0.28 mmol)的2 mL二氯甲烷溶液中, 升温至室温, 搅拌3 h后, 用亚硫酸氢钠猝灭反应. 将猝灭后的混合物用15 mL饱和碳酸氢钠洗涤, 水相用二氯甲烷(15 mL×3)萃取, 合并有机相并用无水硫酸钠干燥, 蒸干溶剂得到氧化中间体粗品. 将反应粗品溶于6 mL四氢呋喃中, 加入4-(吡咯烷-1-基)哌啶(65 mg, 0.42 mmol), 室温下搅拌8 h. 反应完全后, 加入10 mL水稀释, 用二氯甲烷(15 mL×3)萃取, 合并有机相并用饱和碳酸氢钠洗涤, 无水硫酸钠干燥, 过滤蒸干溶剂得到粗品, 粗品经快速柱层析[V(二氯甲烷)∶V(甲醇)∶V(氨水)=200∶1∶10]分离得到目标化合物8a~8p.
所有化合物的理化性质和光谱数据见表1和表2.
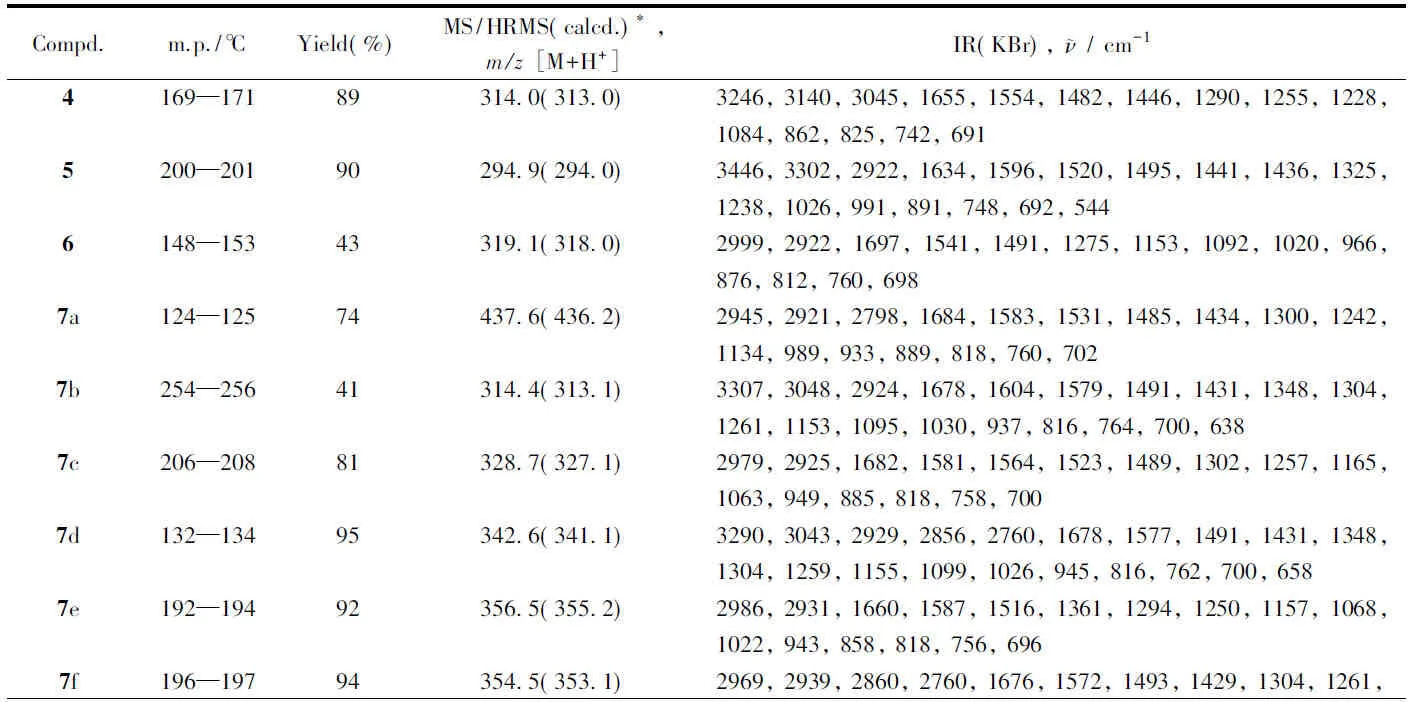
Table 1 Melting points, MS, HRMS and IR data of compounds 4—6, 7a—7p and 8a—8p

Continued

Table 2 1H NMR and 13C NMR data of compounds 4—6, 7a—7p and 8a—8p*

Continued

Continued
2 结果与讨论
2.1 合 成
以2-甲硫基-4,6-二氯嘧啶为起始原料, 通过上羧基、 酰胺化、 氨基亲核取代和关环等4步反应合成了嘧啶并嘧啶酮母核, 开发了一种合成该类化合物的新型有效方法. 然后, 对母核5位氯原子进行取代, 再将7位甲硫基氧化成砜(亚砜)后直接进行亲核取代即可得到化合物8a~8p, 这些新化合物的结构均经1H NMR,13C NMR和MS确证.
2.2 活性结果与讨论
嘧啶并[4,5-d]嘧啶-4(3H)-酮系列化合物的活性测试由合作方美国北卡罗来纳大学教堂山分校(University of North Carolina at Chapel Hill)的Stephen V. Frye课题组完成. 除L3MBTL1蛋白外, 另外选择了7种不同家族的甲基化赖氨酸识别蛋白进行测试: L3MBTL1的同源蛋白L3MBTL3[24]、 MBT家族成员MBTD1[25]、 Chromodomain家族成员CBX7[26]、 PHD家族成员JARID1A[27]、 PHF1[28]、 串联Tudor 域蛋白UHRF1-TTD[29]和53BP1[30].
活性测试采用同质邻近发光放大法(AlphaScreen®)[31]对L3MBTL1, 53BP1, MBTD1, CBX7, JARID1A, PHF1和UHRF1-TTD这7种蛋白进行活性初筛, 实验结果见表3. 由于AlphaScreen®测试方法针对L3MBTL3测试的重复性较差, 故对其采用LANCE®TR-FRET测试方法, 其与AlphaScreen测试方法线性相关(R2=0.8)[32]. 对初筛实验中IC50(半数抑制浓度)<1 μmol/L的化合物, 用等温滴定量热法(ITC)[33]进一步确认其活性.

Table 3 IC50 values determined by AlphaScreen® Assay and LANCE TR-FRET assaya
通过筛选得到活性化合物8a对L3MBTL1的IC50值达到了1.21 μmol/L, 再经过ITC活性测试确认其解离常数Kd=2.4 μmol/L, 对L3MBTL1表现出良好的抑制活性. 在此基础上, 尝试改变其5位基团来提高其活性或者选择性. 首先, 尝试在5位引入不同的氨基取代基团(化合物8b~8k), 其中5位引入甲氨基(化合物8b)、 二甲氨基(化合物8c)、 异丙胺基(化合物8d)和二乙胺基(化合物8e)分别导致3, 15, 14和10倍的活性降低(相比于化合物8a, 下同); 另外, 还尝试引入环状胺基, 当5位上是吡咯烷基(化合物8f)和1-甲基哌啶基-4-氨基(化合物8h)时, 活性分别降低约11倍和6倍; 但当引入吗啡啉环(化合物8g)时, 化合物保持了活性, 且对L3MBTL1表现出良好的选择性[IC50(L3MBTL3)/IC50(L3MBTL1)>37]. 尝试在5位引入链状胺得到化合物8i和8j, 但活性分别降低7和9倍; 5位引入苯胺基(化合物8k)使活性降低了4倍. 然后, 引入非碱性基团(化合物8l~8p), 其中用氢原子代替5位的氨基(化合物8l)导致对L3MBTL1活性降低7倍; 5位引入甲氧基(化合物8m)和2-甲氧基乙氧基(化合物8n)使活性分别降低3倍和4倍. 最后, 母核的5位引入苯酚(化合物8o)和苯基(化合物8p)使活性分别降低了2倍和3倍, 虽然对L3MBTL1活性略有降低, 但对其它甲基化识别蛋白无活性, 即表现出良好的选择性[化合物8o, IC50(L3MBTL3)/IC50(L3MBTL1)>45, 化合物8p, IC50(L3MBTL3)/IC50(L3MBTL1)>29]. 综上所述, 通过对化合物8a的5位进行改造, 得到了3个选择性L3MBTL1络合剂8g, 8o和8p.
3 结 论
以L3MBTL1络合剂1a和1b为基础, 用嘧啶并[4,5-d]嘧啶-4(3H)-酮替换这2个化合物的芳香片段, 得到IC50值为1.21 μmol/L的化合物8a, 通过改变化合物8a的5-位基团合成了15个衍生物, 并在其中找到了选择性络合剂8g, 8o与8p. 通过测试这些化合物对包括L3MBTL1在内的8个甲基化识别蛋白的活性发现, 其活性不仅与化合物1a和1b相当, 而且对其它的甲基化识别蛋白包括与L3MBTL1同源蛋白L3MBTL3没有活性, 这些选择性络合剂的发现有可能用于探索L3MBTL1和其同源蛋白的功能差异, 也为进一步开发L3MBTL1小分子络合剂提供了新的线索.
感谢北卡罗来纳大学教堂山分校(University of North Carolina at Chapel Hill) Stephen V. Frye课题组的Lindsey Ingerman James, Brandi Baughman和Jacqueline Norris在生物活性测试上提供的帮助.
[1] Mai A.,Chem.Med.Chem., 2014, 9(3), 415—417
[2] Arrowsmith C. H., Bountra C., Fish P. V., Lee K., Schapira M.,Nat.Rev.Drug.Discov., 2012, 11(5), 384—400
[3] Dhanak D., Jackson P.,Biochem.Biophys.Res.Commun., 2014, 455, 58—69
[4] Owen D. R., Trzupek J. D.,Drug.Discov.TodayTechnol., 2014, 12, e29—e34
[5] Herold J. M., Ingerman L. A., Gao C., Frye S. V.,Curr.Chem.Genomics, 2011, 5, 51—61
[6] James L. I., Frye S. V.,Clin.Pharmacol.Ther., 2013, 93(4), 312—314
[7] Kalakonda N., Fischle W., Boccuni P., Gurvich N., Hoya-Arias R., Zhao X., Miyata Y., Macgrogan D., Zhang J., Sims J. K., Rice J. C., Nimer S. D.,Oncogene, 2008, 27(31), 4293—4304
[8] Trojer P., Li G. H., Sims R. J., Vaquero A., Kalakonda N., Boccuni P., Lee D., Erdjument-Bromage H., Tempst P., Nimer S. D., Wang Y. H., Reinberg D.,Cell, 2007, 129(5), 915—928
[9] West L. E., Roy S., Lachmi-Weiner K., Hayashi R., Shi X. B., Appella E., Kutateladze T. G., Gozani O.,J.Biol.Chem., 2010, 285(48), 37725—37732
[10] Herold J. M., Wigle T. J., Norris J. L., Lam R., Korboukh V. K., Gao C., Ingerman L. A., Kireev D. B., Senisterra G., Vedadi M., Tripathy A., Brown P. J., Arrowsmith C. H., Jin J., Janzen W. P., Frye S. V.,J.Med.Chem., 2011, 54(7), 2504—2511
[11] Herold J. M., James L. I., Korboukh V. K., Gao C., Coil K. E., Bua D. J., Norris J. L., Kireev D. B., Brown P. J., Jin J., Janzen W. P., Gozani O., Frye S. V.,Med.Chem.Comm., 2012, 3(1), 45—51
[12] Liu J. B., Li Y. X., Chen Y. W., Wu C. C., Wan Y. Y., Wei W., Xiong L. X., Zhang X., Yu S. J., Li Z. M.,Chem.Res.ChineseUniversities, 2016, 32(1), 41—48
[13] Sun B., Yin X. E., Zhang J., Huang J., Xu Y., Zhang F. R., Wang J. H., Wang G. Q., Hu C.,Chem.Res.ChineseUniversities, 2015, 31(6), 936—941
[14] Wang H. Y., Liu J. B., Lu J. R., Ying M., Yang X. Y., Yang S. X., Ma Y.,Chem.J.ChineseUniversities, 2016, 37(2), 246—253(王宏韫, 刘金彪, 卢俊瑞, 应明, 杨旭云, 杨树勋, 马瑶. 高等学校化学学报, 2016, 37(2), 246—253)
[15] Xiang J. B., Geng C., Yi L., Dang Q., Bai X.,Mol.Divers., 2011, 15(4), 839—847
[16] Xiang J. B., Wen D. S., Xie H. X., Dang Q., Bai X.,J.Comb.Chem., 2010, 12(4), 503—509
[17] Yang J. X., Che X., Dang Q., Wei Z. L., Gao S., Bai X.,Org.Lett., 2005, 36(37), 1541—1543
[18] Xie H. X., Xiang J. B., Dang Q., Bai X.,Synlett., 2012, 2012(4), 585—588
[19] Che X., Zheng L. Y., Dang Q., Bai X.,J.Org.Chem., 2008, 73(3), 1147—1149
[20] Seto S., Tanioka A., Ikeda M., Izawa S.,Bioorg.Med.Chem.Lett., 2005, 15(5), 1485—1488
[21] Seto S., Tanioka A., Ikeda M., Izawa S.,Bioorg.Med.Chem., 2005, 13(20), 5717—5732
[22] Dunaiskis A., Staigers T., Keltonic T., Chappie T., Meltz C., Dugger R., Sanner M. A.,Org.Prep.Proced.Int., 1995, 27(5), 600—602
[23] Brinkman J. A., Cheung A. W. H., Firooznia F., Guertin K. R., Marcopulos N., Qi L., Racha J. K., Sarabu R., Tan J., Tilley J. W.,Thiazolo-pyrimidine/pyridineUreaDerivatives, US 20070270433, 2007-05-11
[24] Northcott P. A., Nakahara Y., Wu X., Feuk L., Ellison D. W., Croul S., Mack S., Kongkham P. N., Peacock J., Dubuc A., Ra Y. S., Zilberberg K., Mcleod J., Scherer S. W., Sunil R. J., Eberhart C. G., Grajkowska W., Gillespie Y., Lach B., Grundy R., Pollack I. F., Hamilton R. L., van Meter T., Carlotti C. G., Boop F., Bigner D., Gilbertson R. J., Rutka J. T., Taylor M. D.,Nat.Genet., 2009, 41(4), 465—472
[25] Eryilmaz J., Pan P., Amaya M. F., Allali-Hassani A., Dong A., Adams-Cioaba M. A., Mackenzie F., Vedadi M., Min J.,PLoSOne, 2009, 4(10), e72—e74
[26] Yap K. L., Li S., Munoz-Cabello A. M., Raguz S., Zeng L., Mujtaba S., Gil J., Walsh M. J., Zhou M. M.,Mol.Cell, 2010, 38(5), 662—674
[27] Ditacchio L., Le H. D., Vollmers C., Hatori M., Witcher M., Secombe J., Panda S.,Science, 2011, 333(6051), 1881—1885
[28] Hong Z., Jiang J., Lan L., Nakajima S., Kanno S., Koseki H., Yasui A.,NucleicAcidsRes., 2008, 36(9), 2939—2947
[29] Xie S., Jakoncic J., Qian C.,J.Mol.Biol., 2012, 415(2), 318—328
[30] Botuyan M. V., Lee J., Ward I. M., Kim J. E., Thompson J. R., Chen J., Mer G.,Cell, 2006, 127(7), 1361—1373
[31] Wigle T. J., Herold J. M., Senisterra G. A., Vedadi M., Kireev D. B., Arrowsmith C. H., Frye S. V., Janzen W. P.,J.Biomol.Screen., 2010, 15(1), 62—71
[32] James L. I., Korboukh V. K., Krichevsky L., Baughman B. M., Herold J. M., Norris J. L., Jin J., Kireev D. B., Janzen W. P., Arrowsmith C. H., Frye S. V.,J.Med.Chem., 2013, 56(18), 7358—7371
[33] Wiseman T., Williston S., Brandts J. F., Lin L. N.,Anal.Biochem., 1989, 179(1), 131—137
(Ed.: P, H, F, K)
Design and Synthesis of Pyrimido[4,5-d]pyrimidin-4(3H)-ones as Selective L3MBTL1 Binders†
† Supported by the National Natural Science Foundation of China(No.81072526) and the Science and Technology Development Plan of Jilin Province, China(No.20140309010YY).
ZHOU Hao, CHE Xin, PENG Li, WANG Na, BAI Xu*
(TheCenterforCombinatorialChemistryandDrugDiscovery,SchoolofPharmaceuticalScience,JilinUniversity,Changchun130021,China)
Histone methylation is a key epigenetic mark, and its deregulation is linked to many diseases. Malignant brain tumor(MBT) domain protein is one of the proteins that could read methylated lysine(Kme) of histones. Lethal 3 malignant brain tumor 1(L3MBTL1), a member of the MBT family, is related to transcriptional repression, erythroid differentiation and tumor formation. Developing a potent and selective inhibitor of L3MBTL1 can help explain the regulatory mechanisms and validate its drugability. UNC669 was reported as the first small molecule ligands for a methyl-lysine binding domain. A series of compounds was synthesized by replacing its aromatic component with pyrimido[4,5-d]pyrimidin-4(3H)-ones. Screening these compounds by AlphaScreen® led to a hit compound 8a(IC50=1.21 μmol/L). Exploring 5-position of compound 8a generated three selective and potent L3MBTL1 binders(compounds 8g, 8o and 8p), which showed no significant activity against the other Kme reader proteins in the panel, even its closely related MBT containing proteins, L3MBTL3.
Pyrimido[4,5-d]pyrimidin-4(3H)-ones; Lethal 3 malignant brain tumor 1; Selective binder
2016-04-01.
日期: 2016-06-17.
国家自然科学基金(批准号: 81072526)和吉林省科技发展计划重点项目(批准号: 20140309010YY)资助.
10.7503/cjcu20160203
O621
A
联系人简介: 柏 旭, 男, 博士, 教授, 博士生导师, 主要从事多样性导向合成与创新药物研究. E-mail: xbai@jlu.edu.cn
