四川牡丹和圆裂四川牡丹遗传多样性的ISSR分析
2016-12-17谢登峰曾心美何兴金
童 芬,谢登峰,曾心美,何兴金
(四川大学 生命科学学院 生物资源与环境教育部重点实验室,成都 610064)
四川牡丹和圆裂四川牡丹遗传多样性的ISSR分析
童 芬,谢登峰,曾心美,何兴金*
(四川大学 生命科学学院 生物资源与环境教育部重点实验室,成都 610064)
利用9条ISSR引物对四川牡丹(Paeoniadecomposita)5个居群和圆裂四川牡丹(Paeoniadecompositasubsp.rotundiloba)4个居群的遗传多样性进行了初步检测。结果表明:(1)四川牡丹在物种水平上的多态位百分率(PPB)为89.36%,Nei’s基因多样度(H)为0.291 4,Shannon’s多样性信息指数(I)为0.441 3;圆裂四川牡丹在物种水平上的多态位百分率(PPB)为81.91%,Nei’s基因多样度(H)为0.285 7,Shannon’s多样性信息指数(I)为0.428 5;圆裂四川牡丹的各项多样性参数略低于四川牡丹,而且居群水平上,四川牡丹与圆裂四川牡丹的遗传多样性相对较低。(2)四川牡丹和圆裂四川牡丹居群间遗传分化系数(Gst)分别为0.355 5和0.379 1,2个物种居群内的遗传分化都大于居群间的遗传分化;这2个物种居群间的基因流(Nm)非常有限,其中四川牡丹为0.906 5,圆裂四川牡丹为0.819 0。(3)经Mantel检测,四川牡丹居群间遗传距离与地理距离具有显著正相关关系(r=0.776,P=0.01),而圆裂四川牡丹则不存在显著相关性(r=-0.344,P=0.402)。(4)UPGMA聚类结果显示,四川牡丹与圆裂四川牡丹在分子水平上出现了明显分化,二者为相互独立的物种。研究表明,四川牡丹受到的人为干扰和自然灾害是其濒危的重要原因,与四川牡丹相比,圆裂四川牡丹居群受到的破坏更为严重;建议及时就地保护四川牡丹和圆裂四川牡丹居群的所有个体,而在迁地保护时,因遗传变异主要存在于居群内,应尽可能大量采样,达到最大限度保存其遗传多样性的目的。
四川牡丹;圆裂四川牡丹;遗传多样性;ISSR
四川牡丹(Paeoniadecomposita)和圆裂四川牡丹(Paeoniadecompositasubsp.rotundiloba)隶属于芍药科(Paeoniaceae)芍药属(Paeonia)。四川牡丹是多年生落叶小灌木,花期4~5月,果期8月;野生居群分布在四川西北部的金川大渡河一带,其分布区干旱少雨,温差较大,在阳坡灌木丛中常见,少见于林内。四川牡丹也是中国特有的珍稀濒危物种,1992年被列为国家二级保护植物[1]。而圆裂四川牡丹则分布在四川西北部岷江流域的黑水、松潘、理县、茂县等地,花果期同四川牡丹,在海拔1 600~2 700 m都有分布,其生长环境大多是灌木丛中或林下。其濒危级别目前还未有相关文章报道。
四川牡丹和圆裂四川牡丹两者在形态上十分相似,根据《Flora of China》的描述,它们的差别主要在于心皮数、顶端小叶形状及顶端裂片的长宽比不同。四川牡丹的心皮数大多5枚,顶端小叶椭圆形到卵形;圆裂四川牡丹心皮数3(或2)或4(或5)枚,顶端小叶卵形到卵圆形(图1)。在较早时期只记录有四川牡丹,1997年,洪德元先生根据形态特征的差异,划分出其亚种圆裂四川牡丹[2],随后又根据分子方面证据将圆裂四川牡丹确立为一个独立的种[3]。前人的研究主要集中于四川牡丹,包括分类学[4-5]、生态学[6]、遗传学[7]、种子萌发[8-9]、生理生化等方面[10-11],而对圆裂四川牡丹的研究仅仅涉及到其系统进化方面[12]。牡丹雍容华贵,不仅具有极高的观赏价值,更有巨大的药用价值[13];四川牡丹作为牡丹的一个重要品种,是可贵的栽培资源,现已被引种至河南洛阳、重庆垫江、甘肃天水等地[14];此外,四川牡丹在油用牡丹生产与育种上也具有很大的开发价值[15]。然而作为一种具有重要的观赏和应用价值的物种,其生境已经受到人为和自然的严重破坏,如采挖、放牧、土地利用、滑坡等,这些都严重危及到它们的生存环境,对野生居群的保护和管理迫在眉睫。
拥有足够多的遗传变异性能让一个物种适应环境条件改变而产生的新的选择压力,这样一个物种才能长期存活繁衍下去[16]。因此,检测遗传结构能够帮助我们评估一个物种能否长期存活下去并且是制定保护策略的第一步。ISSR(inter-simple sequence repeat)分子标记又被称作简单重复序列多态性扩增,有样品用量少、快速灵敏和成本低等优点,且稳定性高、实验重复性好[17-18],近年来被广泛运用[19-21]。本研究采用ISSR技术分析四川牡丹5个居群和圆裂四川牡丹4个居群的遗传多样性,以阐明它们的遗传结构和遗传多样性水平,虽然圆裂四川牡丹没有列入濒危名录,但希望通过该研究予以重视,加强保护,为科学制定保护策略提供依据。

图1 四川牡丹和圆裂四川牡丹形态(花期与果期)Fig.1 The morphology of P. decomposita and P. decomposita subsp. rotundiloba(flowering and fruiting stage)
1 材料和方法
1.1 实验材料
从国家标本平台和中国数字植物标本馆查询获得四川牡丹和圆裂四川牡丹分布信息。2015年5~6月野外采集样品材料(表1,图2),本研究的样品按照居群采样,为避免居群内个体被重复取样,个体间相距10 m以上。因为居群较小,每个居群取样数5~16个个体不等。野外采集的植株幼嫩叶片,放于硅胶中及时干燥待用。
1.2 实验方法
1.2.1 ISSR引物筛选和PCR扩增 取叶片1~2片,用植物基因组DNA试剂盒(天根生化科技有限公司,北京)提取总DNA,用1%琼脂糖凝胶电泳检测DNA的质量,紫外分光光度法检测DNA的质量浓度,最后稀释标定到20 ng/μL,放入-20 ℃冰箱保存备用。
所用引物购自华大基因有限公司,源自于哥伦比亚大学University of British Columbia(简称UBC大学)公布的100条ISSR引物。通过查阅四川牡丹及其近缘科属ISSR-PCR实验的相关文献,初步筛选出多态性高、稳定性好的引物9条(表 2)。从每个居群中随机抽选1个样品对这9条引物进行退火温度及扩增条件的优化实验。PCR扩增体系为20 μL,其中含3 μL DNA,引物1 μL,混合MIX(购自康为公司)10 μL,不足的用ddH2O补齐。PCR反应程序为94 ℃预变性3 min;94 ℃变性45 s,47~57 ℃退火45 s,72 ℃延伸90 s,共40个循环;72 ℃延伸7 min。扩增产物以DL2000作为分子量标准,用含有Gold View的1.5%琼脂糖凝胶于120 V恒压电泳80 min。电泳结束后用凝胶成像系统仪(Bio-Rad GelDocXR+imaging system)进行拍照保存。

居群编号同表1图2 四川牡丹和圆裂四川牡丹的实地(中国四川)采样分布图Population codes are given in Table 1Fig.2 The distribution of the samples in Sichuan of China

引物Primer引物序列Sequenceofprimer退火温度Annealingtemperature/℃UBC807(AG)8T50UBC810(GA)8T51UBC811(GA)8C50UBC812(GA)8A50UBC835(AG)8YC48.9UBC840(GA)8YT49UBC844(CT)8RC50.7UBC866(CTC)651UBC873(GACA)453
Note: Y =C/T; R=A/G
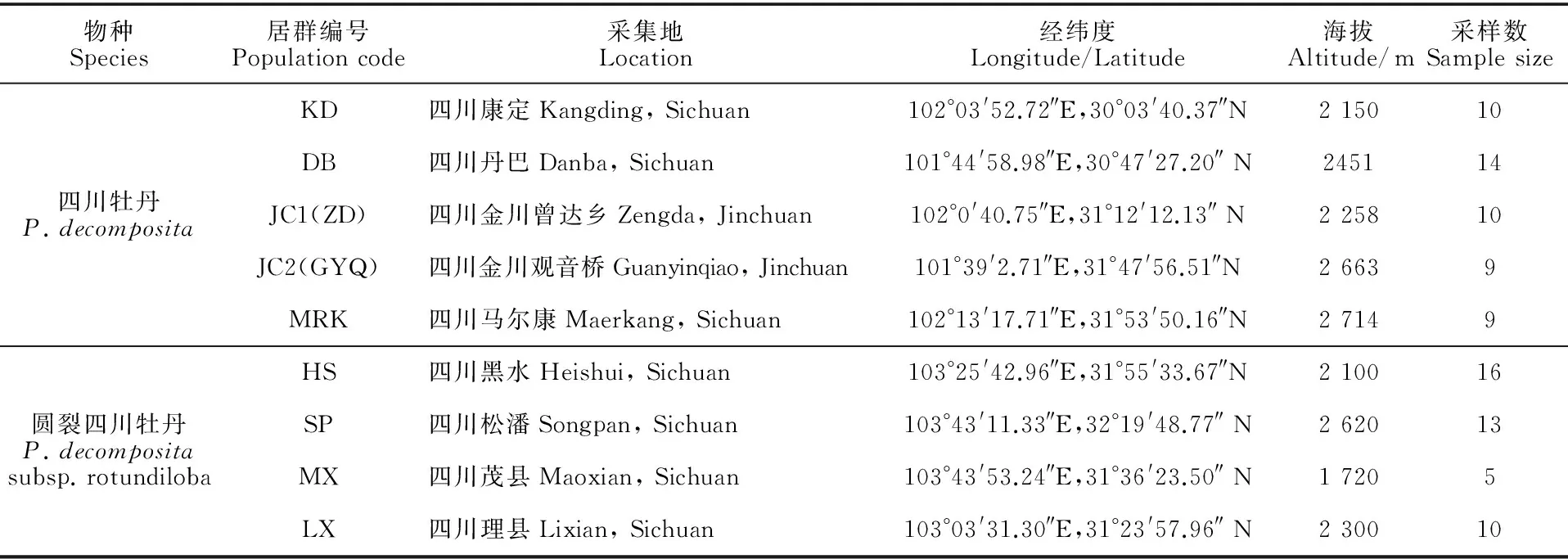
表1 四川牡丹和圆裂四川牡丹居群的地理位置和采样数
1.2.2 数据处理与统计分析 电泳结果根据DL2000(由康为公司提供DNA Marker)判读扩增条带的有无及大小,同一位点有条带赋值“1”,无条带赋值“0”。采用Excel软件对电泳结果进行数据转化,构建0/1数据矩阵。利用POPGENE1.32[22]软件对数据进行分析获得所需的多态性位点等遗传参数,包括多态位百分率(PPB)、基因多样性(H)、Shannon多样性信息指数(I)、平均每个位点上的观察等位基因数(Na)、平均每个位点的有效等位基因(Ne)、总基因多样度(Ht)、居群内的基因多样度(Hs)、居群间的遗传分化系数(Gst)、居群间的基因流(Nm),Nei’s遗传距离(D)和遗传一致度(Hi)[23-25];运用NTSYSpc2.1软件进行UPGMA法聚类分析,构建树状图[26]。同时采用AMOVA1.55[27]软件分析遗传变异在居群内及居群间的分布情况。运用TFPGA1.3软件进行Mantel检测[28],检测居群之间的遗传距离与地理距离之间的关系。为了揭示居群之间的遗传差异及相关性,使用STRUCTUREv2.3软件[29]计算四川牡丹和圆裂四川牡丹居群的遗传结构。软件参数设置为:burn-in,5×104;MCMC,105。并进行6次随机运算(K值设置为2 ~ 8)。
2 结果与分析
2.1 2类牡丹的遗传多样性
用筛选出的9条引物(表2)对居群进行PCR扩增,所得片段在100~2 000 bp之间,共扩出94条清晰可重复的条带,其中多态性条带四川牡丹84条,圆裂四川牡丹77条。结果(表3)显示:在居群水平上,四川牡丹各居群的PPB范围为41.49% ~ 62.77%,平均值为51.06%,表明四川牡丹居群的遗传多样性并不是很低。在物种水平上,四川牡丹的遗传多样性PPB=89.36%,Ne=1.493 9±0.352 5,H=0.291 4±0.170 7,I=0.441 3±0.226 5。假设种群处于Hardy-Weinberg平衡,5个居群平均有效等位基因为1.321 4,平均Nei’s基因多样性指数为0.187 1,各参数均略低于物种水平。Shannon多样性信息指数显示了各居群遗传变异由高到低依次是金川曾达、丹巴、金川观音桥、康定、马尔康,与PPB值分析一致,居群间遗传多样性参数与四川牡丹类似,圆裂四川牡丹遗传多样性水平在物种和居群两个水平表现不同(表3)。圆裂四川牡丹在居群水平上各个居群的PPB为32.98% ~ 63.83%,平均值为45.75%。其中多态位点最高的是黑水居群为63.83%,而理县居群的最低。在物种水平上,圆裂四川牡丹的PPB、Ne、H和I分别是81.91%、1.485 4±0.352 5、0.285 7±0.178 2和0.428 5±0.246 1。同样假设种群处于Hardy-Weinberg平衡,4个居群的平均有效等位基因为1.303 4,平均Nei’s基因多样性指数为0.174 0。Shannon多样性信息指数显示了各居群的遗传变异由高到低依次是黑水、茂县、松潘、理县,Shannon多样性信息指数与PPB结果一致,黑水居群遗传变异最大。
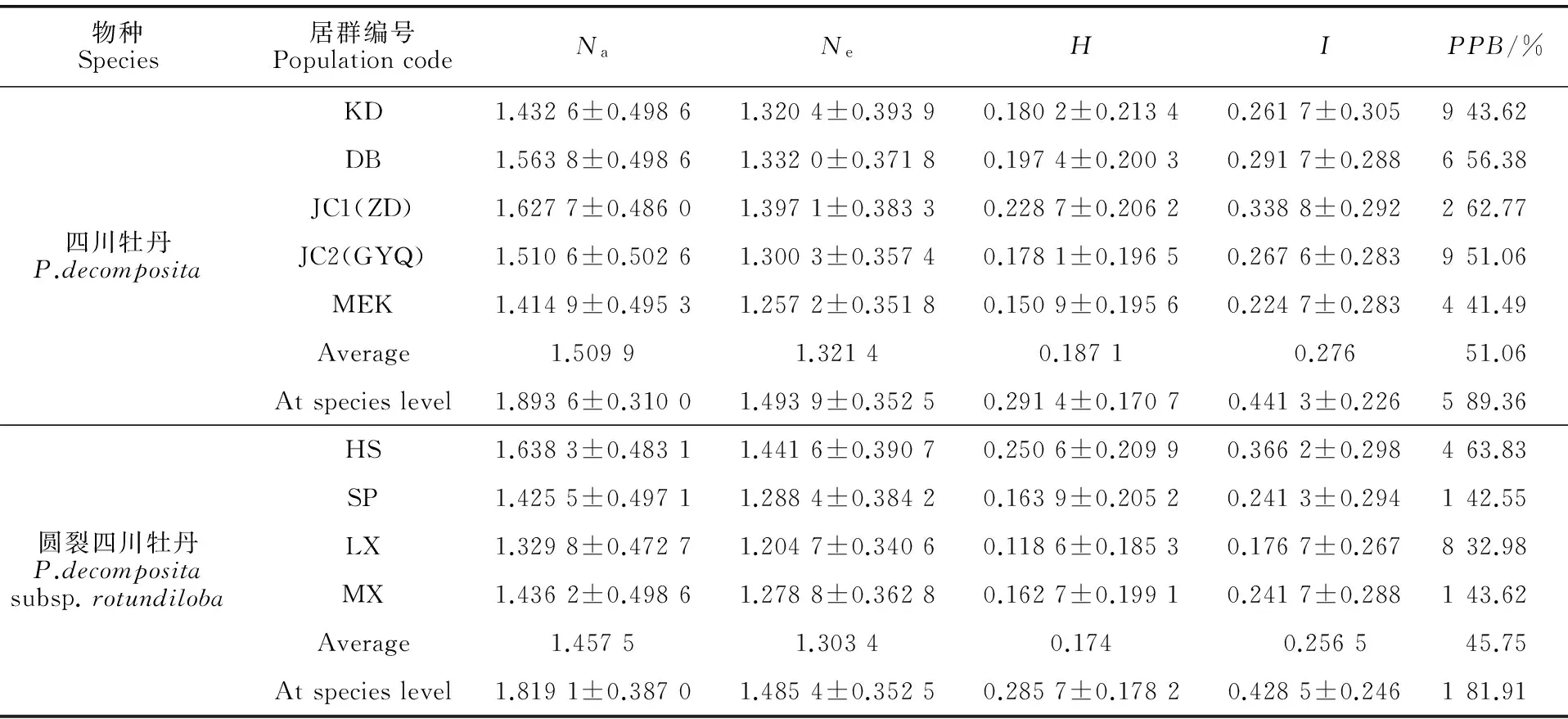
表3 2类牡丹的遗传多样性
注:居群编号同表1;Na.等位基因观测值;Ne.有效等位基因;H. Nei’s基因多样性;I. Shannon’s指数;PPB.多态位百分率
Note: Population codes are identical to Table 1;Na. Observed number of alleles;Ne. Effective number of alleles;H. Nei’s gene diversity;I. Shannon’s information index;PPB. Percentage of polymorphic loci
Ne最大值为(1.397 1±0.383 3),最小值为(1.257 2±0.351 8);H最大值为(0.228 7±0.206 2),最小值为(0.150 9±0.195 6)。表明四川牡丹居群之间存在一定的遗传差异。
2.2 2类牡丹的遗传分化与基因流
用POPGENE1.32算出四川牡丹5个居群Ht为0.289 4,Hs为0.186 5,Gst为0.355 5(表4),即有35.55%的变异存在于居群间,居群内的变异则占64.45%,居群内的遗传分化大于居群间的分化,且居群间基因交流程度低(0.906 5)。利用AMOVA1.55软件分析表明,在总的遗传变异中,有67.43%变异发生在居群内(P<0.001),32.57%的变异在居群间(表5),与POPGENE分析的结果一致,均表明遗传变异主要存在于居群内。
同样,POPGENE1.32计算出圆裂四川牡丹4个居群Ht为0.280 1,Hs为0.173 9,Gst为0.379 1,Nm为0.819 0(表4),基因流小于1,表明居群间基因流少,居群间遗传分化系数为0.379 1,遗传变异主要存在居群内。AMOVA分析结果显示(表5),总变异中34.75%的变异在居群间,表明居群间存在一定程度的遗传分化。
2.3 2类牡丹聚类分析
根据POPGENE计算得出的结果,四川牡丹5个居群两两居群的Nei’s遗传一致度(Hi)范围为0.772 4~0.93 21,遗传距离(D)范围为0.070 3~0.258 3(表6)。
四川牡丹5个居群Nei’s遗传距离都较小,表明地理亲缘很近。金川曾达(ZD)和金川观音桥(GYQ)的遗传一致度最高(Hi=0.932 1)且遗传距离最近(D=0.070 3),聚类分析中也聚在一起;康定(KD)和马尔康(MEK)居群的遗传一致度最低(Hi=0.772 4),遗传距离最远(D=0.258 3),在UPGMA聚类分支中也最远。四川牡丹的Mantel检测揭示居群间地理距离和遗传距离呈正相关(r=0.776,P=0.01),表明居群间有地理上的隔离。

表4 2类牡丹居群多样性Nei’s分析
注:Ht. 总基因多样性;Hs。居群内基因多样性;Gst. 基因分化系数;Nm. 基因流
Note:Ht.Total gene diversity;Hs. Gene diversity within population;Gst. Coefficient of gene differentiation;Nm. Gene flow,Nm=0.5(1-Gst)/Gst[30].

表5 2类牡丹的AMOVA分析
注:*P值表示比观察值的变异大的概率,该概率是通过把居群中的样本经过1 000次随机排列改变计算得到的
Note: *P-values are the probabilities of having a more extreme variance component than the observed values alone, probabilities were caleulated by 1 000 random permutations of individuals across populations

表6 2类牡丹居群的Nei’s遗传一致度(对角线上方)和遗传距离(对角线下方)
用NTSYS软件对圆裂四川牡丹4个居群进行UPGMA聚类分析(图3),松潘(SP)和茂县(MX)居群遗传一致度最高,遗传距离最近,首先聚在一起,而后与黑水居群聚为一支。Mantel分子检测表明圆裂四川牡丹居群的遗传距离和地理距离呈负相关(r=-0.344,P=0.402)。
POPGENE软件计算出四川牡丹和圆裂四川牡丹居群种间总基因多样度为0.351 9,居群内基因多样度为0.180 9,遗传分化系数为0.485 9。说明遗传变异基本存在居群内。四川牡丹和圆裂四川牡丹种间基因流为0.529 1,表明两个种之间的基因交流水平较低;种间分析结果(表5)显示,两个种间的变异百分率是22.01%,小于种内居群间的变异(26.14%)。Mantel检测显示地理距离和遗传距离呈正相关关系(r=0.473,P=0.01),存在隔离,聚类分析结果也支持两者是独立的物种。
基于Nei’s遗传距离的主成分分析中(图4),大体分为两组,四川牡丹为一组,圆裂四川牡丹为另一组;种内的分组为:四川牡丹的康定和丹巴居群为一组,金川曾达、金川观音桥、马尔康居群为一组;圆裂四川牡丹的黑水、松潘、茂县居群为一组,理县居群单独为一组,与UPGMA聚类分析结果基本一致。STRUCTURE软件基于贝叶斯聚类分析显示当K=2时是最合适的分组(图5),即四川牡丹居群为一组,圆裂四川牡丹居群为另一组。
3 讨 论
3.1 遗传多样性
遗传多样性是物种生存、发展、进化的基础[31]。许多研究表明狭域分布的物种或者濒危的物种仍保留着较高的遗传多样性,例如,与四川牡丹同属的滇牡丹[32]、紫斑牡丹[21]。虽然四川牡丹和圆裂四川牡丹仅狭域分布在四川西北部,但是ISSR分子标记揭示了它们在物种水平上遗传多样性较高,分别是89.36%和81.91%,居群水平上的遗传多样性也不太低。
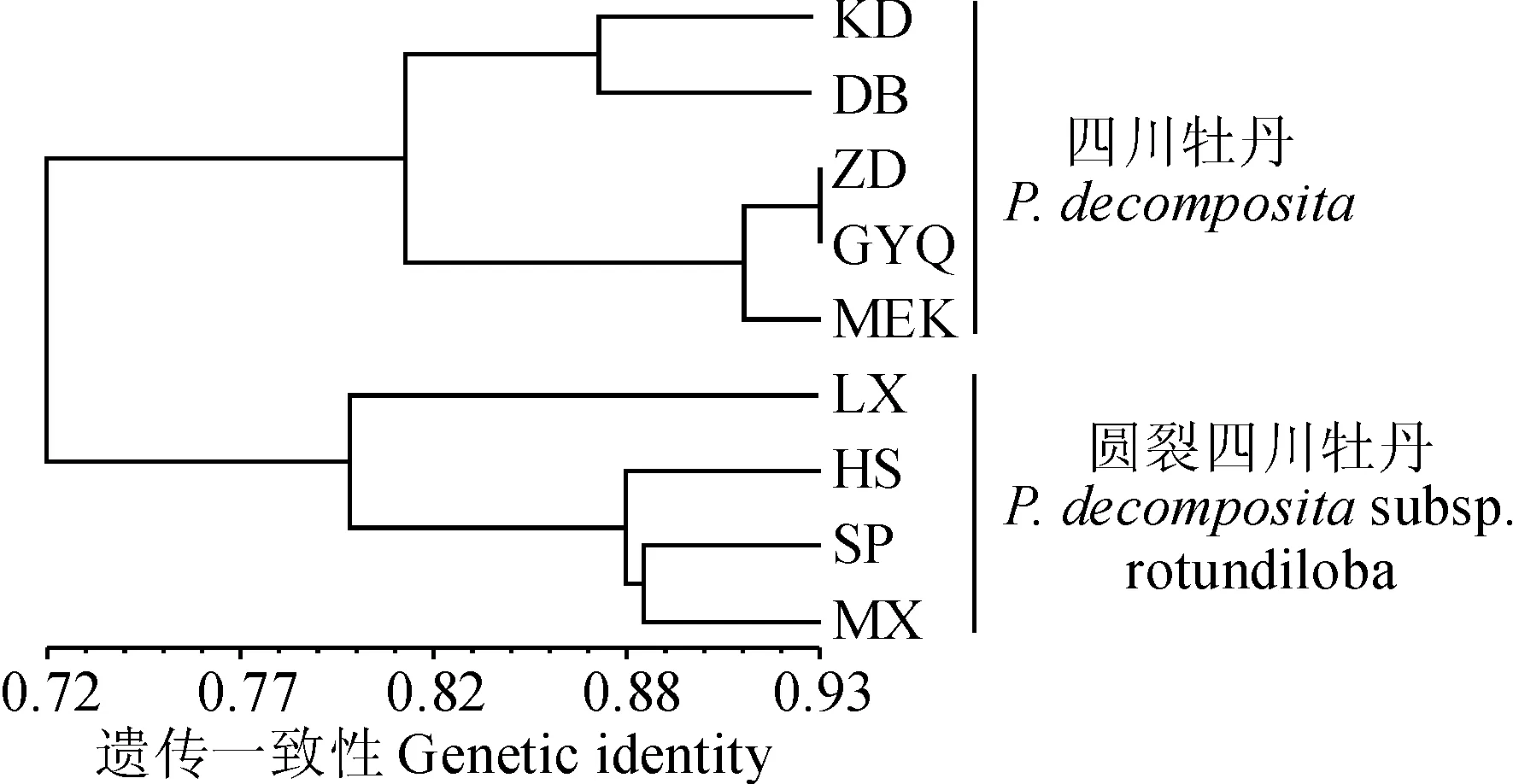
居群编号同表1 图3 2类牡丹居群的UPGMA聚类Population codes are given in Table 1Fig.3 Dendrogram of two tree peonies’ populations

图4 2类牡丹居群主成分分析Fig.4 Principal coordinate analysis(PCoA) of two tree peonies populations

图5 基于Structure的两类牡丹的遗传结构(K=2)Fig.5 Bayesian model-based clustering (Structure) of two tree peonies(optimal value of K=2)
濒危物种遗传多样性高低与很多因素有关,如繁育系统、生活史特性、分布范围、基因流、遗传漂变和自然选择等[33-34]。普遍认为,繁育系统和种子传播方式是影响遗传多样性高低的主要因素。有研究表明长寿命、异交、广布种、动物传播种子都会使物种具有高的遗传多样性[35]。该研究中四川牡丹和圆裂四川牡丹所表现出来的遗传多样性水平可能归于繁育系统、种子传播方式及生活史特性。四川牡丹花朵大,色彩鲜艳,气味浓,具有明显的虫媒传粉特征,骆劲涛在其传粉生物研究中发现四川牡丹的繁育系统以异交为主[36],而这样的繁育系统有利于保持高的遗传多样性和基因流。牡丹类植物种子主要靠重力传播[37],果实成熟后果壳破裂种子散落在母树周围,传播距离有限,而有研究证明靠重力传播种子的特有种遗传多样性偏低[38],也说明了四川牡丹与圆裂四川牡丹在居群水平上遗传多样性相对较低的原因。有文献记载四川牡丹曾广泛分布[39],且四川牡丹和圆裂四川牡丹都是多年生灌木,世代重叠,据此推测它们的遗传基础可能较丰富。尽管它们的繁育系统及生活史特性有一定的抵抗环境压力的优势,但是,随自然(如滑坡、地震)和人为(如采挖、放牧)的干扰而形成了分散的居群,基因交流受到限制,四川牡丹和圆裂四川牡丹仍临着灭绝的危险。
3.2 遗传分化
四川牡丹与圆裂四川牡丹种下居群间的Nei’s基因分化系数Gst分别为0.355 5和0.379 1,与AMOVA分析结果一致,种下居群间出现一定程度的遗传分化,由此认为四川牡丹和圆裂四川牡丹各自的遗传变异主要存在于居群内。
引起物种遗传分化的原因有很多,如繁育系统、遗传漂变、居群间遗传隔离等[40]。一般来说,自交植物的遗传变异大都存在居群间,异交植物的遗传变异大部分存在居群内[41]。Nybom和Bartish得出自交、混合交配、异交植物的平均Gst分别是0.59、0.19、0.23[42],据此推测四川牡丹和圆裂四川牡丹的繁育系统属于异交型,与骆劲涛[36]关于四川牡丹传粉生物学研究一致,并且可能是居群间遗传分化较小的主要原因。如果居群小且居群间存在隔离,遗传漂变就会影响遗传结构并加强居群间遗传分化[43-44]。据野外实地调查且文献记录四川牡丹和圆裂四川牡丹居群内个体数量都极为有限[38],很容易引起遗传漂变,加剧遗传分化的发生。根据Mantel检测结果显示四川牡丹居群间遗传距离和地理距离呈正相关(r=0.776,P=0.01),表明地理隔离对四川牡丹居群遗传分化有一定影响。这可能是由于四川西北特殊的地形如地形复杂、山脉多、海拔高,导致了居群间存在一定的隔离而引起居群间产生分化。基因流对种群的遗传分化也有很重要的影响。居群遗传学理论认为,当基因流小于1时,就不足以抵抗居群内因遗传漂变导致的遗传分化[45],即居群间基因流受到限制时,短距离的居群就会出现遗传分化[46]。本研究表明四川牡丹与圆裂四川牡丹种下居群间的基因流分别是0.906 5和0.819 0,都小于1,表明居群间的基因交流有限,从而导致居群出现遗传分化。
聚类分析结果显示四川牡丹和圆裂四川牡丹种间出现了明显的分化,聚为两大支,可能是长时间的地理隔离导致的,支持两者是独立的物种。由于居群数少且居群内个体少,采集到的样本较少,因此在统计分析中可能存在一些偏差。
3.3 保护策略
经过野外实地调查,发现四川牡丹的自然生长环境已经遭受严重破坏,居群个体数量少,圆裂四川牡丹受到的破坏也较严重,例如,丹巴居群遭受滑坡影响,居群内个体零星分布;马尔康居群受到人为的砍伐;黑水居群遭受修公路破坏;松潘居群受放牧影响,种子大部分被动物啃食,实生苗较少;理县大沟口村居群的破坏更严重,据当地村民介绍,曾有很多圆裂四川牡丹分布,由于修电站,居群个体大面积被淹没。除了自身特性外,推测人为干扰和自然灾害也是其濒危的重要原因,四川牡丹和圆裂四川牡丹分布本就狭窄,居群又少,加上各种程度的人为和自然的破坏,导致濒危程度加剧。它们可能是重要的野生牡丹种质资源,急需制定保护策略保护自然居群,进而保持现有的物种多样性,防止居群退化甚至灭绝[47]。
根据遗传结构及野外考察,建议采取以下保护措施:(1)保护现有居群及生境是保护珍稀濒危植物的最好最有效的措施,减少对四川牡丹和圆裂四川牡丹生态环境的破坏,严禁非法采挖,建立保护区和种质资源库。四川牡丹种子萌发率不是很低,但四川牡丹和圆裂四川牡丹生长在灌木丛中,受到群落内其他优势种的竞争排斥,在减少破坏的情况下,适当降低其他植物的郁闭度。(2)通过对遗传结构的分析,物种水平上仍保留较高的遗传多样性,居群水平上四川牡丹遗传多样性最高的是金川曾达乡居群,最低的是马尔康居群,圆裂四川牡丹遗传多样性最高的是黑水居群,最低的是理县居群,相关部门应对这些居群予以重视,建立保护区或保护点,如若加以重视和保护,四川牡丹及圆裂四川牡丹种群仍有恢复的潜能。(3)四川牡丹和圆裂四川牡丹的遗传变异主要存在居群内,在迁地保护时,应最大限度采样,防止遗传多样性的丢失。
[1] 傅立国,金鉴明.中国植物红皮书——稀有濒危植物[M].北京:科学出版社,1992:530-537.
[2] HONG D Y.Notes onPaeoniadecompositaHand.-Mazz [J].KewBulletin,1997,52(4):957-963.
[3] HONG D Y.Paeoniarotundiloba(D.Y.Hong) D.Y.Hong: A new status in tree peonies (Paeoniaceae) [J].JournalofSystematicsandEvolution,2011,49(5):464-467.
[4] 林启冰,周志钦,赵 宣,等.基于Adh基因家族序列的牡丹组(Sect.MoutanDC.)种间关系[J].园艺学报,2004,31(5):627-632.
LIN Q B,ZHOU Z Y,ZHAO X,etal.Interspecific relationships among the wild species ofPaeoniaSect.MoutanDC. based on DNA sequences ofAdhgene family[J].ActaHorticulturaeSinica,2004,31(5):627-632.
[5] 张金梅,王建秀,夏 涛,等.基于系统发育分析的DNA条形码技术在澄清芍药属牡丹组物种问题中的应用[J].中国科学:C辑:生命科学,2008,38(12):1 166-1 176.
ZHANG J M, WANG J X, XIA T,etal. Application of clarification the species problem ofPaeoniaSect. Moutan DC, based on the phylogenetic analysis of DNA barcode technology[J].ScienceinChina: Series C: Life Science, 2008, 38(12): 1 166-1 176.
[6] 马 莘,夏 颖,马庆庆,等.四川牡丹群落物种生态位及空间关联度分析[J].重庆师范大学学报(自然科学版),2011,28(4):57-61.
MA X, XIA Y, MA Q Q,etal. An analysis on niche and degree of spatial association ofPaeoniadecompositacommunity[J].JournalofChongqingNormalUniversity(Natural Science), 2011, 28(4):57-61.
[7] 王士泉,张大明.四川牡丹和块根芍药第五号染色体异常的减数分裂证据[J].植物分类学报,2008,46 (2):155-162.
WANG S Q,ZHANG D M.Meiotic abnormalities of the fifth pair of chromosomes inPaeoniadecompositaandP.intermedia[J].JournalofSystematicsandEvolution,2008,46(2):155-162.
[8] 景新明,郑光华,裴颜龙,等.野生紫斑牡丹和四川牡丹种子萌发特性及与其致濒的关系[J].生物多样性,1995, 3(2):84-87.
JING X M,ZHENG G H,PEI Y L,etal.A study on the relationship between seed germination of wildPaeoniarockiiandP.szechuanicaand their endangerment[J].ChineseBiodiversity,1995,3(2):84-87.
[9] JING X M,ZHENG G H.The characteristics in seed germination and dormancy of four wild species of tree peonies and their bearing on endangerment[J].ActaPhytophysiologicaSinica,1999,25(3):214-221.
[10] 宋会兴,刘光立,高素萍,等.四川牡丹种子浸提液内源抑制物活性初探[J].园艺学报,2012,39(2):370-374.
SONG H X,LIU G L,GAO S P,etal.Effects of crude extracts ofPaeoniadecompositaseeds on germination and activities of antioxidant enzyme ofBrassicapekinensis[J].ActaHorticulturaeSinica,2012,39(2):370-374.
[11] 杨 勇,刘光立,宋会兴,等.四川牡丹胚乳浸提液对油菜种子萌发与幼苗生长的影响[J].西南农业学报,2013,26(1):89-92.
YANG Y,LIU G L,SONG H X,etal.Effects ofPaeoniadecompositaendosperm extracts on seeds germination and seedling growth ofBrassicacampestris[J].SouthwestChinaJournalofAgriculturalSciences,2013,26(1):89-92.
[12] ZHAO X,ZHOU Z Q.Phylogenetic analysis ofPaeoniasect.Moutan (Paeoniaceae) based on multiple DNA fragments and morphological data [J].JournalofSystematicandEvolution,2008,46(4):563-572.
[13] 李嘉珏.中国牡丹与芍药[M].北京:中国林业出版社,1999.
[14] YUAN J H,CORNILLE A,GIRAUD T,etal.Independent domestications of cultivated tree peonies from different wild peony species[J].MolecularEcology,2014,23(1):82-95.
[15] 杨 勇,刘佳坤,曾秀丽,等.四川牡丹部分野生居群种子脂肪酸组分比较[J].园艺学报,2015,42(9):1 807-1 814.
YANG Y,LIU J K,ZENG X L,etal.A comparative study on composition of seed oil fatty acids of some wild populations ofPaeoniadecomposita[J].ActaHorticulturaeSinica,2015,42(9):1 807-1 814.
[16] VRIJENHOEK R C.Genetic diversity and fitness in small populations[M]// LOESCHCKE V, TOMIUK J. TIAN S K. Conservation Genetics.Basel: Birkhäuser,1994:37-53.
[17] 周亚星,周 伟.ISSR分子标记技术在作物遗传育种中的应用[J].内蒙古民族大学学报(自然科学版),2011,26(6):682-684.
ZHOU Y X,ZHOU W.ISSR marker and its application in crop genetics and breeding[J].JournalofInnerMongoliaUniversityforNationalities(Nat. Sci Edi), 2011,26(6):682-684.
[18] 杨淑达,施苏华,龚 洵,等.滇牡丹遗传多样性的ISSR分析[J].生物多样性,2005,13(2):105-111.
YANG S D,SHI S H,GONG X,etal.Genetic diversity ofPaeoniadelavaias revealed by ISSRs[J].BiodiversityScience,2005,13(2):105-111.
[19] 邹喻苹,葛 颂,王晓东.系统与进化植物学中的分子标记[M].北京:科学出版社,2001:16-17.
[20] 索志立,张会金,张治明,等.紫斑牡丹与牡丹种间杂交后代的DNA分子证据[J].植物分类与资源学报,2005,27(1):42-48.
SUO Z L,ZHANG H J,ZHANG Z M,etal.DNA molecular evidences of the hybrids betweenPaeoniarockiiandP.suffruticosabased on ISSR markers[J].ActaBotanicaYunnanica,2005,27(1):42-48.
[21] 杨美玲,唐 红.紫斑牡丹遗传多样性的ISSR分析[J].西北植物学报,2012,32(4):693-697.
YANG M L,TANG H.Genetic diversity ofPaeoniarockiirevealed by ISSRs[J].ActaBotanicaBoreali-OccidentaliaSinica,2012,32(4):693-697.
[22] YEH F C,YANG R C,BOYLE T B J,etal.POPGENE:the user friendly shareware for population genetic analysis(Ver.1.32)[M].Canada:Moleeular Biology and Biotechnology Centre,University of Alberta,1997.(program available from:http://www.ualbreta.ca/~fyeh/)
[23] NEIM.Analysis of gene frequencies in subdivided populations[J].ProceedingsoftheNationalAcademyofSciences,1973,70 (12):3 321-3 323.
[24] NEIM.Estimation of average heterozygosity and genetic distance from a small number of individuals[J].Genetics,1978,89:583-590.
[25] NEIM.Molecular Evolutionary Genetics [M].New York:Columbia University Press,1987:187-192.
[26] ROHLFFJ.NTSYS-pc2.0 Numerical Taxonomy and Multivariate Analysis System.Exeter Software [CP/DK].New York,1998.
[27] EXCOFFIER L.Analysis of Molecular Variance (AMOVA) Version1.55 [CP/DK].Switzerland:Genetics and Biometry Laboratory,University of Geneva,1995.
[28] MILLER M P.Tools for population Genetic Analysis (TFPGA) Version1.3 [M].Department of biological sciences,Northern Arizona University, Arizona,USA,1997.
[29] PRITCHARD J K,STEPPHENS M,DONNELLY P.Inference of population structure using multilocus genotype data[J].Genetics,2000,7(4):574-578.
[30] MCDERMOTT J M,MCDONALD B A.Gene flow in plant pathosystems[J].AnnualReviewofPhytopathology,1993,31:353-373.
[31] 陈灵芝.中国的生物多样性——现状及其保护对策[M].北京:科学出版社,1993:11-15.
[32] 任秀霞,张 盈,薛璟祺,等.滇牡丹天然居群的遗传多样性分析[J].植物遗传资源学报,2015,16(4):772-780.
REN X X,ZHANG Y,XUE J Q,etal.Genetic diversity analysis of natural populations inPaeoniadelavayi[J].JournalofPlantGeneticResources,2015,16(4):772-780.
[33] HAMRICK J L,GODT M J W,BROWN A H D,etal.Allozyme Diversity in Plant Species[M].1990:43-63.
[34] HE Y P,LIU J Q.A review on recent advances in the studies on plant breeding system[J].ActaPhytoecologicaSinica,2003,27(2):151-163.
[35] HAMRICK J L,LOVELESS M D.The genetic structure of tropical tree populations:Associations with reproductive biology[M]//BOCKJ H,LINHART Y B.Plant Evolutionary Ecology.Westview Press,Boulder,Colorado,1989:129-146.
[36] 骆劲涛.四川牡丹的传粉生物学研究[D].成都:四川农业大学,2013.
[37] 罗毅波,裴颜龙,潘开玉,等.矮牡丹传粉生物学的初步研究[J].植物分类学报,1998,36(2):134-144.
LUO Y B,PEI Y L,PAN K Y,etal.A study on pollination on biology ofPaeoniasuffruticosasubsp.spontanea(Paeoniaceae)[J].ActaPhytotaxonomicaSinica,1998,36(2):134-144.
[38] HAMRICK J L,GODT M J W,SHERMAN-BROYLES S L.Factors influencing levels of genetic diversity in woody plant species[J].NewForest,1992,6(1-4):95-124.
[39] 杨 勇,张姗姗,刘佳坤,等.西南野生牡丹的资源调查、濒危机制及利用分析[C]//张启翔.中国观赏园艺研究进展.北京:中国林业出版社,2015.
[40] BROWN A H D.Enzyme polymorphism in plant populations[J].TheoreticalPopulationBiology,1979,15(1):1-42.
[41] HOGBIN, PATRICIA M, PEAKALL R. Evaluation of the conservation of genetic research to the management of endangered plantZieriaprostrata[J].ConservationBiology, 1999, 13: 514-522.
[42] NYBOM H,BARTISH I V.Effects of life history traits and sampling strategies on genetic diversity estimates obtained with RAPD markers in plants[J].PerspectivesinPlantEcologyEvolutionandSystematics,2000,3(2):93-114.
[43] BARRETT S C H,KOHN J R,etal.Genetic and evolutionary consequences of small population size in plants:implications for conservation[M]//FALK D A, HOLSINGER K E. Genetics and Conservation of Rare Plants. New York: Oxford University Press.,1991: 3-30.
[44] ELLSTRAND N C,ELAM D R.Population genetic consequences of small population size:implication for plant conservation[J].AnnualReviewofEcologyandSystematics,1993,8:238-244.
[45] SLATKIN M.Gene flow in natural populations[J].AnnualReviewofEcologyandSystematics,1985,16:393-430.
[46] HEYWOOD J S.Spatial analysis of genetic variation in plant populations[J].AnnualReviewofEcologyandSystematics,2003,22(1):335-355.
[47] 李 昂,葛 颂.植物保护遗传学研究进展[J].生物多样性,2002,10(1):61-71.
LI A,GE S.Advances in plant conservation genetics[J].BiodiversityScience,2002,10(1):61-71.
(编辑:宋亚珍)
Genetic Diversity of Paeonia decomposita and Paeonia decomposita subsp. rotundiloba Detected by ISSR Markers
TONG Fen, XIE Dengfeng, ZENG Xinmei, HE Xingjin*
(Key Laboratory of Bio-Resources and Eco-Environment of Ministry of Education, College of Life Sciences, Sichuan University, Chengdu 610064, China)
The genetic diversity of 5 populations ofPaeoniadecompositaand 4 populations ofPaeoniadecompositasubsp.rotundilobawas dectected by inter-simple sequence repeat markers(ISSR). (1) ForP.decomposita, the percentage of polymorphic loci(PPB) at the species level, Nei’s gene diversity(H) and Shannon’s information index(I) were 89.36%, 0.291 4, 0.441 3, respectively, while forP.decompositasubsp.rotundiloba, the three indices were 81.91%, 0.285 7, 0.428 5, respectively, all lower than that ofP.decomposita’s. The genetic variation of them were little lower at population level. (2) The coefficient of genetic differentiation among population(Gst) was 0.355 5 forP.decompositaand 0.379 1 forP.decompositasubsp.rotundiloba, suggesting that for both species there is a high genetic differention within population; the gene flow of the two species was 0.906 5 and 0.819 0, respectively and show that gene flow was limited among populations. (3) Mantel test indicated that there was a significant correlation between genetic and geographic distances amongP.decompositapopulations, whereas no significant correlation between genetic and geographic distances ofP.decompositasubsp.rotundilobapopulations. (4) UPGMA analysis showed significant genetic differention between the two species at molecular level, supporting the view that they are two separate species. The study indicated thatP.decompositasuffered human disturbance and natural disaster may be its endangered reasons.P.decompositasubsp.rotundilobapopulations are subjected to severe disruption as well, even more serious thanP.decompositapopulations. So all the individuals of populations should be protectedinsitutimely, and it is as large as possible to gain the sample in order to reserve their genetic diversity owning to the genetic variation mainly exist in population.
Paeoniadecomposita;Paeoniadecompositasubsp.rotundiloba; genetic diversity; ISSR
1000-4025(2016)10-1968-09
10.7606/j.issn.1000-4025.2016.10.1968
2016-07-07;修改稿收到日期:2016-10-13
国家自然科学基金(31470009,31270241,31570198);国家标本平台教学标本子平台(http://mnh.scu.edu.cn/)
童 芬(1990-),女,在读硕士研究生,主要从事生物安全、保护生物学研究。E-mail:tongfen191004@163.com
*通信作者:何兴金,教授,博士生导师,主要从事植物系统和分子进化,保护生物学方面的研究。E-mail:xjhe@scu.edu.cn
Q346.+5;Q789
A
