陆地棉PIP亚家族基因的克隆及表达特征分析
2016-12-17孙雨薇李艳军刘永昌朱守鸿王翔飞
孙雨薇,李艳军*,刘永昌,薛 飞,朱守鸿,王翔飞,孙 杰
(1 石河子大学 农学院,新疆生产建设兵团绿洲生态农业重点实验室,新疆石河子832003;2 石河子大学 药学院,新疆石河子 832003)
陆地棉PIP亚家族基因的克隆及表达特征分析
孙雨薇1,李艳军1*,刘永昌1,薛 飞1,朱守鸿1,王翔飞2,孙 杰1
(1 石河子大学 农学院,新疆生产建设兵团绿洲生态农业重点实验室,新疆石河子832003;2 石河子大学 药学院,新疆石河子 832003)
该研究以陆地棉苯基香豆满苄基醚还原酶( phenylcoumaran benzylic ether reductase,PCBER)氨基酸序列为探针,利用Blastp从陆地棉基因组数据库中发现了6个同源性较高的基因。根据6个基因序列设计引物,利用 RT-PCR技术从陆地棉纤维细胞中克隆出了这6个基因的全长cDNA序列,分别命名为GhPCBER1、GhPCBER2、GhPCBER3、GhIFR、GhPLR1和GhPLR2。多重序列比对和进化树分析发现,6个蛋白均含有PIP类型蛋白的所有保守性基序和活性残基,属于PIP亚家族。实时荧光定量PCR结果显示,除GhPLR1之外其他5个PIP亚家族基因均在纤维细胞中优势或特异表达;在纤维发育过程中,GhPCBER1、GhPCBER2、GhPCBER3和GhIFR的表达均表现为先上升后下降,GhPCBER1和GhPCBER2在花后21 d表达量达到最高,GhPCBER3和GhIFR在花后18 d达到最高,GhPLR1和GhPLR2在纤维中的表达量呈持续上升趋势。根据基因的表达特征,推测PIP亚家族可能在棉纤维的发育过程中发挥着重要作用。
棉花;纤维;PIP;表达分析
棉花是全球性的重要经济作物,是纺织工业最主要的天然纤维来源。成熟棉纤维含有90%以上的纤维素和少量的非纤维素碳水化合物,如木糖葡聚糖、葡聚糖和果胶多糖等[1-2]。纤维素的合成是棉纤维发育过程中主要的代谢途径,近期的研究表明棉纤维中存在苯丙烷代谢途径且在纤维次生壁增厚期优势表达[3-5],是纤维发育过程中仅次于纤维素代谢的第二大代谢途径[4],该途径产生的次生代谢产物可能对棉纤维的发育与品质形成具有重要影响[5-6]。苯丙烷代谢途径是植物最重要的次生代谢途径之一[7],能够产生木质素、木脂素、黄酮、异黄酮、花青素、酚类化合物等次生代谢产物[8-9]。木质素和木脂素是两种不同的物质,木质素是植物体内一种大分子有机物,是具有三维结构的芳香族高聚物,与纤维素、半纤维素合称为“三素”是形成植物骨架的主要成分。木脂素是由苯丙素双分子聚合而成的天然化合物,是一种植物雌激素。在苯丙烷代谢过程中肉桂醛经肉桂醇脱氢酶(cinnamyl alcohol dehydrogenase,CAD)还原为香豆醇(p-Coumaryl,alcohol)、松柏醇(coniferyl alcohol)和芥子醇(sinapyl alcohol)这3种肉桂醇即是合成木质素的主要单体。其中松柏醇能进一步形成木脂素。棉纤维中苯丙烷类物质的发现为棉纤维的品质改良提供了新的思路。利用基因工程手段从棉纤维中克隆苯丙烷物质生物合成相关基因并研究其功能,有助于弄清棉纤维细胞中这类物质的存在情况及其在棉纤维发育及纤维品质形成中的作用,为利用基因工程手段改良棉纤维的品质奠定理论基础。
PIP亚家族是同源性较高的SDR(short-chain dehydrogenase/reductase)家族成员[10],是木脂素和异黄酮等植物次生代谢产物的关键酶。包括苯基香豆满苄基醚还原酶(phenylcoumaran benzylic ether reductase,PCBER),异黄酮还原酶(isoflavone reductase,IFR)和松脂醇-落叶松脂醇还原酶(pinoresinol-lariciresinol reductases,PLR)[11],3种蛋白不仅有很高的氨基酸序列相似性并且都具有一个连续的α/β NADPH结合结构域和较小的底物结合域[12-13]。PCBER是苯丙烷代谢产物木脂素合成过程中的关键酶,在棉花的子叶、茎、叶片、根和发育的纤维中均发现了PCBER的转录产物[14-17],研究发现陆地棉种皮中有高水平的PCBER转录[12],棉花无絮突变体和野生型发育起始期的棉纤维比较蛋白质组研究发现PCBER相应的蛋白点仅存在于野生型棉纤维中[12,18]。PLR是松脂醇-落叶松酯醇还原酶,在8-8’连接结构的木脂素合成途径中,它可以催化松脂醇和落叶松脂醇还原为开环异落叶松树脂酚。近期的研究中发现,拟南芥中的PCBER具有弱PLR酶活性,可催化相同的底物松脂醇和落叶松脂醇还原为开环异落叶松树脂酚[10]。IFR是异黄酮类化合物生物合成途径中的重要酶,参与抵御各种生物和非生物胁迫,银杏中的IFR具有PCBER酶活性[19-20]。目前已经从多种维管植物种中分离出PCBER、PLR和IFR,PIP亚家族基因转化植物后,对于植物中多种代谢产物的含量产生影响[19-23]。目前棉花中尚未见PIP亚家族基因的报道,本研究利用陆地棉PCBER蛋白序列为探针序列同源比对出多个相似性序列,其中包括PCBER、IFR和PLR,为进一步研究PIP亚家族基因在棉纤维发育及品质形成中的功能奠定了基础。
1 材料和方法
1.1 材 料
植物材料为新疆陆地棉(GossypiumhirsutumL.)品种‘新陆早33号’,由石河子大学棉花研究所提供。将‘新陆早33号’播种于石河子大学实验场。在棉田盛花期对当日花挂牌标记,以3 d为一时间节点,摘取3~24 DPA(day post anthesis,开花后天数)棉铃,室内剥取胚珠及纤维;将‘新陆早33号’种子用浓硫酸脱绒后种于花盆中(营养土︰蛭石=2︰1),置于人工气候室,待长至2片真叶时将其小心拔出,水培3 d,取其根、茎、叶,从田间摘取当日花。将上述植物材料液氮速冻后保存于-80℃,用于提取RNA。
1.2 方 法
1.2.1 不同组织RNA提取及cDNA第一链合成 利用CTAB/酸酚法提取棉花组织总RNA[24]。使用ND1000紫外分光光度计定量,根据A260/280和A260/230比值对样品纯度进行鉴定,用1.2%琼脂糖凝胶电泳检测所提RNA的完整性。对质量合格的RNA按照Reverse Transcriptase M-MLV(RNase H-)说明书完成cDNA第一链的合成。

表1 PIP亚家族基因ORF扩增及qPCR引物序列
1.2.2 棉花PIP亚家族基因的克隆 以陆地棉PCBER蛋白序列(GenBank登录号ABN12322.1)作为查找序列,在陆地棉的基因组数据库(https://www.cottongen.org)中进行Blastp比对,获得6个具有较高同源性的基因,利用Blastp比对和进化树分析,发现6个基因包含3个PCBER,1个IFR和2个PLR基因,分别对6个基因进行命名(表1)。每个基因在陆地棉基因组中均具有2个拷贝,分别位于A染色体组和D染色体组上,一一对应(表1)。设计扩增6个基因ORF的引物(表1),以棉纤维cDNA为模板对6个基因进行PCR扩增,反应体系为:10×buffer 2 μL、2.5 mmol/L dNTPs 1.6 μL、5 U/ μLEX-taqDNA 聚合酶0.25 μL、10 μmol/L上下游引物各1 μL,补 ddH2O至20 μL。反应程序为:94 ℃预变性3 min;94 ℃变性30 s, 52~57 ℃退火30 s,72 ℃延伸1 min;重复31个循环;72 ℃延伸10 min。将PCR扩增产物回收后连接至pGEM-T Easy载体,转化大肠杆菌XL1-Blue感受态细胞,挑取单菌落,提取质粒进行PCR和酶切鉴定,正确的质粒送至生工生物工程(上海)股份有限公司测序。
1.2.3 棉花PIP亚家族基因生物信息学分析 利用Expasy中的Protparam软件进行氨基酸基本理化性质分析;用SOPMA软件对蛋白序列进行二级结构预测;用TMHMM2.0在线工具对氨基酸序列跨膜结构进行预测分析,用SignalP 4.1和TargetP 1.1 Server软件进行信号肽预测和亚细胞定位分析;氨基酸序列的同源比对及多序列比对采用Clustal W和DNAMAN进行分析。利用MAGE 6.06软件采用邻近相接法(neighbor-joining method)构建系统进化树,Bootstrap参数设为重复检测1 000次。
1.2.4 实时荧光定量PCR分析 利用qPCR技术分析棉花PIP亚家族基因在棉花不同组织及不同发育时期纤维中的时空表达模式,根据6个基因的cDNA序列,分别设计特异性qPCR引物(表1),以陆地棉His3为内参基因,以棉花幼苗的根、茎、叶、花和不同发育时期(花后3、6、9、12、15、18、21和24 d)的棉纤维cDNA为模板进行qPCR扩增。反应程序为:94 ℃预变性1 min;95 ℃变性15 s,56~57 ℃ 退火20 s,72 ℃延伸30 s,45个循环。每个样设置3个实验重复,将数据输入Excel中按照2-ΔΔCt法分析。
2 结果与分析
2.1 PIP亚家族基因全长cDNA序列克隆
以陆地棉PCBER蛋白序列作为查询序列,在陆地棉基因组数据库中进行Blastp比对,发现了6个同源基因。根据Blastp比对结果,将6个基因分别命名为GhPCBER1、GhPCBER2、GhPCBER3、GhIFR、GhPLR1和GhPLR2。陆地棉基因组中6个PIP亚家族基因的基因组序列全长为3 756~4 779 bp,CDS(coding sequence)长度为921~942 bp,编码306~313个氨基酸(表2)。根据基因的序列设计扩增ORF的引物,以陆地棉纤维cDNA为模板对6个基因进行PCR扩增,结果如图1所示,扩增条带与预期结果相一致。
2.2 PIP亚家族基因编码蛋白的生物信息学分析
根据6个基因的测序结果,运用ProtParam软件预测相应蛋白的基本理化性质(表3),6个蛋白分子量为33 854.9~35 199.6 Da,理论等电点为5.52~7.77,负电荷的氨基酸残基数(Asp+Glu)是35~41,正电荷的氨基酸残基数(Arg+Lys)是32~36;脂肪系数为91.75~101.96,氨基酸残基总的亲疏水性平均系数均为负值,分布在-0.131~ -0.056之间,说明蛋白的亲水性较强。不稳定系数是22.55~43.71,其中GhPLR1和GhPLR2不稳定系数大于40,为不稳定蛋白,其余为稳定蛋白。利用SOPMA在线预测分析蛋白的二级结构(表4),结果表明该蛋白主要由α螺旋、β转角、延伸直链和无规则卷曲4种结构形式构成。其中,GhPCBER1和GhPCBER2蛋白二级结构主要由无规则卷曲组成,GhPCBER3、GhPLR2主要由α螺旋组成,GhIFR和GhPLR1以α螺旋和无规则卷曲为主要组成结构。TMHMM2.0预测表明6个蛋白均未形成跨膜结构域。用SignalP 4.1和TargetP 1.1软件对氨基酸序列跨膜预测结果显示PIP亚家族的6个蛋白均无信号肽,是非分泌蛋白。对6个蛋白的信号肽及亚细胞定位预测显示PIP亚家族蛋白均定位于细胞质中。
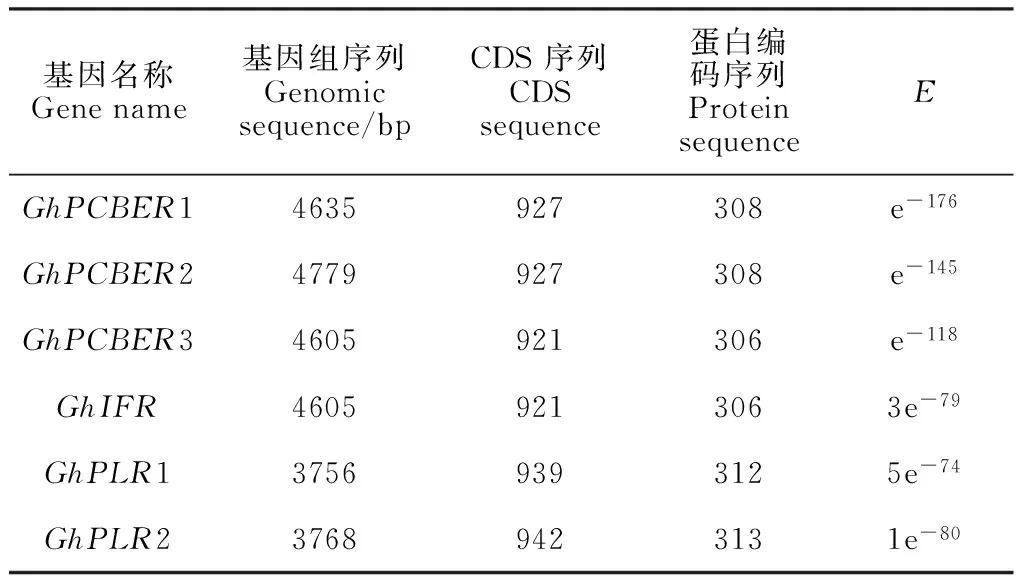
表2 PIP亚家族基因在陆地棉基因组数据库中的序列信息
2.3 PIP蛋白多重序列比对和蛋白进化树分析
为了进一步明确棉花PIP亚家族6个基因的典型结构域及与其他植物PIP亚家族基因的同源性,选取已报道的部分植物PIP亚家族基因,利用Clustal X和DNAMAN软件对不同植物中PIP亚家族基因进行多重序列比对,发现均有与NADPH结合的“GXXGXXG”保守结合域(图2)。利用MEGA6.06软件对不同植物PIP亚家族序列进行进化树分析,结果显示所有基因聚为三大类,大致可分为PCBER、PLR和IFR,而PCBER与IFR的亲缘关系比PLR近。GhPCBER1、GhPCBER2和GhPCBER3与连翘和火炬松的PCBER聚为一类,GhPLR1、GhPLR2与北美红崖柏和亚麻的PLR聚为一类,GhIFR与杨树和琴叶拟南芥的IFR聚为一类(图3)。

M. DNA marker Ⅲ; 1~6. GhPCBER1、GhPCBER2、GhPCBER3、GhIFR、GhPLR1和GhPLR2的 PCR扩增结果图1 陆地棉PIP亚家族基因 PCR 扩增电泳M. DNA marker Ⅲ; 1-6 represent the PCR results of GhPCBER1, GhPCBER2, GhPCBER3, GhIFR, GhPLR1 and GhPLR2, respectivelyFig.1 PCR results of PIP subfamily genes
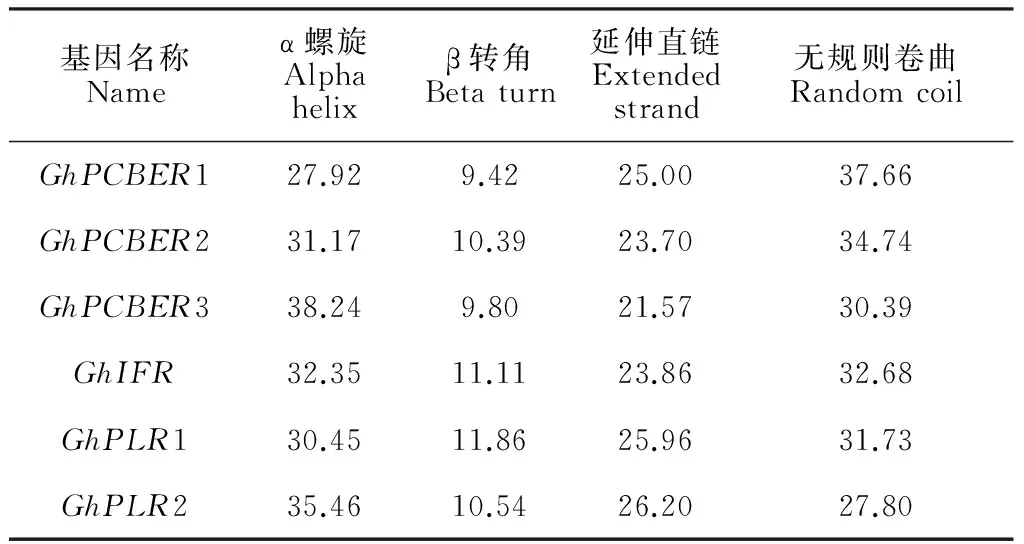
基因名称Nameα螺旋Alphahelixβ转角Betaturn延伸直链Extendedstrand无规则卷曲RandomcoilGhPCBER127.929.4225.0037.66GhPCBER231.1710.3923.7034.74GhPCBER338.249.8021.5730.39GhIFR32.3511.1123.8632.68GhPLR130.4511.8625.9631.73GhPLR235.4610.5426.2027.80

表3 蛋白的基本理化性质

黑色方框为PIP亚家族与NADPH结合的GXXGXXG保守结合域;Fi1PCBER.连翘(AF242491);GhPCBER1、GhPCBER2、GhPCBER3、GhIFR、GhPLR1、GhPLR2.陆地棉(Gh_D01G0977、Gh_A03G0704、Gh_D12G0604、Gh_D02G2151、Gh_A08G1368、Gh_D08G1661);PtIFR.杨树(XP_002313789.1);LuPLR.亚麻(CAH60858)图2 陆地棉PIP与其它PIP蛋白的多重序列比对The black box is a conserved NADPH binding domain of GXXGXXG which is combined with PIP subfamily;Fi1PCBER. Forsythia suspensa(AF242491); GhPCBER1,GhPCBER2,GhPCBER3,GhIFR,GhPLR1,GhPLR2. G. hirsutum L.(Gh_D01G0977,Gh_A03G0704,Gh_D12G0604,Gh_D02G2151,Gh_A08G1368,Gh_D08G1661);PtIFR. Populus trichocarpa (XP_002313789.1); LuPLR. Linum usitatissimum (CAH60858)Fig.2 Multiple sequence alignment of PIP and other PIP proteins

系统树各分支上数字是Bootstrap 1 000次循环检验的置信度;标尺代表遗传距离图3 陆地棉PIP与其他 PIP蛋白的进化树分析The numbers on the tree branches represent bootstrap confidence values as bootstrap is 1 000;The scale bar represents genetic distanceFig.3 Phylogenic relationship of PIP and other PIP proteins

R.根;S.茎;L.叶;F.花;3~24. 花后3、6、9、12、15、18、21、24 d的纤维细胞图4 陆地棉PIP亚家族基因的实时荧光定量 PCR 分析R. Root; S. Stem; L. Leaf; F. Flower; 3-24. Fiber cells at 3, 6, 9, 12, 15, 18, 21 and 24 day post anthesis, respectivelyFig.4 Relative expression of PIP subfamily genes
2.4 PIP亚家族基因在棉花不同组织中的表达特征分析
为研究棉花PIP亚家族6个基因在陆地棉中的表达模式,根据每个基因的cDNA序列设计特异性引物,对其在棉花不同组织和不同发育时期纤维中的表达模式进行实时荧光定量分析。结果(图4)表明,在6个PIP亚家族基因中,GhPLR1在根和茎中表达量明显较高,在纤维中表达无优势,其它5个基因均在纤维中特异或优势表达。GhPCBER3在纤维中特异表达,GhPCBER1、GhPCBER2、GhIFR和GhPLR2在纤维中优势表达,在根和茎中有少量表达,在叶和花中几乎不表达。GhPLR1和GhPLR2在纤维中的表达量呈上升趋势;GhPCBER1、GhPCBER2和GhIFR在纤维中的表达量均呈先升高后降低的趋势。6个基因在纤维发育起始期的表达量均较低,在次生壁合成期表达量较高,推测它们在纤维次生壁合成期可能具有一定的功能。
3 讨 论
前期研究认为棉纤维次生壁中不存在木质素[25]。Fan等[5]利用荧光观察、傅里叶转换红外线光谱(FTER)分析及高效液相色谱(HPLC)分析发现成熟棉纤维中含有木质素,还发现参与单体木质醇生物合成的CAD基因在纤维次生壁合成时期高度表达。Han等[6]克隆了WLIM1转录因子,该基因可以调控苯丙烷代谢相关基因的表达,该基因在棉花中过量表达致使棉纤维木质素含量增多,长度变长,强力增加,推测木脂素对棉纤维的品质具有影响。植物中的次生代谢产物包括木脂素、黄酮、异黄酮、花青素、酚类化合物等[8-9],关于它们在棉纤维中的存在情况及与棉纤维品质的相关性未见报道。PCBER、IFR和PLR是PIP亚家族基因,是合成木脂素和异黄酮过程中重要的酶基因,本研究对陆地棉中PIP亚家族基因进行克隆与分析,对于明确棉纤维中次生代谢产物的存在情况及它们对纤维发育和品质形成的影响具有重要意义。
本研究以陆地棉PCBER蛋白为探针序列,从陆地棉基因组数据库中发现了12个同源基因,根据cDNA序列相似性划分为6对,每对中的两个成员均分别位于A和D染色体组上,且具有较高的序列相似性。由于每对基因中的2个成员序列相似性高(大于97%),难以进行区分,因此将它们视为基因的两个拷贝。本研究共设计6个扩增ORF的引物和6个qPCR引物,克隆了6个基因。
由生物信息学分析发现,6个蛋白的三级结构均具有PIP的典型结构域,即靠近N端区域都存在一段特异性的保守序列“GXXGXXG”即NADPH结合位点,说明6个基因属于PIP亚家族基因。qPCR分析发现,6个PIP亚家族基因均在纤维发育次生壁合成期表达量较高,暗示PIP亚家族在纤维次生壁合成期具有一定的功能。6个基因中,其中GhPCBER3在棉纤维中呈现特异表达,GhPCBER1、GhPCBER2、GhIFR和GhPLR2呈优势表达,PIP亚家族基因编码植物次生代谢产物合成过程中的关键酶,暗示次生代谢产物在棉纤维的发育过程中发挥着关键作用。
[1] MEINERT M C, DELMER D P. Changes in biochemical composition of the cell wall of the cotton fiber during development[J].PlantPhysiology, 1977, 59(6):1 088-1 097.
[2] HUWYLER H R, FRANZ G, MEIER H. Changes in the composition of cotton fiber cell walls during development[J].Planta, 1979, 146: 635-642.
[3] GOU J Y, WANG L J, CHEN S P,etal. Gene expression and metabolite profiles of cotton fiber during cell elongation and secondary cell wall synthesis[J].CellResearch, 2007, 17: 422-434.
[4] ALGHAZI Y, BOUROT S, ARIOLI T,etal. Transcript profiling during fiber development identifies pathways in secondary metabolism and cell wall structure that may contribute to cotton fiber quality[J].PlantCellPhysiology, 2009, 50: 1 364-1 381.
[5] FAN L, SHI W J, HU W R,etal. Molecular and biochemical evidence for phenylpropanoid synthesis and presence of wall-linked phenolics in cotton fibers[J].JournalofIntegrativePlantBiology, 2009, 51: 626-637.
[6] HAN L B, LI Y B, WANG H Y,etal. The dual functions of WLIM1 a in cell elongation and secondary wall formation in developing cotton fibers[J].ThePlantCell, 2013, 25: 4 421-4 438.
[7] BOERJAN W, RALPH J, BAUCHER M. Lignin biosynthesis[J].AnnualReviewPlantBiology, 2003, 54: 519-546.
[8] GRAND C, BOUDET A, BOUDET A M. Isoenzymes of hydroxycinnamate:CoA ligase from poplar stems, properties and tissue distribution[J].Planta, 1983, 158: 225-229.
[9] WALLIS P J, RHODES M J. Mutiple forms of hydroxycinnamate: CoA ligase in etiolated pea seedlings[J].Phytochemistry, 1977, 16: 1 891-1 894.
[10] GANG D R, DINKOVA-KOSTOVA A T, DAVIN L B,etal. Phylogenetic links in plant defense systems: lignans, isoflavonoids, and their reductases[C]// HEDIN P A, HOLLINGWORTH R M, MASTER E P,etal. Phytochemicals for Pest Control. Washington DC: ACS Symposium Series, 1997: 58-89.
[12] TURLEY R. Expression of a phenylcoumaran benzylic ether reductase-like protein in the ovules ofGossypiumhirsutum[J].BiologiaPlantarum, 2008, 52(4): 759-762.
[13] MIN T, KASAHARA H, BEDGAR D L,etal. Crystal structures of pinoresinol-lariciresinol and phenylcoumaran benzylic ether reductases and their relationship to isoflavone reductases[J].JournalofBiologicalChemistry, 2003, 278(50): 50 714-50 723.
[14] DOWD C, WILSON I, MCFADDEN H. Gene expression profile changes in cotton root and hypocotyl tissues in response to infection withFusariumoxysporumf. sp.vasinfectum[J].MolecularPlant-MicrobeInteracttions, 2004, 17: 654-667.
[15] HAIGLER CH, ZHANG D, WILKERSON C G. Biotechnological improvement of cotton fibre maturity[J].PhysiologiaPlantarum, 2005, 124: 285-294.
[16] TALIERCIO E, ALLEN R D, ESSENBERG M,etal. Analysis of ESTs from multipleGossypiumhirsutumtissues and identification of SSRs[J].Genome, 2006, 49: 306-319.
[17] UDALL J A, SWANSON J M, HALLER K,etal. A global assembly of cotton ESTs[J].GenomeResearch, 2006, 16: 441-450.
[18] TURLEY R B, FERGUSON D L. Changes of ovule proteins during early fiber development and a normal and a fiberless line of cotton (GossypiumhirsutumL.)[J].JournalofPlantPhysiology, 1996, 149: 695-702.
[19] CHENG Q, LI N H, DONG L D,etal. Overexpression of soybean isoflavone reductase (GmIFR) enhances resistance toPhytophthorasojaein soybean[J].FrontPlantSci., 2015, 6: 1 024.
[20] CHENG H, LI L L, XU F,etal. Expression patterns of an isoflavone reductase-like gene and its possibleroles in secondary metabolism inGinkgobiloba[J].PlantCellRep, 2013, 32:637-650.
[21] NUOENDAGULA, NAOFUMI K, TETSUYA M,etal. Expression and functional analyses of a putative phenylcoumaran benzylic ether reductase inArabidopsisthaliana[J].PlantCellRep, 2016, 35: 513-526.
[22] NICULAES C, MORREEL K, KIM H,etal. Phenylcoumaran benzylic ether reductase prevents accumulation of compounds formed under oxidative conditions in poplar xylem[J].PlantCell, 2014, 26(9):3 775-3 791.
[23] BANERJEE A, CHATTOPADHYAY S. Effect of over-expression ofLinumusitatissimumpinoresinol lariciresinol reductase. (LuPLR) gene in transgenicPhyllanthusamarus[J].PlantCellTissOrganCult, 2010, 103:315-323.
[24] 蒋建雄,张天真.利用CTAB酸酚法提取棉花组织总RNA[J].棉花学报,2003,15:166-167.
JIANG J X , ZHANG T Z. Extraction of total RNA in cotton tissues with CTAB acidic phenolic method[J].CottonSci., 2003, 15:166-167.
[25] KIM H J, TRIPLETT B A. Cotton fiber growth in plant andinvitro. Models for plant cell elongation and cell wall biogenesis[J].PlantPhysiology, 2001, 127: 1 361-1 366.
(编辑:宋亚珍)
Cloning and Expression Analysis of PIP Subfamily Genes in Gossypium hirsutum
SUN Yuwei1, LI Yanjun1*, LIU Yongchang1, XUE Fei1, ZHU Shouhong1, WANG Xiangfei2, SUN Jie1
(1 College of Agronomy,Shihezi University,Key Oasis Eco-agriculture Laboratory of Production and Construction Group,Shihezi,Xinjiang 832003,China;2 College of Pharmacy,Shehezi University, Shihezi,Xinjiang 832003,China)
Using the protein sequence ofGhPCBERas a probe, six of genes showing high sequence homology were obtained from a genome database ofGossypiumhirsutumL.tetraploid cotton with Blastp search. These sequences were used to design primers, and then six genes were isolated from cotton fiber ofG.hirsutumL. by using RT-PCR technique and designated asGhPCBER1,GhPCBER2,GhPCBER3,GhIFR,GhPLR1 andGhPLR2, respectively. Multiple sequence alignment and phylogenetic tree analysis revealed that all of the 6 proteins belong to PIP subfamily since the conserved motifs and active residues in their sequences. Real time fluorescent quantitative PCR results showed that in addition toGhPLR1, all the other 5 PIP subfamily genes were specifically or preferentially expressed in fiber cells. Of 6 genes, the expression pattern ofGhPCBER1,GhPCBER2,GhPCBER3 andGhIFRshowed increasing firstly and then decreasing trend during cotton fiber developing stage. The expression level ofGhPCBER1 andGhPCBER2 reached a maximum at 21 day post anthesis, andGhIFRandGhPCBER3 reached a maximum at 18 day post anthesis. The expression pattern ofGhPLR1 andGhPLR2 showed an ascendant trend with cotton fiber developing. According to the characteristics of gene expression, it is speculated that the PIP subfamily may play an important role in cotton fiber development.
cotton; fiber; PIP; expression analysis
1000-4025(2016)10-1948-07
10.7606/j.issn.1000-4025.2016.10.1948
2016-06-21;修改稿收到日期:2016-09-07
国家自然科学基金(31460360)
孙雨薇(1990-),女,在读硕士研究生,主要从事棉花分子育种研究。E-mail:sunyuwei2014@aliyun.com
*通信作者:李艳军,博士,副教授,主要从事棉花分子育种研究。E-mail:lyj20022002@sina.com.cn
Q785;Q789
A
