肠道内菌群结构与大肠肿瘤的相关性
2016-12-12付文政张春泽贾岩峰
付文政,张春泽,贾岩峰
·作者须知·
肠道内菌群结构与大肠肿瘤的相关性
付文政,张春泽,贾岩峰
目的:研究肠道内菌群结构与大肠肿瘤的相关性。方法:选取2014年1月—2015年6月年收治的大肠癌患者31例、大肠腺瘤患者31例及同期健康体检者(对照组)31例,收集3组粪便标本,提取粪便DNA,采用荧光定量PCR对DNA指纹图谱进行测序,比较3组肠道菌群结构的差异。结果:3组样本共获得254 271条高质量序列,3组细菌多样性指数比较差异无统计学意义(P>0.05)。对照组拟杆菌属、罗氏菌属、酸杆菌属、真杆菌属、变形菌属所占比例分别为21.00%,4.10%,2.14%,0.61%, 0.49%高于大肠癌组的14.00%,1.56%,0.79%,0.11%,0.12%和大肠腺瘤组的16.03%,1.62%,0.82%,0.12%,0.14%。大肠癌组紫单胞菌属、埃希杆菌、肠球菌属、链球菌属、消化链球菌属所占比例分别为1.75%,2.45%,2.34%,1.19%,0.71%;大肠腺瘤组分别为1.62%,2.32%,2.12%,1.09%,0.69%,均高于对照组的0.09%,0.87%,0.10%,0.45%,0.01%。大肠癌组拟杆菌属、罗氏菌属所占比例低于大肠腺瘤组,肠球菌属、链球菌属所占比例高于大肠腺瘤组荧光定量PCR检测显示对照组拟杆菌占总菌相对含量、丁酸盐产生菌占总菌相对含量为(17.38±3.04)%、(4.38±1.03)%分别高于大肠癌组的(10.85±2.54)%、(1.41±0.89)%与大肠腺瘤组的(12.37±2.05)%、(2.07±1.02)%,大肠癌组含量则少于大肠腺瘤组(P<0.05)。结论:人体肠道内拟杆菌属、丁酸盐产生菌及链球菌等条件致病菌的结构失衡,与大肠肿瘤发生密切相关。
肠道菌群;大肠癌;大肠腺瘤;大肠肿瘤
大肠肿瘤是消化系统常见恶性肿瘤,患病死亡率在我国居第5位。大肠内细菌众多,与人类形成一种共生关系。近年研究发现,肠道内细菌感染与大肠肿瘤的发生密切相关[1]。国外研究证实,链球菌、败毒梭菌感染可诱发大肠癌,而腐蚀柠檬酸杆菌感染对大肠肿瘤的发展起促进作用[2]。为了研究肠道内菌群结构与大肠肿瘤的相关性,我们于2014年1月—2015年6月分别对大肠癌、大肠腺瘤及健康体检者粪便标本中的菌群进行基因测序,现报道如下。
1 资料与方法
1.1临床资料选取大肠癌、大肠腺瘤患者各31例。纳入标准:⑴符合相关诊断标准[3],并经肠镜及病理学检查确诊。⑵基本状况良好,无心、肝、肾及血液系统疾病。⑶未经过辅助放化疗及手术治疗。⑷签署知情同意书。排除标准:⑴病理诊断为非腺瘤性息肉、家族性腺瘤病。⑵近1个月服用过质子泵抑制剂、铋剂、H2受体拮抗剂、抗生素等药物。⑶妊娠或哺乳期妇女。大肠癌组男21例,女10例;年龄38~65岁,平均(53.5±10.3)岁。大肠腺瘤组男20例,女11例;年龄34~67岁,平均(52.2±11.3)岁。选取同期健康体检者31例设为对照组,男21例,女10例;年龄38~66岁,平均(53.8±10.1)岁。3组性别、年龄等一般资料比较差异无统计学意义(P>0.05),具有可比性。
1.2标本采集收集大肠癌组、大肠腺瘤组及对照组新鲜大便3 g,存放于高压灭菌Eppendorff离心管中,-80℃冰箱中冷冻。取0.5 g加入0.1 mol/LPBS 10 mL,振荡混匀。3000 r/min离心处理5 min,取上清液。重复操作3次,取1 mL上清液,加入1.5 mL Eppendorff离心管中。12 000 r/min离心处理10 min,弃上清,留沉淀。在沉淀中再加入1 mL上述上清液,重复操作4次,收集沉淀,置于-20℃冰箱。
1.3DNA提取将收集的沉淀采用磁珠击打破碎细胞结合酚氯仿抽提纯化法[4]提取DNA。取50 μL DNA样品以50 μL TEbuffer做零线对照,采用FLA-5100荧光检测仪(日本生产)测定。用质量体积比为0.8%的琼脂糖凝胶电泳检测DNA片段大小及完整性,电泳完成后在紫外线灯下观察,显示荧光条带则表明有DNA存在。
1.416SrRNA基因V3高变区片段扩增和454测序
以DNA样本为模板进行16SrRNA基因V3高变区片段扩增,扩增产物采用细菌通用引物27f(5'-AGA GTT TGA TCC TGG CTC AG)和 1492r(5'-GGTT ACC TTG TTAC GAC TT)[5]进行DNA指纹图谱分析。
16SrRNA基因V3高变区片段扩增产物用于454测序分析,引物参考文献[5]设计,PCR反应条件严格按照说明书操作。
1.5多变量统计分析对于454测序得到的OTU丰度矩阵,采用各片段或各OTU在样本中的丰度为变量,研究样本的空间分布及与样本间的关系。采用主成分分析(PCA)对样本分布进行无偏见观察,体现数据内部固有结构;再采用基于UniFrac计算的主坐标分析法(PCoA)对OTU丰度矩阵进行分析。对于PCA、PCoA的分析结果进一步做多变量方差分析(MANOVA)以研究不同组样本之间的差异。PCA和MANOVA分析由Matlab软件完成,UniFrac分析由在线工具完成。
随后引入环境因子对样本进行约束以研究不同疾病状态对肠道菌群的影响。以全部OTU在各个样本中的丰度为变量,以全样×本部变量丰度的矩阵为研究对象,采用冗余分析(RDA)分析样本之间的差异。RDA分析中,样本在排序途中的坐标是由环境因子(解释变量)的线性组合决定的。RDA分析由Canoco软件完成。
1.6实时荧光定量PCR采用MJopticon2型实时荧光定量PCR扩增分析仪(美国生产)检测全部样品中的细菌总量、拟杆菌属细菌及丁酸盐产生菌数量,细菌总量与拟杆菌属细菌数量由对应的16S rRNA基因拷贝数表示,丁酸盐产生菌的数量由丁酰-辅酶A转移酶基因A拷贝数表示。细菌总量引物参考文献[6],拟杆菌属细菌引物参考文献[7],丁酸盐产生菌引物参考文献[8]设计,PCR反应条件严格按照说明书操作。用于绘制标准曲线的样品重复做3次反应,用于检测样品的重复2次反应,PCR反应的扩增效率由PCR自带软件进行分析。
1.7统计学分析采用SPSS 17.0软件包对数据行统计学分析,荧光定量PCR结果采用 LightCycler480数据分析软件测得,计量资料用(±s)表示,采用F或t检验;计数资料采用χ2检验,对样本序列采用Shannon-wiener多样性指数分析,P<0.05为差异有统计学意义。
2 结果
2.1测序结果我们提取的部分样本的基因组DNA电泳图见下图1。3组93例样本共检测出254 271条高质量序列,大肠癌组检出84 720条序列,大肠腺瘤组检出86 854条序列,对照组检出82 697条序列。3组检出的序列长度170~230 bp。大肠癌组Shannon-wiener多样性指数为3.64±0.51,大肠腺瘤组为3.58±0.47,对照组为3.62±0.45。3组细菌多样性指数比较差异无统计学意义(F=0.76,P>0.05)。

图1 提取部分样本的基因组DNA电泳图
2.2不同菌属占肠道菌群序列数比例我们以上述DNA样本为模板,PCR扩增16S rRNA基因V3高变区片段,部分样本电泳结果见下图2。对照组拟杆菌属、罗氏菌属、酸杆菌属、真杆菌属、变形菌属所占比例高于大肠癌组、大肠腺瘤组(P<0.05);大肠癌组、大肠腺瘤组紫单胞菌属、埃希杆菌属、肠球菌属、链球菌属、消化链球菌属所占比例高于对照组(P<0.05);大肠癌组、大肠腺瘤组酸杆菌属、真杆菌属、变形菌属、紫单胞菌属、埃希杆菌属、消化链球菌属所占比例比较差异无统计学意义(P>0.05),大肠癌组拟杆菌属、罗氏菌属所占比例低于大肠腺瘤组(P<0.05),肠球菌属、链球菌属所占比例高于大肠腺瘤组(P<0.05),见表1。
图2 部分样本PCR扩增16SrRNA基因V3高变区片段结果电泳图
2.3荧光定量PCR检测荧光定量PCR实验中的扩增曲线和产物溶解曲线见下图3。检测结果显示,3组每ngDNA中总菌16S rRNA基因拷贝数对数值比较差异无统计学意义(P>0.05),对照组拟杆菌属细菌数目、丁酸盐产生菌数目高于大肠癌组与大肠腺瘤组(P<0.05);大肠癌组拟杆菌属细菌数目、丁酸盐产生菌数目均少于大肠腺瘤组(P<0.05),见表2。

图3 部分样本Real-Time PCR的扩增曲线(A)和产物溶解曲线(B)
3 讨论
人类大肠内细菌众多,尤其是在大便内。经过代谢交换与食物代谢等方式,肠道内的细菌与宿主具有紧密的代谢合作关系。一旦肠道菌群结构失衡,则会导致代谢紊乱,进而引发疾病。Li等[9]研究发现,患有克罗恩病、溃疡性结肠炎的成人,与患有过敏性肠炎、乳糜腹泻的婴幼儿,肠道菌群结构相对健康人群的菌群结构发生了整体失衡现象。这一结构失衡主要表现为有害菌的增多和有益菌的减少。近年研究发现,部分有害菌对肠道不同阶段的肿瘤产生促进作用,尤其在慢性感染阶段,正常组织会受到有害菌释放的毒性因子的影响。刘小满等[10]进行的动物研究发现,腐蚀柠檬酸杆菌感染大鼠可引发结肠增生样改变,这种改变最终会导致结肠癌。Tahara等[2]在对人体链球菌属抗体的检查中发现,结直肠癌、大肠腺瘤患者鸟禽类链球菌抗体浓度显著高于健康人群。由此提示,肠道部分菌群的结构变化与大肠肿瘤的发生、发展有关。本研究采用16S rRNA基因克隆文库的方法与454测序方法比较大肠癌、大肠腺瘤患者与健康人群肠道菌群结构的差异,进一步研究肠道菌群结构与大肠肿瘤的关系。3组粪便标本检出的高质量序列数无差异,且细菌多样性指数比较也无统计学意义(P>0.05)。荧光定量PCR结果显示,3组每ngDNA中总菌16S rRNA基因拷贝数对数值比较差异无统计学意义(P>0.05)。由此可知,大肠肿瘤患者,肠道菌群结构改变并不是有益菌的绝对灭失与有害菌的绝对增长,而是有益菌的减少和有害菌的增加。这与国内外文献报道[1,3]基本相符。
对3组粪便标本进行荧光定量PCR检测,发现大肠肿瘤患者肠道菌群变化最显著的特征,是拟杆菌属与丁酸盐产生菌的减少。3组每ngDNA中总菌16S rRNA基因拷贝数对数值比较差异无统计学意义(P>0.05),但对照组拟杆菌属细菌数目、丁酸盐产生菌数目高于大肠癌组与大肠腺瘤组(P<0.05)。拟杆菌属对宿主代谢、营养和健康都发挥重要作用。吴瑞丽等[11]对溃疡性结肠炎患者肠道菌群结构进行ERIC-PCR指纹图谱分析,发现溃疡性结肠炎患者拟杆菌属数目为(2.90±1.95)×103,而对照组为(4.13±2.62)×103。本研究中,大肠癌组、大肠腺瘤组拟杆菌属序列所占比例低于对照组(P<0.05),且大肠癌组拟杆菌属序列所占的比例低于大肠腺瘤组(P<0.05)。由此提示,大肠肿瘤的发生、发展可能与拟杆菌属减少有关。而与拟杆菌属同属于拟杆菌门的紫单胞菌属,却对大肠的疾病产生不同的影响。Yamamoto等[7]进行的动物研究发现,一种能产肠毒素的紫单胞菌,在小鼠肠道内定植可引发肠道组织损伤,诱导白介素-17的产生,并激活转录因子3,进而促进结直肠肿瘤的形成。本研究中,大肠癌组、大肠腺瘤组紫单胞菌序列所占比例显著高于对照组(P<0.01),但大肠癌组、大肠腺瘤组紫单胞菌序列所占比例差异比较无统计学意义(P>0.05),其原因有待进一步研究。
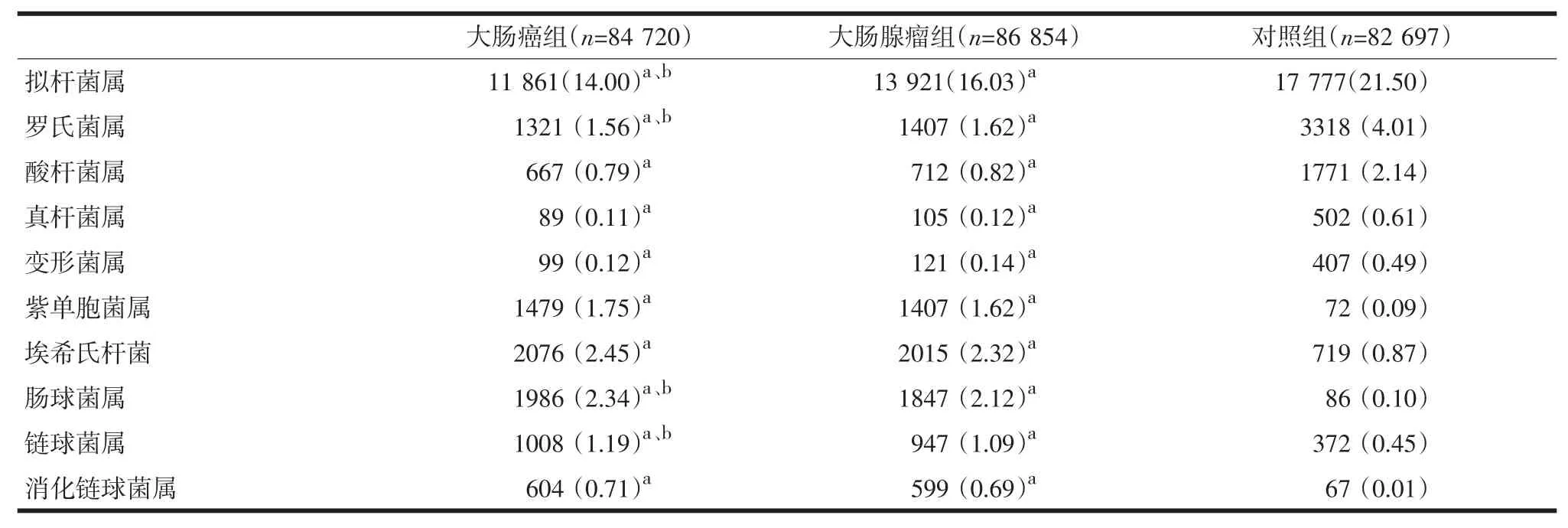
表1 3组不同菌属占肠道菌群序列数比例(n,%)
表2 3组拟杆菌属细菌、丁酸盐产生菌数目比较(±s)
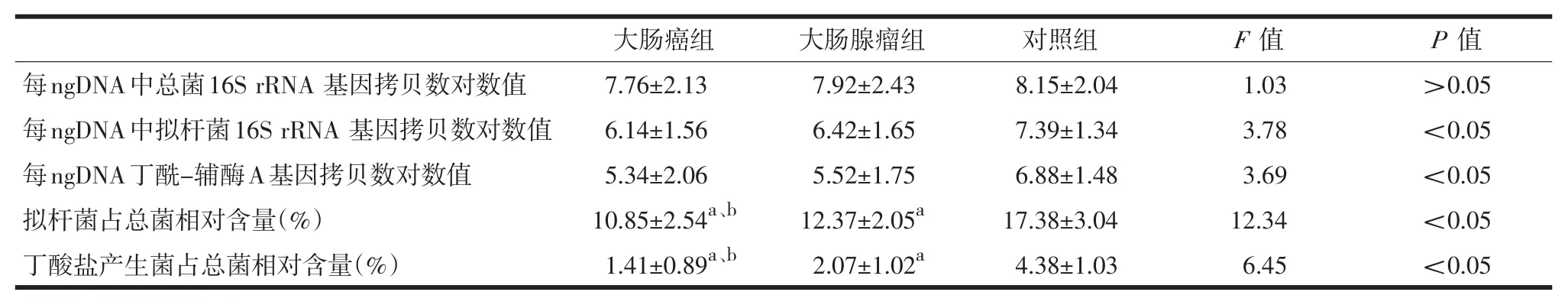
表2 3组拟杆菌属细菌、丁酸盐产生菌数目比较(±s)
注:大肠癌组与对照组比较,aP<0.05;与大肠腺瘤组比较,bP<0.05
每ngDNA中总菌16S rRNA基因拷贝数对数值每ngDNA中拟杆菌16S rRNA基因拷贝数对数值每ngDNA丁酰-辅酶A基因拷贝数对数值拟杆菌占总菌相对含量(%)丁酸盐产生菌占总菌相对含量(%)大肠癌组7.76±2.13 6.14±1.56 5.34±2.06 10.85±2.54a、b 1.41±0.89a、b大肠腺瘤组7.92±2.43 6.42±1.65 5.52±1.75 12.37±2.05a 2.07±1.02a对照组8.15±2.04 7.39±1.34 6.88±1.48 17.38±3.04 4.38±1.03 F值1.03 3.78 3.69 12.34 6.45 P值>0.05<0.05<0.05<0.05<0.05
丁酸盐产生菌对于肠道健康具有重要意义,能为细胞呼吸提供能量,保持肠道上皮完整性,调节肠道免疫应答[8]。丁酸盐产生菌的代表菌种是罗氏菌属。本研究中,大肠癌组、大肠腺瘤组罗氏菌属序列数所占比例低于对照组(P<0.05),且大肠癌组罗氏菌属序列所占比例低于大肠腺瘤组(P<0.05)。与我们研究结果相似的是,国外学者发现,在肠道疾病患者肠道菌群中也发生丁酸盐产生菌减少现象。J Abell等[12]发现,在炎症性肠炎、克罗恩病、大肠癌患者肠道菌群中,一些丁酸盐产生菌如罗氏菌属、直肠真杆菌减少。Kawano等[13]研究还发现,部分因服用抗生素所引发的肠炎、腹泻的患者肠道菌群中,罗氏菌群完全消失。由此可知,大肠肿瘤的发生、发展可能与部分丁酸盐产生菌减少有关。
临床对于细菌在大肠肿瘤发病过程中的影响存在“致癌作用假说”,其机制包括条件致病菌导致炎症的发生,进而产生导致突变的毒性作用而诱发肿瘤。国内外研究也发现,多数大肠癌、大肠腺瘤的发生、发展都伴随有肠道炎症的发生。埃希杆菌、肠球菌属、链球菌属等是临床常见的条件致病菌[14-15]。本研究中,大肠癌组、大肠腺瘤组埃希杆菌属、肠球菌属、链球菌属序列数所占比例高于对照组(P<0.05),且大肠癌组肠球菌属、链球菌属序列数所占比例高于大肠腺瘤组(P<0.05)。董开芯等[1]进行的动物研究发现,肠球菌与链球菌都能通过促进白介素8分泌及细胞分裂来加速癌前病变的进程。Tahara等[2]进行的体外实验也表明,链球菌属细胞壁所分泌的抗原能通过过量表达环加氧酶2而促进人体结肠腺癌上皮细胞系的癌变。由此可知,大肠肿瘤的发生与肠道内条件致病菌的增加有关。
总之,人体肠道内菌群结构失衡与大肠肿瘤的发生、发展密切相关;拟杆菌属、丁酸盐产生菌及链球菌等条件致病菌的结构变化,是大肠肿瘤发生的警示。对这类人群通过针对性的肠道菌群结构优化,减轻菌群变化对宿主的影响,将有助于减少大肠肿瘤的发生。
[1]董开芯,王正强,于新娟,等.胃肠道菌群与肿瘤发生的关系[J].现代生物医学进展,2014,14(11):2196-2200.
[2]Tahara T,Arisawa T.Pathogenesis of CpG island methylator pheno⁃type-positive colorectal cancers:role of genetic alteration and colon⁃ic flora[J].Epigenomics,2014,6(3):249-251.
[3]汤钊猷.现代肿瘤学[M].第3版.上海:复旦大学出版社,2011:125.
[4]Claesson MJ,Cusack S,O’Sullivan O,et al.Composition,variability, and temporal stability of the intestinal microbiota of the elderly[J]. Proc Natl Acad Sci USA,2011,108(1):4586-4591.
[5]Chan AT,Giovannucci EL.Primary prevention of colorectal cancer [J].Gastroenterology,2010,138(6):2029-2043.
[6]Abdulamir AS,Hafidh RR,Mahdi LK,et al.Investigation into the controversial association of Streptococcus gallolyticus with colorec⁃ tal cancer and adenoma[J].BMC Cancer,2009,46(11):403-410.
[7]Yamamoto E,Tahara T,Suzuki H,et al.Fusobacterium in colonic flo⁃ra and molecular features of colorectal carcinoma[J].Cancer Res, 2014,74(5):1311-1318.
[8]罗明,李建明,陈海生,等.大肠癌虚证实证与CD44v6、PCNA表达的相关性研究[J].中国中西医结合外科杂志,2012,18(3):234-237.
[9]Li Y,Zhang X,Wang L,et al.Distribution and gene mutation of enter⁃ic flora carrying β-glucuronidase among patients with colorectal cancer[J].Int J Clin Exp Med,2015,8(4):5310-5316.
[10]刘小满,谭志军.YB-1蛋白在结肠癌组织中的表达部位及意义[J].中国中西医结合外科杂志,2014,20(2):127-130.
[11]吴瑞丽,孙自勤,魏志.溃疡性结肠炎肠道菌群结构ERIC-PCR指纹图谱分析[J].胃肠病学和肝病学杂志,2012,21(9):798-801.
[12]J Abell GC,Christophersen CT,McOrist AL,et al.Dietary resistant and butyrylated starches have different effects on the faecal bacteri⁃al flora of azoxymethane-treated rats[J].Br J Nutr,2011,105(10): 1480-1485.
[13]Kawano A,Ishikawa H,Nakamura T,et al.Evaluation of epidemiolog⁃ical studies of intestinal bacteria that affected occurrence of colorectal cancer:studies of prevention of colorectal tumors by dairy products and lactic acid bacteria[J].Nihon Eiseigaku Zasshi, 2010,65(3):422-446.
[14]沙素梅,吴开春.肠道微生态与大肠肿瘤[J].中华内科杂志,2015, 54(5):402-404.
[15]王姗,曹海龙,鄢芳等.肠道微生态失衡与大肠肿瘤的发生[J].中华内科杂志,2014,53(5):413-415.
(收稿:2015-10-06修回:2016-02-10)
(责任编辑刘洪斌屈振亮)
Relationship of Microbial Community Structure in Intestine with Colorectal Cancer
FU Wen-zheng, ZHANG Chun-ze,JIA Yan-fengDeptment of Anorectal Surgery,Tianjin Peoples′Hospital,Tianjin(300121), China
ObjectiveTo study the relationship ofmicrobial community structure in the intestine with colorectal cancer.MethodsStool specimens were collected in 31 patients with colorectal cancer and 31 pa⁃tients with colorectal adenoma and 31 cases of healthy volunteers from Janurary 2014 to June 2015.Fecal DNA was extracted with fluorescence quantitative PCR for sequencing DNA fingerprinting to compare differences in gut microflora among the three groups.ResultsTotally 254 271 high quality arrays were obtained from the 3 specimen groups,and no statistical differences were found among the 3 groups of multiple polyindices(P>0.05). Proportions of bacteroides genus,Roche genus,acid bacillus,bacillus genera and deformation bacteria in control group were respectively 21.00%,4.10%,2.14%,0.61%and 0.49%,higher than those in colorectal cancer group (14.00%,1.56%,0.79%,0.11%,0.12%and 16.03%)and in coloretal adenomas group(16.03%,1.62%,0.82%, 0.12%,0.14%).Proportions of purple aneromonas spp,escherichia coli,enterococcus,streptococcus and pepto⁃streptococcus in colorectal cancer group were 1.75%,2.45%,2.34%,1.19%and 0.17%respectively.But for colorectal adenomas group,proportions of these bacteria were 1.62%,2.32%,2.12%,1.09%and 0.69%,respec⁃tively.Proportions of these bacteria in above two group were higher than those in control group(they were 0.09%,0.87%,0.10%,0.45%and 0.01%,respectively).Proportions of bacteroides and Roche genus in colorectal group were lower than those in colorectal adenomas group,but for proportions of enterococcus and streptococcus, higher than those in colorectal group.Fluorescence quantitative PCR detection showed that bacteroides relative content and butyric acid salt producing bacteria relative content in control group were(17.38±3.04)%and (4.38±1.03)%,higher than those in colorectal cancer group[(10.85±2.54)%,(1.41±0.89)%]and colorectal ade⁃nomas group[(12.37±2.05)%,(2.07±1.02)%].ConclusionStructural imbalance of gut bacteroides,butyr⁃ate-producing bacteria and opportunistic pathogens Streptococcus is closely related to the occurrence of colorec⁃ tal cancer.
Intestinal flora;colorectal cancer; colorectal adenoma;colon tumor
R735.3+4
A
1007-6948(2016)02-0120-05
10.3969/j.issn.1007-6948.2016.02.004
天津市卫生计生委重点攻关项目(10KG206)
天津市人民医院肛肠科(天津 300121)
付文政,Email:doctor.fwz@gmail.com
