ITS2序列鉴定大蓟、小蓟药材及其近缘混伪品
2016-12-09张景景祁晓婷朱莉敏胡志刚
张景景 祁晓婷 张 超 朱莉敏 杜 康 胡志刚,3
(1 南阳理工学院张仲景国医国药学院,南阳,473000; 2 湖北中医药大学药学院,武汉,430065;3 湖北中医药大学詹亚华名医工作室,武汉,430065)
ITS2序列鉴定大蓟、小蓟药材及其近缘混伪品
张景景1,2祁晓婷1,2张 超2朱莉敏1,2杜 康2胡志刚2,3
(1 南阳理工学院张仲景国医国药学院,南阳,473000; 2 湖北中医药大学药学院,武汉,430065;3 湖北中医药大学詹亚华名医工作室,武汉,430065)
目的:应用ITS2序列鉴别大蓟、小蓟药材及其近缘混伪品,以保障临床用药准确。方法:收集大蓟、小蓟药材的基原物种蓟和刺儿菜及其近缘混伪品,经DNA提取、PCR扩增、测序和序列拼接等步骤,获得ITS2序列,结合GenBank载序列共14个物种54条序列,用MEGA6.0对序列进行比对分析,计算种内种间K2P(Kimura 2-Parameter)遗传距离,构建系统邻接(NJ)树。结果:ITS2序列分析表明,蓟和刺儿菜种内变异位点数均远小于其与混伪品种间变异位点,以上2个物种种内最大(平均)K2P距离均小于其与混伪品种间最小(平均)K2P距离;基于ITS2序列的系统NJ树可以将蓟和刺儿菜及其近缘混伪品很好地区分开,且各物种均单独成支。结论:ITS2序列可以很好地鉴定大蓟、小蓟药材及其近缘混伪品。
大蓟;小蓟;ITS2序列;鉴定;近缘混伪品
《中华人民共和国药典》(2015年版)记载,大蓟、小蓟药材分别为菊科蓟属(CirsiumMill.)植物蓟CirsiumjaponicumFisch.ex DC.和刺儿菜Cirsiumsetosum(Willd.)MB.的干燥地上部分[1],为我国常用凉血止血和祛瘀消肿药,具有重要的药用价值。蓟属植物在我国分布有50余种,其中10余种可作药用[2],其在植物形态和药材性状方面极为相似,难以区分。大蓟和小蓟药材在我国药用历史悠久,始于《名医别录》。两者虽然外形相似,成分相近,但临床应用各有侧重,大蓟解毒疗疮作用较强,而凉血止血作用弱,小蓟凉血止血作用强,而解毒之力弱,故应区别使用,不可混淆[3-4]。据本草考证记载,大蓟药材除来源于同属植物外,还用了飞廉属(CarduusL.)植物,且中药小蓟存在多来源情况[5-6]。目前全国各地药材市场上流通的大蓟和小蓟药材混淆现象时有发生,大蓟药材涉及菊科3属13种植物[7-8],同属多种植物都充作小蓟药材使用,直接影响到临床用药的安全与准确。由于传统的四大鉴定方法主观性强、稳定性差、依赖专业鉴定人才,具有一定的局限性。因此,亟待建立一种快速准确的鉴定方法来区分大蓟、小蓟药材及其近缘混伪品。
DNA条形码(DNA barcoding)是利用基因组中一段通用的标准短序列来实现物种鉴定的新方法,具有操作简易、可重复性强、不受样品形态特征限制等优点,在生物分类学研究中得到广泛应用[9]。陈士林[10]等通过大样本量系统研究,建立了以ITS2序列为主体、psbA-trnH序列为补充的药用植物DNA条形码鉴定体系,中药材DNA条形码分子鉴定指导原则于2010年已列入《中华人民共和国药典》[11]。目前,以ITS2序列为代表的DNA条形码技术在中药材物种鉴定领域已经得到了大量验证[12-16],并成功应用于药材市场真伪检测[17],在中药材物种基原鉴定方面具有广阔的应用前景。本研究运用ITS2序列来鉴别大蓟、小蓟药材及其近缘混伪品,为该属植物的准确鉴定提供依据。
1 材料
本研究收集大蓟、小蓟药材基原物种蓟和刺儿菜及同属混伪品共7个物种20份样品,经中国科学院武汉植物园赵子恩研究员鉴定,凭证标本保存于湖北中医药大学中药标本馆。另从GenBank数据库下载34条相关序列,所有序列经查阅文献确保其准确可靠[18-24]。样本信息及GenBank下载序列信息详见表1。
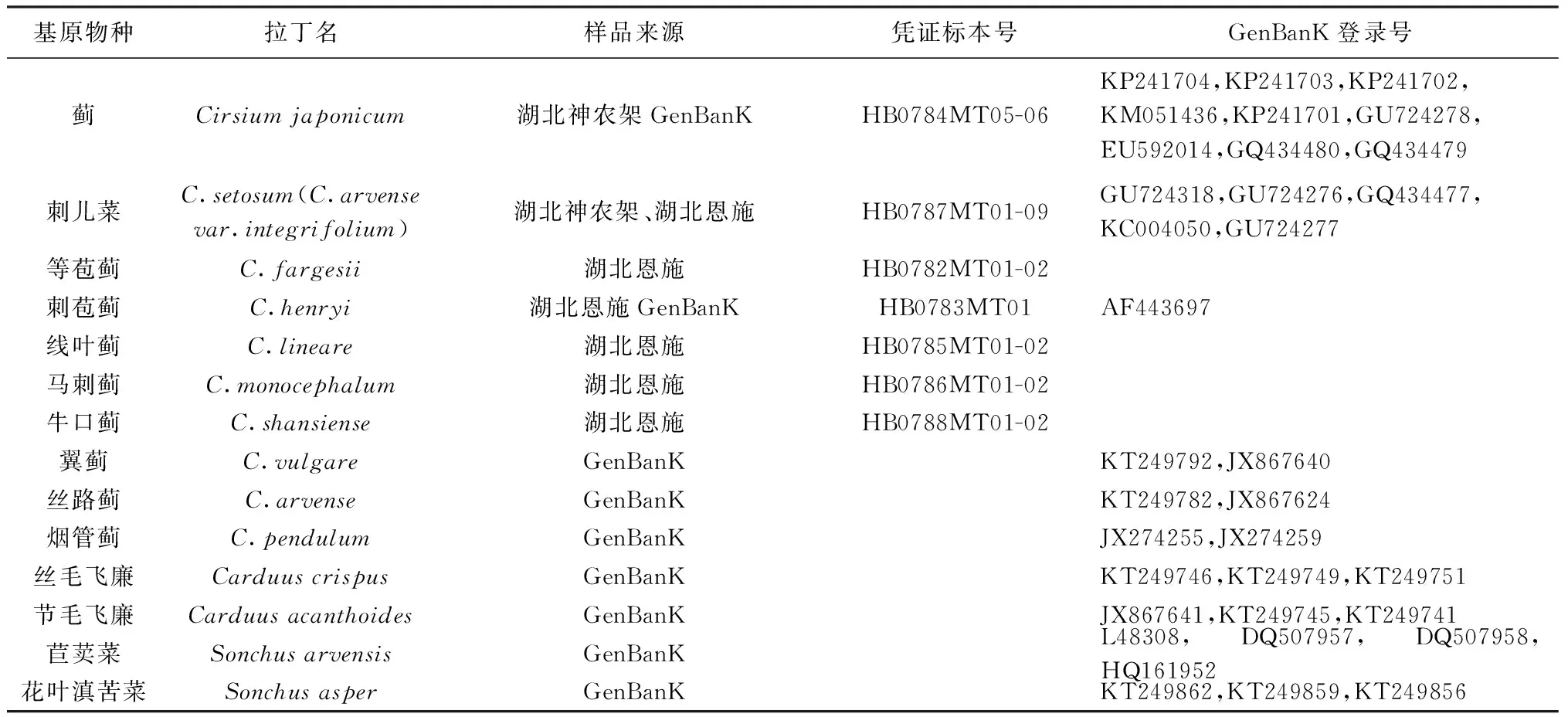
表1 大蓟、小蓟药材及其近缘混伪品样本信息
2 方法
2.1 DNA提取 取干燥叶片经75%乙醇擦拭表面后称取30 mg,用球磨仪(Retsch MM400,Germany)研磨2 min(30 Hz),采用改良的CTAB法提取实验样品总DNA。
2.2 PCR扩增及测序 ITS2通用引物:正向引物ITS2-P3:5′-YGACTCTCGGCAACGGATA-3′,反向引物ITS2-E4:5′-RGTTTCTTTCCTCCGCTTA-3′;反应体系:2×TaqPCR Master Mix 12.5 μL,正反向引物(2.5 μmol·L-1)各1.0 μL,总DNA 2 μL,加ddH2O至25 μL;扩增程序:94 ℃预变性5 min;94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸45 s,进行40个循环;72 ℃延伸10 min[11]。扩增产物经琼脂糖凝胶电泳检测,选择条带单一且清晰的产物进行双向测序。
2.3 数据处理 测序所得峰图采用CondonCode Aligner V4.2.4(CondonCode Co.,USA)软件进行校对拼接,基于隐马尔可夫模型的HMMer注释方法,去除5.8S端和28S端,获得ITS2间隔区序列[25]。使用软件MEGA(Molecular Evolutionary GeneticsAnalysis)6.0进行序列变异分析,计算种内种间K2P(Kimura2-Parameter)遗传距离,构建系统邻接(Neighbor Joining,NJ)树。
3 结果与分析
3.1 序列变异分析 大蓟药材基原物种蓟共获得11条ITS2序列,序列比对后长度为227 bp,有3个单倍型,产生2个变异位点。见表2;小蓟药材基原物种刺儿菜共有14条ITS2序列,序列比对后长度为228 bp,未产生变异位点;蓟属10个物种共41条ITS2序列,序列比对后长度为231 bp,共产生40个变异位点;蓟属及其混伪品14个物种共54条序列,序列比对后长度为222 bp,共产生99个变异位点,远多于以上蓟属物种种间变异位点数(40个)。
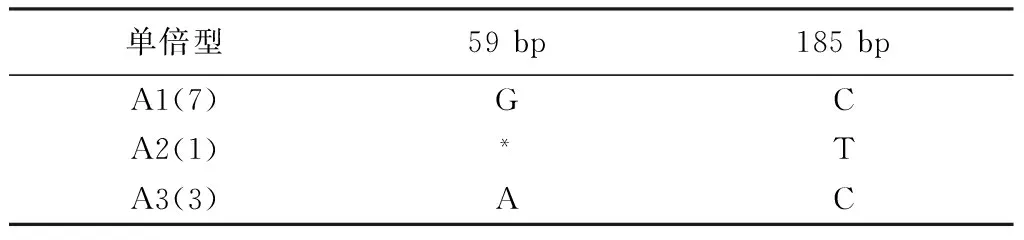
表2 大蓟药材基原物种蓟ITS2序列变异位点
注:括号中数字代表单倍型序列条数,“*”表示与第一行碱基相同。
3.2 遗传距离分析 应用MEGA6.0分析大蓟、小蓟药材及其混伪品的种内种间K2P距离,结果见表3。由结果可知,大蓟药材最大K2P距离(0.009)小于其与混伪品的最小K2P距离(0.019),平均K2P距离(0.003)小于其与混伪品的种间平均K2P距离(0.112);小蓟药材最大K2P距离(0.000)小于其与混伪品的最小K2P距离(0.059),平均K2P距离(0.000)小于其与混伪品的种间平均K2P距离(0.139),这说明K2P距离可很好地区分开大蓟、小蓟药材及其混伪品。
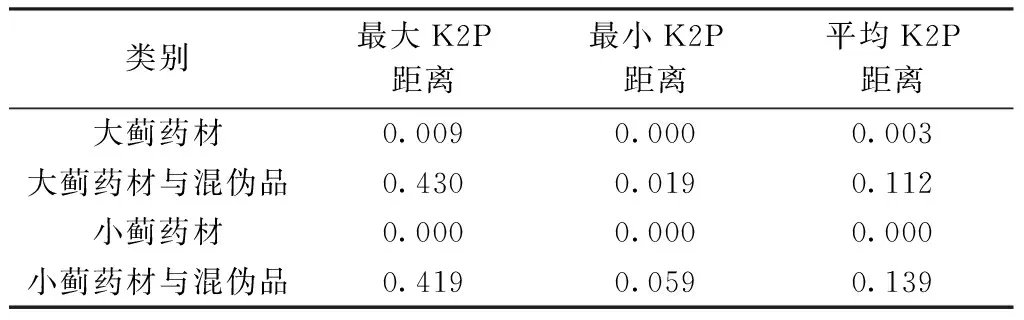
表3 大蓟、小蓟药材及其混伪品K2P距离

图1 基于ITS2序列构建的大蓟、小蓟药材及其近缘混伪品NJ树图
注:Bootstrap 1000次重复,枝上数值仅显示自展支持率≥50%。
3.3 系统聚类分析 基于ITS2序列构建的大蓟、小蓟药材及其混伪品的系统NJ树见图1。从NJ树可知,大蓟药材基原物种蓟11条序列单独聚为一小支,与烟管蓟聚成的一小支共同聚在一大支上,支持率为76%,说明两者的亲缘关系很近;小蓟药材基原物种刺儿菜14条序列单独聚为一支,支持率为100%;同时其余所有近缘混伪品也各自聚为一支,均能很好地区分开。
4 讨论
近年来,中药材市场快速发展,药材掺伪现象屡有发生,为中医用药安全带来隐患。大蓟、小蓟药材因其混伪品主要来自于同科或同属植物,极易产生药材混用情况。中药材的准确鉴定直接影响到临床用药的有效性和安全性,故应选择准确有效的方法以鉴定大蓟和小蓟药材。由于传统的鉴别方法鉴定困难,而DNA条形码技术抛开了植物形态及药材性状上的假象,摆脱了传统鉴定方法依赖专业鉴定人才的障碍,故本研究采用了该技术以鉴定大蓟、小蓟药材及其近缘混伪品。
ITS2序列变异结果可知,大蓟、小蓟药材种内变异位点数(2个和0个)远小于其与混伪品的种间变异,蓟属物种种间变异位点数(40个)远小于该属物种与其近缘混伪品种间变异位点数(99个),此结果与物种分类学的结论相吻合。K2P距离分析结果表明,大蓟和小蓟药材及其混伪品的种内最大(平均)K2P距离均小于其各自与混伪品的种间最小(平均)K2P距离,说明K2P距离可将以上2种药材及其混伪品很好地区分开。基于ITS2序列构建的系统NJ树可知,大蓟和小蓟药材均单独成支,显示良好的单系性,且其余各物种均单独聚为一支。NJ树结果表明,ITS2序列不仅能很好地区分大蓟和小蓟药材,且对本研究涉及的相关近缘混伪品均具有较好的鉴定效果。
本研究结果表明,ITS2序列可以很好地鉴定大蓟、小蓟药材及其近缘混伪品,为这2种药材的临床用药安全提供了可行的物种基原鉴定技术,且从基因水平上为蓟属及近缘物种的分子鉴定分类系统研究及资源开发利用奠定了基础。
[1]国家药典委员会.中华人民共和国药典(一部)[S].北京:中国医药科技出版社,2015:25,48-49.
[2]张鹏云,张耀中.中国植物志[M].北京:科学出版社,2004.
[3]魏彦,邱乃英,欧阳青.大蓟、小蓟的鉴别与临床应用[J].北京中医杂志,2002,21(5):296-297.
[4]倪晓霓.大蓟与小蓟的研究现状及展望[J].时珍国医国药,2005,16(6):548-549.
[5]李媛.大蓟及其混伪品鉴别[J].时珍国医国药,2000,11(8):711.
[6]金延明,李胜华,楼之岑.大蓟与小蓟品种的本草考证[J].中药材,1995,18(3):152-154.
[7]吴蜀星,李治昊,宋良科,等.大蓟的基源调查与鉴定[J].现代中药研究与实践,2013,27(1):21-23.
[8]金延明,李胜华,楼之岑.中药大蓟和小蓟植物资源调查[J].中国中药杂志,1994,19(12):710-711.
[9]陈士林,庞晓慧,姚辉,等.中药DNA条形码鉴定体系及研究方向[J].世界科学技术-中医药现代化,2011,13(5):747-754.
[10]Chen S L,Yao H,Han J P,et al.Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plants species[J].PLoS ONE,2010,5(1):e8613.
[11]陈士林,姚辉,韩建萍,等.中药材DNA条形码分子鉴定指导原则[J].中国中药杂志,2013,38(2):141.
[12]郭力城,胡志刚,凃媛,等.基于ITS2序列鉴定中药材金沸草、旋覆花及其近缘混伪品[J].世界科学技术-中医药现代化,2014,16(2):307-312.
[13]辛天怡,姚辉,罗焜,等.羌活药材ITS/ITS2条形码鉴定及其稳定性与准确性研究[J].药学学报,2012,47(8):1098-1105.
[14]熊超,周红,贺海波,等.基于ITS2序列鉴定谷物芽类药材及其混伪品[J].世界科学技术-中医药现代化,2014,16(11):2343-2348.
[15]明孟碟,张超,严绪华,等.ITS2序列鉴定十大功劳属药用植物[J].中国现代中药,2015,17(10):993-1003.
[16]魏蒙,邬兰,凃媛,等.基于ITS2序列鉴别牡丹皮药材及其混伪品[J].中国中药杂志,2014,39(12):2180-2183.
[17]Han,J.et al.An authenticity survey of herbal medicines from markets in China using DNA barcoding[J].Sci.Rep.2016,6,18723.
[18]Kelch D G,Baldwin B G.Phylogeny and ecological radiation of New World thistles(Cirsium,Cardueae-Compositae)based on ITS and ETS rDNA sequence data[J].Mol Ecol,2003,12(1):141-151.
[19]Choi Y J,Thines M.Host Jumps and Radiation,Not Co-Divergence Drives Diversification of Obligate Pathogens.A Case Study in Downy Mildews and Asteraceae[J].PLoS ONE,2015,10(7):E0133655.
[20]Boto Slotta T A,Horvath D P,Foley M E.Phylogeny of Cirsium spp.In North America:Implications for Biological Control[J].Plants,2012,1(2):61-73.
[21]Chen S,Yao H,Han J,et al.Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species[J].PLoS ONE,2010,5(1):E8613.
[22]Gao T,Yao H,Song J,et al.Evaluating the feasibility of using candidate DNA barcodes in discriminating species of the large Asteraceae family[J].BMC Evol Biol,2010,10:324.
[23]Kim S C,Crawford D J,Francisco-Ortega J,et al.A common origin for woody Sonchus and five related genera in the Macaronesian islands:molecular evidence for extensive radiation[J].Proc Natl Acad Sci USA,1996,93(15):7743-7748.
[24]Kim S C,Chunghee L,Mejias J A.Phylogenetic analysis of chloroplast DNA matK gene and ITS of nrDNA sequences reveals polyphyly of the genus Sonchus and new relationships among the subtribe Sonchinae(Asteraceae:Cichorieae)[J].Mol Phylogenet Evol,2007,44(2):578-597.
[25]keller A,Schleicher T,Schultz J,et al.5.8S-28SrRNA interactionand HMM-based ITS2 annotation[J].Gene,2009,430(1/2):50-57.
(2016-04-12收稿 责任编辑:洪志强)
Identification of Cirsium japonicum,Cirsium setosum and Their Adulterants Based on ITS2 Sequence
Zhang Jingjing1,2,Qi Xiaoting1,2,Zhang Chao2,Zhu Limin1,2,Du Kang2,Hu Zhigang2,3
(1ZhangZhong-jingTraditionalChineseMedicineCollege,NanyangInstituteofTechnology,Nanyang473004,China;2CollegeofPharmacy,HubeiUniversityofChineseMedicine,Wuhan430065,China; 3FamousDoctor′sStudioofZhanYahua,HubeiUniversityofChineseMedicine,Wuhan430065,China)
Objective:To identify C.japonicum, C.setosum and their adulterants through ITS2 Sequence method to ensure clinical medication safety. Methods:DNA was extracted from C. japonicum and C.setosum, which were the original plant samples of Cirsii Japonici Herba and Cirsii Herba together with their adulterant and ITS2 sequences were obtained by PCR, sequencing, and sequence assembly. Totally, 54 sequences of 14 species were downloaded from the GenBank, using MEGA 6.0 to align all sequences, calculating the intraspecific and interspecific Kimura-2-Parameter (K2P) genetic distance and constructing NJ tree. Results:It suggested that the intraspecific variation locis between C.japonicum and C.setosum were much less than that between adulterants. The maximum (average) intraspecific distance of C. japonicum and C.setosum was lower than the minimum (average) interspecific distance of adulterants. Based on ITS2 sequences, this identification method of NJ Tree were effective and clearly distinguished C. japonicum and C.setosum from their adulterant. Also, all species showed obvious monophyly. Conclusion:The sequence of ITS2 identified the herbal materials of C.japonicum, C.setosum and their Adulterants.
Cirsium japonicum;Cirsium setosum;ITS2 sequence;Identification;Adulterants
湖北省科技支撑计划(研发与示范类)(编号:2015BCA275):茯苓等8种湖北优势中药材DNA条形码分子鉴定体系构建,负责人:胡志刚
R282.5
A
10.3969/j.issn.1673-7202.2016.10.052
