几种灵芝rDNA基因间隔序列分析与鉴定*
2016-12-06张凤琴张旺凡李小龙赵彤
张凤琴,张旺凡,李小龙,赵彤
(1.湖南工业大学包装与材料工程学院,湖南 株洲 412007;2.湖南中医药高等专科学校,湖南 株洲 412012)
几种灵芝rDNA基因间隔序列分析与鉴定*
张凤琴1,张旺凡2,李小龙1,赵彤1
(1.湖南工业大学包装与材料工程学院,湖南 株洲 412007;2.湖南中医药高等专科学校,湖南 株洲 412012)
目的基于基因间隔序列(ITS)序列对灵芝进行分类。方法培养灵芝、提取DNA、优化聚合酶链反应(PCR)体系、纯化与克隆、测序、构建遗传进化树、进行遗传分析。结果经过一系列的梯度实验得知优化的PCR体系为25μl反应混合物中含模板DNA 25 ng、Mg2+为2.0 mmol/L、dNTPs 200μmol/L、Taq 1.5 U、引物为20 pmol;最佳反应程序为93℃预变性4 min,93℃变性40 s,59℃退火40 s,72℃延伸1 min,共35个循环,72℃继续延伸7 min,4℃无限循环。所采用的HZ002、DZ003、NZ004和XC005在遗传进化树上面十分接近,说明其可以划分为同一菌属,同属于灵芝菌属。结论基于ITS序列对灵芝进行分类,与其他的分子标记技术所取得的分类结果一致,证实依据ITS序列来对真菌菌属进行分类是十分合理的。
灵芝;聚合酶链反应;基因间隔序列;遗传进化
灵芝是一种名贵的药用真菌,在我国有悠久的药用历史[1]。《神农本草经》将其列为上品[2]。现代医学上也广泛研究和使用[3-6]。灵芝生长的适宜环境为:28~30℃温度、空气湿度70%~80%、二氧化碳CO2的比例<0.1%、黑暗的外界生长条件[7]。以灵芝的形态学特征作为分类标准是灵芝科传统的分类鉴定方法,主要是以其孢子大小和成体的外在的形态学特征作为分类依据[8],也有其他的区分野生和种植的方法[9]。但是子实体的形态特征易受气候、温度、水分、光照等外界条件的影响,很不稳定,才会出现同一个菌种在不同的环境中生长会出现不同的菌体形态的现象[10]。所以对于菌种的准确命名带来很大的障碍,从而导致出现的灵芝菌种的分类以及命名紊乱的现象。基因间隔序列(internal transcribed spacer,ITS)包含核糖体脱氧核糖核酸(ribosomal deoxyri bonucleic acid,rDNA)中介于18 S与5.8 S之间的ITSl,5.8S及5.8S与28S之间的ITS2共3个区域[11]。ITS序列被普遍用于研究属和种间的分类鉴定中,由于进化速率快且长度适宜(在高等真菌中一般<1 000 bp)而被开发成为高等真菌属内种间和种群系统学研究的核基因标记[12]。本实验就是以鉴定出的ITS序列为分析基础,将各种灵芝样品的ITS序列作为目标脱氧核糖核酸(deoxyribonucleic acid,DNA),结合相关的分子生物学技术,得到各个灵芝样品的遗传序列和遗传进化树,并将其与其他的菌种进行对比分析进而得到灵芝的遗传进化现状。
1 材料与方法
1.1实验材料
本实验材料为以下列4种菌种:赤大芝、赤小芝、南韩甜芝和高脚灵芝,其序号为:HZ002、DZ003、NZ004、XC005,均由华中农业大学食用菌研究所提供。实验对其进行ITS序列的测定与分析。
1.2实验方法
1.2.1总DNA的提取采用十六烷基三甲基溴化铵法(cetyltrimethylammonium Ammonium bromide,CTAB)。取长势较好的灵芝菌丝将其放入到低于80℃的高温干燥箱进行干燥,再取其大约20 mg干燥的菌丝用液氮研磨成粉末,加入到1.5 ml离心管中;加入已经65℃预热的CTAB提取液800μl,加入10%β-巯基乙醇20μl,颠倒混匀,放在65℃的恒温水浴锅里面水浴40 min(每隔10 min对其进行振荡摇匀1次),取出,静置冷却至室温。加入400μl Tris饱和酚,在65℃的水浴锅中进行水浴15 min(样品要进行颠倒混匀几次),取出后,10 000 r/min离心10 min,取上清液。加等体积的Tris饱和酚,充分混匀,10 000 r/min离心10 min,取上清液。加入等体积的体积比为25∶24∶1的酚/氯仿/异戊醇,充分混匀,10 000 r/min离心5 min,取上清液。注意吸取的时候,不要碰到上、下分层的中间隔层。将步骤5重复1遍。加入相等体积的异丙醇,在室温下静置10min后,12 000 r/min冷冻离心10 min,丢弃其上清液。注意倾倒其上清液时,要小心不要将管底的微小沉淀倒出去。加入1 ml的70%乙醇洗涤沉淀,使得沉淀处于悬浮状态后即可,弃去乙醇。沉淀用50μl由Tris碱和EDTA配制而成的缓冲液溶解。若未能及时溶解,可在37℃的恒温水浴锅中水浴约30 min左右即可将其完全溶解。取10μl溶液进行1.5%琼脂糖凝胶电泳检测。用752型紫外分光光度计测定各个DNA样品的浓度和纯度,置入-20℃冰箱冷冻保存备用。
1.2.2聚合酶链反应(polymerase chain reaction,PCR)扩增及产物检测在整个PCR体系中,所需要添加的反应物主要有双脱氧的无菌水、10 Taq酶的配套缓冲液、MgCl2溶液、4dNTPs溶液、引物、DNA模板和Taq DNA聚合酶。在整个PCR体系中,Mg2+浓度、dNTPs浓度,以及PCR循环的退火温度是对于扩增结果影响较大的主要因子。因此,本实验对每一个主要变量采用梯度实验的方法以获得最优的反应体系,变量的设置分别为:退火温度57、59、61、63、65和67℃梯度;Mg2+浓度1.0、1.5、2.0、2.5和3.0 mmol/L;梯度:dNTPs浓度100、150、200、250和300μmol/L梯度。取10μl溶液进行1.5%的琼脂糖凝胶电泳检测,溴化乙锭染色。在凝胶成像系统上观察、分析与记录。
1.2.3ITS序列克隆与分析将样品DNA导入到载体上,与经冷氯化钙处理过的感受态细胞进行目的DNA转化,进行蓝白斑筛选。将得到的呈现为白斑的单菌落挑取出来,与PCR产物一起送至上海生物工程股份有限公司进行序列测定。利用遗传分析软件MEGA 6.06与Clustalx两种工具软件对4种灵芝进行遗传分析以及建立遗传进化树。
2 结果
2.1PCR扩增的优化体系
通过一系列的梯度实验得知,最佳的25μl PCR体系为:10 Taq配套缓冲液2.0 mmol/L MgCl2、200μmol/L 4dNTP溶液、0.4μmol/L ITS1和ITS2、25 ng DNA模板和1.5 U Taq DNA聚合酶。反应程序为:94℃预变性4 min,94℃变性40 s,59℃退火40 s,72℃延伸2 min,共计35个循环,72℃继续延伸7 min,4℃无限循环。
2.2ITS序列的PCR结果
通过使用特异性的引物进行PCR扩增后得到的电泳图像见图1。该实验研究的4种灵芝样品的PCR扩增出来的ITS序列在600~700 bp,经查阅相关的资料,符合一般的rDNA的ITS序列的典型长度。
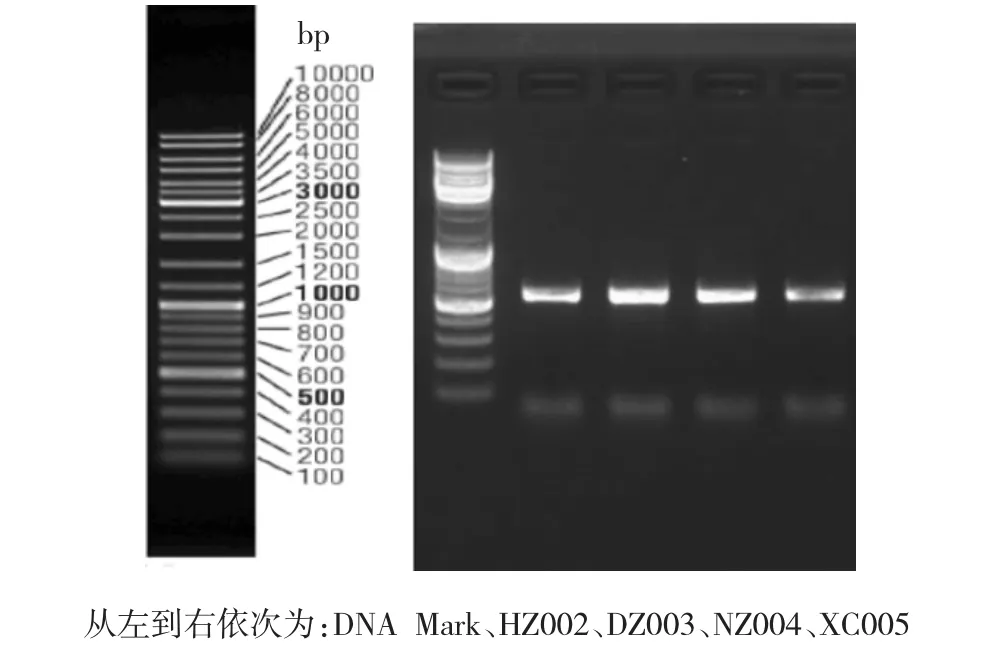
图14 种灵芝样品的ITS序列
2.3ITS序列分析
经测序最终得到包括ITS1、5.8 S及ITS2的长度分别为600(HZ002)、561(DZ003)、601(NZ004)和595 bp(XC005)的序列。见图2。
2.4聚类分析
DZ003、HZ002、NZ004、XC005与其他的真菌的ITS序列数条在美国国立生物技术信息中心的GenBank中记录为KU145513.1(艾隐球菌)、JN164961.1(复本栓菌)、KX147237.1(锗栓孔菌)、KM822753.1(绒毛栓孔菌)、KF963616.1(小薄孔菌)、KC907401.1(香栓孔菌)、KT876604.1(云芝孔菌)、JN164978.1(多带革孔菌)等。通过对比数据库中的相关数据,发现ITS序列的相似度>99%,可将其分类为1个菌属。见图3。


图2 ITS序列图
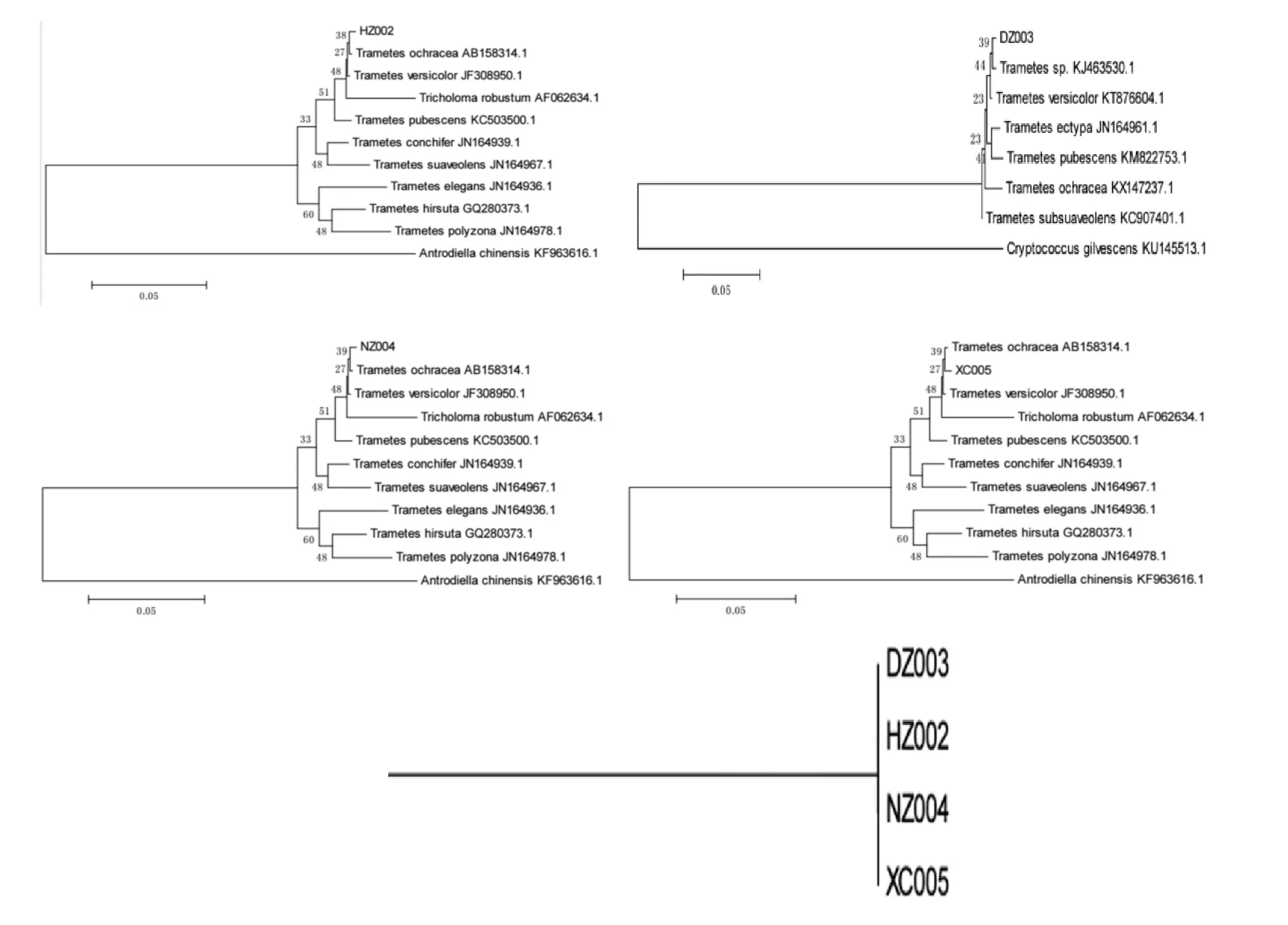
图34 种灵芝的遗传进化树
3 讨论
DZ003与艾隐球菌的遗传距离比较远,说明其进化过程有一定的差距,有不相似的地方,而与复本栓菌、锗栓孔菌、绒毛栓孔菌、栓菌属真菌、香栓菌、云芝孔菌菌种的遗传距离比较近,说明在进化的过程中有很大程度的重叠性,形态学特征和性质特征有一定的相似性。HZ002、NZ004、XC005与小薄孔菌的遗传距离比较远,说明在有些方面具有一定的差异性,而与盘栓孔菌、雅致栓菌、毛栓孔菌、锗栓孔菌、多带革孔菌、绒毛栓孔菌、香栓孔菌、粗毛栓菌、粗壮口蘑菌的遗传距离较近,在其生理学特征及形态学特征有很大程度的相似性,特别是与锗栓孔菌的遗传距离最近,有很大程度的相似性。4种实验样品的相似性很大,遗传距离很近,在很多方面都有极大地相似性,说明可将其归属于同一菌属,都属于灵芝菌属,与使用形态学特征为依据的传统分类学和其他的分子标记技术的分类情况一致[7]。综上所述,利用ITS序列分析的结果与传统的分类学和其他的分子标记技术所呈现的结果十分接近,甚至出现一些传统分类学不能精确分类的结果,再次验证使用ITS序列对灵芝菌属分类的合理性。
[1]金鑫,刘宗敏,黄羽佳,等.我国灵芝栽培现状及发展趋势[J].食药用菌,2016,1:33-37.
[2]邓叔群.中国的真菌[M].北京:科学出版社,1963:445.
[3]MENESES M E,MARTÍNEZ-CARRERA D,TORRES N,et al. Hypocholesterolemic properties and prebiotic effects of mexican ganoderma lucidum in C57BL/6 Mice[J].PLoS One,2016,11(7): DOI:10.1371/journal.pone.0159631.
[4]GUOS S,CUIX L,RAUSCHW D.Ganodermalucidum polysaccharides protect against MPP(+)and rotenone-induced apoptosis in primary dopaminergic cell cultures through inhibiting oxidative stress[J].Am J Neurodegener Dis,2016,5(2):131-144.
[5]JIAO Y,XIE T,ZOU L H,et al.Lanostane triterpenoids from Ganoderma curtisii and their NO production inhibitory activities of LPS-induced microglia[J].Bioorg Med Chem Lett.2016,26(15): 3556-3561.
[6]LEE Y H,KIM J H,SONG C H,et al.Ethanol extract of ganoderma lucidum augments cellular anti-oxidant defense through activation of Nrf2/HO-1[J].J Pharmacopuncture,2016,19(1):59-69.
[7]林志彬.灵芝的现代研究[M].北京:北京医科大学出版社,2001:1-327.
[8]ZHU Y,TAN T L.Penalized discriminant analysis for the detectionofwild-grownandcultivatedganodermalucidumusing Fourier transform infrared spectroscopy[J].Spectrochim Acta A Mol Biomol Spectrosc,2016,159:68-77.
[9]FRANCESCA T,ELENA F,PAOLA P,et al.Exploring the diversity of the bifidbacterial population in the hum an intestinal tract[J].Applied and Environment Microbiology,2009,75(6): 1534-1545.
[10]王磊,宿红艳,吕少磊.八个灵芝属菌株的分子标记与亲缘关系鉴定[J].新乡学院学报,2011,28(3):236-239.
[11]黄龙花,杨小兵,张智,等.基于ITS序列分析鉴定灵芝属菌种[J].中国食用菌,2010,29(1):55-57.
[12]徐凯,唐传红,王天娇,等.基于SRAP、ISSR和RAPD分析灵芝G0130菌株单核体多态性[J].工业微生物,2014,4:39-45.
(童颖丹 编辑)
Analysis and identification of rDNA ITS sequences of severalGanodermaspecies*
Feng-qin Zhang1,Wang-fan Zhang2,Xiao-long Li1,Tong Zhao1
(1.College of Materials Science and Engineering,Hunan University of Technology, Zhuzhou,Hunan 412007,China;2.Hunan Traditional Chinese Medical College, Zhuzhou,Hunan 412012,China)
Objective To classifyGanodermabased on internal transcribed spacer(ITS)sequences.Methods Several species ofGanodermawere cultivated.DNA was extracted.PCR system was optimized followed by purification and cloning,and sequencing.Then phylogenetic tree was constructed,and genetic analysis was made.Results The whole PCR system was optimized which included 25 μl reaction mixture containing template DNA 25 ng,Mg2+2.0 mmol/L,dNTPs 200 μmol/L,Taq 1.5 U,primer 20 pmol.The optimum reaction program was pre-denaturation at 93℃for 4 min,denaturation at 93℃for 40 s,annealing at 59℃for 40 s, prolongation at 72℃for 1 min for 35 cycles;then final prolongation at 72℃for 7 min followed by 4℃infinite loop.Adopted HZ002,DZ003,NZ004 and XC005 were very close in the phylogenetic tree,indicating that they belonged to the same genus,Ganoderma.Conclusions ITS sequence classification is consistent with the traditional taxonomy and classification obtained with other molecular markers,which is very reasonable for classification of fungal genus.
Ganoderma lucidum;polymerase chain reaction;internal transcribed spacer;genetic evolution
R282
A
10.3969/j.issn.1005-8982.2016.21.007
1005-8982(2016)21-0035-05
2016-06-23
湖南省科技厅项目(No:2014sk3026)
张旺凡,Tel:13707338091;E-mail:zwf8866@163.com
