基于分子对接技术预测人面子叶中黄酮成分抗菌作用靶点Δ
2016-12-05夏厚林周厚琴陆小华赵艳玲
李 洋,夏厚林,周厚琴,2,陆小华,2,张 璐,2,赵艳玲
(1.成都中医药大学药学院,四川 成都611137; 2.解放军第302医院药学部,北京 100039)
基于分子对接技术预测人面子叶中黄酮成分抗菌作用靶点Δ
李 洋1,2*,夏厚林1,周厚琴1,2,陆小华1,2,张 璐1,2,赵艳玲2#
(1.成都中医药大学药学院,四川 成都611137; 2.解放军第302医院药学部,北京 100039)
目的:木犀草素、槲皮素、儿茶素、表儿茶素是人面子叶中主要的黄酮成分,研究显示人面子具有抗菌活性,利用Autodock软件,模拟并预测黄酮成分的抗菌作用靶点。方法:采用中医药化学数据库和Autodock 4.2软件进行分子对接。结果:模拟分析得出木犀草素、槲皮素、儿茶素、表儿茶素与青霉素结合蛋白3、青霉素结合蛋白5、DNA聚合酶的对接结果及其功能区域的相互作用关系。结论:木犀草素是人面子叶中潜在活性成分,青霉素结合蛋白5和DNA聚合酶是人面子叶中黄酮成分抗菌的潜在靶点。本研究预测了人面子叶的抗菌作用靶点,为探索其抗菌机制奠定了基础,也为以木犀草素为先导化合物开发新型药物提供了理论基础。
人面子叶; 黄酮; Autodock; 青霉素结合蛋白; DNA聚合酶
人面子(Dracontomelondao)是漆树科人面子属常绿大乔木植物,生长于中国台湾、海南、广西、广东、云南等热带地区。人面子叶即人面子的干燥叶片,是华南地区民间常用的、传统的用于治疗人体皮肤感染特别是皮肤炎症和疼痛的中草药。民国时期萧步丹所著《岭南采药录》记载:“人面子,……解毒,治偏身风毒痛痒”。文献报道,人面子叶的乙醇提取物对金黄色葡萄球菌和枯草芽孢杆菌具有抑制作用,人面子叶的乙酸乙酯提取物对大肠埃希菌(Escherichiacoli.)具有较强的抑制作用[1-2]。此外,人面子的挥发油还具有抗肿瘤的作用[2],人面子叶的提取物具有抗病毒的作用。但是,人面子叶的抗菌作用靶点尚不明确,作用机制有待于进一步研究。计算机辅助药物设计是以计算机为主要工具,结合化合物、生物蛋白数据库,利用药物和相关靶标的知识,通过理论模拟、算法计算,预测药物与受体生物大分子之间的关系,实现合理药物设计的现代药物设计方法。在药物设计中,分子对接技术处于关键性地位[3-6]。分子对接即通过计算机模拟药物分子产生药效反应的过程,药物分子与靶蛋白相互结合,产生相互作用,发挥药效。通过分子对接技术,可确定复合物中靶点与配体正确的相对位置和取向,了解两者的相互作用,确定药物作用机制与设计新药的基础[7-8]。青霉素结合蛋白(penicillin binding protein,PBP)是广泛存在于细菌表面的一种膜蛋白,在异种细菌之间其种类及含量均不相同,但各种菌种的PBP又有许多类似的结构与功能,在细菌生长、繁殖中发挥着重要作用[9-10]。DNA聚合酶是细胞复制DNA的重要作用酶,其以DNA为复制模板,将DNA由5’端点复制至3’端,其主要活性是催化DNA的合成[11]。本研究采用计算机分子对接技术,研究人面子叶中四种主要的黄酮成分槲皮素、儿茶素、表儿茶素、木犀草素与PBP和DNA聚合酶的相互作用关系,探索人面子叶抗菌作用靶点及机制。
1 材料与方法
1.1 材料来源
Autodock 4.2软件来源于美国斯克利普斯研究所(http://autodock.scripps.edu),为免费授权的软件。PBP蛋白和DNA聚合酶蛋白晶体结构来源于蛋白质数据库(http://www.rcsb.org/pdb)。黄酮化合物结构来源于中医药数据库(http://tcm.cmu.edu.tw)。软件运行于Linux操作系统平台下。相关软件均是已授权或免费授权。
1.2 方法
1.2.1 结构预处理:在蛋白质数据库中,搜索并下载PBP蛋白和DNA聚合酶蛋白晶体结构文件。DNA聚合酶,基源为大肠杆菌,蛋白号:4ckl;PBP-5蛋白,基源为大肠杆菌,蛋白号:3beb;PBP-3蛋白,基源为大肠杆菌,蛋白号:4bjp。利用对接软件,去除各蛋白晶体结构中溶剂和相关配体,得到洁净的DNA聚合酶、PBP-3和PBP-5蛋白结构,并加入电荷和极性氢原子,保存。应用Chem3D软件,对从中医药数据库下载的槲皮素、木犀草素、儿茶素、表儿茶素的分子结构进行能量优化,保存。
1.2.2 Autodock分子对接:在Linux终端下运行Autodock 4.2软件,依次加载洁净蛋白和能量优化后的化合物(配体),用Autogrid计算格点能量,对接运算采用拉马克遗传算法,各化合物与蛋白之间的结合情况用半经验的自由能评价方法评价。参数设置过程中,将参数“能量最大评价次数”确定为2 500 000,运行次数确定为100,其他参数采用软件的默认设置。根据相关文献报道,确定各蛋白活性口袋的氨基酸残基和中心点坐标(X,Y,Z):4ckl氨基酸残基为HIS45、LYS42、LEU41、GLY40、ASN165、LEU98、SER171、ASN169、GLY170、ILE182、PRO184、PRO183、TYR266,坐标为(23.204,-34.491,-23.884);3beb氨基酸残基为ASN199、THR214、GLY215、HIS216、TYR222、ASN223、LEU224、GLU249、ARG248、GLY247、THR224、VAL239、GLY242、ARG243、ALA17、ASP16、GLU251,坐标为(41.739,-5.728,26.979);4bjp氨基酸残基为PRO425、MET422、SER261、VAL531、GLU304、VAL302、PHE303、SAP301、ARG297、ALA498、ILE512、ASN533、SER259、LYS500、VAL501、LYS499,坐标为(-16.564,32.895,14.509)[12]。
2 结果
经过Autodock软件的运算分析,得到人面子叶中黄酮类化合物槲皮素、木犀草素、儿茶素、表儿茶素分别与PBP-3、PBP-5、DNA聚合酶蛋白的对接信息。对接信息包括半经验化的自由能评价评分、抑制常数值(inhibition constant,Ki)、对接模拟温度。对于多构象的对接结果,选取前10位的对接构象结果。PBP-3蛋白与表儿茶素、槲皮素、木犀草素、儿茶素相互作用,其最佳结合自由能分别为-26.25、-26.16、-27.08、-26.66 kJ/mol,与之相对应的Ki分别为25.38、26.34、18.19、21.46 μmol/L,见表1。通过比较Ki,发现木犀草素对PBP-3蛋白具有较低的抑制常数和较强的抑制作用。
通过Autodock自带的分析程序,分析得到了PBP-3蛋白和相关黄酮配体的最佳对接构象及其相关作用方式。如图1(A)所示,表儿茶素与PBP-3蛋白结合的空间构型,对应表1中表儿茶素等级1的对接结果。可见,表儿茶素与PBP-3蛋白中GLU258、GLU292、ARG295、THR300的氨基酸残基有相应的相互作用,结合图2(A)发现,表儿茶素与ARG295具有氢键的相互作用,氢键键长是2.62 A。此外,还有数个氨基酸残基与表儿茶素之间具有疏水作用,但尚未形成相关配位键或氢键。图1(B)对应表1中槲皮素等级1的对接结果。可见,槲皮素与PBP-3蛋白中ILE532、ALA513、GLY502、PRO305的氨基酸残基有相应的相互作用,结合图2(B)发现,槲皮素与VAL501、ILE532、ALA513、GLU304具有氢键的相互作用,氢键键长分别是2.62、3.16、2.58、2.64 A。此外,还有数个氨基酸残基与槲皮素之间具有疏水作用,但尚未形成相关配位键或氢键。图1(C)对应表1中木犀草素等级1的对接结果。可见,木犀草素与ASN197的氨基酸残基有相应的相互作用,结合图2(C)发现,木犀草素与ASN197、TYR183、SER252具有氢键的相互作用,其中与ASN197具有多氢键交互作用,但仍有数个氨基酸残基与木犀草素之间具有疏水作用但尚未形成相关配位键或氢键。图1(D)对应表1中儿茶素等级1的对接结果。可见,儿茶素与PBP-3蛋白中PRO305的氨基酸残基有相应的相互作用,结合图2(D)发现,儿茶素与GLU304具有氢键的相互作用,氢键键长是3.04 A。

表1 PBP-3(4bjp)的对接信息

A.表儿茶素;B.槲皮素;C.木犀草素;D.儿茶素A.L-Epicatechin;B.Quercetin;C.Luteolin;D.Cianidanol图1 PBP-3(4bjp)的对接空间位置示意图Fig 1 Space position of PBP-3(4bjp) molecular docking

A.表儿茶素;B.槲皮素;C.木犀草素;D.儿茶素A.L-Epicatechin;B.Quercetin;C.Luteolin;D.Cianidanol图2 PBP-3(4bjp)的对接交互作用二维平面图Fig 2 2D planar graph of PBP-3 (4bjp) molecular docking interaction
PBP-5蛋白与表儿茶素、槲皮素、木犀草素、儿茶素相互作用,其最佳结合自由能分别为-35.62、-31.94、-31.69、-33.49 kJ/mol,与之相对应的Ki分别为0.58、2.55、2.80、1.36 μmol/L,见表2。通过比较Ki发现,这4种黄酮对PBP-5蛋白具有较强的抑制作用,特别是表儿茶素的Ki为582 nmol/L,说明表儿茶素能较强抑制PBP-5蛋白,这可能是人面子叶抑菌作用潜在靶点。

表2 PBP-5(3beb)的对接信息
图3—4对应表2中各黄酮类物质等级为1的对接结果。如图3(A)表儿茶素与PBP-5蛋白结合的空间构型可见,表儿茶素与PBP-5蛋白中ASN197、LEU201、ILE212、VAL225、ARG198的氨基酸残基有相应的相互作用,结合图4(A)发现,表儿茶素与ASN223、LYS213、GLY211、ILE212、ASN197具有氢键的相互作用,氢键键长分别是2.65、3.13、2.94、2.92、2.97 A。此外,还有数个氨基酸残基与表儿茶素之间具有疏水作用但尚未形成相关配位键或氢键。由图3(B)可见,槲皮素与PBP-5蛋白中TYR183、ASN197的氨基酸残基有相应的相互作用,结合图4(B)发现,槲皮素与ILE212具有氢键的相互作用,氢键键长为3.07 A。此外,还有数个氨基酸残基与槲皮素之间具有疏水作用,但尚未形成相关配位键或氢键。由图3(C)可见,木犀草素与ASN197的氨基酸残基有相应的相互作用,结合图4(C)发现,木犀草素与ASN197、TYR183、SER252具有氢键的相互作用,其中与ASN197具有多氢键交互作用。由图3(D)可见,儿茶素与PBP-5蛋白中ASN197、ARG198的氨基酸残基有相应的相互作用,结合图4(D)发现,儿茶素与TYR183具有氢键的相互作用,氢键键长为3.24 A。此外,ASN197是4种黄酮类化合物共同作用的氨基酸残基,说明这是潜在的抗菌作用靶点。木犀草素对PBP-3和PBP-5的作用位点均相同,表明木犀草素的作用位点较为稳定,值得进一步探索。
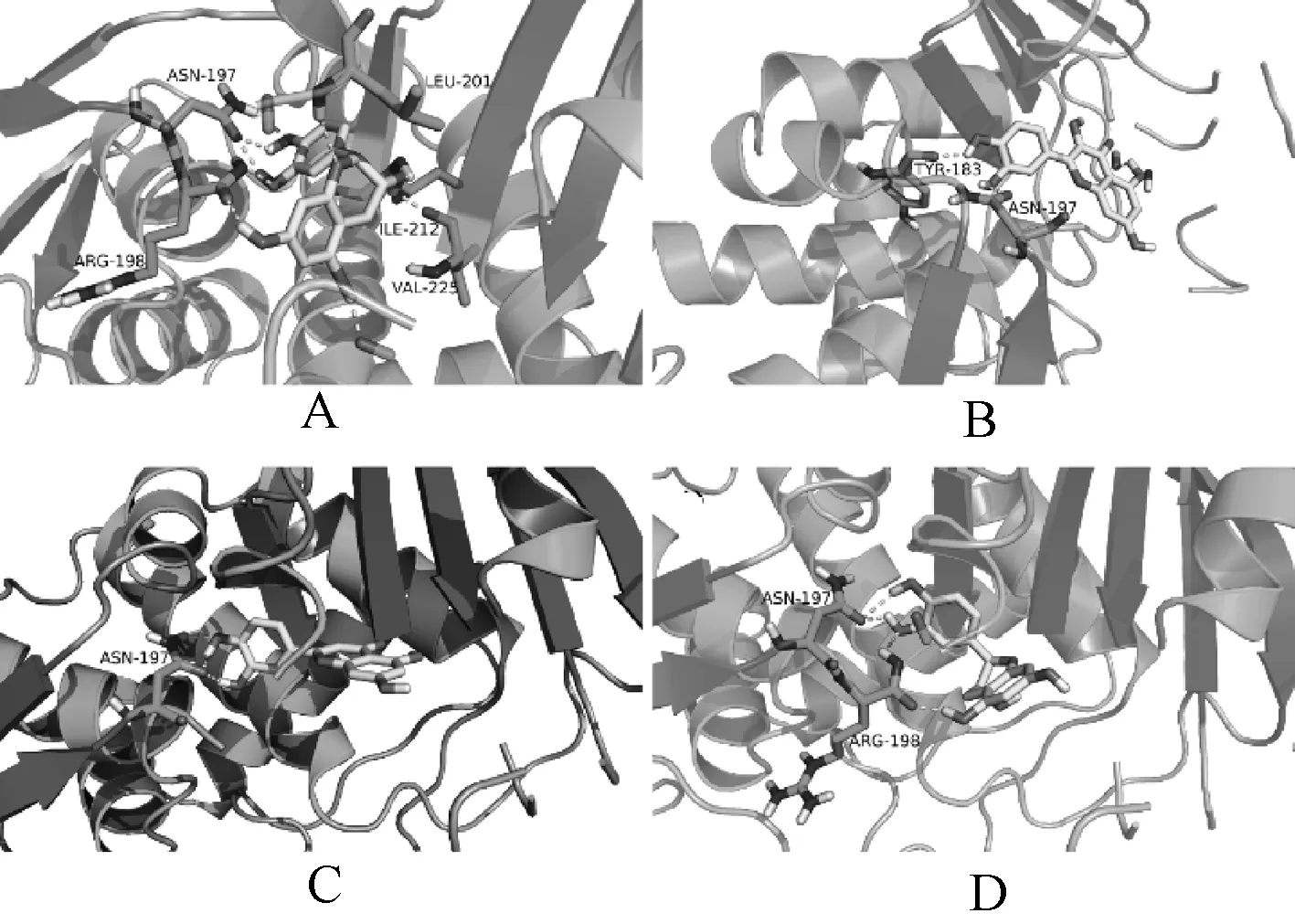
A.表儿茶素;B.槲皮素;C.木犀草素;D.儿茶素A.L-Epicatechin;B.Quercetin;C.Luteolin;D.Cianidanol图3 PBP-5(3beb)的对接空间位置示意图Fig 3 Space position of PBP-5(3beb) molecular docking
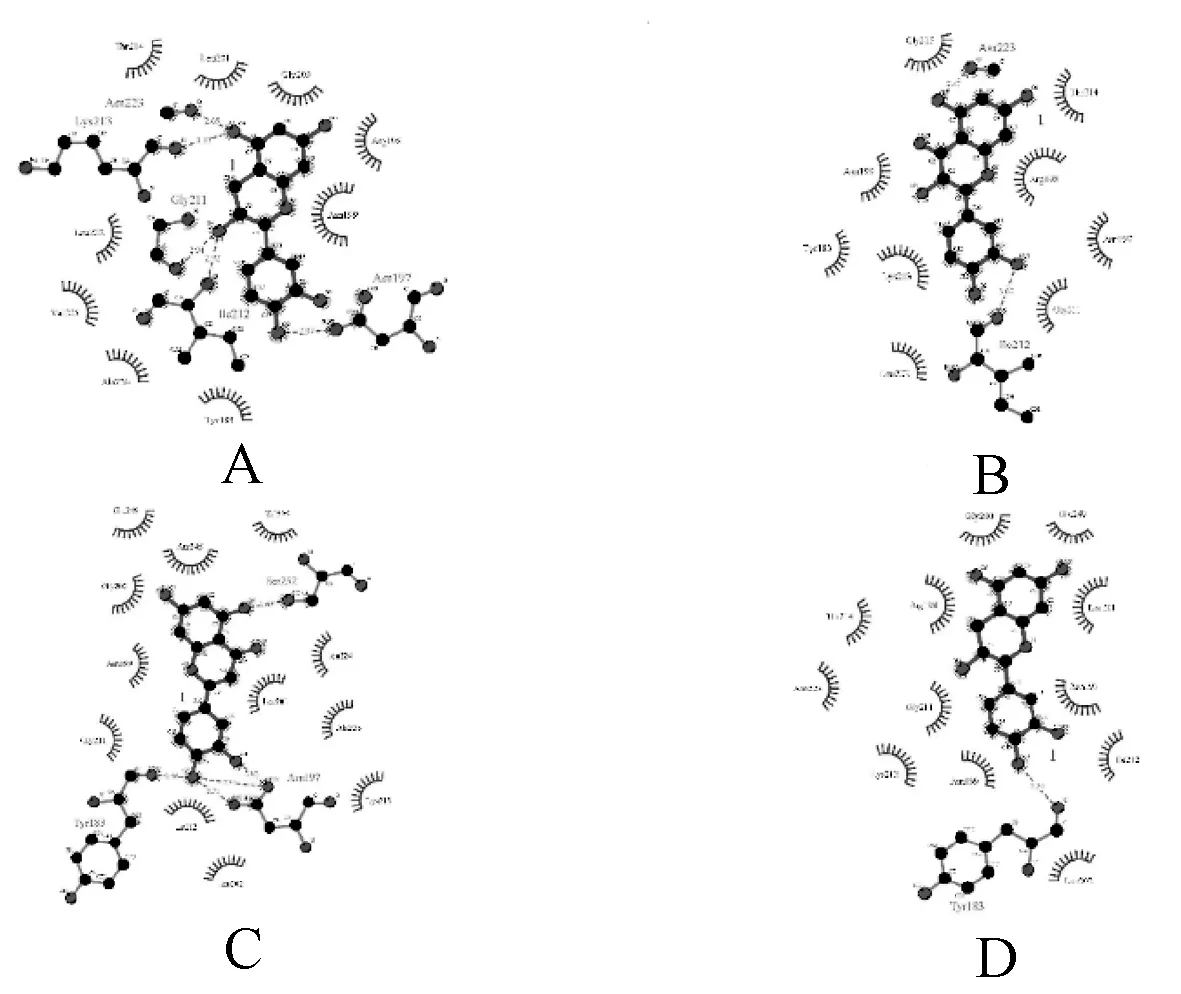
A.表儿茶素;B.槲皮素;C.木犀草素;D.儿茶素A.L-Epicatechin;B.Quercetin;C.Luteolin;D.Cianidanol图4 PBP-5蛋白(3beb)的对接交互作用二维平面图Fig 4 2D planar graph of PBP-5(3beb) molecular docking interaction
DNA聚合酶蛋白与表儿茶素、槲皮素、木犀草素、儿茶素相互作用,其最佳结合自由能分别是-31.23、-30.31、-31.81、-26.83 kJ/mol,与之相对应的Ki分别为3.40、4.96、2.69、20.18 μmol/L,见表3。通过比较Ki发现,表儿茶素、槲皮素、木犀草素对DNA聚合酶蛋白具有较强的抑制作用,特别是木犀草素的Ki为2.69 μmol/L,说明木犀草素能较强抑制DNA聚合酶,这可能是人面子叶抑菌作用潜在靶点。

表3 DNA聚合酶蛋白(4ckl)的对接信息
图5—6对应表3中各黄酮类物质等级为1的对接结果。由图5(A)表儿茶素与DNA聚合酶结合的空间构型可见,表儿茶素与DNA聚合酶中ARG46、ASP39、ILE89的氨基酸残基有相应的相互作用,结合图6(A)发现,表儿茶素与ARG39和ARG42具有氢键的相互作用,键长分别为2.67、3.11 A。由图5(B)可见,槲皮素与DNA聚合酶中SER97、ARG91、ARG99的氨基酸残基有相应的相互作用,结合图6(B)发现,槲皮素与ARG91和ARG99具有氢键的相互作用,氢键键长分别是2.73、3.15 A。由图5(C)可见,木犀草素与SER172、GLY170、PHE96、THR219的氨基酸残基有相应的相互作用,结合图6(C)发现,木犀草素与GLY170、TYR219、SER172具有氢键的相互作用,其中与GLY170具有多氢键交互作用,但仍有数个氨基酸残基与木犀草素之间具有疏水作用但尚未形成相关配位键或氢键。由图5(D)可见,儿茶素与DNA聚合酶中THR219、GLY170的氨基酸残基有相应的相互作用,结合图6(D)发现,儿茶素与GLU304具有氢键的相互作用,氢键键长为3.04 A。
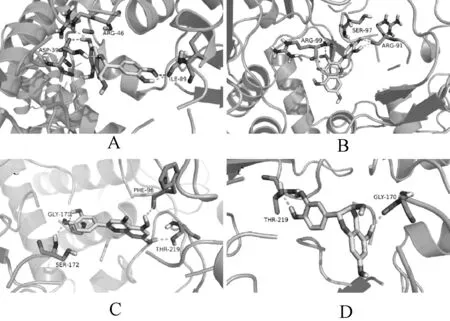
A.表儿茶素;B.槲皮素;C.木犀草素;D.儿茶素A.L-Epicatechin;B.Quercetin;C.Luteolin;D.Cianidanol图5 DNA聚合酶蛋白(4ckl)的对接空间位置示意图Fig 5 Space position of DNA polymerase (4ckl) molecular docking

A.表儿茶素;B.槲皮素;C.木犀草素;D.儿茶素A.L-Epicatechin;B.Quercetin;C.Luteolin;D.Cianidanol图6 DNA聚合酶蛋白(4ckl)的对接交互作用二维平面图Fig 6 2D planar graph of DNA polymerase (4ckl) molecular docking interaction
3 讨论
分子对接是依据酶的锁-钥匙原理,从已知结构的化合物及蛋白出发,通过计算机模拟、化学计量学计算,识别并预测受体-配体结合的方法,在预测蛋白质复合物结构和结合位点方面对探索作用机制具有重要的意义,并已广泛用于药物虚拟筛选、定量构效关系研究。
从单个化合物对蛋白的影响和Ki绝对值大小的角度而言,表儿茶素对PBP-5的Ki最低,为582 nmol/L,表明以Ki绝对值大小为参数,表儿茶素对PBP-5的抑制强度最大。其次分别为儿茶素(其对PBP-5的Ki为1.36 μmol/L)、槲皮素(其对PBP-5的Ki为2.55 μmol/L)、木犀草素(其对DNA聚合酶的Ki为2.69 μmol/L)。但综合3种目标蛋白,木犀草素的算术平均Ki值最低,为7.89 μmol/L(其对PBP-3、PBP-5、DNA聚合酶的Ki分别为18.19、2.80、2.69 μmol/L),其后依次是表儿茶素(9.78 μmol/L)、槲皮素(11.28 μmol/L)、儿茶素(14.33 μmol/L),表明木犀草素是人面子叶中潜在活性成分。
从单个蛋白的作用强度而言,4种黄酮类化合物对PBP-5蛋白的Ki普遍较低,最佳结合构象均<3 μmol/L,说明PBP-5蛋白是人面子叶抗菌作用的潜在靶点。其次为4种黄酮类化合物对DNA聚合酶蛋白的Ki,除儿茶素外其余化合物最佳结合构象均<5 μmol/L,说明DNA聚合酶蛋白也是人面子叶抗菌作用的潜在靶点。相比较而言,4种黄酮类物质对PBP-3蛋白的Ki较高,均>18 μmol/L,说明PBP-3蛋白可能不是人面子叶抗菌作用的靶点。
此外,通过空间位置示意图发现,ASN197是4种黄酮类化合物对PBP-5蛋白共同作用的氨基酸残基。但是在交互作用二维平面图中,4种黄酮类化合物对PBP-5蛋白ASN197氨基酸残基的作用方式各异,表儿茶素和木犀草素是氢键的作用方式,槲皮素和儿茶素是具有疏水作用但尚未形成相关配位键或氢键,说明木犀草素是人面子叶抗菌活性作用的潜在靶点。比较木犀草素的对接信息发现,木犀草素对PBP-3和PBP-5具有相同作用位点,均与TYR183、ASN197、SER252氨基酸残基有氢键作用,表明木犀草素的作用位点较为稳定,值得进一步探索。
[1]苏秀芳,农万延.人面子叶提取物抑菌作用研究[J].中国药房,2010,21(23):2115-2117.
[2]Liu SY,Zhao YL,Zeng N,et al.Anti-bacterial effect of four extracts from leaves ofDracontomelondaoonEscherichiacoligrowth using microcalorimetry coupled with principal component analysis[J].J Therm Anal Calorim,2014,116(1):491-497.
[3]苏秀芳,梁振益,张一献.人面子茎皮挥发油化学成分的研究[J].时珍国医国药,2008,19(7):1640-1641.
[4]Wang SQ,Du QS,Chou KC.Study of drug resistance of chicken influenza A virus (H5N1) from homology-modeled 3D structures of neuraminidases[J].Biochem Biophys Res Commun,2007,354(3):634-640.
[5]Wang SQ,Du QS,Huang RB,et al.Insights from investigating the interaction of oseltamivir (Tamiflu) with neuraminidase of the 2009 H1N1 swine flu virus[J].Biochem Biophys Res Commun,2009,386(3):432-436.
[6]Chou KC,Wei DQ,Zhong WZ.Binding mechanism of coronavirus main proteinase with ligands and its implication to drug design against SARS[J].Biochem Biophys Res Commun,2003,308(1):148-151.
[7]Chou KC.Insights from modelling the 3D structure of the extracellular domain of alpha-7-nicotinic acetylcholine receptor[J].Biochem Biophys Res Commun,2004,319(2):433-438.
[8]Wadood A,Riaz M,Jamal SB,et al.Molecular docking study of P4-Benzoxaborole-substituted ligands as inhibitors of HCV NS3/4A protease[J].Bioinformation,2003,9(6):309-314.
[9]Goto J,Kataoka R,Muta H,et al.ASE Dock-docking based on alpha spheres and excluded volumes[J].J Chem Inf Model,2008,48(3):583-590.
[10] Sauvage E,Powell AJ,Heilemann J,et al.Crystal structures of complexes of bacterial DD-peptidases with peptidoglycan-mimetic ligands: the substrate specificity puzzle[J].J Mol Biol,2008,381(2): 383-393.
[11] Sauvage E,Derouaux A,Fraipont C,et al.Crystal structure of penicillin-binding protein 3 (PBP3) from Escherichia coli[J].PloS One,2014,9(5):e98042.
[12] Hearnshaw SJ,Edwards MJ,Stevenson CE,et al.A new crystal structure of the bifunctional antibiotic simocyclinone D8 bound to DNA gyrase gives fresh insight into the mechanism of inhibition[J].J Mol Biol,2014,426(10):2023-2033.
Prediction of Target Points of the Antibacterial Activity of Flavonoids From the Leaves of Dracontomelon Dao Based on Molecular Docking TechnologyΔ
LI Yang1,2, XIA Houlin1, ZHOU Houqin1,2, LU Xiaohua1,2, ZHANG Lu1,2, ZHAO Yanling2
(1.College of Pharmacy, Chengdu University of Traditional Chinese Medicine,Sichuan Chengdu 611137, China; 2.Dept.of Pharmacy, 302 Military Hospital of PLA, Beijing 100039, China)
OBJECTIVE:Luteolin, Cianidanol, Quercetin, PBP-3(4bjp)were the major flavonoids from the leaves ofDracontomelondao. It was reported thatDracontomelondaohad antibacterial activity. Autodock software was adopted to simulate and predict the target points of the antibacterial activity of flavonoids. METHODS: Chinese medicine chemistry database and Autodock 4.2 software were used for the molecular docking. RESULTS: The simulation analysis showed the correlation of docking results and functional area betwwen Luteolin, Cianidanol, Quercetin, L-Epicatechin and PBP-3, PBP-5, DNA polymerase. CONCLUSIONS: Luteolin is the potential active component inDracontomelondao. PBP-5 and DNA polymerase are the potential targets for the antibacterial activity of flavonoids from the leaves ofDracontomelondao. In this study, antibacterial targets was predicted by Autodock softwave which lays the foundation for exploring the antibacterial mechanism and provide theory basis for the development of new drugs with luteolin as lead compounds.
Dracontomelondao; Flavonoids; Autodock; PBPs; DNA polymerase
国家重大新药创制专项(No.2015ZX09J15102-004);国家自然科学基金资助项目(No.81342571)
R96
A
1672-2124(2016)10-1303-05
2016-08-29)
*在读硕士研究生。研究方向:中药质量控制、计算机辅助药物设计。E-mail:liyang0838@qq.com
#通信作者:主任医师,博士,研究员。研究方向:中药药理学。E-mail:zhaoyl2855@126.com
DOI 10.14009/j.issn.1672-2124.2016.10.003
