利用SSR分子标记研究国内外黍稷地方品种和野生资源的遗传多样性
2016-12-01乔治军刘敏轩王瑞云
连 帅,陆 平,乔治军,张 琦,张 茜,刘敏轩,王瑞云
(1山西农业大学农学院,山西太谷 030801;2中国农业科学院作物科学研究所,北京 100081;3山西农业科学院品种资源研究所,太原 030031)
利用SSR分子标记研究国内外黍稷地方品种和野生资源的遗传多样性
连 帅1,2,陆 平2,乔治军3,张 琦2,张 茜2,刘敏轩2,王瑞云1
(1山西农业大学农学院,山西太谷 030801;2中国农业科学院作物科学研究所,北京 100081;3山西农业科学院品种资源研究所,太原 030031)
【目的】从分子水平研究国内外黍稷种质资源的遗传多样性差异,为黍稷种质资源的研究、保护和利用提供依据。【方法】用不同地理来源且性状差异显著的6份黍稷种质资源对来自高通量测序技术开发的黍稷基因组SSR引物进行筛选,从而获得条带清晰,稳定性好的63对SSR黍稷基因组引物,利用这63对SSR多态性引物对来自国内外的192份黍稷地方品种和野生种质进行遗传多样性分析。统计各试材在同一引物中的条带情况,并以此来分析试材的遗传多样性与所在群体间的亲缘关系。【结果】63对SSR引物共检测出161个等位变异位点,平均每个SSR位点2.56个;平均Shannon-Weaver指数(I)为0.6275,平均基因多样度(Nei)为0.3874,平均PIC值为0.4855。10个不同地理来源群体间表现出显著的遗传多样性差异,各群体的有效等位变异变化范围较窄,最小的是南方群体,为1.2407±0.4315;最大的是内蒙古高原群体,为1.8846±0.4892。国内群体Shannon-Weaver指数为内蒙古高原>东北地区>黄土高原>西北地区>南方地区,而国外Shannon-Weaver指数排序依次为前苏联>欧洲>蒙古>印度>美国。从Nei’s基因杂合度分析,观察杂合度(Ho)最小的是印度群体,为0.2372±0.2962,最大的是内蒙古高原群体,为0.3966±0.3250。期望杂合度(He)最小的是美国群体,为0.3114±0.2203;最大的是内蒙古高原群体,为0.4622±0.1862。从国外种、国内栽培种和国内野生种3个大群体来看,野生种质资源有效等位基因数(1.9285±0.5101)、Shannon-Weaver指数(0.6948±0.2852)、Nei基因多样性指数(0.4373± 0.1773)远大于国外种和国内栽培种。而对国内外两大群体而言,国内资源的有效等位基因数(1.8145±0.4519)、Shannon-Weaver指数(0.6657±0.2413)和Nei基因多样性指数(0.412±0.1574)均大于国外资源(1.6862± 0.4527、0.5897±0.2469、0.3652±0.1655)。UPGMA聚类分析结果显示,10个地理群聚为三大类,内蒙古高原地区、黄土高原地区、东北地区、西北地区、蒙古地区聚为一类,前苏联、美国、印度、欧洲地区聚为一类,南方地区单独聚为一类。其中,来自东北黑龙江齐齐哈尔的泰来小野糜(34号)在截距0.37处被独立分为一支,来自甘肃的野黍子(19号)在截距0.34处被分为独立个体,表明这两个材料与其他材料遗传差异较大。但从整体遗传多样性上来看192份材料国内外群体遗传分化不明显,群体间的亲缘关系较近,且不同群体间材料存在着互相渗透。【结论】内蒙地区、东北地区、黄土高原地区种质资源遗传多样性最丰富,是遗传关系最为复杂的地区,进一步印证了中国是黍稷起源的中心。
黍稷;地方品种;野生种;SSR标记;遗传多样性
0 引言
【研究意义】黍稷(Panicum miliaceum L.)起源于中国[1],属于禾本科(Gramineae)黍属(Panicum L.),一年生草本植物,具有早熟、耐旱、耐瘠的特性[2]。全世界黍稷栽培面积约600万公顷,主产区分布在欧洲和亚洲,主产国为原苏联和中国,中国主要分布在西北、华北、东北地区,南方有零星种植[3]。黍稷能适应多种土壤,对肥力较差的砂土有较强的适应能力,耐盐碱能力也较强,常作为盐碱地和开荒沙漠的先锋作物。因其生育期短,也作为自然灾害后的补救作物[4]。中国虽是黍稷的主产国,并在种质资源方面取得了一些研究成果,但基于DNA水平分子标记的研究开发却相对较少,应用分子标记的报道则更少,尤其是随着黍稷种质资源研究的不断深入,对黍稷的国外种,野生种和野生近缘植物的搜集、研究和保护更为需要。【前人研究进展】近年来,DNA分子标记发展迅速,已作为研究遗传变异的重要手段应用于各种作物的遗传多样性研究中,如谷子[5]、玉米[6]、陆地棉[7]、野生稻[8]等。其中,SSR可直接检测DNA分子结构变异[9]、并能反映参试材料差异、灵敏度较高、稳定性较好、操作较简单[10],因而在遗传多样性分析[11]、种质鉴定、DNA指纹图谱构建[10]、基因定位和分子标记育种[12]中广泛应用。此外,SSR分子标记为共显性,具有多态性丰富、PCR结果重现性高和易于鉴别基因型的优点[13],是群体遗传标记变异的最好标记之一[14]。国内外关于黍稷种质资源遗传多样性的研究一直匮乏。M'RIBU等[15]应用RAPD技术研究4种黍稷的遗传多样性以判定其来源,根据原产地的地理区域对黍稷品种进行了划分。KARAM等[16-17]对12份来源于美国和加拿大的黍稷利用AFLP标记检测其遗传多样性。LÁGLER等[18]选用9个来自草莓属植物的ISSR标记检测了21份匈牙利黍稷,检测到15个等位基因,其中只有7个引物扩增出DNA片段。HUNT等[19]利用禾本科作物(水稻、小麦、燕麦、大麦)的46对SSR引物对118份材料进行SSR多样性分析,得出黄土高原生态型材料的遗传多样性比较丰富,有可能是黍稷的起源中心的结论。CHO等[20]通过一个SSR富集文库黍基因组DNA开发了25个多态性SSR标记并分析了50份黍稷材料的遗传多样性;连帅等[21]利用5个黍稷特异性SSR标记对5个黍稷栽培生态区的40份黍稷进行遗传多样性分析,发现SSR标记表现出了一定的地域性;LI等[22]通过核糖体DNA(rDNA)分析内部转录间隔区(ITS)和外部转录间隔区(ETS),发现黍稷在ITS具有特异性的特点,并通过对新疆墓地出土的古DNA和现代品种黍稷材料的分析,得到黍稷遗传多样性随时间流逝而部分丧失,还进一步推出新疆在黍稷驯化和传播中东西方联系的十字路口的重要地位。【本研究切入点】由于可应用的分子标记很少且多态性不高,已有研究不足以全面反映黍稷的遗传多样性水平。小宗作物课题组利用二代测序技术大规模开发了黍稷基因组SSR引物,并分析了黍稷育成品种的遗传多样性(文章已接受待出版)。【拟解决的关键问题】本研究采用前期筛选得到的162对多态性SSR标记进一步对保存在国家种质库国内不同生态区的地方栽培品种、野生种以及从国外收集引进来自不同国家的黍稷种质资源进行遗传多样性分析。以揭示黍稷种质资源群体间的遗传多样性关系与遗传多样性地理分布的特点,为黍稷种质资源的研究、保护和利用提供依据,同时也为后续进行黍稷的起源、演化、传播研究提供数据。
1 材料与方法
1.1 供试材料
试验材料包括来自国家种质资源库的50份国内材料和近年新收集引进的142份国外材料(电子附表)。其中国内资源包括31份栽培种(山西2份、内蒙3份、黑龙江3份、甘肃5份、新疆3份、陕西4份、吉林3份、宁夏3份、河北2份、辽宁1份、海南1份、青海1份)和19份野生资源(山西9份、内蒙5份、黑龙江3份、甘肃1份、新疆1份)。142份国外资源分别来自蒙古(81份)、印度(28份)、美国(11份)、苏联(8份)、土耳其(4份)、波兰(3份)、德国(3份)、匈牙利(1份)、吉尔吉斯斯坦(1份)、哈萨克斯坦(1份)、韩国(1份)。根据参试材料的来源地,首先将其分成国内种和国外种两大群体;再进一步将国内栽培种和野生种质分开,分为国外种,国内栽培种和国内野生种3个组群;最后根据栽培区、地理来源将来自国内(12个省(区))和国外(11个国家)的材料分为10亚群,分别为内蒙古高原区(内蒙古)、东北区(黑龙江、吉林、辽宁、河北承德)、黄土高原区(山西、陕西、宁夏)、西北区(甘肃、新疆、青海)、南方区(海南)、蒙古(蒙古、韩国)、印度、美国、前苏联、欧洲(土耳其、波兰、德国、匈牙利、吉尔吉斯斯坦、哈萨克斯坦)。
1.2 DNA提取与检测
每个参试材料随机选取20个单株中的幼嫩叶片组织60 mg混合后,用新型植物基因组DNA快速提取试剂盒(北京鼎国昌盛生物有限公司)提取基因组DNA,采用1%的琼脂糖凝胶电泳检测其质量,之后再用NanoDropND-1000(NanoDrop,Wilmington,DE,USA)测定浓度后加入100 mL双纯水将其稀释成DNA母液于-80℃低温下保存备用。
1.3 SSR扩增及电泳
采用162对小宗作物课题组通过高通量测序技术开发的黍稷基因组SSR引物,选用6份不同地理来源、性状差异显著的黍稷种质资源(国外3份,国内3份)为DNA模板进行引物的PCR扩增和多态性筛选,最终筛选出63对条带清晰,多态性稳定的SSR引物用于后续的遗传多样性分析。
SSR扩增反应在MY-CYCLERPCR仪上进行。20 μL反应体系中含1.2 μL基因组DNA、0.6 μL引物、0.4 μL Taq酶(2.5 U·μL-1)、2 μL 10×buffer、0.8 μL dNTP(10 mmol·L-1)和ddH2O 15.0 μL。PCR程序为94℃ 5 min;94℃ 30 s,55℃ 30 s,72℃ 1 min,30个循环;72℃ 5 min。然后于16℃保存。PCR扩增产物在8%聚丙烯酰胺凝胶上进行电泳分离,之后用硝酸银染色显影。
1.4 数据统计分析
分别记录同一SSR引物扩增电泳条带(等位变异)在各参试材料中的有无。若有,根据分子量从大到小的顺序,记录为A、B、C……;若无,记为..。不同群体间和群体内的某一位点的观测等位变异数(observed number of alleles,Na)、有效等位变异数(effective number of alleles,Ne)、观测杂合度(observed heterozygosity,Ho)、期望杂合度(expected heterozygosity,He)及Shannon-Weaver指数等遗传参数的统计计算和不同群体间的Nei78遗传距离在Popgen1.32软件包[23]中完成;在PowerMarkerV3.25软件包和MEGA6软件包[24]中完成参试材料间多态性信息含量指数(polymorphism information content index,PIC)和Nei83遗传距离频率计算及群体间遗传距离频率的聚类图绘制。
2 结果
2.1 SSR多态性引物的筛选
利用随机选择的6份材料对162对SSR引物进行PCR退火温度和引物扩增多态性筛选,有77对SSR引物能扩增出清晰、稳定、可辨识的谱带,有效扩增比率为48.1%,其中63对SSR引物具有多态性,多态性引物比率为77.8%。最终选用63对具有稳定多态性的引物进行全部参试材料的遗传多样性分析。
2.2 SSR位点多样性分析
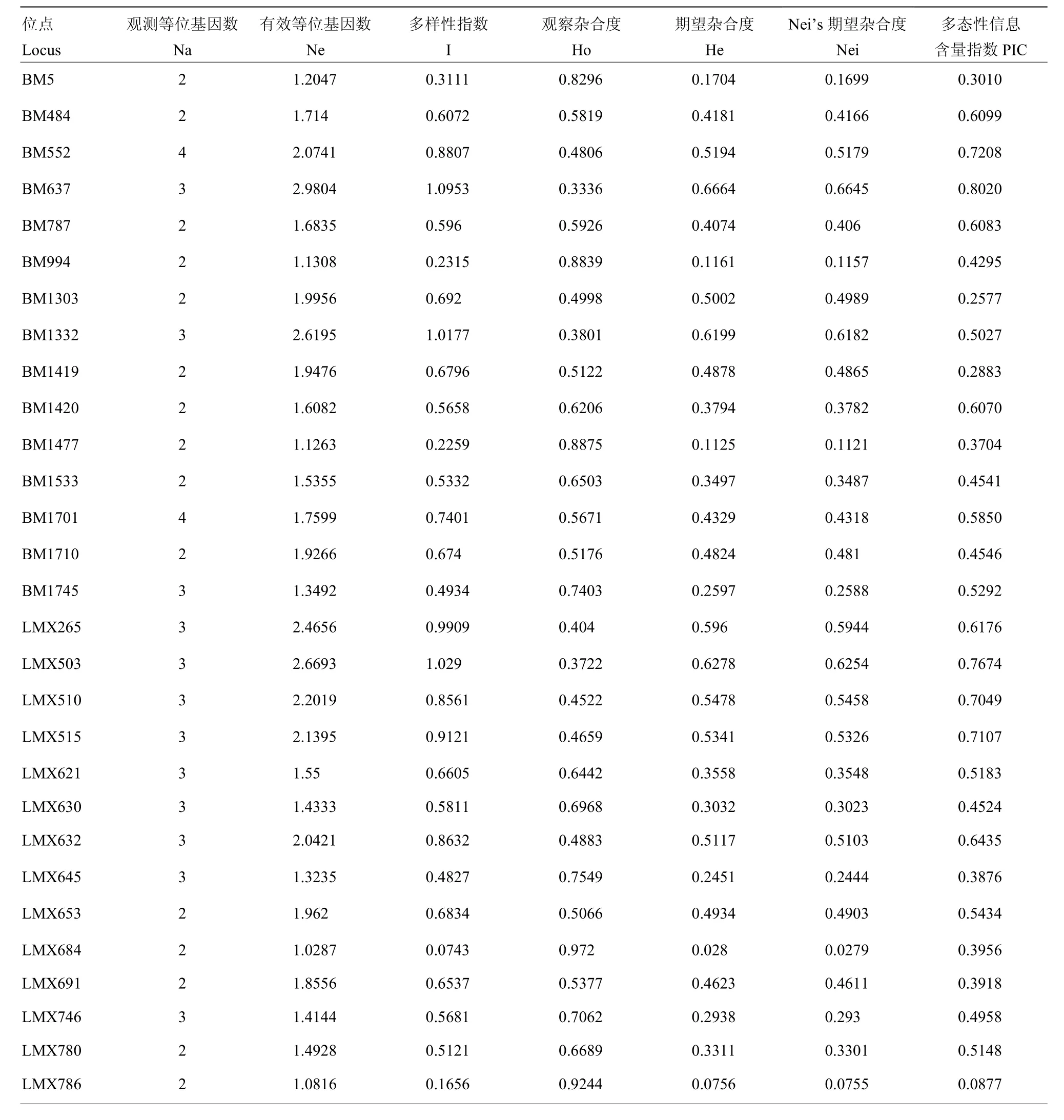
表1 63对SSR引物测定的遗传参数Table 1 Genetic parameters from the 63 polymorphic SSR markers used in this study

续表1 Continued table 1
192份参试材料在63对SSR引物上共检测出161个等位变异(表1),变幅为2—4个,平均每对SSR引物扩增出2.56个等位变异,其中有效等位变异数1.74,有效等位变异所占比重为67.97%。其中,引物BM552、BM1701、LMX1251等位变异最多,为4个,其有效等位变异数分别为2.0741、1.7599和1.1824。多态性信息量(PIC)的变幅介于0.0877—0.8020,平均值为0.4855,PIC指数最大的标记是BM637,最小的标记是LMX786。观测杂合度(Ho)在0(LMX1071、1387)—0.9405(LMX1332)变化,平均为0.2940。期望杂合度(He)的平均值为0.3887,最大是0.6664(BM637),最小为0.028(LMX684)。Nei基因多样性指数的变化范围是0.0279(LMX684)—0.6645(BM637)。Shannon-Weaver指数值最大的位点是BM637,最小的为LMX684,Shannon-Weaver指数变化范围为0.0743—1.0953,平均0.6275。结果表明,获得的SSR位点的遗传多样性比较丰富,且不同指标能从不同角度揭示黍稷SSR引物在遗传多样性方面存在的巨大差异,其中,效等位变异数、Shannon-Weaver指数与Nei基因多样性指数呈现正相关,能够反映群体遗传多样性。因此,有效等位变异数、Nei基因多样性指数和Shannon-Weaver指数这三个参数对于遗传多样性分析意义重大。
2.3 不同来源黍稷资源群体间的遗传变异分析
由表2可知,各参试群体的有效等位变异变化范围较窄,最小的是南方群体,为1.2407±0.4315;最大的是内蒙古高原群体,为1.8846±0.4892;总体看来,有效等位变异,国内5个群体大于国外各群体。国内群体间的有效等位变异依次为内蒙古高原>东北地区>黄土高原>西北地区>南方地区,变化范围为(1.2407±0.4315)—(1.8846±0.4892)。国外群体间有效等位变异依次是前苏联>欧洲>蒙古>印度>美国。变化范围为(1.5522±0.4658)—(1.7409±0.4662)。分析有效等位变异结果可知,国内外群体间的有效等位变异差异不大,但国内群体间的遗传丰富度总体大于国外群体。
国内群体的Shannon-Weaver指数最小值为0.1669±0.2991;最大值为0.6694±0.2799,均值为0.5319± 0.2888,分布也较国外更为宽泛。国内群体Shannon-Weaver指数为内蒙古高原>东北地区>黄土高原>西北地区>南方地区,其中内蒙古高原群体与东北地区群体的Shannon-Weaver指数接近,只差0.007。国外群体Shannon-Weaver指数范围为(0.4584±0.3102)—(0.5916±0.2722),均值为0.5358±0.2849。Shannon-Weaver指数排序依次为前苏联>欧洲>蒙古>印度>美国。由Shannon- Weaver指数分析结果可知,国内群体间的Shannon- Weaver指数即遗传均匀度差异不大,但国内群体总体高于国外。
从Nei’s基因杂合度分析,观察杂合度(Ho)最小的是印度群体,为0.2372±0.2962,最大的是内蒙古高原群体,为0.3966±0.3250;前苏联群体次之,为0.3539±0.3347。期望杂合度(He)最小的是美国群体,为0.3114±0.2203;最大的是内蒙古高原群体,为0.4622±0.1862。总体来看,Ho值国内群体比国外群体略小,而He值国内群体却比国外群体稍大。
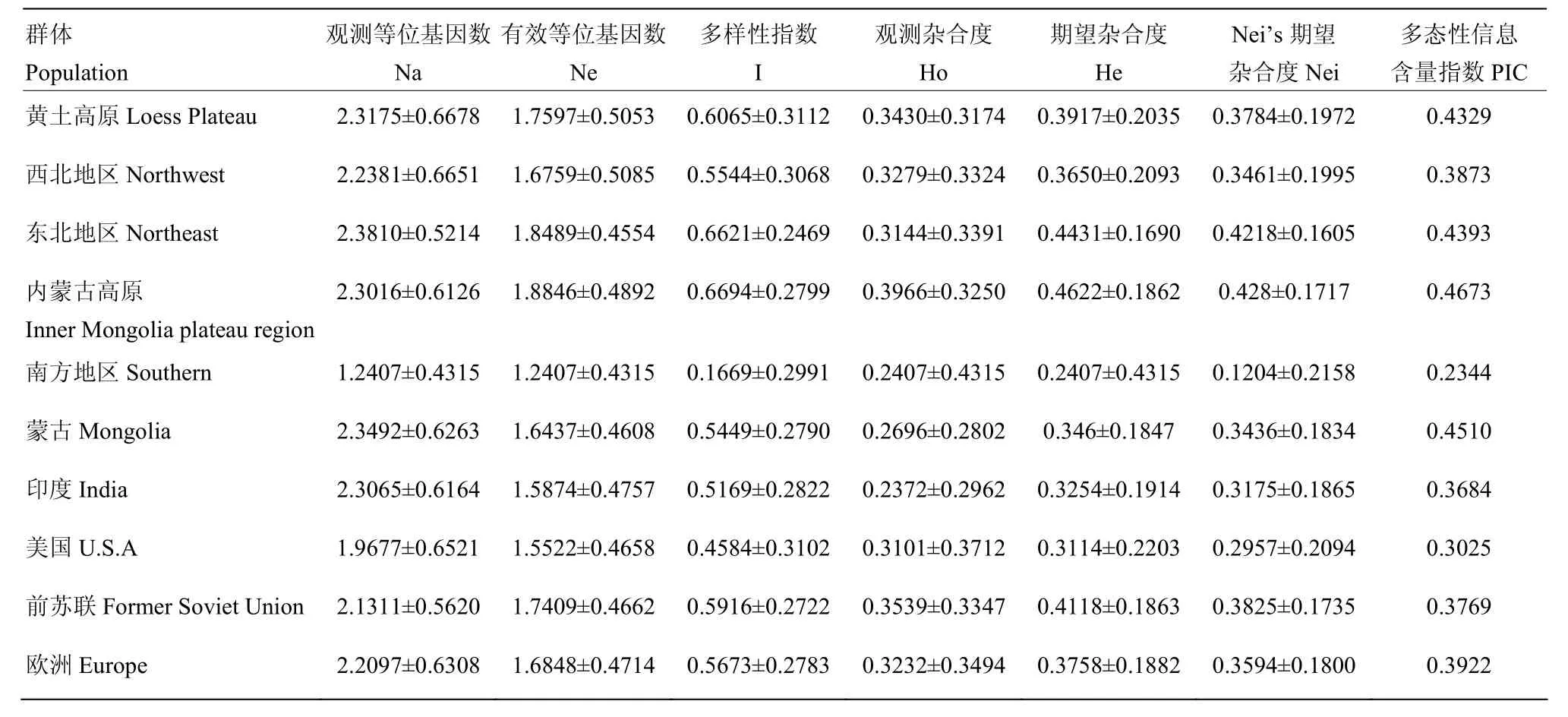
表2 10个群体的遗传多样性分析Table 2 Estimates of genetic diversity within 10 populations of the main cultivation areas of broomcorn millet
对于有效等位变异数和Shannon-Weaver指数,不同群体的变化趋势是一致的。如国内内蒙古高原、东北群体都远大于国外群体。而对于观察杂合度和期望杂合度来说,变化趋势却并不完全一致,但总的结果是国内群体的Nei基因多样性指数是高于国外群体。因此,等位变异数、有效等位变异数以及有效等位变异所占比重等指标,可从不同侧面揭示各群体材料的特点。Nei基因多样性指数、Shannon-Weaver指数则综合反映了各群体的遗传多样性。
据此,综合各遗传参数可知,国内黍稷种质遗传多样性总体高于国外种质。国外群体中,前苏联、欧洲的遗传多样性较高。而在国内群体中,内蒙古高原、东北地区的遗传多样性较高,除南方地区外基本均高于国外群体。南方地区因不是黍稷栽培的主载区,取材少,类型单一,多样性低。
就国内资源和国外资源两大群体而言(表3),国内资源的有效等位基因数(1.8145±0.4519)、Shannon-Weaver指数(0.6657±0.2413)和Nei基因多样性指数(0.412±0.1574)均大于国外资源(1.6862±0.4527、0.5897±0.2469、0.3652±0.1655)。因此,认为此次分析的国内黍稷资源的遗传多样性大于国外种质资源。而从国外种、国内栽培种和国内野生种3个大群体来看(表4),野生种质资源有效等位基因数(1.9285±0.5101)、Shannon-Weaver指数(0.6948±0.2852)、Nei基因多样性指数(0.4373± 0.1773)远大于国外种和国内栽培种,而由于缺少了野生种的国内栽培种也同时因材料过少,有效等位基因数(1.6727±0.4612)、Shannon-Weaver指数(0.5528± 0.2877)、Nei基因多样性指数(0.352±0.1925)略低于国外种(1.6862±0.4527、0.5897±0.2469、0.3652± 0.1655),由此可知,国内野生资源在黍稷遗传多样性中的重要地位。
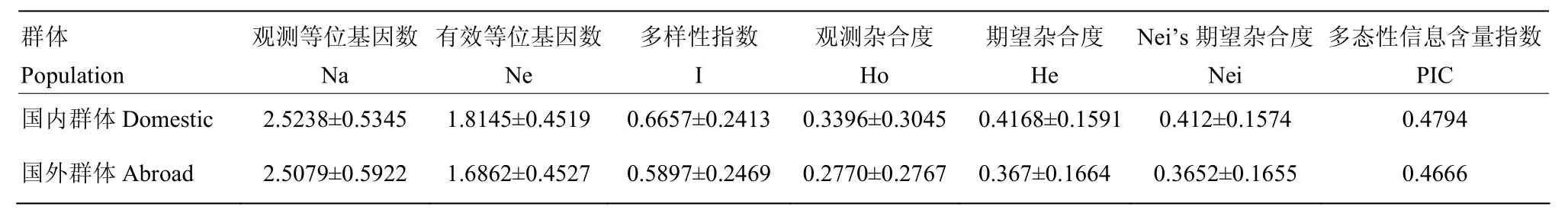
表3 国内外群体遗传多样性分析Table 3 Estimates of genetic diversity of populations of broomcorn millet from home and abroad
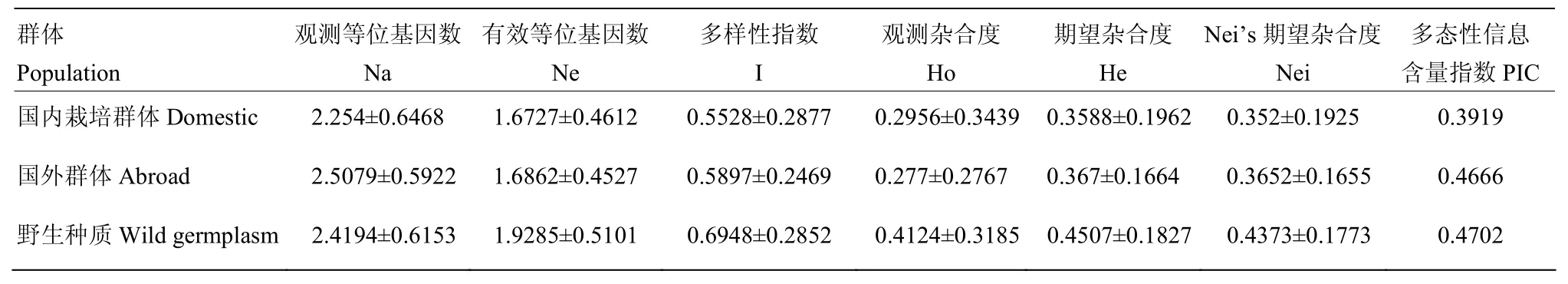
表4 野生种与国内外群体遗传多样性分析Table 4 Estimates of genetic diversity of wild species and populations of broomcorn millet from home and abroad
2.4 遗传相似性分析
采用POPGENE1.32计算不同群体黍稷种质间的遗传相似度(表5),结果显示遗传距离的变化范围为0.0207—0.2948,平均值为0.1030,遗传一致度的变化范围0.7447—0.9796,平均值0.9046。其中,南方群体与国内外群体间遗传距离均达0.2以上,内蒙古高原群体和印度、美国遗传距离也达0.1水平,而遗传距离最小的是黄土高原和西北群体,只有0.0207,遗传距离越大,说明亲缘关系越远,遗传相似性越低,而各群体间遗传距离变化区间只有0.27左右,遗传一致度接近于1,表明各群体间遗传距离大不,亲缘关系都很近,遗传相似性高。
2.5 参试群体的UPGMA聚类分析
利用10个参试群体绘制的基于UPGMA法的树状聚类图,从遗传距离频率0.09处可将所有参试群体聚类为3大组群,分别命名为组群Ⅰ、组群Ⅱ和组群Ⅲ(图1)。
组群I由来自于国内黄土高原、东北、西北、内蒙古高原生态区和蒙古的材料组成。聚类分析表明,遗传关系与地理分布有关,整个组群I地理分布上紧密连接,由国内黍稷主栽区与接壤的蒙古构成。
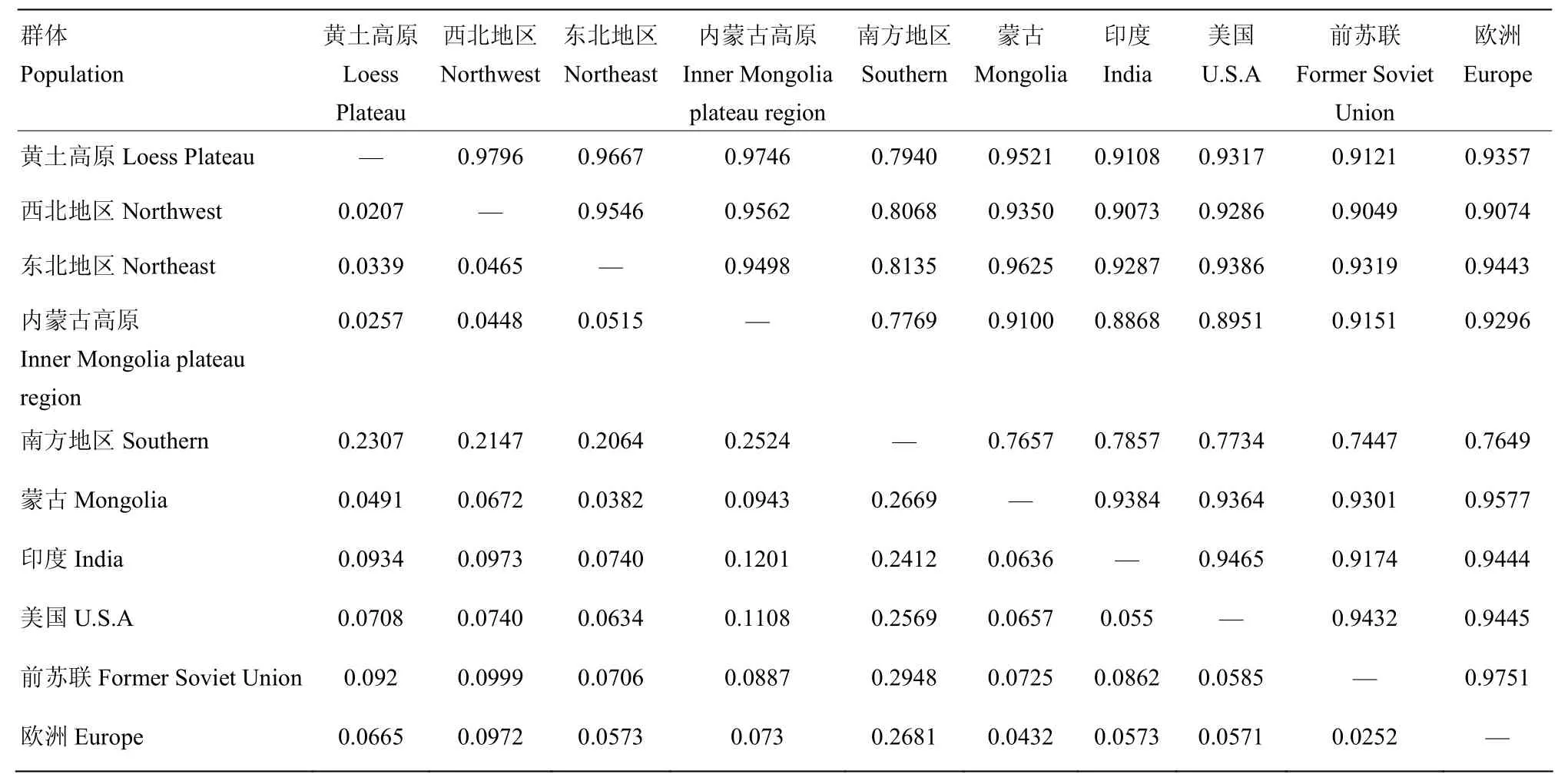
表5 10个群体遗传距离与遗传一致度Table 5 Nei’s unbiased measures of genetic identity and genetic distance

图1 基于SSR标记数据的种质资源群体间聚类分析图Fig. 1 Dendrogram generated by UPGMA cluster analysis of 10 populations of the main cultivation areas of broomcorn millet accessions based on data from 63 SSR markers
组群Ⅱ全部由国外群体组成。前苏联与欧洲群体遗传关系较近,美国和印度群体遗传关系较近,前苏联和欧洲群体地理分布相连,而美国和印度材料遗传关系近,可能因为引种地相似一致和长年驯化的结果。总体上,组群Ⅰ和组群Ⅱ有可能是长期地理隔离引起的生殖隔离造成的。
组群Ⅲ由南方群体构成,南方群体自成一支与国内外群体遗传关系均较远,从地理分布上看,它离黍稷主栽区较远,长期的自然条件、人为影响或许是造成与大部分群体差异较大的原因。但因南方群体的取材较少,地区、品种单一,具体原因有待进一步研究。
从192个参试个体绘制的基于UPGMA法的树状聚类图(图2)来看,所有材料的遗传距离频率在0.06—0.37变化,其中来自东北黑龙江齐齐哈尔的泰来小野糜(34号)在截距0.37处被独立分为一支,来自甘肃的野黍子(19号)在截距0.34处被分为独立个体,表明这两个材料与其他材料遗传差异较大。其余材料在截距0.31处被分为若干个小群体,个体与个体间差异不大,没有分成大的聚类,整体分布与地理分布有一定关联,也存在区域间和国内外间的互相渗透。

图2 基于SSR标记数据的192份材料聚类图Fig. 2 Dendrogram generated by UPGMA cluster analysis of 192 broomcorn millet accessions based on data from 63 SSR markers
综上,基于遗传距离频率的UPGMA法构建的树状聚类图与遗传距离、遗传一致度的分析结果,相互印证、吻合一致。
3 讨论
3.1 黍稷遗传多样性
黍稷虽栽培历史久远,但作为小宗作物,种植面积狭小,食用人群较少,所关注程度一直较低,所以研究水平也一直落后于水稻、小麦、玉米主要作物。尽管国家种质库中保存着8 600多份黍稷种质资源,但开展的研究主要集中于形态学水平的遗传多样性评价和抗旱耐盐资源的初步鉴定等方面,而对分子水平的遗传多样性则所知甚少。如胡兴雨等[24]对来自国家种质资源库中的8 016份黍稷种质资源进行了株高、千粒重、生育期等11个农艺性状进行主成分分析和聚类分析;WANG等[25]选择《中国黍稷(糜)品种资源目录》中山西省1 192份黍稷种质,分析了黍稷种质资源穗型与主要农艺性状的关系;LIU等[26]研究了不同耐性黍稷在混合盐胁迫下的生理应答机制并分析了195份黍稷核心种质的耐盐性[27],为后续开展耐盐基因挖掘奠定了基础;王纶等[28]选用山西省有代表性的500份黍稷种质资源采用反复干旱法进行了抗旱性鉴定评价;刘晓欢等[29]研究了黍稷品种间农艺性状的形态解剖差异;张盼盼等[30]对黍稷苗期PEG胁迫下抗旱指标进行了鉴选研究。而在分子水平,黍稷中已有AFLP[16-17]、RAPD[15]和ISSR[18]等标记应用于遗传多样性研究。KARAM等[16-17]检测了黍稷遗传多样性用AFLP银染标记;LÁGLER等[18]选用9个来自草莓属植物的ISSR标记检测了21份匈牙利黍稷;2008年,HUNT等[19]对来源于中国不同生态区的118份黍稷材料选取了水稻、小麦、大麦和燕麦的46个SSR标记进行了遗传变异分析;2010年,CHO等[20]通过构建黍稷基因组DNA的富SSR文库,以此开发了25个多态性微卫星标记。上述研究所用标记,特别是SSR标记的数量较少,很难全面反映黍稷种质资源的多样性,因此从黍稷自身基因组开发SSR标记对促进其遗传多样性的研究具有十分重要的意义。随着测序技术的成熟发展及其价格降低,应用二代测序技术与磁珠富集法相结合大规模的开发了谷子[31]、燕麦[32]、蚕豆[33]和山黧豆[34]等作物的基因组SSR标记,大大促进了相关小宗作物分子水平的研究进展。本研究对来自国内外不同生态区的的192份种质(栽培种和野生种)利用前期开发的黍稷基因组SSR引物进行遗传多样性分析,共检测出161个等位变异,其中每对引物有效等位变异数1.74,有效等位变异所占比重为67.97%。多态性信息量PIC介于0.0877—0.8020,Nei基因多样性指数的变化范围是0.0279—0.6645,Shannon-Weaver指数变化范围为0.0743—1.0953,其数值结果均高于先前研究,基因组SSR标记的成功应用,对黍稷种质资源的遗传多样性有了更全面多样的认识,并有助于黍稷遗传资源的进一步挖掘利用。同时本研究建立的SSR标记在分子水平也为国内黍稷资源研究打下了坚实基础。
3.2 黍稷起源、演化与传播
黍稷是人类最早驯化栽培的谷物之一,中国是目前已知的黍稷栽培历史最悠久的国家,研究中国黍稷和国外黍稷的遗传多样性水平,对于探讨黍稷的起源、演化与传播也具有重要的意义。本研究通过对来自黍稷主栽区192份黍稷品种进行SSR遗传多样性的系统分析,揭示了各群体间遗传多样性差异。其中,内蒙古高原、东北、黄土高原群体在有效等位变异、Shannon-Weaver指数和Nei基因多样性指数都高于其他群体,结合其他各参数而言,国内黍稷资源的遗传多样性也大于国外种质资源,同时还发现不同群体黍稷种质间的遗传距离变化范围为0.0207—0.2948,遗传一致度变化范围为0.7447—0.9796,更进一步显示出10个群体来源的黍稷资源群体间的遗传距离不大,亲缘关系都很近,遗传相似性高。
通过分析各栽培区的遗传多样性可以为黍稷的起源和演化研究提供依据和思路。在先前研究中,中国、中亚和中欧都被提出为黍和其野生祖先驯化的可能起源中心,但具体位置尚有待于进一步研究确认[35]。而中国黍稷品种资源群体间的显著差异,作为全世界大面积栽培黍稷的国家,栽培环境、自然气候条件千差万别,同时还可能隐含着自然和人工优胜选择、栽培资源与野生资源的互相基因渗透,这也是世界的其他地方不能比拟的。从研究中可知,中国黍稷种质的遗传多样性高于国外品种,拥有更多样多元的遗传多样性,此结果与胡兴雨等[24]的研究结果一致。其中,内蒙古高原群体和东北群体品种的遗传多样性要高于其他地区,该区域有可能是黍稷起源地,并以此向周边扩展,此结果与LI等[22]推得新疆是黍稷驯化和传播的十字路口,是由中国北方通过新疆向中亚、西亚进而进入欧洲的结果一致。同时,蒙古与国内归为一组,俄罗斯与欧洲归为一组的研究互相佐证,表明群体聚类与驯化、传播及地理分布有一定关联。
3.3 特殊群体与特殊个体
黑龙江齐齐哈尔的泰来小野糜(34号)和甘肃的野黍子(19号)在聚类分析中非常独特,远远分离于其他黍稷材料之外。这有可能是由于野生种质没有经过人为驯化不受人为环境影响又或是与其他野生近缘种杂交影响所致。而对于南方种群虽仅有一份材料但在群体聚类中却自成一个独立的群体,与国内外其他各群体遗传距离均达0.2以上,亲缘关系较远,可能与自然环境,所在的气候区有关,真实原因还需利用植物学、细胞生物学和分子生物学等手段对其他性状进行深入研究。
同时通过将国内栽培种和野生种分别分析,进一步发现了国内野生种质的丰富遗传多样性与其在黍稷种质资源中的重要地位,此次野生种质只有19份,但其有效等位基因数、香农信息值数、Nei基因多样性指数均远高于其他群体,充分显示其遗传多样性的丰富。所以,野生种质和南方零星种质资源有必要在今后的种质保存、资源管理和基础研究工作中重点关注。
4 结论
中国黍稷种质资源具有较丰富的遗传多样性,普遍高于国外材料,而国内资源又以内蒙古高原、东北地区材料的遗传多样性最高,进一步验证了“中国是黍稷起源中心”的论点。同时应进一步加强野生材料、南方种质的收集和国外资源的引进,丰富现有资源的遗传多样性,并促进黍稷遗传改良、演化传播等研究。
[1] CRAWFORD G W. Agricultural origins in North China pushed back to the Pleistocene–Holocene boundary. Proceedings of the National Academy of Sciences of the USA, 2009, 10(6): 7271-7272.
[2] 王星玉, 王纶. 黍稷种质资源描述规范和黍稷标准. 北京: 中国农业出版社, 2006: 5-15. WANG X Y, WANG L. Proso Millet Germplasm Resources Description and Panicum Standard. Beijing: China Agriculture Press, 2006: 5-15. (in Chinese)
[3] 王星玉. 中国黍稷. 北京: 中国农业出版社, 1996: 2-6. WANG X Y. China Panicum. Beijing: China Agriculture Press, 1996: 2-6. (in Chinese)
[4] 柴岩. 糜子. 北京: 中国农业出版社, 1999: 71-86. CHAI Y. Broomcorn Millet (Panicum miliaceum L.). Beijing: China Agriculture Press, 1999: 71-86. (in Chinese)
[5] LI Y, JIA J Z, WANG Y R. Intraspecific and interspecific vari2 ation in Seta ria revealed by RAPD analysis. Genetic Resources And Crop Evolution, 1998, 45(3): 279-285.
[6] YANG L X, FU J H, WARBURTON M, LI X H, ZHANG S H, KHAIRRALLAH M, LIU X Z, PENG Z B, LI L C. Comparison of genetic diversity among maize inbred lines based on RFLPs, SSRs, AFLPs and RAPDs. Journal of Genetics and Genomics, 2000, 27(8): 725-733.
[7] WU Y Y, ZHANG T Z, YIN J M. Genetic diversity detected by DNA markers and phenotypes in upland cotton. Journal of Genetics and Genomics, 2001, 28(11): 1040-1050.
[8] QIAN W, GE S, HONG D Y. Assessment of genetic variation of Oryza granulata detected by RAPDs and ISSRs. Journal of Integrative Plant Biology, 2000, 42(7): 741-750.
[9] WEBER J L. Informativeness of human (dC-dA)n (dG-dT)n polymorphism. Genomics, 1990, 7(1): 524-530.
[10] ZHANG X L, TANG K X. Plant Biotechnology. Beijing: Science Press, 2004: 401-500.
[11] D′ENNEQU M L T, PANAUD O, TOUPANCE B. Assesment of genetic relationships between Setaria italica and its wild relative S.virid is using AFL Pmarkers. Theoretical and Applied Genetics, 2000, 100(7): 1061-1066.
[12] 吴大鹏, 房嫌嫌, 崔闰根, 陈进红, 祝水金. 国内外陆地棉品种资源的亲缘关系和遗传多态性研究. 棉花学报, 2011, 23(4): 291-299. WU D P, FANG X X, CUI Y G, CHEN J H, ZHU S J. Genetic relationship and diversity of the upland cotton germplasms from different cotton producing countries using SSR markers. Cotton Science, 2011, 23(4): 291-299. (in Chinese)
[13] 徐先良, 赖勇, 王鹏喜, 范贵强, 汪军成, 王晋, 孟亚雄, 李葆春,马小乐, 王化俊. 大麦亲本材料农艺性状鉴定及遗传多样性分析.麦类作物学报, 2013, 33(4): 640-646. XU X L, LAI Y, WANG P X, FAN G Q, WANG J C, WANG J, MENG Y X, LI B C, MA X L, WANG H Y. Identification of agronomic traits and genetic diversity analysis of barley parent materials. Journal of Triticeae Crops, 2013, 33(4): 640-646. (in Chinese)
[14] 王晋, 王世红, 赖勇, 孟亚雄, 李葆春, 马小乐, 尚勋武, 王化俊.大麦SSR标记遗传多样性及群体遗传结构分析. 核农学报, 2014, 28(2): 177-185. WANG J, WANG S H, LAI Y, MENG Y X, LI B C, MA X L, SHANG X W, WANG H J. Genetic diversity and population structure analysis by using SSR markers in barley. Journal of Nuclear Agricultural Sciences, 2014, 28(2): 177-185. (in Chinese)
[15] M'RIBU H K, HILU K W. Detection of interspecific and intraspecific variation in Panicum millets through random amplified polymorphic DNA. Theoretical and Applied Genetics, 1994, 88(3): 412-416.
[16] KARAM D, WESTRA P, NISSEN S J, WARD S M, FIGUEIREDO J E F. Genetic diversity among proso millet (Panicum miliaceum) biotypes assessed by AFLP technique/Diversidade genética entre biótipos de proso millet (Panicum miliaceum) revelada pela técnica de AFLP. Planta Daninha, 2004, 22(2): 167-174.
[17] KARAM D, WESTRA P, NIESSEN S J. Assessment of silver-stained AFLP markers for studying DNA polymorphism in proso millet (Panicum miliaceum L.). Revista Brasileira de Botanica, 2006, 29(4): 609-615.
[18] LÁGLER R, GYULAI G, HUMPHREYS M, SZABÓ Z, HORVÁTH L, BITTSÁNSZKY A, KISS J, HOLLY L, HESZKY L. Morphological and molecular analysis of common millet (P. miliaceum) cultivars compared to an aDNA sample from the 15th century (Hungary). Euphytica, 2005, 146: 77-85.
[19] HUNT H V, CAMPANA M G, LAWES M C, PARK Y J, BOWERM A, HOWE C J, JONES M K. Genetic diversity and phylogeography of broomcorn millet (Panicum miliaceum L.) across Eurasia. Molecular Ecology, 2011, 20(7): 4756-4771.
[20] CHO Y, CHUNG J W, LEE G A, MA K H, DIXIT A, GWAG J G, PARK Y J. Development and characterization of twenty-five new polymorphic microsatellite markers in proso millet (Panicummiliaceum L.). Genes & Genomics, 2010, 3(2): 267-273.
[21] 连帅, 王瑞云, 马跃敏, 刘笑瑜, 季煦. 不同生态区糜子种质资源的遗传多样性分析. 山西农业大学学报(自然科学版), 2015, 35(3): 225-227. LIAN S, WANG R Y, MA Y M, LIU X Y, JI X. Genetic diversity of broomcorn millet (Panicum miliaceum L.) germplasms of different ecotype zone of China. Journal of Shanxi Agricultural University (Natural Science Edition), 2015, 35(3): 225-227. (in Chinese)
[22] LI C X, DONG Y, LIU M X, LU P, LI W Y, WANG Y N, CUI X Y, ZHOU H, XU Y. Ancient DNA analysis of Panicum miliaceum (broomcorn millet) from a Bronze Age cemetery in Xinjiang, China. Vegetation History and Archaeobotany, 2016, 25(5): 469-477.
[23] KUMAR S, TAMURA K, NEI M. MEGA3:Integrated software for molecular evolutionary genetics analysis and sequence alignment. Briefings in Bioinformatics, 2004, 5: 150-163.
[24] 胡兴雨, 陆平, 贺建波, 王纶, 王玉星, 张红生, 张宗文, 吴斌. 黍稷农艺性状的主成分分析与聚类分析. 植物遗传资源学报, 2008, 9(4): 492-496. HU X Y, LU P, HE J B, WANG L, WANG Y X, ZHANG H S, ZHANG Z W, WU B. Principal components and cluster analysis of agronomic traits of Proso Millet (Panicum miliaceum). Journal of Plant Genetic Resources, 2008, 9(4): 492-496. (in Chinese)
[25] WANG L, WANG X Y, WEN Q F. Research and utilization of proso millet germplasm resource in China. Journal of Plant Genetic Resources, 2005, 6(5): 471-474.
[26] LIU M X, QIAO Z J, ZHANG S, WANG Y Y, LU P. Response of broomcorn millet (Panicummiliaceum L.) genotypes from semiarid regions of China to salt stress. The Crop Journal, 2014, 8(6): 57-66.
[27] 刘敏轩, 张宗文, 吴斌, 陆平. 黍稷种质资源芽、苗期耐中性混合盐胁迫评价与耐盐生理机制研究. 中国农业科学, 2012, 45(18): 3733-3743. LIU M X, ZHANG Z W, WU B, LU P. Evaluation of mixed salt-tolerance at germination stage and seedling stage and the related physiological characteristics of Panicum miliaceum L.. Scientia Agricultura Sinica, 2012, 45(18): 3733-3743. (in Chinese)
[28] 王纶, 温琪汾, 曹厉萍, 王玉星. 黍稷抗旱种质筛选及抗旱机理研究. 山西农业科学, 2007, 35(4): 31-34. WANG L, WEN Q F, CAO L P, WANG Y X. Drought-resistant germplasm screening and drought-resistance mechanism in proso millet. Journal of Shanxi Agricultural Sciences, 2007, 35(4): 31-34. (in Chinese)
[29] 刘晓欢, 王瑞云, 杜海娥, 李红英, 王爱萍, 王海岗, 张海平, 秦香,张彬. 糜子品种间农艺性状的形态解剖差异. 山西农业大学学报(自然科学版), 2013, 33(4): 295-298. LIU X H, WANG R Y, DU H E, LI H Y, WANG A P, WANG H G, ZHANG H P, QIN X, ZHANG B. The study on the morphological and anatomical structures of agronomic traits among different cultivars of common millet (Panicum miliaceum L.). Journal of Shanxi Agricultural University (Natural Science Edition), 2013, 33(4): 295-298. (in Chinese)
[30] 张盼盼, 冯佰利, 王鹏科, 高晓丽, 高金峰, 宋慧, 张晓东, 柴岩. PEG胁迫下糜子苗期抗旱指标鉴选研究. 中国农业大学学报, 2012, 1: 53-59. ZHANG P P, FENG B L, WANG P K, GAO X L, GAO J F, SONG H, ZHANG X D, CHAI Y. Study on identification of drought-resistance indexes at seedling stage in broomcorn millet under PEG stress. Journal of China Agricultural University, 2012, 1: 53-59. (in Chinese)
[31] JIA X P, ZHANG Z B, LIU Y H, ZHANG C W, SHI Y S, SONG Y C, WANG T Y, LI Y. Development and genetic mapping of SSR markers in foxtail millet [Setariaitalica(L.) P.Beauv.]. Theoretical and Applied Genetics, 2009, 11(8): 821-829.
[32] WU B, LU P, ZHANG Z W. Recombinant microsatellite amplification: A rapid method for developing simple sequence repeat markers. Molecular Breeding, 2009, 2(9): 53-59.
[33] YANG T, BAO S Y, FORD R, JIA T J, GUAN J P, HE Y H, SUN X L, JIANG J Y, HAO J J, ZHANG X Y, ZONG X X. High-throughput novel microsatellite marker of faba bean via next generation sequencing. BMC Genomics, 2012, 1(3): 602-608.
[34] JIANG J Y, BURLYAEVA M, HU J G, COYNE C J, KUMAR S, REDDEN R, SUN X L, WANG F, CHANG J W, HAO X P, GUAN J P, ZONG X X. Large-scale microsatellite development in grasspea (Lathyrussativus L.), an orphan legume of the arid areas. BMC Plant Biology, 2014, 14: 65-69.
[35] HUNT H V, BADAKSHI F, ROMANOVA O, HOWE C J, JONES M K, HESLOP-HARRISON J S P. Reticulate evolution in Panicum (Poaceae): The origin of tetraploid broomcorn millet P.miliaceum. Journal of Experimental Botany, 2014, 30: 109-122.
(责任编辑 李莉)
Genetic Diversity in Broomcorn Millet (Panicum miIiaceum L.) from China and Abroad by Using SSR Markers
LIAN Shuai1,2, LU Ping2, QIAO Zhi-jun3, ZHANG Qi2, ZHANG Qian2, LIU Min-xuan2, WANG Rui-yun1
(1College of Agriculture, Shanxi Agricultural University, Taigu 030801, Shanxi;2Institute of Crop Science, Chinese Academy of Agricultural Sciences, Beijing 100081;3Institute of Crop Genetic Resources, Shanxi Academy of Agricultural Sciences, Taiyuan 030031)
【Objective】The objective of this study is to assess the genetic diversity of broomcorn millet accessions which collected from China and abroad.【Method】Five hundred pairs of SSR primers developed in the authors’ laboratory by high-throughput sequencing were used to identify polymorphisms in six representatives randomly selected from the total of accessions. A total of 63 primer pairs produced clear and reproducible polymorphic fragments among the six accessions and then were used to analyze the genetic diversity and relationship of 192 broomcorn millet landraces and wild accessions.【Result】A total of 161 alleles were detected with an average of 2.56 alleles per locus, and the mean Shannon-Weaver index (I), mean Nei and mean PIC were 0.6275, 0.3874 and 0.4855, respectively. The results indicated that there is a significant difference among the 10 populations of broomcorn millet resources in genetic diversity from diverse geographic origins. The variance range of effective alleles number is 1.2407 (South region) - 1.8846 (Inner Mongolia). In domestic populations, the rank of Shannon-Weaver index is Inner Mongolia Plateau>Tohoku>Loess Plateau>Northwest>southern regions, and the rank of foreign populations is the former Soviet Union>Europe>Mongolia>India>United States. The results of Nei’s genetic heterozygosity analysis showed that the minimum and maximum of observed (Ho) and expected heterozygosity (He) is 0.2372 from India and 0.3966 from Inner Mongolia as well as 0.3114 from the Unite State and 0.4622 from Inner Mongolia Plateau, respectively. The effective number of alleles (1.9285±0.5101), Shannon-Weaver index (0.6948±0.2852) and Nei gene diversity index (0.4373±0.1773) of the wild germplasm are much higher which in domestic and foreign accessions. For domestic population and alien population, the effective number of alleles (1.8145±0.4519) of domestic resources, Shannon-Weaver index (0.6657±0.2413), and Nei gene diversity index (0.412± 0.1574) of domestic accessions were higher than that in foreign resources (1.6862±0.4527, 0.5897±0.2469, 0.3652±0.1655). UPGMA cluster analysis showed that the 10 geographic populations could be clustered into three categories, the accessions from the Inner Mongolia Plateau, the Loess Plateau, northeast, northwest, Mongolia area were clustered as one group, the former Soviet Union, the United States, India, Europe together as one group, and southern region of China clustered as one independent group. The wild millet (34) which is from Qiqihaer is separated from others at 0.37, the wild millet from Gansu (19) was divided into an independent individual at 0.34, indicating that there are significant genetic variances between the two wild accessions and others. In general, genetic division of population is not significant for 192 domestic and foreign accessions, and there have material interpenetration among different groups.【Conclusion】The Inner Mongolian Plateau, northeast area and the Loess Plateau with the most abundant genetic diversity is the most complex area of genetic relationship, which further confirms that China is the origin center of Panicum miliaceum.
Panicum miliaceum L; landraces; wild species; SSR markers; genetic diversity
2016-03-31;接受日期:2016-05-20
农业部谷子糜子产业体系(CARS-07-12[1].5-A1)、国家自然科学基金青年基金(31301386)、国家自然科学基金(31271791)
联系方式:连帅,Tel:15534988123;E-mail:lianshuaisxnd@163.com。通信作者刘敏轩,Tel:01062159962;E-mail:liuminxuan@caas.cn。通信作者王瑞云,Tel:15234420135;E-mail:wry925@126.com
