大豆5个新发现ERF基因的生物信息学及表达分析
2016-11-24高士童孙婉姝任巍巍王秀文王玉书
翟 莹,张 军,赵 艳,高士童,孙婉姝,张 闯,任巍巍,王秀文,王玉书
(1.齐齐哈尔大学 生命科学与农林学院,黑龙江 齐齐哈尔 161006; 2.黑龙江省兽医科学研究所,黑龙江 齐齐哈尔 161005)
大豆5个新发现ERF基因的生物信息学及表达分析
翟 莹1,张 军2,赵 艳1,高士童1,孙婉姝1,张 闯1,任巍巍1,王秀文1,王玉书1
(1.齐齐哈尔大学 生命科学与农林学院,黑龙江 齐齐哈尔 161006; 2.黑龙江省兽医科学研究所,黑龙江 齐齐哈尔 161005)
ERF转录因子广泛存在于植物中并且参与植物对生物及非生物胁迫的响应。从NCBI数据库中获得5个新的ERF基因,GmERFa/b/c/d/e。蛋白序列分析显示,5个ERF基因均含有一个保守的AP2/ERF结合域。进化分析表明,GmERFe与GmERF3和GmERF7同源性最高,GmERFb、GmERFc与GmERF5的同源性最高,GmERFd与GmEREBP1的同源性较高,而GmERFa与其他ERF蛋白的同源性均较低。实时荧光定量PCR结果显示,5个基因都主要在大豆的根和叶中表达。逆境处理后的实时荧光定量PCR结果显示,GmERFd/e主要对乙烯信号和低温产生响应,GmERFb主要对干旱产生响应,而GmERFa主要对低温产生响应。
大豆;ERF转录因子;生物信息学分析;表达分析
乙烯响应因子(ethyleneresponsefactor,ERF)基因以家族形式广泛存在于植物中,隶属于AP2/ERF超家族。它们功能多样,不仅在植物的生长和发育过程中发挥功能,更在植物应对不良环境时发挥重要作用[1-3]。ERF转录因子家族的成员都含有一段由58或59个氨基酸残基组成的保守序列,即ERF的DNA结合域。ERF转录因子能够识别并结合GCC-box、DRE/CRT等顺式作用元件就取决于DNA结合域中特定的氨基酸序列,而这些顺式作用元件则通常位于胁迫相关的启动子中[4-7]。除此以外,ERF蛋白还可以与其他蛋白相互作用,包括bZIP转录因子蛋白,共同调控相关基因的表达[8-9]。多数ERF都是转录激活子调控相关基因的表达[10-12],少数ERF则作为转录抑制子,它们不仅可以抑制相关基因的表达,还能够降低其他转录因子的转录激活活性[10,13]。ERF抑制子的转录抑制活性通常与位于ERF蛋白C末端的EAR(ERF-associatedamphiphilicrepressor)元件相关,这是一段保守的L/FDLNL/F(X)P序列。EAR元件中的保守氨基酸若发生突变或整体失去,将导致抑制活性的丧失[14]。
研究表明,ERF转录因子可以被多种逆境及逆境相关的信号分子诱导表达,例如高盐、干旱、低温、脱落酸(ABA)、乙烯(ET)、茉莉酸和水杨酸等[12-13]。将它们在植物中过量表达可以提高转基因植株对多种生物及非生物胁迫的综合抗性[12-13,15]。植物中存在大量的ERF基因,仅大豆中就发现了98个包含有完整AP2/ERFDNA结合域的独立基因[16]。但这些基因在功能上存在冗余性,因此有必要对大豆ERF家族中其他成员作进一步鉴定。本研究从基因数据库中搜索到5个功能未鉴定的大豆ERF转录因子基因,对它们进行生物信息学分析并对它们在不同组织及逆境胁迫下的表达模式进行鉴定,以期揭示大豆中ERF的抗逆机制并丰富大豆抗逆基因资源,为后续植物ERF基因的研究提供理论依据。
1 材料与方法
1.1 试验材料及处理
大豆品种合丰46由齐齐哈尔大学遗传研究室保存。对盆栽的四叶期大豆幼苗进行以下逆境处理。参照Zhai等[13]的方法用ET和ABA处理大豆幼苗;将大豆幼苗置于含20%PEG8000的MS营养液中进行干旱处理;置于含200mmol·L-1NaCl的MS营养液中进行高盐处理;置于4 ℃培养箱中进行低温处理。分别在处理0、1、2、5、10和24h取样,剪取0.1g叶片并迅速置于液氮中保存备用。同时剪取大豆的根、茎、花和未成熟胚置于液氮中保存备用。
1.2 生物信息学分析
用已知的ERF基因保守序列[12]搜索NCBI数据库(http://www.ncbi.nlm.nih.gov/)获得功能未知的大豆ERF基因完整序列;用在线软件PSORT(http://psort.hgc.jp/)进行核定位信号的预测;用DNAMAN软件构建ERF蛋白系统进化树。
1.3 表达模式分析
根据5个ERF的基因序列,使用Primer5软件设计实时荧光定量PCR引物(表1)。在BIO-RADCFX96Real-TimePCR仪上,以大豆组成型表达基因β-Tubulin (GenBank登录号为GMU12286)作为内参,以大豆不同组织的cDNA和不同处理时间点叶片cDNA为模板。反应体系为2×SYBRPremixExTaq (TaKaRa) 10μL、cDNA2μL、10μmol·L-1正反向引物各0.4μL、ROXReferenceDyeⅡ 0.2μL,补水至总体积20μL。
表1 所用引物序列
Table 1 Sequences of primers
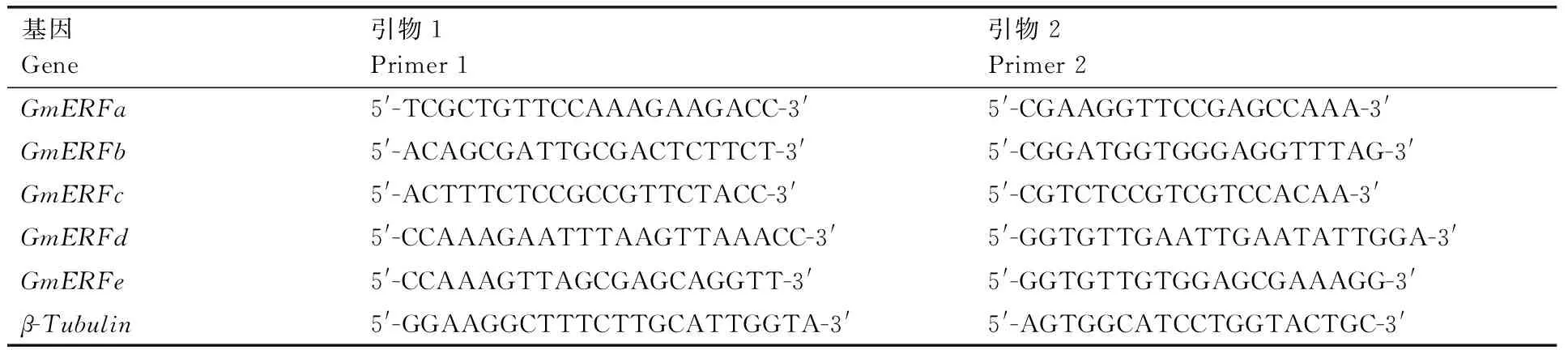
基因Gene引物1Primer1引物2Primer2GmERFa5'-TCGCTGTTCCAAAGAAGACC-3'5'-CGAAGGTTCCGAGCCAAA-3'GmERFb5'-ACAGCGATTGCGACTCTTCT-3'5'-CGGATGGTGGGAGGTTTAG-3'GmERFc5'-ACTTTCTCCGCCGTTCTACC-3'5'-CGTCTCCGTCGTCCACAA-3'GmERFd5'-CCAAAGAATTTAAGTTAAACC-3'5'-GGTGTTGAATTGAATATTGGA-3'GmERFe5'-CCAAAGTTAGCGAGCAGGTT-3'5'-GGTGTTGTGGAGCGAAAGG-3'β-Tubulin5'-GGAAGGCTTTCTTGCATTGGTA-3'5'-AGTGGCATCCTGGTACTGC-3'
反应程序:95 ℃预变性10 s;95 ℃变性 20 s,58 ℃退火20 s,72 ℃延伸30 s,循环40次。所有处理均做3次重复,采用2-△△Ct法分析数据,计算出基因的相对表达量。利用BIO-RAD CFX Manager软件对试验结果进行分析。
2 结果与分析
2.1 五个ERF基因的生物信息学
用ERF基因保守序列在NCBI数据库中搜索获得5条功能未知的大豆ERF基因序列,它们都包含完整的开放阅读框(open reading frame, ORF),将它们分别命名为ERFa/b/c/d/e。GmERFa(GenBank登录号:HM219231) ORF全长828 bp,编码275个氨基酸,蛋白序列中部含有一个由59个氨基酸组成的AP2/ERF结合域(图1),结合域外含有2个预测的核定位信号(R203KRRR和P219VVKKEK)。GmERFb(GenBank登录号:BT096447) ORF全长648 bp,编码215个氨基酸,蛋白序列中部含有一个由58个氨基酸组成的AP2/ERF结合域(图1),结合域内含有两个预测的核定位信号(R31KRP和P45WKKQRV)。GmERFc(GenBank登录号:XM003551818) ORF全长639 bp,编码212个氨基酸,靠近蛋白序列N端含有一个由58个氨基酸组成的AP2/ERF结合域(图1),结合域内含有2个预测的核定位信号(R32KRP和P46LKKARV)。GmERFd(GenBank:XM003538704) ORF全长810 bp,编码269个氨基酸,靠近蛋白序列C端含有一个由59个氨基酸组成的AP2/ERF结合域(图1),结合域外含有1个预测的核定位信号(K262KRKK)。GmERFe(GenBank登录号:NM001254494) ORF全长903 bp,编码300个氨基酸,靠近蛋白序列N端含有一个由58个氨基酸组成的AP2/ERF结合域(图1),结合域外含有1个预测的核定位信号(P66VKRQRK)。

图1 五个ERF蛋白的AP2/ERF结合域Fig.1 AP2/ERF binding domains of 5 ERF proteins
将这5个ERF蛋白与GenBank中功能已经鉴定的6个大豆ERF蛋白构建系统进化树,进行系统发育分析。结果如图2所示,GmERFe与GmERF3和GmERF7同源性最高,在它们蛋白的N端均含有一个功能未知的MCGGAI(I/L)元件,推测GmERFe功能上与GmERF3和GmERF7相似,均为转录激活子;GmERFb、GmERFc与GmERF5的同源性最高,在它们蛋白的C端均含有一个EAR抑制元件,推测GmERFb和GmERFc功能上与GmERF5相似,均为转录抑制子;此外,GmERFd与GmEREBP1的同源性较高,而GmERFa与其他ERF蛋白的同源性均较低。
2.2 五个ERF基因的组织表达
实时荧光定量PCR检测5个ERF基因在大豆不同组织中的表达情况。结果显示,5个基因都主要在大豆的根和叶中表达,其中GmERFb/c/e在根中的表达量高于叶,而GmERFa/d在叶中和根中的表达量无明显差异(图3)。此外,GmERFb在胚中基本不表达;GmERFc在花中的表达量也比较高,仅次于根中的表达量。以上结果表明ERF转录因子基因可能主要在大豆的根部和叶片中行使一定的调控功能。
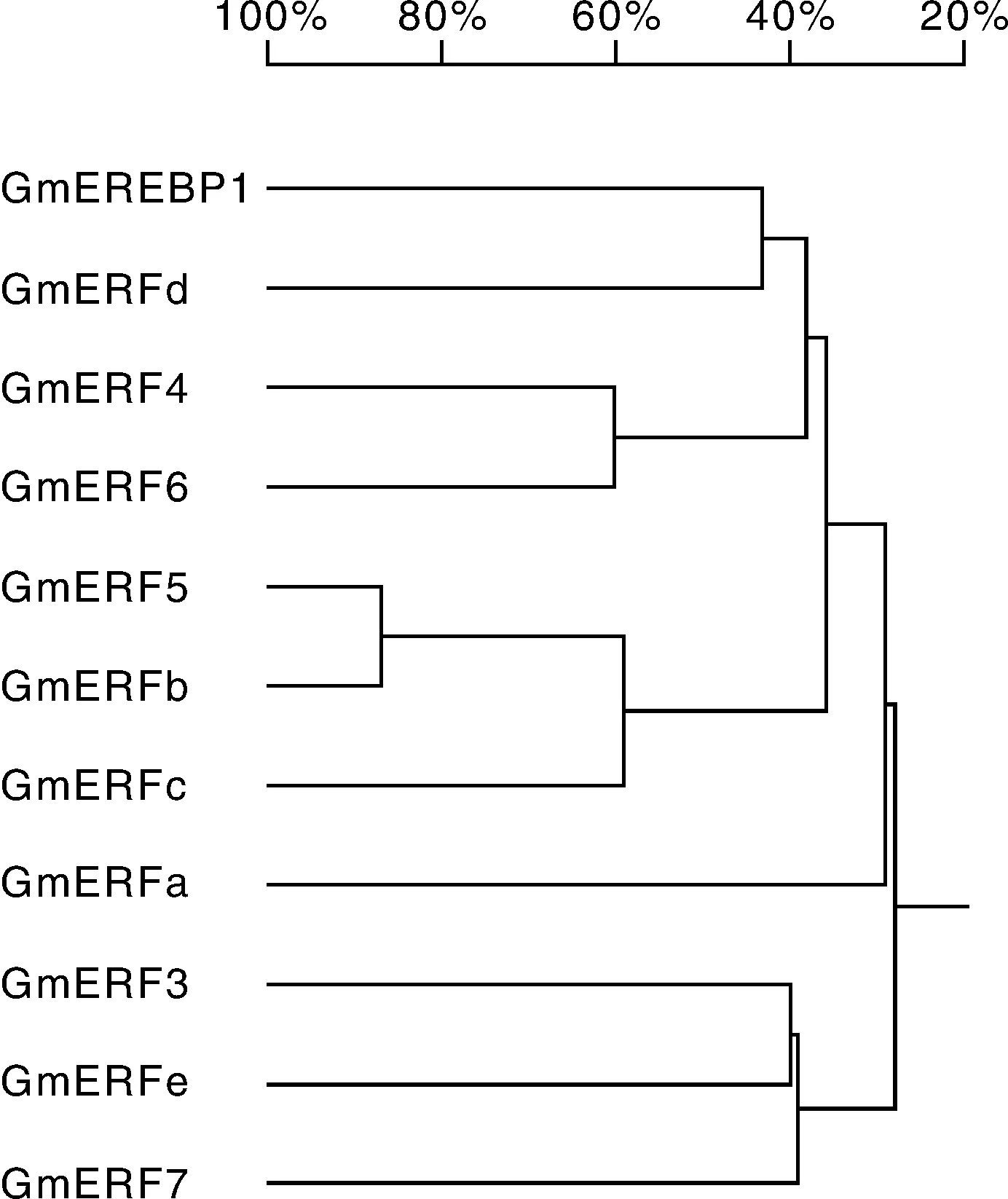
GmEREBP1: AAM45475; GmERF3: NP001238300; GmERF4: ACE76905; GmERF5: AEX25891; GmERF6: AEQ55267; GmERF7: AEQ55266图2 系统进化树Fig.2 Phylogenetic tree

图3 五个ERF基因在大豆不同组织中的表达Fig.3 Expression of 5 ERF genes in soybean tissues
2.3 五个ERF基因在逆境处理条件下的表达
用ABA、ET、干旱、高盐和低温处理大豆幼苗,通过实时荧光定量PCR检测5个ERF基因在逆境下的表达动态。结果显示,ABA处理后,除了GmERFa和GmERFc在1 h有升高外,其他均有所下降(图4-A);ET处理后,5个基因的表达量均有所升高,GmERFa/b/c的表达量升高不明显,GmERFd和GmERFe的表达量升高较为明显,均在处理2 h达到最大值,分别是对照的20倍和11倍(图4-B);干旱处理后,5个基因的表达趋势较为一致,均是先降低后升高,在处理24 h时达到最大值(图4-C);高盐处理后,5个基因的表达量均随时间延长有所下降(图4-D);低温处理后,5个基因均呈现先升高后降低的趋势,GmERFa/b/c的表达量在1 h达到最大值,GmERFd和GmERFe的表达量在2 h达到最大值(图4-E)。
3 讨论
植物在一生中总会遇到各种各样的生物及非生物胁迫(例如病原侵染、干旱、极端温度和盐碱等)。作为对不良环境的应答,植物会在染色体DNA、转录及转录后3个水平上精确调控相关基因的表达[17]。转录因子通过与启动子中顺式作用元件的特异结合可以在转录水平上调控下游基因的表达。ERF转录因子是一个庞大的基因家族,不同的ERF转录因子对于基因的转录调控既存在差异,又存在功能冗余。

图4 五个ERF基因在ABA(A)、ET(B)、干旱(C)、盐分(D)、低温(E)处理下的表达Fig.4 Expression of 5 ERF genes in ABA (A), ET (B), drought (C), salt (D) and low temperature (E) treatment
根据Tournier等[18]的分类方法,ERF转录因子分4个亚类,第Ⅱ亚类中的ERF均为转录因抑制子,都含有EAR抑制元件。所以推测本研究中GmERFb和GmERFc应为转录抑制子,而GmERFa/d/e不含EAR元件,应为转录激活子。核定位预测结果显示5个ERF基因均含有1~2个预测的核定位信号,这与以往鉴定的ERF转录因子定位于细胞核中的结果相一致[7,19]。这是因为转录因子虽在细胞质中合成,但却在细胞核中发挥转录调控作用。
植物在遭受生物和非生物胁迫时最先感知的就是根和叶片,所以逆境相关的基因主要在根和叶片中发挥作用,这与本研究中5个ERF主要在根和叶中表达相一致。本文中的5个ERF基因对逆境胁迫的应答并不一致,这与前人发现的拟南芥中不同的ERF成员在胁迫下具有不同表达模式的研究结果相一致[10]。表明不同的ERF基因对相同或不同的逆境均存在不同的应答机制。转录因子之所以会对不同的逆境信号产生不同的响应,可能是由于DNA结合特性不同或者是存在翻译后水平调控, 也有可能是与不同蛋白相互作用的结果[20]。根据实时荧光定量PCR的结果,5个ERF基因中,GmERFd/e主要对乙烯信号和低温产生响应,GmERFb主要对干旱产生响应,而GmERFa主要对低温产生响应,可进行进一步研究。
[1] 崔喜艳, 陈众峰, 陈展宇. AP2/ERF转录因子对植物非生物胁迫应答的研究进展[J]. 吉林农业大学学报, 2015, 37(4): 417-423.
CUI X Y, CHEN Z F, CHEN Z Y. Advances of researches on AP2/ERF transcription factor response to plant abiotic stress[J].JournalofJilinAgriculturalUniversity, 2015, 37(4): 417-423. (in Chinese with English abstract)
[2] UPADHYAY R K, SONI D H, SINGH R, et al.SIERF36, an EAR-motif-containing ERF gene from tomato, alters stomatal density and modulates photosynthesis and growth[J].JournalofExperimentalBotany, 2013, 64(11): 3237-3247.
[3] 张淑珍, 华彩峰, 董利东, 等. ERF转录因子及在大豆中的研究进展[J]. 大豆科学, 2015, 34(3): 512-517.
ZHANG S Z, HUA C F, DONG L D, et al. ERF transcription factors and their research advancement in soybean[J].SoybeanScience, 2015, 34(3): 512-517. (in Chinese with English abstract)
[4] THIRUGNANASAMBANTHAM K, DURAIRAJ S, SARAVANAN S, et al. Role of ethylene response transcription factor (ERF) and its regulation in response to stress encountered by plants[J].PlantMolecularBiologyReporter, 2015, 33(3): 347-357.
[5] OHME-TAKAGI M, SHINSHI H. Ethylene-inducible DNA binding proteins that interact with an ethylene-responsive element[J].PlantCell, 1995, 7(2): 173-182.
[6] HAO D, OHME-TAKAGI M, SARAI A. Unique mode of GCC box recognition by the DNA-binding domain of ethylene-responsive element binding factor(ERF domain) in plants[J].JournalofBiologicalChemistry, 1998, 273(41): 26857-26861.
[7] PARK J M, PARK C J, LEE S B, et al. Overexpression of the tobacco Tsi1 gene encoding an EREBP /AP2-type transcription factor enhances resistance against pathogen attack and osmotic stressing tobacco[J].PlantCell, 2001, 13(5): 1035-1046.
[8] BUTTNER M, SINGH K B. Arabidopsis thaliana ethylene-responsive element binding protein (AtEBP), an ethylene-inducible, GCC box DNA-binding protein interacts with an ocs element binding protein[J].ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 1997, 94(11): 5961-5966.
[9] XUE G P, LOVERIDGE C W. HvDRF1 is involved in abscisic acid mediated gene regulation in barley and produces two forms of AP2 transcriptional activators, interacting preferably with a CT-rich element[J].PlantJournal, 2004, 37(3): 326-339.
[10] FUJIMOTO S Y, OHTA M, USUI A, et al. Arabidopsis ethylene-responsive element binding factors act as transcriptional activators or repressors of GCC box-mediated gene expression[J].PlantCell, 2000, 12(3): 393-404.
[11] XU Z S, XIA L Q, CHEN M, et al. Isolation and molecular characterization of theTriticumaestivumL. ethylene-responsive factor 1 (TaERF1) that increases multiple stress tolerance[J].PlantMolecularBiology, 2007, 65(6): 719-732.
[12] ZHAI Y, WANG Y, LI Y J, et al. Isolation and molecular characterization ofGmERF7, a soybean ethylene-response factor that increases salt stress tolerance in tobacco[J].Gene, 2013, 513(1): 174-183.
[13] ZHAI Y, LI J W, LI X W, et al. Isolation and characterization of a novel transcriptional repressorGmERF6 from soybean[J].BiologiaPlantarum, 2013, 57(1): 26-32.
[14] OHTA M, MATSUI K, HIRATSU K, et al. Repression domains of class II ERF transcriptional repressors share an essential motif for active repression[J].PlantCell, 2001, 13(8): 1959-1968.
[15] DONG L D, CHENG Y X, WU J J, et al. Overexpression ofGmERF5, a new member of the soybean EAR motif-containing ERF transcription factor, enhances resistance to Phytophthora sojae in soybean[J].JournalofExperimentalBotany, 2015, 66(9): 2635-2647.
[16] ZHANG G Y, CHEN M, CHEN X P, et al. Phylogeny, gene structures, and expression patterns of the ERF gene family in soybean (GlycinemaxL.) [J].JournalofExperimentalBotany, 2008, 59(15): 4095-4107.
[17] ZHANG Y Y, YANG C W, LI Y, et al. SDIR1 is a RING finger E3 ligase that positively regulates stress-responsive abscisic acid signaling inArabidopsis[J].PlantCell, 2007, 19(6): 1912-1929.
[18] TOURNIER B, SANCHEZ-BALLESTA M T, JONES B, et al. New members of the tomato ERF family show specific expression pattern and diverse DNA-binding capacity to the GCC box element[J].FEBSLetters, 2003, 550(1-3): 149-154.
[19] ZHANG G Y, CHEN M, LI L C et al. Overexpression of the soybeanGmERF3 gene, an AP2/ERF type transcription factor for increased tolerances to salt, drought, and diseases in transgenic tobacco[J].JournalofExperimentalBotany, 2009, 60(13): 3781-3796.
[20] ONATE-SANCHEZ L, SINGH K B. Identification of Arabidopsis ethylene-responsive element binding factors with distinct induction kinetics after pathogen infection[J].PlantPhysiology, 2002, 128(4): 1313-1322.
(责任编辑 张 韵)
Bioinformaticsandexpressionanalysisof5newfoundERFgenesinsoybean
ZHAIYing1,ZHANGJun2,ZHAOYan1,GAOShi-tong1,SUNWan-shu1,ZHANGChuang1,RENWei-wei1,WANGXiu-wen1,WANGYu-shu1
(1. College of Life Science and Agro-forestry, Qiqihar University, Qiqihar 161006, China;2. Heilongjiang Institute of Veterinary Science, Qiqihar 161005, China)
ERFtranscriptionfactorsarewidespreadinplants,whicharewidelyinvolvedinplantresponsetobioticandabioticstress.Inthisstudy,fivenovelERFgenesequences, ERFa/b/c/d/e,wereobtainedbyblastfromNCBIdatabase.ProteinsequencesanalysisshowedthatthefiveERFproteinsallcontainedatypicalAP2/ERFbindingdomain.PhylogeneticanalysisindicatedthatGmERFewashighlyhomologoustoGmERF3andGmERF7;GmERFbandGmERFcwerehighlyhomologoustoGmERF5;GmERFdwashomologoustoGmEREBP1;whileGmERFahadlowerhomologywithotherERFproteins.TheexpressionpatternsoffiveERFgenesinsoybeantissuesandunderstresstreatmentswereanalyzedbyreal-timequantitativePCR.TheresultsshowedthatthefiveERFgenesexpressedhighlyinrootandleaf.GmERFd/erespondedmainlytoethylenesignalandlowtemperature,GmERFbrespondedmainlytodrought,andGmERFarespondedmainlytolowtemperature.
soybean;ERFtranscriptionfactor;bioinformaticsanalysis;expressionanalysis
http://www.zjnyxb.cn
10.3969/j.issn.1004-1524.2016.10.03
2016-02-16
国家自然科学基金项目(31301335);黑龙江省教育厅科学技术研究项目(12541889);黑龙江省自然科学基金项目(C201458)
翟莹(1982—),女,吉林吉林人,讲师,博士,主要从事植物分子育种研究。E-mail: fairy39809079@126.com
S
A
浙江农业学报ActaAgriculturaeZhejiangensis, 2016,28(10): 1644-1649
翟莹,张军,赵艳,等. 大豆5个新发现ERF基因的生物信息学及表达分析[J].浙江农业学报,2016,28(10): 1644-1649.
