HBV核苷(酸)类似物耐药突变及其检测技术的临床应用
2016-11-15巢薇何宝玉
巢薇何宝玉
·综述·
HBV核苷(酸)类似物耐药突变及其检测技术的临床应用
巢薇★何宝玉
目前,可供临床应用的抗慢性乙型肝炎(chronic hepatitis B,CHB)的核苷(酸)类似物[nucleos(t)ide analogs,NAs]包括拉米夫定(lamivudine,LAM)、替比夫定(telbivudine,LdT)、阿德福韦酯(adefovir,ADV)、恩替卡韦(entecavir,ETV)及替诺福韦酯(tenofovir,TDF),NAs作为病毒逆转录酶抑制剂,可抑制HBV DNA复制扩增。由于HBV属于高基因变异率病毒,NAs长期治疗后,选择压力下使耐药病毒株成为优势株,影响治疗效果。因此,为防治CHB抗病毒治疗中耐药的发生,弄清楚其耐药机制及变异的监测及诊断十分必要。本文总结了近年来发现的常见突变位点,并比较了几种临床应用的突变监控及检测技术,对耐药突变进行早期、快速检测具有重要临床意义,使慢性HBV感染的监测达到最优化。
核苷(酸)类似物;耐药突变;乙型肝炎病毒;慢性乙型肝炎;常见耐药变异;突变位点检测
乙型肝炎病毒(hepatitis B virus,HBV)是一种高危致病性病毒,慢性乙型肝炎(chronic hepatitis B,CHB)是HBV最常引起的一种慢性疾病,1/3以上的感染者在中国[1]。抗病毒治疗是CHB的根本治疗措施,核苷(酸)类似物[Nucleos(t)ide analogs,NAs]作为目前临床抗病毒治疗的主流药物,主要包括拉米夫定(lamivudine,LAM)、替比夫定(telbivudine,LdT)、阿德福韦酯(adefovir,ADV)、恩替卡韦(entecavir,ETV)及替诺福韦酯(tenofovir,TDF)5种常用药物,它们均可抑制病毒DNA复制[2]。但随着应用时间的推移和应用群体的扩大,NAs耐药问题日趋严重。启动NAs抗病毒治疗后,需定期监控评估NAs常见耐药突变位点变异率,根据应答和耐药情况调整药物和疗程[3]。这已成为CHB抗病毒治疗中的重要监测措施。新突变位点的不断发现及检测方法的不断更新和广泛应用,将为提高CHB抗病毒的治疗疗效提供重要的理论和技术支持。本文综述了近年来报道的基因多位点耐药相关突变,这有助于临床及时发现乙型肝炎患者是否存在HBV耐药。同时,文章比较了几种临床应用的突变监控及检测技术,结合其原理及技术发展,对耐药检测技术的现状进行分析比较,为临床动态监测HBV变异病毒株、指导合理用药奠定了基础。
1 HBV NAs的耐药机制
HBV吸附脱壳感染肝细胞后,在HBV DNA聚合酶作用下,以负链DNA为模板修补延长正链,形成的共价闭合环状的双链DNA(covalently closed circular DNA,cccDNA)进入细胞核[4-5]。在核内以cccDNA为模板转录产生的mRNA,含有HBV全部遗传信息,故称为前基因组RNA。前基因组RNA出核后逆转录为负链DNA,继续以负链为模板合成正链,双链环化形成完整的HBV DNA[4,6]。NAs治疗则通过抑制逆转录酶活性,阻止前基因组RNA逆转录形成新的病毒DNA,从而发挥抑制病毒复制的作用。HBV前基因组RNA是以HBV的cccDNA为模板合成的,即NAs的药效靶点在cccDNA的下游,见图1。所以NAs不能直接清除已经存在的cccDNA[7-8],因此,为了维持HBV的低水平复制,NAs治疗需很长时间。其次,由于HBV DNA复制过程必需的逆转录酶缺乏3′-5′外切活性,极易导致碱基错配,病毒基因自发突变频率高。因此,一旦病毒对长期服用的某种NAs药物产生选择压力,此种药物便失去了疗效,从而导致耐药的发生。
体内出现耐药变异后,继续维持相同NAs治疗,野生敏感病毒株被抑制,变异株加快复制取代野生株成为优势株,从而导致对NAs耐药。随着研究的深入,耐药机制已由传统的定性研究向精确的定量研究跨越,随之而来的一系列问题需要研究者继续去探索:变异株达到病毒群的多少百分比可以称为优势株?继续使用NAs治疗,变异株积累成优势株,最后成为耐药株的速率与不同NAs的服用剂量有何关系?优势株占多少比例时会出现病毒学突破?在检测到耐药突变后,如何进一步明确患者体内变异株的比率或者变异株的拷贝数与临床的关系?新突变位点的不断发现及检测方法的不断更新,将对这些问题的深入研究和探讨提供必要的保障与支持。
2 HBV NAs常见的耐药突变位点
HBV耐药基因突变发生于病毒逆转录酶基因序列区域。根据2001年提议的耐药突变位点命名规则,将HBV耐药变异统一从逆转录酶区(各基因型均为344个氨基酸)的第一个氨基酸数起并加前缀rt,分别为rt1~rt344(例如YMDD变异被命名为rtM 204V)[9]。
2.1 LAM和LdT耐药变异位点
LAM是最早应用于临床的核苷类似物,迄今仍有CHB患者使用LAM,因此,其耐药突变位点最多,而且耐药发生率以每年约30%的速率增加[10]。LAM耐药突变主要发生在逆转录酶基因C区的保守序列YMDD区域,碱基发生置换时,第204位的蛋氨酸(M)变为亮氨酸(V)或异亮氨酸(I)即为rtM 204V/I/S。在rtM 204V/I出现后,若继续维持LAM治疗,则会出现耐药性更强的突变—rtL180V/I/M[11-12]。rtM 204V/I和rtL180V/I/M的患者会进一步引发第3个常见的突变位点rt173L。近年来报道的LAM耐药突变位点还包括rtA181T及rtQ215S。长期的LAM治疗通常会导致许多组合型耐药突变,rtL180M+rtM 204V、rtL180M+ rtM 204I、rtV173L+rtL180M+rtM 204V、rtM 204I是LAM耐药变异的主要组合方式[13]。
LdT和LAM同属于L-核苷,故二者具有交叉耐药突变位点,包括M 240及L180[14]。由于存在交叉耐药,故不可在出现LAM耐药后继续使用LdT治疗[15]。因此,对于结构相似会出现交叉耐药现象时,需综合考虑耐药位点及相关变异情况,适时进行耐药监控,结合临床症状,选择正确的抗病毒治疗药物。
2.2 ADV耐药变异位点
ADV耐药突变率较LAM低,初治患者(指未用过NAs抗HBV药物者)第1年未见耐药,HBeAg阴性初治患者ADV治疗的基因耐药率前5年分别为0、3%、7%、8%、11%[11]。N236T氨基酸置换是ADV标志性耐药变异位点,继续ADV治疗,引起YMDD临近区域的耐药变异rtA181V/T。常见的ADV耐药突变还有C区的rtV214A、D区的rtQ215S、rtI233V及rtL217R等。ADV药物结构与LAM不同,rtN236T变异不会改变LAM的敏感性,但rtA181V/T、rtV214A及rtQ215S变异株会与LAM出现交叉耐药[16]。
2.3 ETV耐药变异位点
ETV是鸟嘌呤核苷类似物,ETV的耐药位点是在拉米夫定耐药位点(rtL180M+rtM 204I/V/S)基础上同时出现A区rtI169T、B区rtT184C/G、rtS202G/I和D区rtM 250V等突变。ETV对初治患者基本不发生耐药,1~5年的耐药突变率均为<1%[17]。而发生拉米夫定耐药患者中,ETV治疗2年的累积基因耐药率为16%,病毒学反弹超过10%,较初治患者明显升高。ETV具有高基因屏障的特点,多位点突变才可能出现耐药现象,这也是ETV耐药发生率低的重要原因。
2.4 TDF耐药变异位点
TDF与ADV结构相似,两者具有类似的抗病毒活性。研究显示,TDF治疗3年后并未出现明确的TDF耐药,有报道第194位丙氨酸至苏氨酸的替换(rtA194T)与TDF耐药相关,但出现该耐药后仍对ETV敏感[18]。体外实验发现,rtA181/T或rtN236T ADV耐药株对TDF的敏感性下降3~4倍,推测可能是TDF潜在突变位点[19]。
耐药位点的提出与确立是一个长期的、需要重复验证的过程。在验证过程中,由于表型耐药检测体系的差别可能会出现结果的不一致。单个变异位点的确定需要结合HBV基因型、全基因组序列及患者的临床指标等各方面因素[20-21]。此外,我们需要深入了解耐药变异与临床耐药的关系,结合临床症状及个体化因素,综合分析耐药变异引起的临床改变,从而确定准确可靠耐药变异位点。随着研究的不断深入,越来越多的突变位点被发现,表1总结了近几年发现报道的NAs常见的耐药突变位点,可目前应用于临床指导药物选择的位点有限。因此,我们需要在探索新位点的同时,建立更加成熟的突变检测技术,制定合理的抗病毒治疗方案,对患者服用药物的相关耐药突变位点实时监控,及时调整治疗方案。
3 HBV NAs的耐药突变检测
建立准确、灵敏和经济可行的耐药突变检测方法对于慢性HBV感染抗病毒治疗中耐药性的监测和确定具有重要意义。目前已报道的HBV耐药突变的检测方法根据原理的不同大致可分为测序法、反向杂交法及PCR相关技术方法等,这些方法各有利弊。随着临床耐药形势日渐严峻,发展并推广特异性强、灵敏度高及价格适中的检测手段成为当务之急。
3.1 测序法
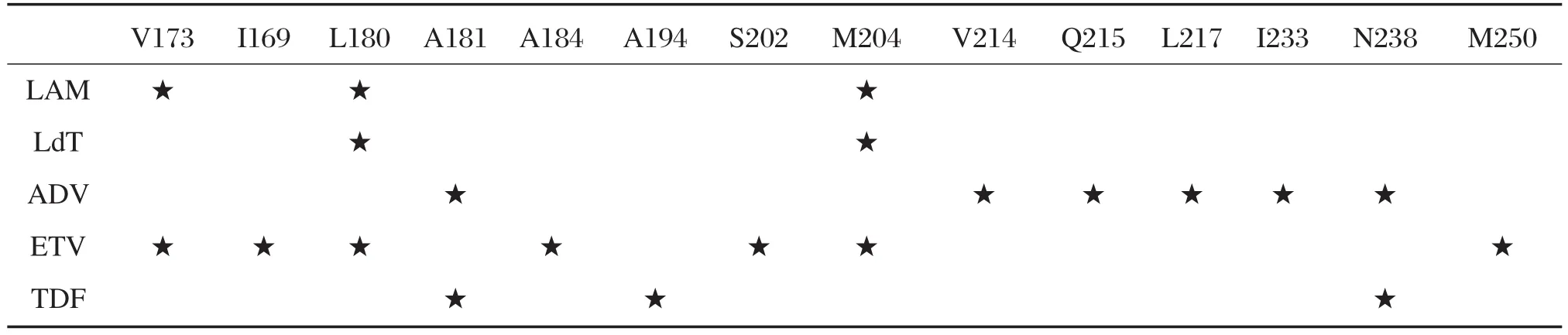
表1 HBV NAs常见的耐药突变位点Table 1 Common drug resistance mutations of NAs against HBV
测序法主要包括经典直接测序法、克隆测序法、焦磷酸测序法、SNaPshot技术等。随着生物科学的迅猛发展,作为最准确的耐药检测方法,测序法不断发展完善。与其他耐药检测方法相比,测序法精准度和特异性都较高,且能对多个位点同时进行检测。测序法与其他检测手段进行比较,可作为检测功效的衡量标准。
3.1.1 直接测序法(direct sequencing,DS)与SNaPshot单核苷酸多态性检测法
DNA直接测序法一直被认为是检测基因突变的金标准,研究者曾利用DS检测出HBV YMDD和rtA181T/V位点变异[22]。DS最大的优势是可检测新发突变位点,DS的检测结果通常与还可与其他突变检测技术比较,作为检测功效的衡量标准。由于DS要求将目标序列进行扩增并连接到载体再行检测,因此无法满足HBV快速诊断和大规模检测的要求。另外,对于多种基因型同时存在的病毒群,DS只能检测DNA含量高的优势病毒株。而对于病毒含量低标本,DS灵敏度不高,可能会漏检低比例(<1%)耐药突变[23]。
SNapshot单核苷酸多态性检测法,即微测序法,是在DNA直接测序基础上发展出来的一种新的检测方法。该技术依据待检测序列设计特异性引物,在反应体系中加入荧光标记的ddNTP,依据峰的颜色确定样本的基因型,依据峰的位置确定延伸产物对应的SNP位点。Salpini等[24]利用SNaPshot技术对204例CHB患者血清检测耐药位点检测,结果与测序结果一致,证实了SNaPshot技术高度的准确性。与直接测序法相比该技术操作简便,经济快速。但是此类方法对引物的要求较高,并且一般只可以检测已知突变位点。
3.1.2 焦磷酸深度测序
焦磷酸深度测序技术(pyro-sequencing,PyroS)是近年来临床应用较广的一种实时DNA序列分析技术。该技术敏感度较高,最少可检测0.1%比例的耐药突变,因此可作为耐药变异的早期检测方法。Ciftci等[25]运用该技术对42位CHB患者的耐药位点进行扫描,发现了rtL180I、rtQ215P、rtI233Y、rtN238D和rtM 250S 5个独立的耐药突变位点,作者进一步研究发现rtI266G突变会与LAM耐药有关,推测可能是LAM潜在的耐药变异位点。目前,以高灵敏度及特异性为特点的焦磷酸深度测序已超越经典普通测序方法,尤其是在早期监控及检测阶段,发挥了重要的作用。
3.2 杂交法
杂交法是使样本的扩增产物与荧光标记探针特异性结合,通过对鉴别不同荧光检测突变位点的方法。由于扩增产物可同时与多个探针结合,故杂交法可一次性检测多个突变模式。
3.2.1 线性探针技术
线性探针技术用于检测HBV耐多药,具有成本低、特异性高等特点,为耐多药突变的早发现、早治疗提供了诊断依据。Yoshida等[26]针对HBV野生型和已知位点(rtM 204V/I)的耐药型毒株基因序列分别设计探针,固定于PVDF膜,将待检测样本经PCR扩增后杂交,结果在109名CHB患者中,检出了90名rtM 204V/I突变,检出率为88.2%。线性探针技术的缺点是只可检测已知突变。近年来,以线性探针为基础发展起来的交联扩增技术(ligation amplification assay,LigAmp)在YMDD变异检测时具有较高敏感度,可在LAM耐药突变进行早期监控[27]。
3.2.2 基因芯片技术
基因芯片突变检测技术是将特定的基因片段或寡核苷片段以特定顺序密集排列于某种固相载体上(如硅片或玻片),形成基因芯片,与荧光标记样本杂交,通过激光共聚焦系统检测杂交信号强度,读取样本的数量和序列信息。已有研究显示,芯片检测法准确率达96%,敏感性达95%,特异性为100%,对比传统的DNA序列分析法(准确率87%,敏感性82%,特异性100%),该技术有较高的检测灵敏度与准确率[28]。基因芯片技术与传统杂交技术相比,具有检测效率高、系统微型化、能够同时对多个位点同步检测的优势,符合现代分子生物医学发展的趋势,但HBV的高突变率缩短了寡核苷探针的使用寿命,偏差的出现导致一定比例的假阴性,制约了该技术的发展及应用[29]。
3.3 PCR相关技术
3.3.1 实时荧光定量PCR(realtime fluorescence quantitative PCR,RTFQ-PCR)熔解曲线分析法
RTFQ-PCR技术利用荧光信号实时积累监控整个PCR过程,SYBR GreenⅠ非特异性地嵌入DNA双链间,发出强烈荧光信号,若存在其他双链寡核苷酸,便会产生假阳性信号,降低了检测的精确性[30]。Zeng等[31]利用实时荧光PCR技术检测HBV YIDD耐药突变,与市售优秀HBV耐药突变测序检测试剂盒检测结果的完全一致率91.2%(93/102),部分一致率8.8%(9/102),未发现完全不一致(0/102),两法一致性好(Kappa=0.676,P= 0.000)。克隆测序检结果与RTFQ-PCR完全一致。RTFQ-PCR检测技术可通过Ct数值确定变异病毒含量及其与野生毒株的比值,具有重复性好、灵敏度高、无扩增后处理等优点,是临床应用时间最长、最成熟的一种分子检测技术,目前已成为临床实验室普遍采用的检测技术。但该方法也有其局限性,如HBV的高突变性导致探针和引物设计出现偏差,出现假阴性。
3.3.2 聚合酶链式反应微板核酸杂交-酶联免疫吸附法(PCRmicroplate nucleic acid hybridization-enzyme linked immunosorbent assay,PCRmnh-ELISA)
PCRmnh-ELISA检测技术将基因扩增、核酸杂交和酶联显色3种诊断技术结合,通过设计特定的组合引物将突变基因和正常基因分辨出来。PCR-mnh增强了核酸杂交的特异性,而与ELISA技术结合后则提高了检测方法的灵敏度。PCRmnh-ELISA可以检测出已知变异的基因序列,可同时从1 000个正常基因序列中检测出1个变异位点,且检测结果不受正常基因序列干扰。但体系中的非特异性探针杂交及过度敏感的信号放大系统均会导致假阳性的产生。此外,此法操作复杂,需在开放环境中进行,可能会对实验室造成污染。目前该技术仅用于科学研究,临床普及难度大。
3.4 其他检测方法
变性高效液相色谱(denaturing high performance liquid chromatography,DHPLC),也称为温度调节的杂合双链分析技术,是近年来发展起来的一项新的分析技术。该技术利用部分变性条件下同源—异源双链靶DNA解链特征的差异性进行突变位点检测。该检测方法具有快速、高特异性、高灵敏、高通量、廉价省时及操作简单等优点。但是,该技术对待检样本的PCR技术要求很高,对PCR引物设计、体系优化及最适扩增片断等都有较高要求,还需保证PCR产物具有较高的含量和质量。当PCR片段扩增特异性较差时,可能会导致结果假阳性,以致检测结果不准确。
4 展望
随着对NAs耐药机制研究的不断深入,耐药管理的观念也在不断更新。在整个用药期间对病患定期随访、精确监控耐药位点以便第一时间诊断出病毒耐药成为临床最行之有效的耐药管理方案,精确严谨的实验室检测则是上述方案取得成功的有力保证。
随着生物医学的快速发展,越来越多的新技术开始应用于HBV耐药突变的检测。敏感性和特异性越来越高、可检测位点(尤其是未知变异位点)越来越多、样本量越来越大、操作简化、成本低廉,这些特点都是NAs耐药突变检测新技术的发展趋势。经典的直接测序法特异性强、敏感度高,可检测未知变异位点,一直被视为变异位点检测的金标准;荧光定量PCR技术灵敏度较高,而且荧光定量分析仪目前也已普及,价格相对适中,因此目前认为该方法在临床上应用前景最为广泛,适合大标本量检测;以焦磷酸测序技术和SNaPshot为代表的第三代测序技术兼顾了高敏感性、高特异性等特点,高通量的检测又可大幅度降低成本,SNaPshot技术更可直观地检测基因片段碱基序列,但技术尚未成熟,且对引物和实验条件要求较高,临床普及开展难度大,相信未来会有所改善。
[1]Qi FH,Wang ZX,Cai PP.Traditional Chinese medicine and related active compounds:a review of their role on hepatitis B virus infection[J].Drug Discov Ther,2013,7(6):212-224.
[2]Fan J,Zhang Y,Xiong H,etal.Nucleotide analogueresistant mutations in hepatitis B viral genomes found in hepatitis B patients[J].JGen Virol,2015,96(Pt 3):663-670.
[3]Koumbi L.Current and future antiviral drug therapies of hepatitis B chronic infection[J].World JHepatol,2015,7(8):1030-1040.
[4]Yim HJ,Hwang SG.Options for the management of antiviral resistance during hepatitis B therapy:reflections on battles over a decade[J].Clin Mol Hepatol,2013,19(3):195-209.
[5]Kim DY,Chang HY,Lim SM,et al.Quasispecies and pre-existing drug-resistant mutations of hepatitis B virus in patients with chronic hepatitis B[J].Gut Liver,2013,7(3):329-334.
[6]Zoulim F.Mechanism of viral persistence and resistance to nucleoside and nucleotide analogs in chronic hepatitis B virus infection[J].Antiviral Res,2004,64(1):1-15.
[7]Shaw T,Bartholomeusz A,Locarnini S,HBV drug resistance:mechanisms,detection and interpretation[J].Hepatol,2006,44(3):593-606.
[8]Gupta N,GoyalM,Catherine T,et al.Themolecularand structural basis of HBV-resistance to nucleos(t)ide analogs[J].Clin Transl Hepatol,2014,2(3):202 -211.
[9]Suppiah J,Mohd ZR,Haji NS,et al.Drug-resistance associated mutations in polymerase(p)gene of hepatitis B virus isolated from malaysian HBV carriers[J]. HepatMon,2014,14(1):e13173.
[10]LiX,Liu Y,Zhao P,etal.Investigation into drug-resistant mutations of HBV from 845 nucleoside/nucleotide analogue-naive Chinese patients with chronic HBV infection[J].Antivir Ther,2015,20(2):141-147.
[11]Lei J,Wang Y,Wang LL,etal.Profile of hepatitis B virus resistance mutations against nucleoside/nucleotide analogue treatment in Chinese patientswith chronic hepatitis B[J].Virol J,2013,10:313.
[12]Liu Y,Xu ZH,Wang Y,et al.rtM 204Q may serve as a novel lamivudine-resistance-associated mutation of hepatitis B virus[J].PlosOne,2014,9(2):e89015.
[13]Parvez MK,Arab AH,Al-DosariMS,et al.Antiviral natural products against chronic hepatitis B:recent developments[J].Curr Pharm Des,2016,22(3):286-293.
[14]Cai S,Yu T,Jiang Y,et al.Comparison of entecavir monotherapy and de novo lamivudine and adefovir combination therapy in HBeAg-positive chronic hepatitis B with high viral load:48-week result[J].Clin Exp Med,2015,Epub ahead of print.
[15]Pan HY,Chen L,Yang DH,et al.Ten-year follow up of hepatitis B relapse after cessation of lamivudine or telbivudine treatment in chronic hepatitis B patients[J].Clin Microbiol Infect,2015,21(12):1123.
[16]Kim JH,Ko SY,Choe WH,et al.lamivudine plus adefovir combination therapy for lamivudine resistance in hepatitis-B-related hepatocellular carcinoma patients[J].Clin Mol Hepatol,2013,19(3):273-279.
[17]Takamatsu Y,Tanaka Y,Kohgo S,et al.4′-modified nucleoside analogs:potent inhibitors active against entecavir-resistant hepatitis B virus[J].Hepatology,2015,62(4):1024-1036.
[18]Murakam i E,Tsuge M,Hiraga N,et al.Effect of tenofovir disoproxil fumarate on drug-resistant HBV clones[J].Infect,2016,72(1):91-102.
[19]Hann HW,Chae HB,Dunn SR.Tenofovir(TDF)has stronger antiviral effect than adefovir(ADV)against lamivudine(LAM)-resistant hepatitis B virus(HBV)[J].Hepatol Int,2008,2(2):244-249.
[20]Liu C,Lin JP,Chen HJ,et al.Detection of hepatitis B virus genotypic resistance mutations by coamplification at lower denaturation temperature-PCR coupled with sanger sequencing[J].Clin M icrobiol,2014,52(8):2933-2939.
[21]Bhattacharya D,Thio CL.Review of hepatitis B therapeutics[J].Clin Infect Dis,2010,51(10):1201-1208.
[22]JardiR,ButiM,Frias FR,etal.Rapid detection of lamivudine-resistant hepatitis B virus polymerase gene variants[J].Virol Methods,1999,83(1-2):181-187.
[23]Lowe CF,Merick L,Harrigan PR,et al.Implementation of next-generation sequencing for hepatitis B virus resistance testing and genotyping in a clinical microbiology laboratory[J].Clin Microbiol,2016,54(1):127-133.
[24]SalpiniR,AlteriC,Cento V,etal.Snapshoton drugresistance rate and profiles in patientswith chronic hepatitis B receiving nucleos(t)ide analogues in clinical practice[J].JMed Virol,2013,85(6):996-1004.
[25]Ciftci S,Keskin F,Cakiris A,et al.Analysis of potential antiviral resistance mutation profiles within the HBV reverse transcriptase in untreated chronic hepatitis B patients using an ultra-deep pyrosequencing method[J].Diagn Microbiol Infect Dis,2014,79(1):25-30.
[26]Yoshida S,Hige S,Yoshida M,et al.Quantification of lamivudine-resistant hepatitis B virus mutants by type-specific TaqManminor groove binder probe assay in patients with chronic hepatitis B[J].Ann Clin Biochem,2008,45(Pt1):59-64.
[27]Ismail AM,Sachithanandham J,Eapen CE,etal.Performance of LigAmp assay for sensitive detection of drug-resistant hepatitis B virusminor variants in comparison with standard nucleotide sequencing[J].Mol Diagn Ther,2014,18(6):655-663.
[28]Chan K,Yam I,Yuen J,et al.A comprehensive HBV array for the detection of HBV mutants and genotype[J].Clin Biochem,2011,44(14-15):1253-1260.
[29]HuaW,Zhang G,Guo S,etal.Microarray-based genotyping and detection of drug-resistant HBV mutations from 620 Chinese patients with chronic HBV infection[J].Braz J Infect Dis,2015,19(3):291-295.
[30]He X,Wang F,Huang B,et al.Detection and analysis of resistance mutations of hepatitis B virus[J].Int JClin Exp Med,2015,8(6):9630-9639.
[31]Zeng Y,Li D,Wang W,et al.Establishment of real time allele specific locked nucleic acid quantitative PCR for detection of HBV YIDD(ATT)mutation and evaluation of its application[J].Plos One,2014,9(2):e90029.
Hepatitis B virus nucleos(t)ide analogues drug resistance mutations and the clinical application of variation loci detection
CHAOWei★,HEBaoyu
(Medical Clinical Laboratory,The Fourth Hospital Affiliated to Guangxi Medical University,Liuzhou,Guangxi,China,545005)
At present,nucleos(t)ide analogues(NAs)available for clinical application of Chronic hepatitis B(CHB)include lamivudine(LAM),telbivudine(LdT),adefovir(ADV),entecavir(ETV),and tenofovir(TDF).As virus reverse transcriptase inhibitors,NAs can inhibit HBV DNA replication.Because of the highmutation rate of HBV,long-term treatment with NAs resulting in drug-resistant strains will reduce the effect of NA treatments.Therefore,in order to control the occurrence of drug resistance in CHB antiviral treatment,figuring out the resistance mechanism and monitoring and diagnosis of variation is essential.In this paper,we summarized common mutations discovered in recent years,and compared the clinical application of several technologies monitoring and detecting drug-resistant mutations,which is important for early and rapid mutation detection and optimizing the monitoring of chronic HBV infection.
Nucleos(t)ide analogues;Drug resistance mutations;Hepatitis B virus;Chronic hepatitis B;Common drug-resistance variation;Variation loci detection
国家自然科学基金(81160269);柳州市肿瘤疾病防治重点实验室建设(2014G020403)
广西医科大学第四附属医院医学检验科,广西,柳州545005
★通讯作者:巢薇,E-mail:2312969581@qq.com
