基于线粒体COI基因的雷州半岛马尾藻属常见种DNA条形码鉴定及系统进化分析
2016-11-12全秋梅谢恩义刘楚吾
贺 亮,廖 健,全秋梅,梁 忠,谢恩义,刘 丽,刘楚吾
(广东海洋大学水产学院 南海水产经济动物增养殖广东普通高校重点实验室,广东 湛江 524025)
基于线粒体COI基因的雷州半岛马尾藻属常见种DNA条形码鉴定及系统进化分析
贺亮,廖健,全秋梅,梁忠,谢恩义,刘丽,刘楚吾
(广东海洋大学水产学院 南海水产经济动物增养殖广东普通高校重点实验室,广东 湛江 524025)
采用DNA条形码技术辅以传统形态分类方法,对雷州半岛马尾藻属(Sargassum)6常见种进行物种鉴定和系统进化分析。测得6种马尾藻线粒体COI基因序列,该序列与GenBank和BOLD数据库中马尾藻序列同源性均大于或等于99%,该结果与形态分类学鉴定结果一致。COI基因序列特征分析表明,6种马尾藻保守位点421个,变异位点58个,简约信息位点21个,单一突变位点37个,变异率7.7%,T、C、A、G平均含量分别为39.1%、18.9%、19.1%、22.9%,A+T含量(58.2%)高于C+G含量(41.8%),UUU所编码的苯丙氨酸(F)使用频率最高,达12.5%,最大似然法估算的转换颠换比值为2.42,种间遗传距离多在0.030±0.013~0.100±0.016之间。聚类分析结果与形态学鉴定、同源性分析结果一致。
马尾藻属;mtDNA-COI基因;物种鉴定;分子系统进化;DNA条形码
马尾藻属(Sargassum C.Agardh)隶属于褐藻门(Phaeophyta)无孢子纲(Phaeophyta)墨角藻目(Fucales)马尾藻科(Sargassaceae),是热带及温带沿海常见的大型海藻,主要分布于印度-西太平洋和澳大利亚、加勒比海等沿海海域[1],多数种类生活在低潮带和潮下带浅水区的岩石上,是重建海藻床和实施海洋生境修复的重要构成物种,具有重要的生态价值。该属藻类株型较大,种类甚多,曾记录的种、变种及变型共 878种,目前已证实有 340种,我国海域有130种[1]。目前,已有基于形态特征的马尾藻系统学研究[2-7],但分子生物学角度的相关研究相对欠缺。近年来,随着分子生物学的快速发展,基于DNA条形码技术广泛应用于生物分类学、系统学以及种群遗传学等方面研究,分子数据结合传统形态学分类数据使难以界定的物种分类得以较好地解决[8]。每种生物特有的基因序列是进行分子生物学鉴定的基础,核基因、质体基因、线粒体基因等可能成为分子标记的潜在对象。线粒体DNA (mitochondrial DNA,mtDNA) 作为真核生物的核外遗传物质,具有基因组结构简单、呈母性遗传、编码效率高、进化速率快且不发生重组等特性,应用线粒体DNA及其片段的测序分析技术对马尾藻进行物种鉴定与分类分析将更加客观、科学,其中,细胞色素C氧化酶亚基I (cytochrome c oxidase subunit I,COI) 基因序列是线粒体基因的重要部分,具有进化速度较快、易扩增等优点,可作为良好的分子标记用于种内、属间系统进化研究[9]。目前,线粒体COI作为DNA条形码已广泛应用于动物分类鉴定与系统进化研究[10-12],而有关海洋藻类的DNA条形码研究多集中在硅藻以及红藻上,但关于褐藻研究相对比较薄弱,有待进一步的发展,如Saunders[13]于2005年首次利用线粒体COI基因对红藻3个群体的46个样品进行分析,结果表明,线粒体COI基因可准确区分形态相似的红藻,并发现一些新种;Evans等选取COI、18Sr DNA、rbc L、ITSrDNA等4个基因作为DNA条形码对硅藻属的34个体进行评价研究,认为线粒体COI比rbc L和18sr DNA的变异率均较高,ITSr DNA的种内变异率太高,且现有数据库中硅藻的ITSr DNA序列信息极少,线粒体COI基因较适于作为硅藻的DNA条形码进行研究[14];Mattio和Payri在分析马尾藻属不同条形码基因时也暗示了线粒体基因 mtsp(mitochondrial targeting signal peptide)、COI和COIII作为褐藻条形码基因的可能性[15]。本研究获取6种马尾藻线粒体COI部分序列,从分子水平对其进行DNA条形码鉴定,并探讨其系统进化,为马尾藻物种鉴定和分子系统学研究提供分子生物学依据,为雷州半岛地方性马尾藻的种质资源保护与可持续利用提供基础数据。
1 材料与方法
1.1材料采集
马尾藻样品于2015年11月至2016年3月采自湛江硇洲岛海域、徐闻四塘和海安湾。低温条件下迅速带回实验室,选择生长良好藻体,用抽滤海水洗净藻体表面附着物后,暂养于1 000 mL烧杯内,暂养条件为:温度(20±0.5)℃,盐度为28,光照周期12L:12D,照度4 000~6 000 lx,翌日提取基因组DNA。
1.2形态学分类
[1]对藻样鉴定至种。
1.3分子生物学鉴定
1.3.1基因组DNA提取每个样本剪取幼嫩的叶片组织约100 mg,于研钵中与石英砂、碳酸钙充分研磨后,采用Ezup柱式植物基因组DNA抽提试剂盒(上海生工生物工程有限公司)提取DNA,提取步骤参照说明书。经10 mg/mL琼脂糖凝胶电泳定性检测和Nanodrop 2000定量分析后,合格样品于4℃下保存备用。
1.3.2mtDNA-COI 序列PCR扩增与测序选取检测合格的基因组DNA进行PCR扩增,扩增引物参考文献[16],正反向序列为:CNCOI-F,5′-TCA ACAAATCATAAAGATATTGG-3′; CNCOI-R,5′-ACT TCTGGATGTCCAAAAAAVCA-3′,由上海生工生物工程有限公司合成。PCR反应条件:94℃ 2min;94℃ 30 s,50℃ 30 s,72℃ 1min,35个循环;72℃下再延伸10min,4℃下保存。反应体系采用25μL,含2.5 mmol/L dNTP 2μL、10×Buffer(含Mg2+) 2.5μL、2.5 mmol/L dNTPs 2μL、5μmol/L引物各1μL、模板DNA 1μL、500 u/L Taq DNA聚合酶0.2μL,ddH2O补足体积。扩增产物用10 mg/mL琼脂糖凝胶电泳检测,合格产物送至上海生工生物工程有限公司测序。
1.3.3数据分析
原始数据经人工校正,利用 Bio-Editor和DNAMAN软件对所测序列进行峰图人工校正和序列拼接后,与此同时,从GenBank中下载褐藻门9种马尾藻的线粒体COI基因序列(GenBank序列号及详细信息见表1),共16条序列,利用Mega 5.0进行多序列比对,并计算碱基组成及序列变异各项参数,其中包括保守位点(C)、变异位点(V)、简约信息位点(Pi)和单一突变位点(S),利用Mega 5.0中Cumpute codon usage Bias程序分析密码子使用频率,计算Kimura 2-parameter遗传距离;并以邻位连接(Neighbor-Joining,NJ)构建系统进化树,Bootstrap 置信值重复抽样1 000次,以最大似然法(ML) 估算转换颠换比R值。
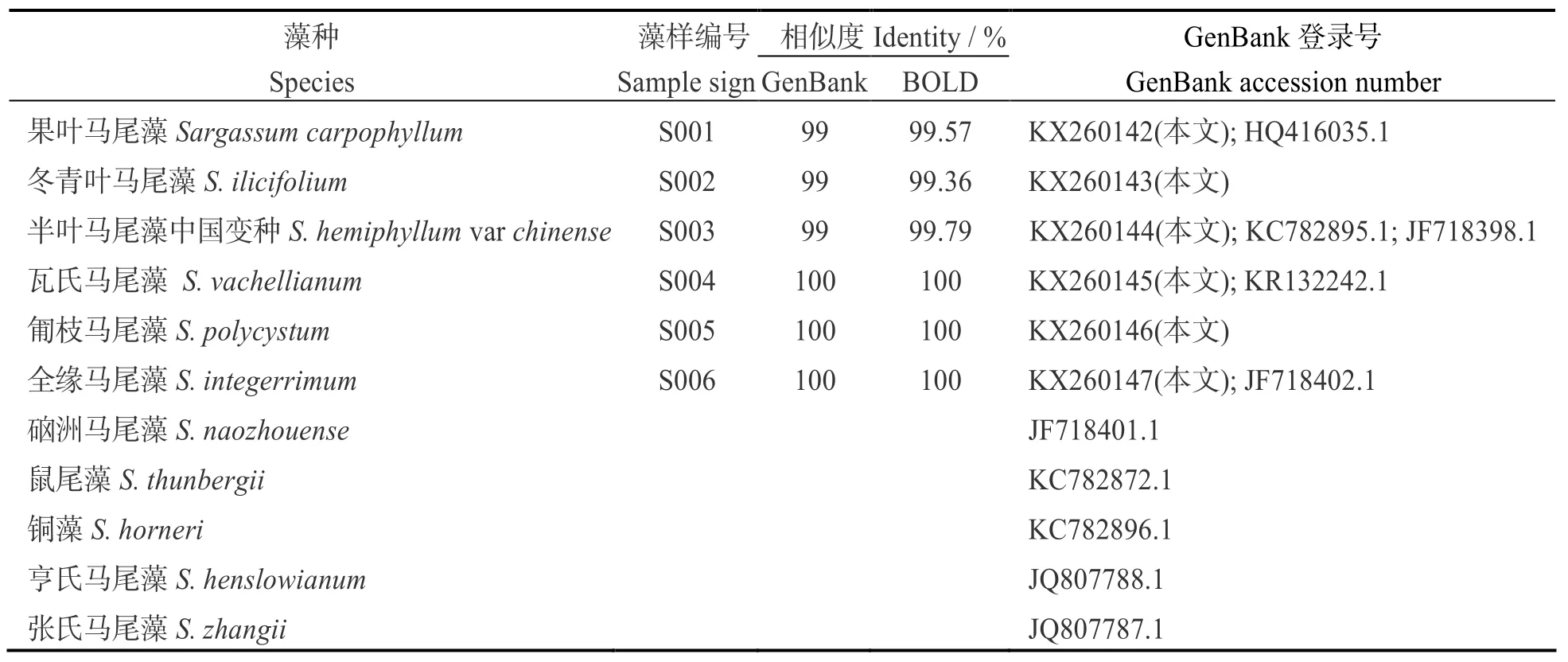
表1 本研究所涉及线粒体COI基因序列基本信息Table 1 Information about mtDNA-COI gene sequences
2 结果与分析
2.1形态学分类与分子鉴定
藻样形态学特征见表2,依据文献[1]鉴定为果叶马尾藻(Sargassum carpophyllum)、冬青叶马尾藻(S.ilicifolium)、半叶马尾藻中国变种(S.hemiphyllum var chinense)、瓦氏马尾藻(S.vachellianum)、匍枝马尾藻(S.polycystum)、全缘马尾藻(S.integerrimum)。其线粒体COI基因序列经人工矫正和剪切后长度为479 bp,与GenBank、BOLD数据库中已有数据比对发现,与其同源性最高的分别是果叶马尾藻、冬青叶马尾藻、半叶马尾藻中国变种、瓦氏马尾藻、匍枝马尾藻以及全缘马尾藻,相似度均大于或等于99%,与形态学鉴定结果一致。
2.2线粒体COI基因片段序列特征分析
多序列比对结果表明,6种马尾藻线粒体COI基因序列保守位点421个,变异位点58个,简约信息位点21个,单一突变位点37个,变异率7.7%。碱基组成分析揭示,T、C、A和G的平均含量分别为39.1%、18.9%、19.1%、22.9%,其中A+T含量(58.2%)明显高于C+G(41.8%)。碱基在密码子的第1、2、3位分布不均匀,具有明显偏倚性,其中碱基C偏向于密码子第1位(28.1%),其在第2、3位的含量显著降低,分别为9.8%和18.8%。密码子使用偏向性结果表明,UUU所编码的苯丙氨酸(F)使用频率最高,达12.5%。479 bp线粒体COI基因部分序列共编码159个氨基酸,且无插入、缺失及终止密码子。估算的转换颠换R比值为2.42,表明序列中转换明显多于颠换。
2.3遗传距离
11种马尾藻Kimura双参数进化模型的种间平均遗传距离见表3。结果显示,藻样S001、张氏马尾藻(S.zhangii)与铜藻(S.horneri)种间平均遗传距离最大,均为 0.112±0.016,表明三者亲缘关系最远,而样品S002和样品S006种间平均遗传距离最小(0.002±0.002),表明两者亲缘关系最近,多数种间平均遗传距离在 0.030±0.013~0.100± 0.016之间。可见,11种马尾藻种间遗传距离较小,这与11种马尾藻同属马尾藻属存在一定联系。
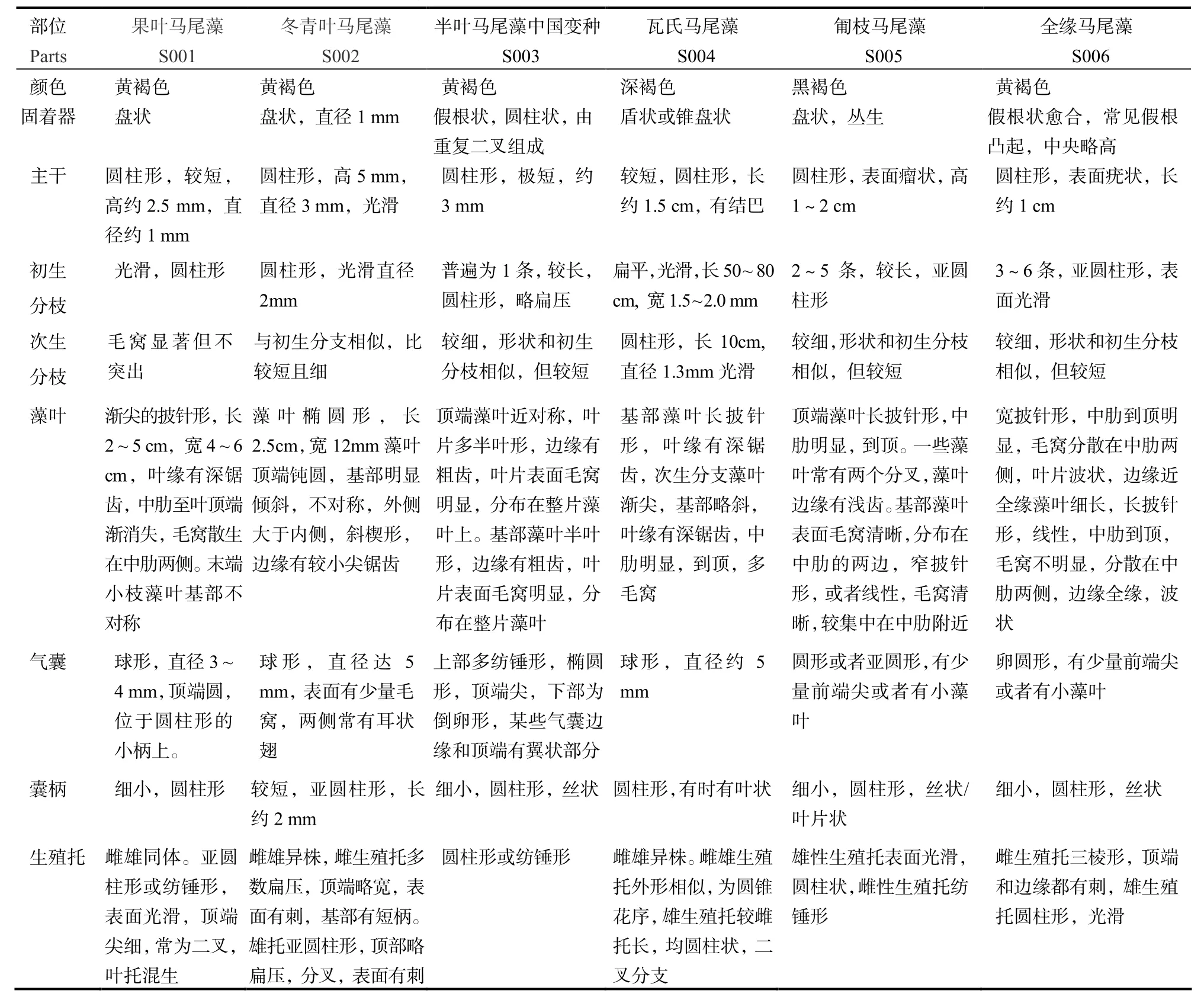
表2 6种马尾藻主要部位形态特征描述Table 2 Morphological description of the main parts of 6 species of Sargassum
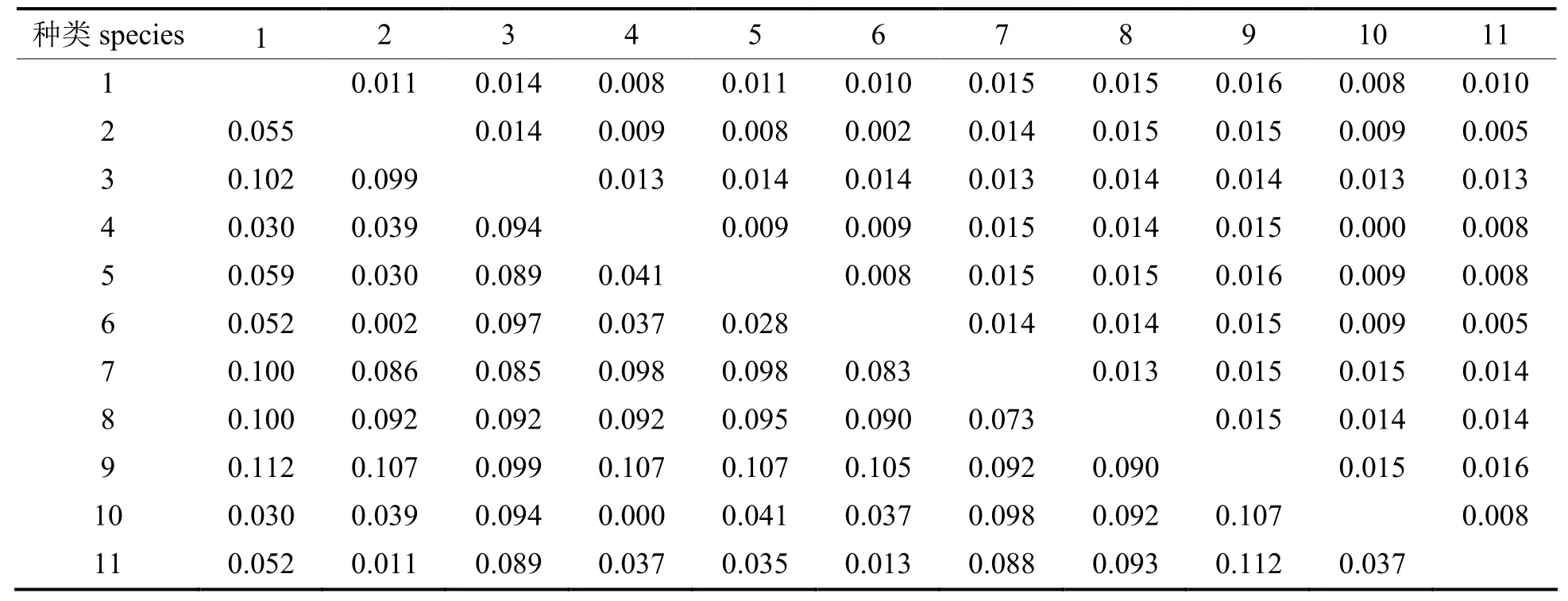
表3 基于线粒体COI基因序列11种马尾藻的16条序列种间遗传距离Table 3 Interspecific genetic distance based on 16 sequences of mtDNA-COI gene from 11 species of Sargassum
2.4聚类分析
采用邻接法(Neighbor-Joining,NJ),以海带目海带科的海带(Saccharina japonica,AB775228.2)为外群,构建11种马尾藻的系统发生树(图1)。由图1可见,马尾藻属的11个物种与外群分开,其中,藻样S004、S001、S003与GenBank数据库下载瓦氏马尾藻(S.vachellianum)、果叶马尾藻(S.carpophyllum)、半叶马尾藻中国变种(S.hemiphyllum var chinense)分别以高达99%、100%、100%置信值在发生树上各聚为一支,表明藻样S004、S001、S003与GenBank数据库下载数据为同种,与形态学、同源性分析结果保持一致,证实此三藻样分别为瓦氏马尾藻(S.vachellianum)、果叶马尾藻(S.carpophyllum)、半叶马尾藻中国变种(S.hemiphyllum var chinense),而藻样 S006与GenBank数据库下载全缘马尾藻(S.integerrimum)聚为一小支,且与冬青叶马尾藻(S.ilicifolium)聚为一大枝,表明三者亲缘关系较近,藻样 S005在发生树上单独聚为一支,与其他藻类相对较远。
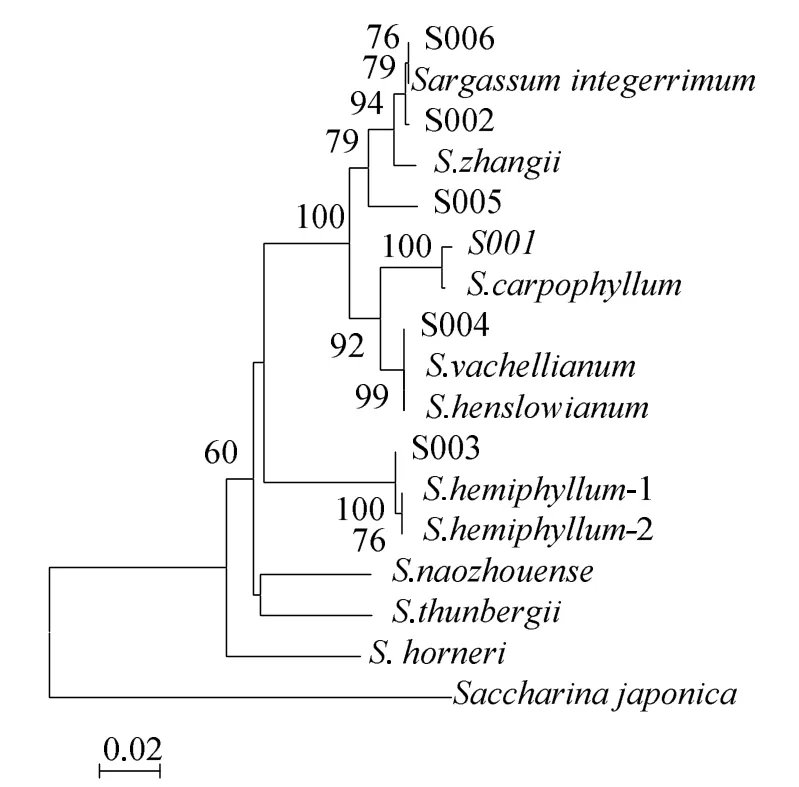
图1 11种马尾藻线粒体COI基因片段聚类分析Fig.1 Phylogenetic analysis of mtDNA-COI gene fragments of 11 species of Sargassum
3 讨 论
长期以来,复杂多样的海洋藻类分类问题一直都是困扰国内外藻类学家的一项重大难题,而其复杂的系统进化关系也成为学者们常关注的焦点。目前,对于马尾藻分类鉴定主要依据藻体颜色、固着器、主干、初次生分枝、藻叶、气囊和生殖托的形态以及毛窝是否存在等形态学特征,但这些性状受外界环境、物种性别以及发育阶段等因素影响变化较大,仅依靠形态特征对某些外观较为相似的马尾藻,很难对其进行准确区分,因此在进行藻类分类鉴定时利用DNA条形码技术辅助研究显得尤为重要[17-18]。线粒体COI基因是线粒体内13种氨基酸编码基因之一,在种间变异适宜,可提供比较丰富的变异位点,已渐渐被应用于马尾藻属等样品的物种鉴定及系统进化等研究[19-21]。本研究通过对马尾藻样品线粒体 COI基因中 479 bp片段并结合GenBank和BOLD数据库中已有数据进行同源性分析,利用 Kimura双参数进化模型的种间平均遗传距离,并以NJ法构建系统发生树,鉴定出所获取马尾藻样品分别为果叶马尾藻(S.carpophyllum)、冬青叶马尾藻(S.ilicifolium)、半叶马尾藻中国变种(S.hemiphyllum var chinense)、瓦氏马尾藻(S.vachellianum)、匍枝马尾藻(S.polycystum)以及全缘马尾藻(S.integerrimum),与形态结果一致。说明线粒体 COI基因用于马尾藻物种鉴定具有较高的可行性与高效性,通过传统形态学与DNA条形码技术相结合,可较好实现对马尾藻样本的准确鉴定。
经过十几年的发展,海藻分子系统学研究已建立了基本方法,在分类、系统进化和生物地理学研究方面取得了一定的研究成果。但是到目前为止,多数工作仍限于积累信息、利用孤立的分子证据构建局部的分子系统树,分子方法在藻类方面尚无统一的研究标准,而整体的系统发生关系研究仍然很模糊,仅是对一些物种的传统分类地位提出质疑和分类修订建议,此外,NCBI数据库里藻类的基因序列并不全面,缺乏很多藻类的基因数据,准确完整的基因序列还有待补充[23]。因此,藻类物种鉴定目前仍以形态学鉴定为主,相信随着对马尾藻分子标记研究的深入以及分子数据的补充与完善,将有助于促进马尾藻DNA条形码的建立,进而使物种繁多、形态多变的马尾藻物种鉴定和种间系统发生关系研究更加科学。
[1]曾呈奎,陆保仁.中国海藻志·第3卷·褐藻门[M].北京:科学出版社,2000:1-237.
[2]黄冰心,丁兰平,孙忠良,等.中国海洋褐藻门新分类系统[J].广西科学,2015,22(4):189-200.
[3]田淑娴,陈万东,林利,等.繁殖期半叶马尾藻中国变种的形态结构观察[J].海洋渔业,2014,36(2):107-298.
[4]孙修涛,王飞久,张立敬,等.鼠尾藻生殖托和气囊的形态结构观察[J].海洋水产研究,2007,28(3):125-131.
[5]赵素芬,孙会强,刘东超,等.雷州马尾藻Sargassum leizhouense (Fucales,Phaeophyta)形态结构及分类研究[J].海洋与湖沼,2014,45(2):291-115.
[6]潘金华,张全胜,许博.鼠尾藻有性繁殖和幼孢子体发育的形态学观察[J].水产科学,2007,26(11):589-592.
[7]赵素芬,刘丽丝,孙会强,等.湛江海域浒苔属Enteromorpha种类的形态与显微结构[J].广东海洋大学学报,2013,33(6):2-8.
[8]张凤英,马凌波,施兆鸿,等.3种鲳属鱼类线粒体COI基因序列变异及系统进化[J].中国水产科学,2008,15(3):392-399.
[9]董丽娜,黄梓荣,艾红,等.北部湾3种金线鱼属鱼类COI基因序列的比较分析[J].中国水产科学,2011,18(3):508-514.
[10]HEBERT P D,PENTON E H,BURNS J M,et al.Ten species in one:DNA barcoding reveals cryptic species in the neotropical skipper butterfly astraptes fulgerator [J].Proceedings of the National Academy of Sciences of the United States of America,2004,101(41):14 812 - 14 817.
[11]PARK H Y,YOO H S,JUNG G,et al.New DNA barcodes for identification of Korean birds [J].Genes& Genomics,2011,33(2):91-95.
[12]WARD R D,ZEMLAK T S,INNES B H,et al.DNA barcoding Australia's fish species[J].Philosophical Transactions of the Royal Society B:Biological Sciences,2005,360(1462):1847-1857.
[13]SAUNDERS G W.Applying DNA barcoding to red macroalgae a preliminary appraisal holds promise for future applications[J].Philosophical Transaction of the Royal Society B,2005,360:1879-1888.
[14]EVANS K M,WORTLEY A H,MANN D G.An assessment of potential diatom“Barcode” Genes (COI,rbc L,18S and ITS r DNA) and their effectiveness in determining relationships in Sellaphora (Bacillariophyta)[J].Protist,2007,158(3):349-364.
[15]MATTIO L,PAYRI C.Assessment of five markers as potential barcodes for identifying Sargassum subgenus Sargassum species (Phaeophyceae,Fucales) [J].Cryptogamie Algologie,2010,31(4):467-485.
[16]GARY W S.Applying DNA barcoding to red macroalgae:a preliminary appraisal holds promise for future applications[J].The Royal Society,2005,360(1462):1879-1888.
[17]陈军,王寅初,余秋瑢,等.绿潮暴发期间我国青岛漂浮铜藻的分子鉴定[J].生物学杂志,2016,33(1):39-42.
[18]刘鑫鑫,朱军,黄惠琴,等.海南2种马尾藻的分类鉴定[J].热带生物学报,2014,5 (4):357-367.
[19]MC DEVIT D C,SAUNDERS G W.On the utility of DNA barcoding for species differentiation among brown macroalgae (Phaeophyceae) including a novel extraction protocol[J].Phycological Research,2009,57(2):131-14.
[20]CLARKSTON B E,SAUNDERS G W.A comparison of two DNA barcode markers for species discrimination in the red algal family Kallymeniaceae (Gigartinales,Florideophyceae),with a description of Euthora timburtonii sp.nov.[J].Botany,2010,88(2):119-131.
[21]MC DEVIT D C,SAUNDERS G W.A DNA barcode examination of the Laminariaceae (Phaeophyceae) in Canada reveals novel biogeographical and evolutionary insights[J].Phycologia,2010,49(3):235-248.
[22]郜星晨.基于线粒体COI序列的中国沿海鲀形目鱼类DNA条形码研究[D].广州:暨南大学,2015.
[23]徐涤,秦松.海洋褐藻分子系统学研究进展[J].海洋科学,2002,26(2):19-22.
(责任编辑:刘庆颖)
DNA Barcoding and Phylogenetic Analysis of Common Species of Sargassum from Leizhou Peninsula Based on mtDNA-COI Gene Sequences
HE Liang,LIAO Jian,QUAN Qiu-mei,LIANG Zhong,XIE En-yi,LIU Li,LIU Chu-wu
(Key Laboratory of Aquaculture in South China Sea for Aquatic Economic Animal of Guangdong Higher Education Institutes,Fisheries College,Guangdong Ocean University,Zhanjiang 524025,China)
Species identification and phylogenetic relationship in 6 common species of Sargassum collected from Leizhou Peninsula were analyzed by appling DNA barcoding technology assisted traditional morphological classification method.Mt-DNA COI gene sequences of 6 species of Sargassum and existing data in GenBank and BOLD database for homology analysis showed that the similarity is 99% or more,the molecular data were consistent with the morphological identification results.In addition,the analyses on characteristics of COI gene sequence shows that COI gene sequence in 6 species Sargassum including 421 conserved sites,58 variable sites,21 Parsim-Informative sites,and 37 Singleton sites,accounted for 7.7%.Nucleartides composition results showed:T,C,A,G average contents were 39.1%,18.9%,19.1%,22.9%,respectively,and A+T content (58.2%) was significantly higher than C+G content (41.8%).The highest frequency of amino acid was Phenylalanine(F) which coded by UUU(12.5%).The rate of transition and transversion based on maximum likelihoodwas 2.42.Interspecific genetic distances were from 0.001 ± 0.001 to 0.113 ± 0.016.The result of cluster analysis was consistent with that of morphological identification and homology analysis.
Sargassum; mtDNA-COI; species identification; molecular phylogenetic; DNA barcoding
Q78;Q949
A
1673-9159(2016)04-0017-06
10.3969/j.issn.1673-9159.2016.04.004
2016-06-07
2016年度广东大学生科技创新培育专项资金立项项目(pdjh2016b0236);中央分成海域使用金支出项目(2011-2-2-09-3);广东省海洋渔业科技推广专项(A201308E02);广东省科技计划项目(2012B020307006)
贺亮(1990-),男,硕士研究生,从事海洋生境修复与生物资源保护研究。E-mail:gdhydxhl1990@foxmail.com
刘丽(1974-),女,教授,主要从事海洋生物保护及遗传多样性研究。E-mail:zjouliuli@163.com
