马氏珠母贝养殖群体早期生长阶段的遗传多样性与有效亲本数量估计
2016-11-12李俊辉刘青云杜晓东邓岳文王庆恒
李俊辉,刘青云,,杜晓东,,邓岳文,,王庆恒,
(1.广东海洋大学水产学院,广东 湛江 524088;2.广东省珍珠养殖与加工工程技术研究中心,广东 湛江 524088;3.广西水产科学研究院,广西 南宁530021)
马氏珠母贝养殖群体早期生长阶段的遗传多样性与有效亲本数量估计
李俊辉1,刘青云1,3,杜晓东1,2,邓岳文1,2,王庆恒1,2
(1.广东海洋大学水产学院,广东 湛江 524088;2.广东省珍珠养殖与加工工程技术研究中心,广东 湛江 524088;3.广西水产科学研究院,广西 南宁530021)
从大亚湾野生群体选择亲本繁育1个养殖群体,受精第6天、23天与80天分别进行优选,按50%选留比率选留快生长个体,分别在受精后第3天、30天120天取样,利用8对SSR标记分析养殖群体遗传多样性与估计有效亲本数量。结果表明,在3个生长阶段每个位点的平均等位基因数分别为3.750、3.875和3.625,平均观察杂合度分别为0.324 2、0.527 3和0.372 4,平均期望杂合度分别为0.551 1、0.561 3和0.504 8。有效亲本数量的估计分别为38、28和30,3个生长阶段估算的有效亲本数量均比实际所用到的亲本数量少。选优管养措施导致养殖群体的遗传多样性与有效亲本数量降低。
马氏珠母贝;遗传多样性;有效群体数量;SSR标记
有效群体数量是指群体内所具有的相当于理想群体(含量在世代间保持恒定、群体内随机交配、无世代重叠、无其它遗传效应影响的群体)繁殖个体的数目[1-2]。目前,有效群体数量是具有相同遗传变异损失速率和近亲繁殖速率的理想种群大小,即真实种群的有效种群大小。苗种培育过程中,如果亲本数量偏少,导致子代群体的有效群体数量减少,因多代累积导致的近交效应,致使子代养殖的性状退化。对于怀卵量高的养殖动物,在育苗过程中若常使用较少数量亲本繁育苗种,并且使用性别比例不平衡的亲本等,将引起对子代群体的有效亲本及遗传多样性的不利影响,这已经在很多水产养殖物种的研究中得到了证明[3-4]。此外,高产卵量的养殖动物早期发育过程中死亡率也会较高,这样也会导致子代群体的有效亲本数量出现明显的变化[5]。
马氏珠母贝(Pinctada fucata martensii)是我国南方重要的经济养殖贝类,主要用于培育海水有核珍珠。近几年马氏珠母贝珍珠产量与质量均明显下滑,主要原因在于养殖群体生长与育珠性状退化与海区环境的污染,其中养殖群体生长与育珠性状退化可能与其遗传多样性的降低有关。由于成熟的马氏珠母贝雌性个体的产卵量高,为节约成本,在育苗过程中常利用少数量亲本繁育子代群体,由此因少数量亲本引起的瓶颈效应导致养殖群体遗传多样性降低,同时,在幼体培育与海区养殖过程中常进行多次选优淘汰生长较慢的个体,这种管养措施对马氏珠母贝的养殖群体的遗传多样性有什么影响?本论文通过建立1个养殖群体,在幼体期、变态期与稚贝期分别进行选优,利用SSR标记分析选优管养措施对养殖群体遗传多样性与有效亲本数量的影响,以期望为遗传育种提供参考。
1 材料与方法
1.1养殖群体繁育与取样
2009年5月从大亚湾收集野生马氏珠母贝群体,运回湛江承梧海区养殖。2012年4月,挑选性腺成熟的个体作为繁育亲本,其中雌、雄亲本分别为30、20只,解剖性腺,人工授精。在幼体期(第6天)、变态期(第23天)与稚贝期(第80天)分别进行选优,选留生长最快的个体,每次优选比例均约为50%,按照生产常见技术进行幼体培育与稚贝海区养殖。
在幼体期、变态附着期与稚贝期共3个生长阶段进行取样,取样时间点设为受精后第3、30和120天,利用75%(φ)酒精溶液固定样品,4℃保存。
1.2DNA提取以及SSR分析
参照文献[6]利用吐温法提取第3天的样品DNA,对第30天的样品先将其外壳剥离,再用吐温法进行DNA的提取,两个取样时间点样品数量均为96个。吐温法提取 DNA的过程:在 1.5 mL离心管加入15μL裂解液(10 mmol Tris-HCl,50 mmol KCl,0.5%Tween-20,0.3 mg/mL蛋白酶K,pH=8.0),轻微离心;在55℃恒温裂解3h,85℃15min灭活蛋白酶K;3000r/min离心5min,保留上清液备用。
稚贝 DNA的提取采用上海生工生物工程公司的Universal Genomic DNA Mini-Isolation Kit,参照试剂盒说明书进行,样品数量均为96个。
选用本实验室开发的8对SSR引物,引物的详细信息见表 1。PCR扩增反应体系和条件见刘青云(2013)[7],具体如下:PCR反应体系15 µL,包括:DNA 2μL,10×PCR buffer(10 mM)1.5μL,dNTPs(4 mM)1μL,MgCl2(2.5 mM)1.5μL,Taq polymerse(2.5u/mol)1μL,引物1μL,ddH2O 7μL,然后根据各对引物条件的不同进行PCR扩增,反应程序为:94℃预变性5min,94℃变性42 s,相应退火温度下退火50 s,72℃延伸45 s,30个循环,72℃延伸5min。
1.3数据统计
利用Popgene32计算等位基因频率、有效等位基因数(ne)、等位基因数(na)、观察杂合度(Ho)以及期望杂合度(He);根据各位点的等位基因频率通过PIC-CALC进行计算每个位点的多态信息含量(PIC)。
利用COLONY2.0软件的同胞重建法估算3个不同生长阶段养殖群体的有效亲本数量。COLONY的原理是根据孟德尔遗传法则运用最大似然法将子代划分为全同胞和半同胞,并推断亲本的基因型。根据公式ΔF=1/2Ne计算近交率。
2 结果与分析
2.13个生长阶段养殖群体遗传多样性
利用8对SSR引物对3个生长阶段养殖群体进行遗传多样性分析。结果表明,在第3、30和120天分别检测到30、31和29等位基因。各生长阶段养殖群体各位点等位基因频率见表 2。不同取样点3M-10位点的扩增图谱见图1。
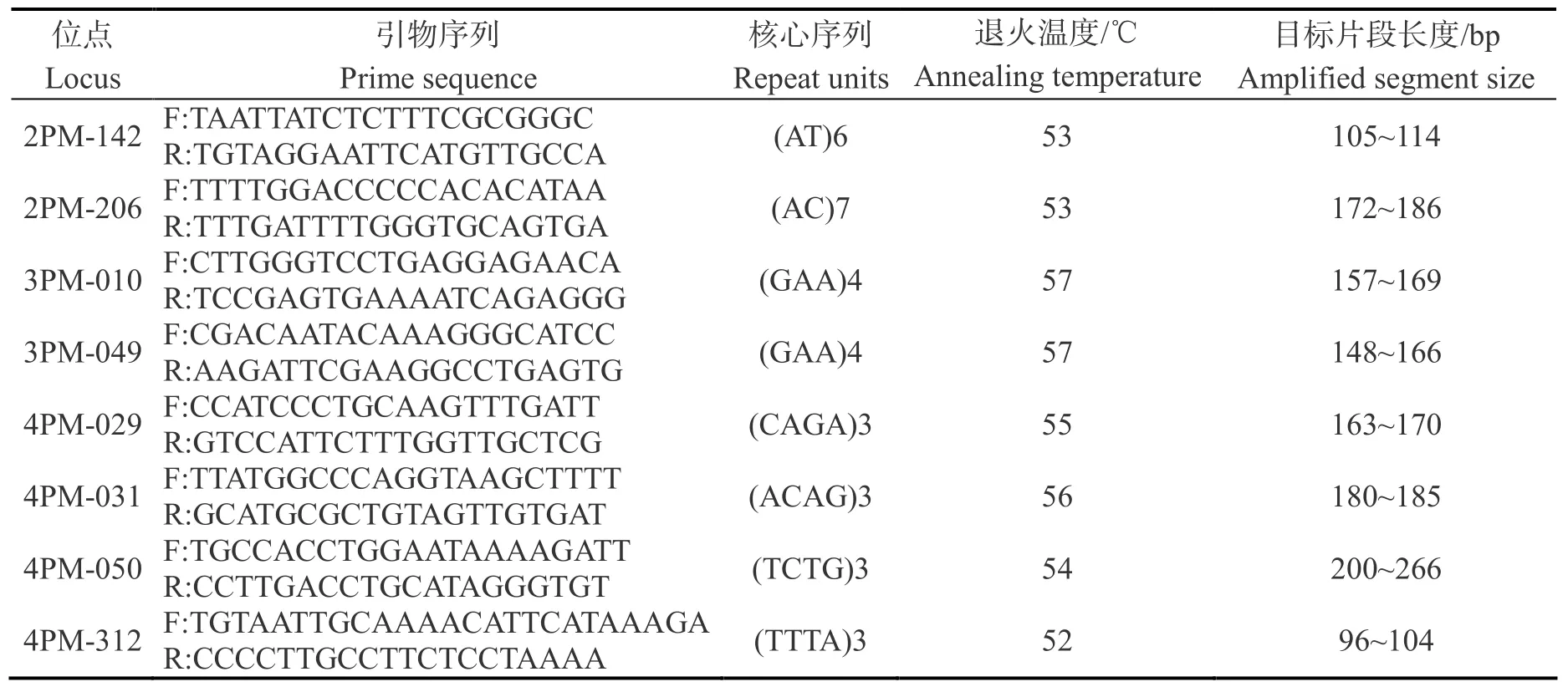
表1 微卫星引物序列及退火温度Table 1 Primer sequence and annealing temperature of microsatellite DNA

表2 不同生长阶段养殖群体各位点等位基因频率Table 2 Allele frequency in the loci in the stock of pearl oyster Pinctada fucata martensii at three different growth stages
3个生长阶段养殖群体的平均观察杂合度分别为 0.3242、0.5273与 0.3724,期望杂合度分别为0.5511、0.5613与0.5048;3个阶段养殖群体平均多态信息含量分别为0.4770、0.4846与0.4445。

表3 马氏珠母贝养殖群体3个生长阶段遗传多样性的比较Table 3 Genetic variability of the stock of pearl oyster Pinctada fucata martensii at three different growth stages

图1 3PM-10引物在第120天样品的扩增图谱Fig.1 The silver staining PAGE image of locus 3PM-10 amplified by the samples at days 120 after fertilization
2.2养殖群体3个生长阶段有效亲本数量估计
根据8个SSR位点在3个生长阶段各96个个体的基因型数据,进行同胞重建的分析;将第3天的样品划分为 25对全同胞,426对半同胞;将第30天样品划分为31对全同胞,594对半同胞;将第120天样品划分为16对全同胞,578对半同胞。有效亲本数量在 3个生长阶段分别推算为 38、28和30,分别占实际亲本比例为76%,56%和60%。根据公式ΔF=1/2Ne算出每个阶段的近交率分别为0.013、0.018和0.017(表4)。
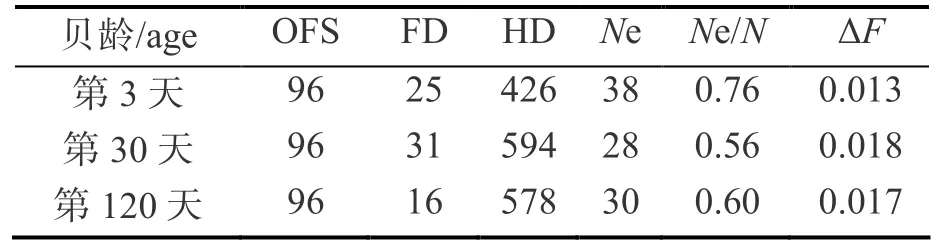
表4 马氏珠母贝养殖群体3个生长阶段幼虫的同胞重建信息Table 4 Sib identification in the stock of pearl oyster Pinctada fucata martensii at three different growth stages
3 讨论
本研究中采用的SSR标记均为多态性型标记,在3个不同生长阶段都能检测到较高的多态信息含量(PIC)。依据Botstein等[8]的研究,当PIC>0.5时,表示该位点为高度多态性位点;当 0.25<PIC<0.5时,表示该位点为中度多态性位点;当 PIC<0.25时,表示该位点为低度多态性位点。在第1个取样时间点,8个SSR包含4个高度、3个中度与1个低度多态性位点;在第2个取样时间点,8个SSR位点包含5个高度与3个中度多态性位点;在第3个取样时间点,8个SSR位点包含中3个高度、4个中度与1个低度多态性位点;3 个阶段养殖群体平均多态信息含量分别为 0.477 0、0.484 6与0.444 5(表3),表明本研究中所用的8个微卫星位点均包含较高的信息含量,多态性较好。
杂合度是评价遗传多样性常用指标,若群体的杂合度越高,说明群体的遗传多样性越丰富。现有研究表明,利用SSR标记分析马氏珠母贝野生群体与养殖群体的观测杂合度为为0.15~0.72,期望杂合度为0.38~0.75[9-13]。例如,赵晓霞等[10]利用SSR标记分析了利用不同数量亲本繁育的4个养殖群体的遗传多样性,结果表明这4个养殖群体的观测杂合度为0.473 7~0.714 3,期望杂合度为0.527 9~0.592 6;王学颖等[12]利用SSR标记马氏珠母贝金黄壳色第3代选育群体与基础群体的遗传多样性,结果表明选育群体与基础群体平均观测杂合度分别为 0.411 9和 0.440 3,平均期望杂合度分别为 0.533 3 和0.546 4。本研究结果表明,3个不同生长阶段养殖群体的观测杂合度与期望杂合度分别为 0.324 3 ~0.527 3和0.504 8~0.561 3,与上述研究比较,本实验群体具有中等大小的遗传多样性。
本研究结果表明,不同生长阶段养殖群体的遗传多样性出现了明显变化。从第3天到第30天平均观察杂合度从0.324 2增加到了0.527 3,这说明了到第 30天养殖群体的遗传多样性有所提高,这个可能与生长早期部分幼虫具有引起适合度降低隐性基因有关,具有这些基因的个体容易死亡,从而使得第 30天养殖群体杂合个体数量所占的比例有所增加。关于纯合致死现象已经在其他贝类有过相关的报道[14]。
在亲本的基因型信息未知情况下的家系分析中,基于分子标记的同胞重建法获得了广泛的应用[5,15]。在应用分子标记进行有效亲本数量估算时,为了提高准确率,常使用很多种方法联合估算,然而基于家系分析的对有效亲本数量的估算被当作是最准确的一种方法[16-17]。本研究通过对马氏珠母贝3个生长阶段养殖群体进行同胞重建分析,估算了马氏珠母贝有效亲本数量,第3天养殖群体有效亲本数量为38个,占到实际亲本数量的76%,亲本数量已经出现了明显下降,第30天与120天养殖群体的有效亲本数量分别为28和30,分别占实际亲本数量的56%和60%。第3天养殖群体有效群体亲本数量变小可能与人工繁殖所用到的亲本性别比例不平衡有关,本研究中雌雄亲本数量不等比例,分别为30与20;在第30天与120天养殖群体有效亲本数量的减少可能是跟养殖方式有关,在幼体培育与稚贝培育过程中共进行了3次选优,即淘汰生长缓慢的个体,留下快速生长的个体。已有文献报道了养殖过程中的优选操作对养殖群体的遗传多样性的影响,并表明附着较慢的幼虫对亲本繁殖成功变异的缩小具有很重要的作用,因而那些在分级筛选中剔除掉的附着较慢的幼虫使得亲本繁殖成功出现偏差,从而对有效亲本数量产生影响[18]。
群体选择是贝类遗传育种常见的技术手段,其核心问题是选育群体的有效亲本数量。在群体选择过程中,如果繁育子代群体的亲本数量偏少,导致子代群体近交水平增加,影响选育进展[19]。子代群体有效亲本数量的影响因素包括产卵方式、性别比例与管养措施等方面。本研究结果表明,在苗种培育与海区养殖过程中选优的管养模式会导致子代有效亲本数量减少;同时,研究也表明利用SSR标记能有效估计子代群体有效亲本数量,这与笔者的前期结果[20]相一致。
[1]WRIGHT S.Evolution in mendelian populations[J].Genetics,1931,16(2):97-159.
[2]WRIGHT S.Size of population and breeding structure in relation to evolution[J].Science,1938,87:430-431.
[3]TARIS N,ERNANDE B,MCCOMBIE H,et al.Phenotypic and genetic consequences of size selection at the larval stage in the Pacific oyster(Crassostrea gigas)[J].Journal of Experimental Marine Biology and Ecology,2006,333(1):147-158.
[4]LIND C E,EVANS B S,KNAUER J,et al.Decreased genetic diversity and a reduced effective population size in cultured silver-lipped pearl oysters(Pinctada maxima)[J].Aquaculture ,2009,286(1-2):12-19.
[5]LALLIAS D,TARIS N,BOUDRY P,et al.Variance in the reproductive success of flat oyster Ostrea edulis L.assessed by parentage analyses in natural and experimental conditions[J].Genetics Research,2010,92(3):175-187.
[6]刘广绪,包振民,胡景杰,等.栉孔扇贝、华贵栉孔扇贝及其种间杂交子代、种内交配子代的ISSR分析[J].中国海洋大学学报,2006,31(1):71-75.
[7]刘青云.2013.马氏珠母贝群体有效亲本数量估计与近交系遗传分析[D].湛江:广东海洋大学.
[8]BOTSTEIN D,WHITER R L,SKOLNICK M,et al.Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J].American Journal of Human Genetics,1980,32(3):314.
[9]闫学春,佟广香,匡友谊,等.两个马氏珠母贝养殖群体遗传多样性微卫星分析[J].水产学杂志,2009,22(1):5-9.
[10]赵晓霞,邓岳文,杜晓东,等.利用微卫星标记分析马氏珠母贝 4 个养殖群体遗传结构[J].基因组学与应用生物学,2010,29(5):879-884.
[11]王爱民,王嫣,顾志峰,等.马氏珠母贝2个地理群体杂交子代的杂种优势和遗传变异[J].海洋与湖沼,2010,41(1):140-147.
[12]王学颖,高远镇,杜晓东,等.马氏珠母贝金黄壳色系F3和基础群体遗传结构比较[J].海洋通报,2012,31(3):324-328.
[13]张嘉丽,罗燕秋,黄怡,等.马氏珠母贝近交与杂交家系的遗传结构比较[J].基因组学与应用生物学,2015,34(4):723-730.
[14]LAUNEY S,HEDGECOCK D.High genetic load in the Pacific oyster Crassostrea gigas[J].Genetics,2001,159(1):255-265.
[15]KANNO Y,VOKOUN J C,LETCHER B H.Sibship reconstruction for inferring mating systems,dispersal and effective population size in headwater brook trout(Salvelinus fontinalis)populations[J].Conservation Genetics,2011,12(3):619-628.
[16]CERVANTES I,PASTOR J M,GUTIÉRREZ J P,et al.Computing effective population size from molecular data:The case of three rare Spanish ruminant populations[J].Livestock Science,2011,138(1):202-206.
[17]LIU QY,CHEN WY,LIU JL,et al.Genetic variation and an estimation of effective population size in the pearl oyster Pinctada martensii[J].Biochemical Systematics and Ecology,2014,54:53-58.
[18]LAING I,EARl N H.The lipid content,spatfall and subsequent growth of early and late settling hatchery-reared Pacific oyster,Crassostrea gigas Thunberg,larvae[J].Aquaculture Research,1998,29(1):19-25.
[19]邓岳文,符韶,杜晓东,等.马氏珠母贝选系 F2早期选择反应和现实遗传力估计[J].广东海洋大学学报,2008,28(4):26-29.
[20]高远镇,王学颖,卢婧,等.马氏珠母贝选系F4遗传结构和亲缘关系建分析[J].中国农学通报,2012,28(32):100-106.
(责任编辑:陈庄)
Genetic Diversity and Effective Population Size Estimate in the Stock of Pearl Oyster Pinctada fucata martensii at Early Growth Stages
LI Jun-hui1,LIU Qing-yun1,3,DU Xiao-dong1,2,DENG Yue-wen1,2,WANG Qing-heng1,2
(1.Fisheries College,Guangdong Ocean University,Zhanjiang 524088,China;2.Pearl Breeding and Processing Engineering Technology Research Center of Guangdong Province,Zhanjiang 524088,China;3.Guangxi Academy of Fishery Sciences,Nanning 530021,China)
In this paper the effects of selection for faster growth on genetic diversity and effective population size in the stock of pearl oyster Pinctada fucata martensii is reported.In May of 2012,a stock was established by the breeders collected from Daya Bay wild population.The female and male breeder numbers were 30 and 20,respectively.Selection practices were performed at days 6,23 and 80 after fertilization.The selection percentage was 50% at each time.Animals were sampled at days 3,30 and 120 after fertilization.Genetic diversity and effective population size were analyzed by using eight SSR primer pairs.The results showed that the average numbers of alleles at each sampling time were 3.750,3.875 and 3.625,respectively.The average observed heterozygosities in the stock at each sampling time were 0.324 2,0.527 3 and 0.372 4,respectively.The average expected heterozygosities in the stock at each sampling time were 0.551 1,0.561 3 and 0.504 8,respectively.Effective population sizes at each sampling time were 38,28 and 30,respectively.A decrease in effective population size at each sampling time relative to the number of breeders used to develop the stock was detected.Thepresent results suggested that selection practices could result in decreases in genetic diversity and effective population size in the stock of pearl oyster P.fucata martensii.
Pinctada fucata martensii;Genetic diversity;Effective population size;SSR marker
S917.4
A
1673-9159(2016)04-00012-05
10.3969/j.issn.1673-9159.2016.04.003
2016-03-31
广东省海洋与渔业局科技专项 (A201308A10,Z2014009) ;广东省科技专项(2015A030302079)
李俊辉(1976—),女,讲师,专业方向为无脊椎动物增养殖。Email:lijunh@21cn.com
杜晓东(1962—),男,教授,专业方向为珍珠贝基础生物学。Email:zjdxd@21cn.com
