木麻黄EST序列中SSR的分布特征及有效SSR标记的筛选
2016-11-10仲崇禄姜清彬
胡 盼 仲崇禄 张 勇 姜清彬 陈 羽 陈 珍 韩 强
Khongsak Pinyopusarerk2 Didier Bogusz3
(1.中国林业科学研究院热带林业研究所,广州 510520; 2.澳大利亚联邦科学与工业研究组织植物所,阿克顿 2600,澳大利亚; 3.法国发展研究院(IRD),蒙彼利埃,BP 64501,法国)
木麻黄EST序列中SSR的分布特征及有效SSR标记的筛选
胡 盼1仲崇禄1张 勇1姜清彬1陈 羽1陈 珍1韩 强1
Khongsak Pinyopusarerk2Didier Bogusz3
(1.中国林业科学研究院热带林业研究所,广州 510520;2.澳大利亚联邦科学与工业研究组织植物所,阿克顿 2600,澳大利亚;3.法国发展研究院(IRD),蒙彼利埃,BP 64501,法国)
从NCBI的EST数据库中获得的木麻黄EST序列共有34 752条,进行拼接后得到全长7 278.578 kb的非冗余序列(Unigene)12 062条,并从中检索得到分布于353条Unigene的367个SSR位点,SSR检出率为2.93%,平均分布距离为19.83 kb,包括39种重复基序类型。其中,以二核苷酸和三核苷酸为主要类型,在总SSRs中所占比例分别为57.77%和34.60%;而二核苷酸重复基序中,AG/CT所占比例最高,为93.87%;在三核苷酸重复基序中AAG/CTT所占比例最高,为44.09%。对检索出的EST-SSR位点设计得到97对引物,其中32对为可有效扩增引物。Blastx分析发现77.3%的含SSR位点的EST序列与非冗余蛋白序列数据库中功能序列具有同源性,而功能已知的序列中葡萄来源的序列占有最大比例(10.4%)。GO功能分类发现,含有SSR位点的EST序列中有47.3%至少具有1个GO注释,归入细胞组分的序列最多,而其中细胞质和细胞核的功能项所占比例较大。
木麻黄;EST;SSR
SSR(Simple Sequence Repeat,SSR)是由2~6个核苷酸为重复单位组成的串联重复序列。大量研究表明,SSR广泛存在于各类物种中,且具有极高的多态性,是许多遗传学研究如遗传多样性研究[1~4]、构建连锁图谱[5~7]、基因图位克隆[8~9],以及进化学研究[10~12]中广泛应用并极为有效的标记。然而,传统的利用基因组DNA文库中的同源DNA片段开发SSR标记的过程极为耗时和昂贵,所以迫切需要更有效、经济的开发SSR标记的方法。自人类基因组计划启动以来,公共数据库中表达序列标签(Expressed Sequence Tags,EST)成百上千倍的增长。这些庞大的EST数据成为了开发SSR标记的新的来源。EST-SSR标记在大量作物研究中发挥了巨大的作用,如拟南芥(Arabidopsisthaliana),玉米(Zeamays),水稻(Oryzasativa)和小麦(Triticumaestivum)[13~15]。林木遗传育种研究中,利用EST-SSR标记进行重要的林木经济性状,如生长、材性和抗病性等的QTL定位和选择也越来越受到人们重视。在广泛引种的桉树遗传图谱上就定位了大量的数量性状位点,如生长[16~17]、材性[17]和耐寒性[18]。
木麻黄科(Casuarinaceae)植物包括4属,由86个种和13个亚种组成,为常绿乔木或灌木,天然分布于大洋洲、东南亚和天平洋群岛[19]。该树种具有速生、抗风沙、耐干旱和耐贫瘠等特点,被广泛引种至热带和亚热带沿海地区,在沿海防护林生态体系建设中发挥着重要作用[20]。目前利用等位酶技术[21]、RAPD[22~23]、AFLP[24~25]和ISSR[26]等分子标记在木麻黄科植物中开展的研究多集中于遗传多样性和遗传结构的分析,本研究通过对公共数据库NCBI中的木麻黄科植物的EST序列进行拼接和注释,发掘有效的SSR标记,以期为木麻黄遗传多样性分析和重要生态性状的定位分析提供参考。
1 材料与方法
1.1 试验材料
以中国林科院热带林业研究所(广州)苗圃内2年生木麻黄为材料,采集12个单株的新鲜小枝,根据改良的CTAB法提取基因组DNA[27]。
1.2 木麻黄EST序列的来源及下载
登陆美国国立生物信息中心网站(http://www.ncbi.nlm.nih,gov/),在dbEST数据库中,采用Entrez检索系统,输入词条“Casuarina”来搜索木麻黄的EST序列。在木麻黄科目录下共有2种,其中包括粗枝木麻黄Casuarinaglauce(34 700),短枝木麻黄Casuarinaequisetifoliasubsp.equisetifolia(52)。截止2013年12月31日,木麻黄EST序列总数为34 752条。获得搜索结果后,将所有序列以FASTEN格式下载到本地计算机。
1.3 木麻黄EST序列的预处理
从公共数据库中直接获得的EST序列包含有一些低质量的较短片段(<100 bp)和带有少量载体序列及末端带有polyA/T尾的序列,这些均对相关信息的分析有较大影响,因此,需进行EST序列的预处理。首先,利用est_trimmer.pl(http://pgrc.ipk-gatersleben.de/misa/download/est_trimmer.pl)软件去除5′端或3′端50 bp的polyA或polyT,同时去除过短(<100 bp)的序列,过长(>700 bp)的EST序列则保留其5′端700 bp;然后,利用seq_trim软件以通用载体序列为参考序列,去除载体系列[28];最后,利用Phrap软件对EST序列进行聚类拼接,以去除冗余序列,拼接标准为:最小匹配碱基数(minmatch)为20,最小分值(minscore)为40[29~30]。
1.4 木麻黄EST-SSR位点的搜索
利用在线软件SSRIT(Simple Sequence Repeat Identification Tool,SSRIT)(http://www.gramene.org/db/searches/ssrtool)对聚类后的非冗余EST序列进行SSR搜索。搜索条件为:二核苷酸的最少重复次数为9,三核苷酸的最少重复次数为6,四、五和六核苷酸的最少重复次数为5。
1.5 木麻黄EST序列比对和注释
利用程序Blast2GO(https://www.blast2go.com/)对含有SSR位点的木麻黄EST序列与NCBI数据库中的非冗余蛋白数据库(non-redundant)进行同源性比对,参数设置为:e-value=10-3,hits=20,HSP length cutoff=33[31]。利用Blast2GO对比对结果进行mapping映射获得GO terms,然后从细胞组分、分子功能、生物过程3个方面进行序列的功能注释和分类。
1.6EST-SSR引物设计和有效EST-SSR标记的验证
根据SSR位点两侧的序列,用Primer Premier5.0软件来设计引物,引物长度一般为18~24个核苷酸,GC含量在40%~60%,退火温度为50~65℃,正向引物和反向引物的退火温度相差在2℃以内,预期扩增片段长度在100~500 bp。
试验所有引物的合成由北京华大基因完成。引物筛选PCR反应体系为10 μL,即:1×buffer(100 mmol·L-1Tris-HCl pH9.0,80 mmol·L-1(NH4)2SO4,100 mmol·L-1KCl,0.5% NP-40),2.0 mmol·L-1MgCl2,200 μmol·L-1dNTP,0.5 μmol·L-1正向引物,0.5 μmol·L-1反向引物,1 U Taq DNA聚合酶,0.5 μmol·L-1模板DNA;扩增程序为:94℃预变性5 min,接以94℃ 30 s,退火温度30 s,72℃ 1 min,循环数为35次,最后在72℃补平10 min,4℃保温24 h;PCR扩增产物经2%琼脂糖凝胶于120 v,30 min电泳检测片段是否存在单一明亮条带,有单一明亮条带的即为可有效扩增的ESR-SSR标记。
2 结果与分析
2.1 木麻黄EST序列的冗余性
从NCBI的dbEST数据库获得的木麻黄EST序列共有34 752条(截止2013年12月31日)。首先,用est_trimmer.pl软件和进行seq_trim软件进行序列预处理,去掉5′端或3′端50 bp的polyA或polyT;然后,运用Pharp软件进行序列聚类拼接,去除冗余序列;最后,共获得12 062条非冗余序列(即Unigenes),总长度为7 278.578 kb,序列冗余率达到65.29%。Unigene中,67.13%为单拷贝序列Singlets(8 098条),平均长度为533.4 bp,变化范围为46~1 066 bp;Contigs为3 965条,平均长度达到746.5 bp,变化范围为130~2 637 bp。Unigene长度分布如图1所示。长度大于500 bp的Unigene占73.0%,长度为200~500 bp的Unigene占22.5%。
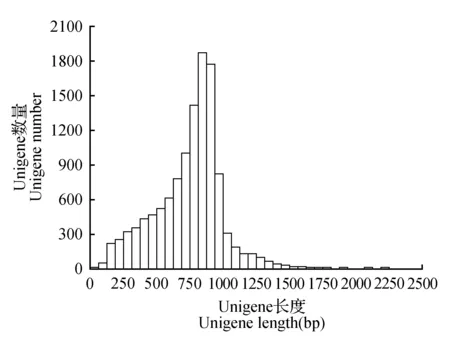
图1 木麻黄Unigene长度分布Fig.1 Length distribution of casuarina Unigene
2.2 木麻黄EST-SSR的分布频率和基序特征
利用SSRIT软件对12,062个木麻黄Unigenes进行SSR位点搜索,共获得367个SSR位点,这些SSR位点分布于353条Unigenes序列中。EST序列中含有SSR位点的频率仅为2.93%,平均分布距离为19.83 kb。其中含有2个SSR位点的EST序列为12条,含有3个SSR位点的EST序列仅有1条,其余序列均为含有1个SSR位点的EST序列。
木麻黄EST-SSR类型丰富,包含二核苷酸、三核苷酸、四核苷酸、五核苷酸,以及六核苷酸5种类型(图2)。其中,以二核苷酸和三核苷酸为主,所占比例分别为57.77%和34.60%,四核苷酸和六核苷酸所占比例相近,分别为3.27%和3.54%,五核苷酸所占比例极少,仅为0.82%。

图2 木麻黄EST-SSR的分布频率Fig.2 Distribution frequency of EST-SSR in Casuaria
共发现39种不同类型的重复基序,其中二核苷酸和五核苷酸重复基序的类型为3种,三核苷酸和四核苷酸重复基序的类型为10种,六核苷酸重复基序的类型最多,为13种;而二核苷酸重复基序数量最多,三核苷酸重复基序次之,其他重复基序的数量均较少。其中,在二核苷酸重复基序中,AG/CT数量最多,为199,占二核苷酸重复基元数的93.87%,AT/AT和AC/GT的数量相近,均较少,分别为7和6;在三核苷酸重复基序中AAG/CTT最多,为56,占三核苷酸重复基元数的44.09%,AGT/ATC和AGG/CCT的数量相近,分别为14和13,分别占三核苷酸重复基序数的11.02%和10.24%(表1)。
2.3 木麻黄EST-SSR的同源性比对分析
通过Blastx程序,对353条含有SSR的EST序列与NCBI非冗余蛋白序列数据库(Nr)进行同源性比对分析。结果表明,共有273条EST序列具有同源序列,占序列总数的77.3%,其中有3条EST序列与未知功能蛋白同源,其余80条EST序列无同源序列(图3:A)。在270条已知功能的EST序列中,主要比对到巨桉(14条)、麻疯树(18条)、葡萄(28条)、可可(17条)、毛果杨(11条)、乌梅(15条)、川桑(11条)、蓖麻(13条)、胡杨(7条)、金钱橘(12条)、梨(12条)、苹果(9条)、桃(12条),其他植物91条,其中,所占比例最大的是葡萄、其次是麻疯树和可可,巨桉、毛果杨、乌梅的比例相近(图3:B)。
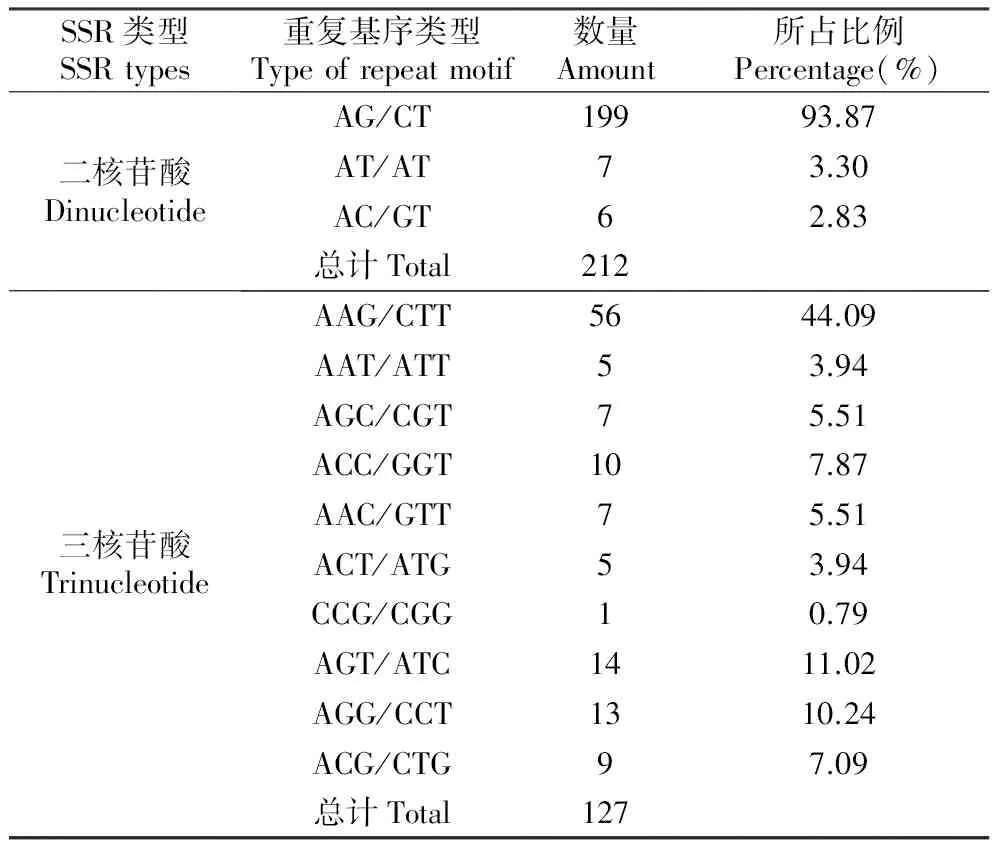
表1 SSR中重复基序的比例
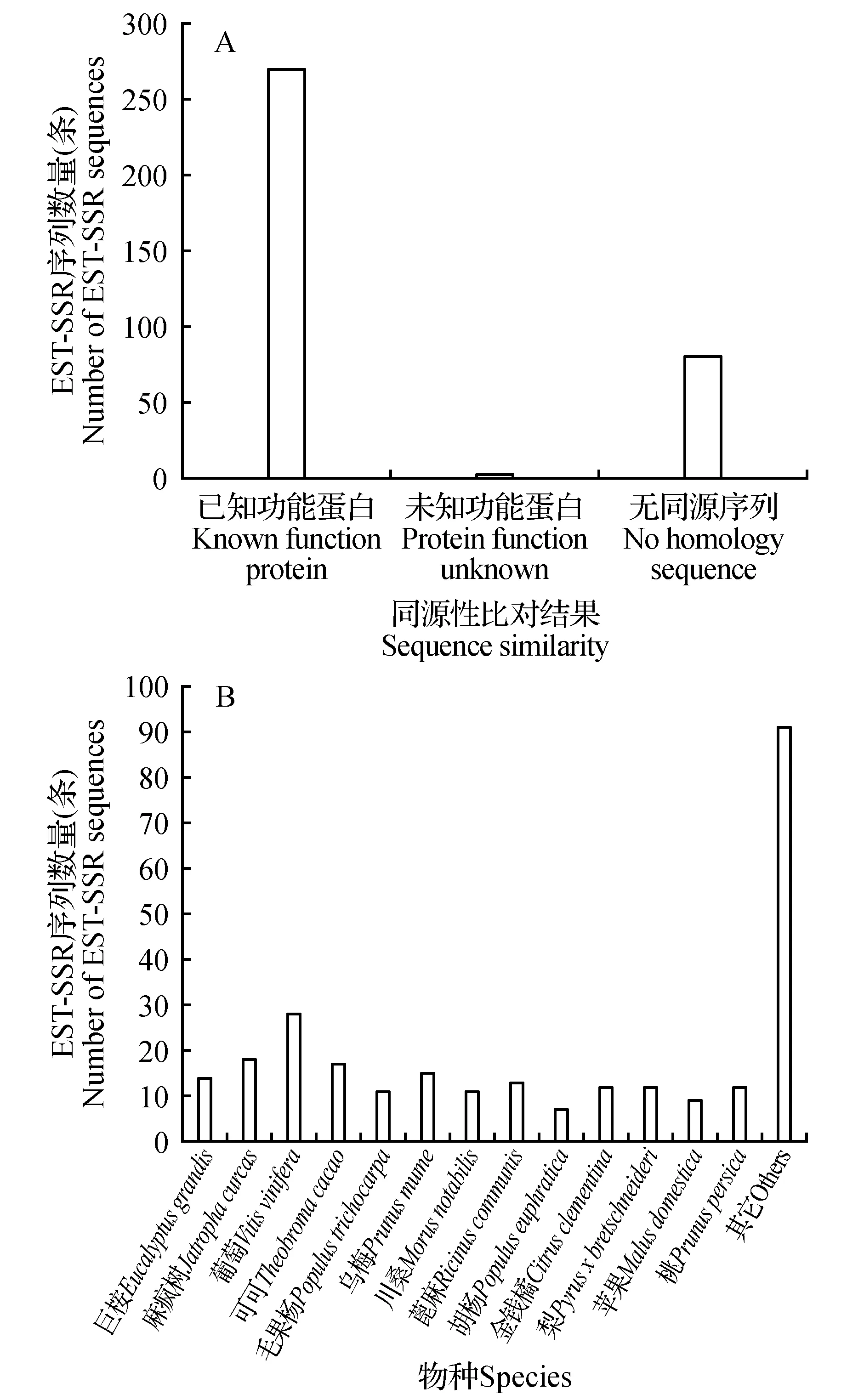
图3 木麻黄EST-SSR序列同源性比对分析Fig.3 Blast analysis of EST-SSR in Casuaria
2.4 木麻黄EST-SSR的功能注释及分类
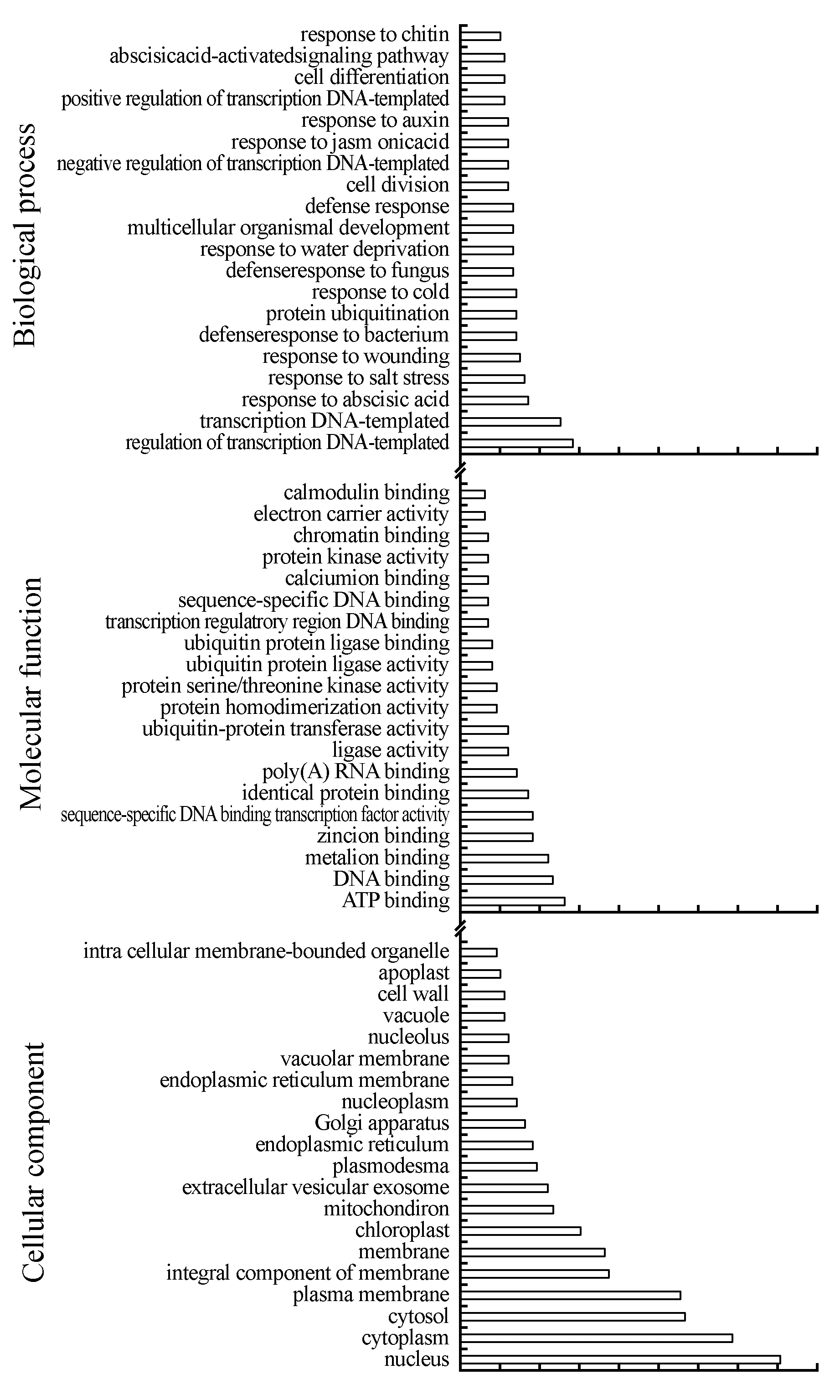
图4 木麻黄EST-SSR序列GO功能注释Fig.4 GO functional ontology of EST-SSR in Casuaria
为了对含有SSR位点的EST序列进行功能分类,对这些序列进行GO注释。在353条含有SSR的EST序列中共有167条(47.3%)序列至少具有1个GO功能注释,并且包含在60个功能项。其中,生物过程(Biological processes,BP)159条,细胞组分(Cellular components,CC)160条,分子功能(Molecular function,MF)148条(图4)。在生物过程中,归入DNA转录调节(regulation of transcription DNA-templated)和DNA转录(transcription DNA-templated)的序列最多;在细胞组分中,归入细胞核(nucleus)和细胞质(cytoplasm)的序列最多,细胞溶质(cytosol)和细胞质膜(plasma membrane)次之;在分子功能中,归入ATP结合(ATP binding)的最多,DNA结合(DNA binding)和金属离子结合(metal ion binding)次之;其他功能类别包括脱落酸响应、盐胁迫响应、防御响应、蛋白激酶活性、液泡膜和细胞壁等。

表2 EST-SSR引物信息
2.5 EST-SSR的有效筛选
对所获得的367个SSR位点设计引物,其中由于存在退火温度过高或过低、包含引物二聚体等情况,有256个位点在所设定的引物设计条件中无法获得适合的引物,获得97对待筛选SSR引物。利用12个木麻黄个体对这97个待筛选引物进行PCR验证,有32个引物可获得单一明亮条带(图5),其引物序列和退火温度信息见表2。

图5 引物筛选PCR检测Fig.5 PCR detection of primer selection
3 讨论
随着植物功能基因组研究的迅速发展,公共数据库中的表达序列标签(ESTs)呈指数趋势增长,通过搜索EST序列中的SSR位点开发新的EST-SSR标记成为新标记开发的焦点。本研究中通过搜索公共数据库中34 752条EST序列中的SSR位点,得到位点总数367个,检出率仅为2.93%。Areshchenkova等在27 000条番茄EST序列中搜索SSR位点时,发现SSR位点数250个,检出率仅为1.2%[32];吴春太等在39 227条油棕EST序列中检索得到699个SSR位点,检出率4.45%[33];而韩明利等利用NCBI数据库中与番茄果实性状相关的83 613条EST序列检索得到2 382个SSR位点,检出率高达10.8%[34];另外,本文中木麻黄EST序列中SSR的平均分布距离为19.83 kb,远低于大豆(7.40 kb)、玉米(8.10 kb)等作物,但与棉花(20.00 kb)中SSR的平均分布距离相近[35]。这可能是由于两个方面的原因造成的:一,由于公共数据库中的EST序列存在冗余性,这将会影响精确估计EST序列中SSR的发生频率;二,所用处理软件以及设置参数的不同,也将影响EST序列中SSR位点的检出率。
EST-SSR标记广泛应用于遗传图谱构建、基因定位及比较基因组学研究中。安泽伟等利用10 778条橡胶树EST序列,搜索得到430个SSR位点,针对这些EST-SSR位点,在所设计的148对引物中,筛选得到15对可有效扩增且在橡胶树无性系中表现出较高多态性的标记[36];本文中,对367个木麻黄EST-SSR位点设计了97对引物,其中有32对为有效扩增引物,说明利用公共数据库中的EST序列开发SSR标记,进行木麻黄遗传学研究是可行的,当然这些EST-SSR标记的多态性有待于进一步分析。因设置的引物设计参数较为严苛,本文中设计得到的引物偏少,而通过设置适当的软件参数,以适中的搜索条件和设计要求进行SSR位点搜索和引物设计,可获得更多的SSR位点,并以此得到更多的可筛选引物。木麻黄EST-SSR标记的开发,将有利于木麻黄遗传多样性研究,对于拓宽木麻黄种质资源遗传基础也具有积极意义。
EST-SSR的多态性主要源于重复次数和重复长度[34]。一般认为DNA复制或修复过程中DNA链的滑动和错配,或者有丝分裂或减数分裂过程中姐妹染色单体的不均等交换,是造成SSR长度变异的主要原因。木麻黄EST序列中SSR重复基序的重复次数在5~52变化,最大的重复次数存在于二核苷酸中。詹少华等研究认为大豆中SSR基序重复次数变异范围越大,SSR标记的多态性就越高[37]。而李淑娴等在研究桉树属(Eucalyptus)SSR长度变化时获得桉树属SSR位点1 775个,SSR序列长度变化范围为12~64 bp,平均长度18.5 bp,其发现桉树EST-SSR的长度变异速率、SSR位点的丰度均与重复基元的长度呈负相关关系,且二核苷酸重复SSR的理论多态性最高[38],与本研究结果一致。
EST是cDNA克隆测序产生的部分序列,通过与公共数据库中非冗余蛋白质或核酸序列进行相似性比对,可进行EST-SSR的功能分析。吴春太等对公共数据库中油棕(ElaeisguineensisJacg.)的EST序列进行聚类拼接处理后,搜索油棕非冗余序列中的SSR位点,通过对含有SSR位点的EST序列进行BLASTx比对,发现与细胞相关的SSR-EST序列占有较大比例(76.47%)[33];本文中,353条含有SSR位点的EST序列经过同源性比对发现,270条与已知功能序列同源,3条与未知功能序列具有同源性,另外80条无同源序列,而在已知功能的EST序列中,主要比对到葡萄、麻疯树、可可以及巨桉等树种(图3:B),且同源性较高,与葡萄、可可及巨桉序列的相似性均为84%,与麻疯树序列的相似性为83%。分析发现,许多序列涉及多重功能,如转录调节、代谢过程、细胞生理过程等。
植物中有关EST序列的功能研究进展缓慢,但大量动物和人类基因研究认为,SSR可能参与基因表达调控以及基因重排和变异等重要的生命活动,SSR长度的变化对生命活动有显著影响[14,39~40]。当关联比较基因组遗传信息和质量性状信息时,运用EST-SSR标记可有效的进行功能基因的定位和鉴别,以及遗传图谱和物理图谱等的构建[41~42]。本研究通过搜索木麻黄总EST序列中的SSR,分析了木麻黄EST-SSR的分布频率和特点,以期为木麻黄重要生态性状相关标记的开发,木麻黄重要生态及经济性状的遗传连锁图谱的构建,以及木麻黄种质资源分析和分子辅助育种提供参考资料。
1.Chen X,Cho Y G,McCouch S R.Sequence divergence of rice microsatellites inOryzaand other plant species[J].Mol Gen Genomics,2002,268:331-343.
2.Fahima T,Röder M S,Grama A,et al.Microsatellite DNA polymorphism divergence inTriticumdicoccoidesaccessions highly resistant to yellow rust[J].Theor Appl Genet,1998,96:187-195.
3.Huang X,Börner A,Röder M,et al.Assessing genetic diversity of wheat(TriticumaestivumL.)germplasm using microsatellite markers[J].Theoretical and Applied Genetics,2002,105(5):699-707.
4.Loudet O,Chaillou S,Camilleri C,et al.Bay-0×Shahdara recombinant inbred line population:a powerful tool for the genetic dissection of complex traits inArabidopsis[J].Theoretical and Applied Genetics,2002,104(6-7):1173-1184.
5.Röder M S,Korzun V,Wendehake K,et al.A microsatellite map of wheat[J].Genetics,1998,149(4):2007-2023.
6.Ma X F,Wanous M K,Houchins K,et al.Molecular linkage mapping in rye(SecalecerealeL.)[J].Theoretical and applied genetics,2001,102(4):517-523.
7.Gupta P K,Varshney R K.The development and use of microsatellite markers for genetic analysis and plant breeding with emphasis on bread wheat[J].Euphytica,2000,113(3):163-185.
8.Korzun V,Röder M,Worland A J,et al.Intra-chromosomal mapping of genes for dwarfing(Rht12) and verbalization response(Vrn1) in wheat by using RFLP and microsatellite markers[J].Plant Breeding,1997,116(3):227-232.
9.Liu X,Smith C,Gill B.Identification of microsatellite markers linked to Russian wheat aphid resistance genes Dn4 and Dn6[J].Theoretical and Applied Genetics,2002,104(6-7):1042-1048.
10.Buchanan F C,Adams L J,Littlejohn R P,et al.Determination of evolutionary relationships among sheep breeds using microsatellites[J].Genomics,1994,22(2):397-403.
11.Li C D,Rossnagel B G,Scoles G J.The development of oat microsatellite markers and their use in identifying relationships among Avena species and oat cultivars[J].Theoretical and Applied Genetics,2000,101(8):1259-1268.
12.Matsuoka Y,Mitchell S E,Kresovich S,et al.Microsatellites in Zea-variability,patterns of mutations,and use for evolutionary studies[J].Theoretical and Applied Genetics,2002,104(2-3):436-450.
13.Cardle L,Ramsay L,Milbourne D,et al.Computational and experimental characterization of physically clustered simple sequence repeats in plants[J].Genetics,2000,156(2):847-854.
14.Tóth G,Gáspári Z,Jurka J.Microsatellites in different eukaryotic genomes:survey and analysis[J].Genome research,2000,10(7):967-981.
15.Morgante M,Hanafey M,Powell W.Microsatellites are preferentially associated with non-repetitive DNA in plant genomes[J].Nature genetics,2002,30(2):194-200.
16.Bundock P C,Potts B M,Vaillancourt R E.Detection and stability of quantitative trait loci(QTL) inEucalyptusglobulus[J].Tree Genetics & Genomes,2008,4(1):85-95.
17.Freeman J S,Whittock S P,Potts B M,et al.QTL influencing growth and wood properties inEucalyptusglobulus[J].Tree Genetics & Genomes,2009,5(4):713-722.
18.Byrne M,Murrell J C,Owen J V,et al.Mapping of quantitative trait loci influencing frost tolerance inEucalyptusnitens[J].Theoretical and Applied Genetics,1997,95(5-6):975-979.
19.Wilson K L,Johnson L A S.Casuarinaceae.Flora of Australia:Volume 3 Hamamelidales to Casuarinales[M].Canberra:Australian Government Publishing Service,1989.
20.仲崇禄.木麻黄遗传变异规律的研究[D].北京:中国林业科学研究院,2000.
21.葛菁萍,林鹏.厦门三种木麻黄属(Casuarina)植物的种内遗传变异和种间亲缘关系分析[J].黑龙江大学自然科学学报,2002,19(4):110-114.
22.郭启荣,林益明,周涵滔.4种木麻黄亲缘关系的RAPD分析[J].厦门大学学报:自然科学版,2003,42(3):378-383.
23.何坤益,萧如英,邓书麟.应用RAPD分子指纹研究木贼叶木麻黄国际种源之遗传变异[J].台湾林业科学,2001,16(4):285-293.
24.黄桂华.短枝木麻黄种质资源遗传多样性的AFLP分析[D].北京:中国林业科学研究院,2006.
25.Huang G H,Zhong C L,Su X H,et al.Genetic variation and structure of native and introducedCasuarinaequisetifolia(L.Johnson) provenances[J].Silvae Genetica,2009,58(1-2).
26.Ho Kuen-yih,Lee Shih-chieh.ISSR-based genetic diversity ofCasuarinaspp.in coastal windbreaks of Taiwan[J].African Journal of Agriculture Research,2011,6(25):5664-5671.
27.Doyle J J.Trees within trees:genes and species,molecules and morphology[J].Systems Biology,1997,46:537-553.
28.Falgueras J,Lara A J,Fernández-Pozo N,et al.SeqTrim:a high-throughput pipeline for pre-processing any type of sequence read[J].BMC bioinformatics,2010,11(1):38.
29.Gordon D,Abajian C,Green P.Consed:a graphical toolfor sequence finishing[J].Genome Res,1998,8:195-202.
30.Gordon D,Desmarais C,Green P.Automated finishingwith auto-finish[J].Genome Res,2001,11:614-625.
31.Conesa A,Götz S,Carcía-Gómes JM,et al.Blast2go:a universal tool for annotation,visualization and analysis in functional genomics research[J].Bioinformatics,2005,21(18):3674-3676.
32.Areshchenkova T,Ganal M W.Comparative analysis of polymorphism and chromosomal location of tomato microsatellite markers isolated from different sources[J].Theoretical and Applied Genetics,2002,104(2-3):229-235.
33.吴春太,陈青,刘锐,等.油棕EST序列中SSR的分布特征分析[J].中国油料作物学报,2012,34(1):101-105.
34.韩明利,崔娜,于志海,等.番茄果实EST资源SSR信息分析[J].华北农学报,2011,26(4):213-217.
35.李永强,李宏伟,高丽锋,等.基于表达序列标签的微卫星标记(EST-SSRs)研究进展[J].植物遗传资源学报,2004(5):91-95.
36.安泽伟,赵彦宏,程汉,等.橡胶树EST-SSR标记的开发与应用[J].遗传,2009,31(3):311-319.
37.詹少华,盛新颖,樊洪泓,等.大豆EST序列长度与SSR特性的关系[J].大豆科学,2009,28(2):204-209.
38.李淑娴,张新叶,王英亚,等.桉树EST序列中微卫星含量及相关特征[J].植物学报,2010,45(3):363-371.
39.Thibodeau S N,Bren G,Schaid D.Microsatellite instability in cancer of the proximal colon[J].Science,1993,260(5109):816-819.
40.Aaltonen L A,Lauri A,Leach F S,et al.Clues to the pathogenesis of familial colorectal cancer[J].Science,1993,260(5109):812-816.
41.张新叶,宋丛文,张亚东,等.杨树EST-SSR 标记的开发[J].林业科学,2009,45(9):53-59.
42.Ellis J R,Burke J M.EST-SSRs as a resource for population genetic analyses[J].Heredity,2007,99:125-132.
DistributionandMolecularMarkerofSSRinESTResourceofCasuarinaceae
HU Pan1ZHONG Chong-Lu1ZHANG Yong1JIANG Qing-Bin1CHEN Yu1CHEN Zhen1HAN Qiang1Khongsak Pinyopusarerk2Didier Bogusz3
(1.Research Institute of Tropical Forestry,Chinese Academy of Forestry,Guangzhou 510520;2.CSIRO National Research Collections Australia,Acton,ACT 2600,Australia;3.Institut de Recherche pour le Développement(IRD),BP 64501,Montpellier,France)
There are 34 752 ESTs of Casuarinaceae in the database of NCBI, resulting in 12 062 non-redundant sequences(Unigene) with total length of 7 278.578 kb. 367 SSR were discovered to distribution on 353 Unigenes, the frequency and repeat motif types number of these EST-SSRs was 2.93% and 39, and the mean distance was 19.83 kb. Dinucleotide and trinucleotide repeats were the dominant types among the EST-SSRs, accounting for 57.77% and 34.06%, respectively. AG/CT(93.87%) and AAG/CTT(44.09%) were the most abundant motifs for dinucleotide and trinucleotide. There were 97 pairs of primers designed for the obtained EST-SSR, and 32 of all could be effectively amplified. By Blast analysis, 77.3% ESTs sequences containing SSR locus were homologous with functional sequences in non-redundant protein sequences databases. The homology with functions from Vitis vinifera had the greatest proportion(10.4%). By GO functional classification, 47.3% EST sequences containing SSR locus had at least one GO ontology, the sequences relevant to cellular components were most, and the functional items relevant to cytoplasm and nucleus were highest.
Casuarinaceae;EST;SSR
十二五林业科技支撑计划任务(2012BAD01B0603);广东省林业科技创新专项资金项目(2014KJCX017);国家自然科学基金项目(31470634)和福建省林木种苗科技攻关四期项目的子课题4:木麻黄改良代种子园营建技术研究
胡盼(1987—),女,博士研究生,主要从事木麻黄遗传改良研究。
* 通信作者:E-mail:zcl@ritf.ac.cn
2015-06-08
S792.93
A
10.7525/j.issn.1673-5102.2016.01.016
