不同种源马尾松ISSR遗传结构及影响因素分析
2016-10-28杜明凤丁贵杰
杜明凤, 丁贵杰
(1. 贵州大学 林学院, 贵阳 550025; 2. 贵州省森林资源与环境研究中心,贵阳 550025; 3. 贵州师范大学 喀斯特研究院, 贵阳 550001 )
不同种源马尾松ISSR遗传结构及影响因素分析
杜明凤1,2,3, 丁贵杰1,2*
(1. 贵州大学 林学院, 贵阳 550025; 2. 贵州省森林资源与环境研究中心,贵阳 550025; 3. 贵州师范大学 喀斯特研究院, 贵阳 550001 )
应用ISSR分子标记技术,对来自广西、贵州3个种源的马尾松开展遗传多样性、遗传结构及遗传距离等研究。结果表明:从100条引物中筛选出12条引物,共扩增出92个条带,86条具有多态性。POPGENE分析显示:马尾松群体水平上的Nei’s基因多样性指数的变化范围为0.182 4~0.206 5,Shannon遗传多样性指数的变化范围为0.281 8~0.317 8,3个群体的多态性水平差异不大;物种水平上的多态性百分率为93.48%,Nei’s基因多样性指数为0.284 2,Shannon信息指数为0.438 1;表明马尾松在物种水平上具有较高水平的遗传多样性。遗传结构分析显示:马尾松的基因分化系数(Gst)为0.315 3,表明遗传变异主要来源于群体内;基因流Nm为1.085 3 ,表明不同群体间存在一定的基因流动。AMOVA分析显示:马尾松的遗传分化指数(Fst)为0.246 (P=0.001),表明群体间已出现明显的遗传分化。UPGMA聚类和Mantel检测结果显示:每个群体内的个体均能很好地首先聚集为一个分支,群体间的遗传距离与地理距离之间存在显著相关性(r=0.972,P=0.001)。这说明马尾松在裸子植物界中具有较高水平的遗传多样性,遗传变异主要分布于群体内,群体间已出现了明显的遗传分化,这种分化并非由遗传漂变引起,可能与地理生境的差异有关。
马尾松, ISSR, 遗传多样性, 遗传结构, 遗传距离
马尾松(Pinusmassoniana)属裸子植物松科(Pinaceae)松属(PinusLinn),具有速生、丰产、适应能力强、分布广泛、全树综合利用程度最高、经济价值高等优良特性,是我国南方最主要的优质针叶用材树种之一,在我国森林资源发展、林纸一体化、松香林产化工业及森林生态服务功能中占有重要地位(丁贵杰等,2006)。目前,马尾松在造林栽培技术(丁贵杰等,2002)、经营管理技术及应用(丁贵杰和周政贤,1996;丁贵杰,1998)等方面已取得成果。随着分子标记技术的发展及应用,RAPD(万爱华等,2008)、ISSR(张一等,2009)、SSR(谭小梅等,2012)等标记先后用于马尾松种子园(万爱华等,2008;张薇等,2008;谭小梅等,2012)、亲本与子代之间(张一等,2010)的遗传多样性、遗传改良等方面的研究,为马尾松分子育种研究奠定了一定基础。
由于马尾松分子育种研究起步较晚,许多遗传性状机理、遗传结构形成机制及相关因素等方面的研究相对缺乏;同时,高温、干旱等异常气候频发加剧了生境的恶化,人为的强烈干扰致使马尾松资源不可避免遭致破坏,其遗传多样性、基因流格局是否发生改变等问题,需要进一步深入研究。为此,本研究采用ISSR分子标记,对广西、贵州3个不同种源马尾松的遗传多样性、遗传结构进行研究,以揭示马尾松群体的遗传分化水平及基因交流程度,并探究地理距离对遗传结构的影响,为马尾松引种、遗传改良及资源保护提供参考,同时对认识马尾松遗传结构的形成机制具有一定的意义。
1 材料与方法
1.1 材料
材料来源于广西和贵州3个不同的马尾松人工林,均是广西林科院选育的半同胞优良家系,每个种源选择24株生长良好的单株当年生嫩叶,共72个单株针叶样品(具体信息见表1),液氮速冻保存带回实验室置于-80 ℃冰箱中。
1.2 DNA提取
分别取-80 ℃下的每株嫩叶,于液氮中研磨成细粉状,利用天根公司DNA secure Plant Kit(DP320-02)提取马尾松基因组,经1%琼脂糖凝胶电泳和紫外分光光度计检测其质量和浓度后,用TE缓冲液稀释DNA至25 ng·μL-1,-20 ℃保存备用。
1.3 PCR扩增与检测
参照加拿大哥伦比亚UBC公司公布的ISSR引物序列,利用BIO-RAD t100 PCR扩增仪从100条引物 (上海生工合成) 中筛选出12条ISSR引物及退火温度进行PCR扩增 (表2)。10 μL反应体系: 4 μL (25 ng·μL-1) DNA模板,0.4 μL (100 ng·μL-1) 引物,0.6 μL ddH2O, 5.0 μL Mix (Mix购自北京天根公司),含0.1 U·μL-1Taq聚合酶,500 μmol·L-1dNTPs,20 mmol·L-1Tris-HCl (pH8.3),100 mmol·L-1KCl,3 mmol·L-1MgCl2,其他稳定剂和增强剂)。PCR反应程序:(1)预变性:94 ℃ 5 min;(2)扩增循环(40个循环):预变性94 ℃ 45 s,退火1 min,72 ℃ 45 s;(3)延伸:72 ℃ 10 min。设不加DNA模板的空白作为对照。扩增产物经2.0%琼脂糖凝胶 (内含GoldView I型核酸染色剂) 电泳分离,在紫外分析仪上检查。

表 1 材料及来源
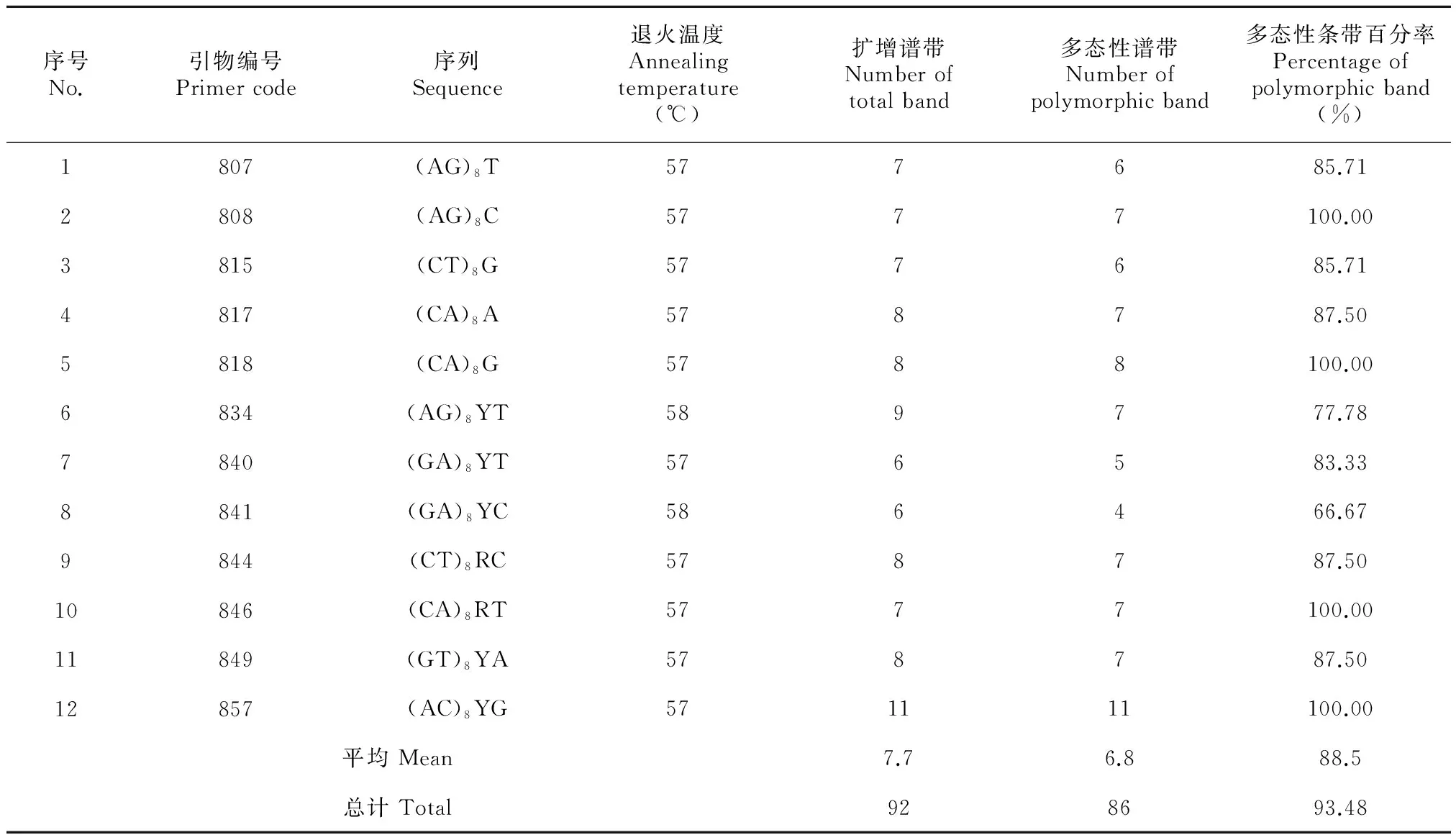
表 2 ISSR引物序列与扩增情况
1.4 数据分析
每条引物PCR扩增重复3次,数据统计遵循以下原则:模糊不清、重现性差的谱带不计,与阴性对照有相同迁移率的谱带不计,分子量小于200 bp的谱带不计。ISSR-PCR产物在凝胶的同一迁移率上,有带赋值“1”(强带和弱带同记),无带赋值“0”, 得到原始数据矩阵。利用POPGENE 32.0软件估算多态性百分率(PPB)、等位基因数(Ao)、有效等位基因数(Ae)、Shannon信息指数(I)、Nei’s基因多样性指数(H)、Nei’s基因分化指数(Gst)、基因流(Nm)等遗传参数分析;利用AMOVA 1.55软件(张富民等,2002)对群体间、群体内的遗传变异进行分析;利用NTSYS-pc2.10e软件根据遗传距离构建UPGMA聚类图;利用ARCGIS软件计算不同种源的地理距离;利用R软件对遗传距离与地理距离进行Mantel检验。
2 结果与分析
2.1 ISSR引物的扩增片段效果
从100条引物中筛选出12条稳定清晰、重复性好、 多态性高的引物(表2), 对3个种源72份材料进行ISSR-PCR扩增,共扩增出92个条带, 86个条带具有多态性,多态位点百分率为93.48%,条带大小200~1 500 bp不等,500~1 000 bp内的多态性片段最多。平均每条引物扩增7.7条,其中引物857扩增条带最多,为11条,引物841扩增条带最少,为6条;不同引物扩增条带的多态位点百分率在66.67%~100%之间,其中引物808、818、846、857检测效率最高,其检测位点的多态性百分率为100%,引物841最低,为66.67%。

图 1 ISSR引物857扩增DNA片段 M. 分子量标记; 1-24. GX1居群; 25-48. GX2居群; 49-72. GZ居群。Fig. 1 ISSR amplified bands of primer 857 M. Marker; 1-24. Population GX1; 25-48. Population GX2; 49-72. Population GZ.
2.2 马尾松遗传多样性
由表3可知,在物种水平上,72份材料的多态位点百分率为93.48%、观测等位基因数为1.934 8、有效等位基因数为1.461 9、Nei’ s基因多样性指数为0.284 2、Shannon信息指数为0.438 1。群体水平上,3个群体的多态位点百分率在64.13%~71.74%之间,平均为66.67%;观测等位基因数变幅为1.641 3~1.717 4,平均为1.666 7;有效等位基因数变幅为1.300 3~1.345 8,平均为1.322 2; Nei’ s基因多样性指数在0.182 4~0.194 8之间,平均为0.194 6;Shannon信息指数为0.281 8~0.317 8之间,平均为0.299 6。根据Nei’s指数和Shannon指数的大小变化可知,物种水平上的遗传多样性高于群体水平;不同群体间,GX2的遗传多样性水平最高,GZ其次,GX1最低。

表 3 不同种源马尾松ISSR遗传多样性

表 4 不同种源马尾松的遗传结构

表 5 不同种源马尾松遗传变异的AMOVA分析
2.3 马尾松遗传分化
从表4可知,由Nei’s指数估算得出的群体间基因分化系数Gst为0.315 3,群体间的基因流为1.085 3,表明群体间存在基因交流;由Shannon指数估算得出总遗传变异中,68.37%源于群体内,31.61%源于群体间,均表明马尾松的遗传多样性主要来自于群体内不同个体的遗传差异。运用AMOVA对马尾松3个群体的遗传分化系数进行分析,结果显示(表5):马尾松的77.28%遗传变异来源于群体内,22.72%的遗传变异来源于群体间,与Nei’ s指数和Shannon指数的估算结论一致;遗传分化指数Fst表明,马尾松群体间的遗传分化虽只占总遗传分化的小部分,但其遗传分化水平表现出极显著差异(Fst=0.246,P=0.001)。
2.4 马尾松遗传距离与聚类分析
由表6可知,马尾松3个群体间的遗传距离范围为0.096 0~0.269 7,其中GX1和GX2的遗传距离最小(0.096 0),遗传相似度最大;GX1与GZ的遗传距离最大(0.269 7),遗传相似度最小。利用遗传相似系数(Gs)构建的UPGMA聚类图(图2)显示,3个群体内的个体植株均能较好地聚在一起;当阈值取0.68时,GX1与GX2聚为一支,GZ单独聚成一支;说明GX1与GX2亲缘关系较近,两者与GZ的亲缘关系较远。通过Mantel检验发现,3个马尾松群体间的遗传距离与其地理距离具有显著相关性(r=0.972,P=0.001),说明马尾松群体间的遗传多样性具有明显的地域分布规律。
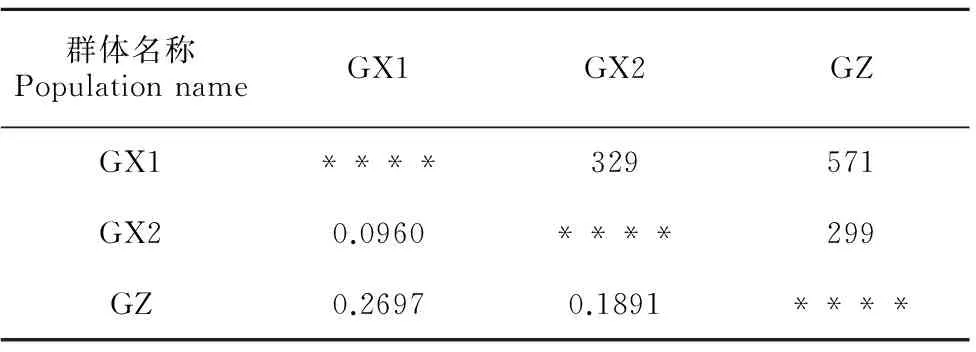
表 6 不同种源马尾松遗传距离 (对角线下方)

图 2 不同种源马尾松的ISSR聚类图Fig. 2 Clustering of different populations of P. massoniana
3 讨论与结论
3.1 遗传多样性
遗传多样性是物种生存和发展的前提,物种的遗传多样性水平越高意味着适应环境变化的能力就越强,遗传变异的大小决定其进化速率的快慢(Barrett & kidwell,1998)。本研究通过对广西、贵州的3个种源72份材料的ISSR分析得出,马尾松在物种水平上的多态位点百分率、Nei’s指数、Shannon指数分别为93.48%、0.284 2、0.438 1,与朱亚艳等(2014)、王茜等(2013)、张一等(2009)的研究结果(多态位点百分率为82.25%~94.35%、Nei’s指数为0.276 5~0.378 9、Shannon指数为0.427 8~0.550 3)相类似,表明马尾松在物种水平上具有丰富的遗传多样性,这与Chen et al(2013)、Liao et al(2012)的研究结论一致,即自然分布较广的物种一般都具有较高的遗传多样性;这也是其在恶劣环境下具有较强生存适应能力的重要原因之一。与裸子植物相比,略低于油松(郝真真,2009)、华山松(赵杨等,2012) (多态位点百分率为92.81%~100%、Nei’s指数为0.334 1~0.402 9、Shannon指数为0.448 0~0.585 5),高于云南松(杨章旗,2014)、红松(贾俊玲,2011)、红豆杉(李乃伟等,2011)、台湾杉(李江伟等,2014) (多态位点百分率为74.62%~82.19%、Nei’s指数为0.207 6~0.256 0、Shannon指数为0.322 9~0.377 8);整体而言,马尾松的遗传变异丰富,在裸子植物界中处于较高水平。
3.2 遗传结构
掌握物种的遗传结构即遗传变异在种群间的分布,是物种保护繁育的重要基础和前提。影响物种遗传分化的主要因素包括迁移、突变、重组、自然选择、地理隔离、遗传漂变、基因流等(Viki et al,2013)。本研究发现,Nei’s基因多样性指数、Shannon信息指数及AMOVA分析结果相似,即马尾松遗传多样性主要分布于群体内(Gst=0.3153),群体间存在着一定的基因交流(Nm=1.0853)。这与Nybom & Bartish(2000)认为寿命长、异交的植物类群其遗传变异主要来源于群体内部的研究结论一致,同时也表明其群体内个体选优的潜力很大。但与谭小梅等(2012)、艾畅等(2006)的研究数据存在差异(其基因分化系数Gst分别为0.050 4、0.076 7);上述研究针对种子园内不同群体展开,各群体的生态环境相对一致;马尾松是典型风媒异交物种,其花粉小,能借助风媒传播,加之人工授粉辅助,可加大种子园内不同群体间的基因流动,因而表现出其群体间的分化很小,这与本研究来自不同省份的种源材料在地理距离上、生态环境等方面差异较大。
由于多数马尾松的研究结果均显示其遗传变异主要来源于群体内,因此对于马尾松群体间的遗传分化情况关注很少。本研究通过AMOVA分析发现,马尾松群体间的遗传分化虽然仅占其小部分,但已出现显著差异(Fst=0.246,P=0.001),这与油松(孟翔翔等,2013)、台湾杉(李江伟等,2014)等研究结果相似,暗示马尾松种群在长期进化过程中可能产生了分化。Wight(1931)认为Nm>1时,基因流能防止由遗传漂变引起的群体间的遗传分化;本研究中Nm=1.085 3,说明马尾松群体间存在基因交流,其遗传分化并非由遗传漂变引起。
3.3 遗传距离
Fischer(2000)认为物种的遗传距离与地理距离之间不相关,意味着遗传漂变可能是导致群体间遗传分化的主导因素。本研究发现,遗传距离与地理距离之间具有显著相关性(r=0.972,P=0.001),生境来源相近的家系QG83和QG111首先聚在一起,两者与贵州的QM01群体具有较远的亲缘关系;相比而言,QG83与QM01的遗传距离比QG111与QM01之间的更远,与其地理差异一致,表明马尾松群体间的遗传多样性呈现一定的地理分布规律。综合上述结果,可初步推断引起马尾松群体间遗传分化的主要因素并非遗传漂变而可能是地理差异。本研究还发现,广西2个种源均为低纬度地区;防城港临海,属于海洋性季风气候,海拔在100 m以内;百色市地处珠江水系上游,典型山区,属于亚热带季风湿润气候,平均海拔500 m;贵州种源麻江地处云贵高原向湘桂丘陵过渡的斜坡地带,以山地为主,属亚热带季风湿润气候,平均海拔930 m。各自的地理位置、气候特征、海拔、地形、土壤以及水热条件等生态环境均有差异,各自形成不同的小生境。尽管马尾松具有风媒、异交等生物学特性,研究结果亦显示群体间存在一定基因交流,但面临长距离、海拔梯度大的实际情况,其后代向外扩散的能力是非常有限的。因此,马尾松很可能在不同生境下经过长期的进化,逐渐形成不同的适应机制,因而开始呈现出群体间的遗传分化。
AI C,XU LA,LAI HL,et al, 2006. Genetic diversity and paternity analysis of a seed orchard inPinusmassoniana[J]. Sci Silv Sin,42(1): 146-150. [艾畅,徐立安,赖焕林,等, 2006. 马尾松种子园的遗传多样性与父本分析 [J]. 林业科学,42(1): 146-150.]
BARRETT BA, KIDWELL KK, 1998. AFLP-based genetic diversity assessment among wheat cultivars from the Pacific Northwest [J]. Crop Sci , 38(5):1 261-1 271.
CHEN SX, ZHOU J, CHEN Q, et al, 2013. Analysis of the genetic diversity of garlic (AlliumsativumL. ) germplasm by SRAP [J]. Biochem Syst Ecol,50:139-146.
DING GJ, 1998. Study on rational cutting age of building timber of masson pine plantation in Guizhou Province [J]. Sci Silv Sin,34(3):40-46. [丁贵杰, 1998. 贵州马尾松人工建筑材林合理采伐年龄研究 [J]. 林业科学,34(3):40-46.]
DING GJ,WU XB,WANG PC, 2002. A study on management model system and optimum cultivation patterns ofPinusmassonianafor pulpwood stand [J]. Sci Silv Sin,38(5):7-13. [丁贵杰,吴协保,王鹏臣, 2002. 马尾松纸浆材林经营模型系统及优化栽培模式研究 [J]. 林业科学,38(5):7-13. ]
DING GJ,ZHOU ZC,WANG ZR, et al, 2006. Cultivation and utilization of pulpwood stand forPinusmassoniana[M]. Beijing: China Forestry Publishing House: 1-34. [丁贵杰,周志春,王章荣, 等, 2006. 马尾松纸浆用材树种培育与利用 [M]. 北京: 中国林业出版社: 1-34. ]
DING GJ,ZHOU ZX, 1996. The economic results analysis on masson pine at various planting density and utilization ways [J]. J Nanjing For Univ, 20(2):24-29. [丁贵杰,周政贤, 1996. 马尾松不同造林密度和不同利用方式经济效果分析 [J]. 南京林业大学学报, 20(2):24-29. ]
FISCHER P, 2000. Time dependent flow in equimolar micellar solutions: transient behaviour of the shear stress and first normal stress difference in shear induced structures coupled with flow instabilities [J]. Rheol Acta, 39(3): 234-240.
GAUDETT M, SALOMON B, SUN G, 2005. Molecular variation and population structure inElymustrachycaulusand comparison with its morphologically similarE.alaskanus[J]. Plant Syst Evol ,250(1):81-91.
HAO ZZ, 2009. Genetie variation and spatial genetic strueture of Chinese pine (PinustabulaeformisCarr.) [D]. Taiyuan: Shanxi University:1-63. [郝真真, 2009. 油松遗传多样性与空间遗传结构研究 [D]. 太原:山西大学:1-63.]
JIA JL, 2011. Analysis of genetic diversity of Jilin naturalPinuskoraiensispopulation by ISSR marker [D]. Dalian: Liaoning Normal University:1-50. [贾俊玲, 2011. 吉林天然红松林遗传多样性的ISSR分析 [D]. 大连:辽宁师范大学:1-50. ]
LI JW,YANG QJ,LIU XQ,et al, 2014. Genetic diversity ofTaiwaniacryptomerioidesdetected by ISSR analysis [J]. Sci Silv Sin,50(6): 61-66. [李江伟,杨琴军,刘秀群,等, 2014. 台湾杉遗传多样性的ISSR分析) [J]. Sci Silv Sin (林业科学),50(6): 61-66.]
LI NW,HE SA,SHU XC,et al, 2011. Genetic diversity and structure analyses of wild and ex-situ conservation populations ofTaxuschinensisvar.maireibased on ISSR marker [J]. J Plant Resour Environ, 20(1): 25-30. [李乃伟,贺善安,束晓春,等, 2011. 基于ISSR标记的南方红豆杉野生种群和迁地保护种群的遗传多样性和遗传结构分析 [J]. 植物资源与环境学报. 20(1): 25-30.]
LIAO L, GUO QS, WANG ZY, et al, 2012. Genetic diversity analysis ofPrunellavulgarisin China using ISSR and SRAP markers [J]. Biochem Syst Ecol, 45:209-217.
MENG XX,DI XY,WANG MB,et al, 2013. Analysis of relationship between genetic structure of Chinese pine and mountain barriers [J]. Acta Ecol Sin, 33 (19) :6 382-6 388. [孟翔翔,狄晓艳,王孟本,等, 2013. 油松遗传结构与地理阻隔因素的相关性 [J]. 生态学报, 33 (19) :6 382-6 388. ]
NYBOM H, BARTISH IV, 2000. Effects of life history traits and sampling strategies on genetic diversity estimates obtained with RAPD markers in plants [J]. Persp Plant Ecol Evol Syst, 3(2):93-114.TAN XM,ZHOU ZC, JIN QG,et al, 2012. Genetic diversity and mating system analysis ofPinusmassonianain a second-generation clonal seed orchard [J]. Sci Silv Sin,2(2):69-74. [谭小梅,周志春, 金国庆,等, 2012. 马尾松二代无性系种子园遗传多样性和交配系统分析 [J]. 林业科学,2(2):69-74.]
VIKI M, SUMAN K, PRAMOD T, 2013. SRAP methods revealed high genetic diversity within populations and high gene flow ofVandacoeruleaGriffex Lindl(Blue Vanda),an endangered orchid species [J]. Gene, 519(1): 91-97.
WANG AH,XU YM, GUI LH,et al, 2008. Genetic structure of clonal seed orchard of masson pine by RAPD technology [J]. J NE For Univ,36(1):18-19. [万爱华,徐有明,管兰华,等, 2008. 马尾松无性系种子园遗传结构RAPD分析 [J]. 东北林业大学学报,36(1):18-19.]
WANG Q,ZHANG DL,YANG MH,et al, 2013. ISSR analysis of genetic distance ofPinusmassonianaLamb. Elite clones [J]. J Cent S Univ For Technol,33(12):72-76. [王茜,张冬林,杨模华,等, 2013. 马尾松优良家系优株ISSR遗传距离分析 [J]. 中南林业科技大学学报,33(12):72-76.]
WIGHT S, 1931. Evolution Mendelian population [J]. Genetics, (16):97.
YANG ZQ,FENG YH,WU DS, 2014. Analysis of genetic diversity ofPinusyunnanensisvar.tenuifolianature populations by SSR marker [J]. Guihaia, 34(1): 10-14. [杨章旗,冯源恒,吴东山, 2014. 细叶云南松天然种源林遗传多样性的SSR分析 [J]. 广西植物, 34(1): 10-14.]
ZHANG FM,GE S, 2002. Data analysis in population genetics. I. analysis of RAPD data with AMOVA [J]. Biodivers Sci,10(4):438-444. [张富民,葛颂, 2002. 群体遗传学研究中的数据处理方法1. RAPD数据的AMOVA分析 [J]. 生物多样性,10(4):438-444.]
ZHANG W,GONG J, JI KS, 2008. Genetic diversity for seedling orchard of Masson′s pine [J]. Mol Plant Breed,6(4):717-723. [张薇,龚佳,季孔庶, 2008. 马尾松实生种子园遗传多样性分析 [J]. 分子植物育种,6(4):717-723.]
ZHANG Y,CHU DY, JIN GQ,et al, 2010. Relationship between hybrid performance and genetic distances amongPinusmassonianaclones based on ISSR molecular marker [J]. For Res,23(2):215-220. [张一,储德裕, 金国庆,等, 2010. 马尾松亲本遗传距离与子代生长性状相关性分析 [J]. 林业科学研究,23(2):215-220.]
ZHANG Y,CHU DY, JIN GQ,et al, 2009. Molecular characterization of elite genotypes within a first generation breeding population ofPinusmassonianausing ISSR [J]. For Res, 22(6):772-778. [张一,储德裕, 金国庆,等, 2009. 马尾松1代育种群体遗传多样性的ISSR分析 [J]. 林业科学研究, 22(6):772-778.]
ZHAO Y,DAI Y,LI YP, 2012. Genetic diversity for clonal seed orchard ofPinusarmandii[J]. J NE For Univ, 40(10): 4-11. [赵杨,代毅,李玉璞, 2012. 华山松无性系种子园遗传多样性分析 [J]. 东北林业大学学报, 40(10): 4-11.]
ZHU YY,HE H,QIN X, et al, 2014. Analysis on genetic diversity for second-generation breeding parents ofPinusmassoniana[J]. J Cent S Univ For Technol, 34(9):65-69. [朱亚艳,何花,秦雪,等, 2014. 马尾松二代育种亲本遗传多样性分析 [J]. 中南林业科技大学学报, 34(9):65-69.]
Analysis of genetic structure and its related factors ofPinusmassonianafrom different populations by ISSR marker
DU Ming-Feng1,2,3, DING Gui-Jie1,2*
( 1.CollegeofForestry,GuizhouUniversityGuiyang, Guizhou 550025, China; 2.ResearchCentreofForestResourcesandEnvironment,Guiyang 550025, China; 3.SchoolofKarstScience,GuizhouNormalUniversity, Guizhou 550001, China )
ISSR markers were used to study the genetic diversity, genetic structure and genetic distance of three populations ofPinusmassonianafrom Guangxi Zhuang Autonomous Region and Guizhou provinces. Of the 100 primers screened,12 primers were selected and they generated 92 stable bands, among which 86 bands were polymorphic. The result of POPGENE indicated that Nei’ s gene diversities (h) at population level were from 0.182 4 to 0.206 5, Shannon’s information indexes (I) at population level were from 0.281 8 to 0.317 8, which suggested that the polymorphism level of three populations had small differences. At species level, the percentage of polymorphic bands (PPB) were 93.48%, Nei’s gene diversities (H) was 0. 284 2, Shannon’s information index (I) was 0.438 1. All those showed thatP.massonianahad a higher genetic diversity at the species level. The analysis of genetic structure indicated that the average coefficient gene differentiation (Gst) was 0.315 3, which implied most of genetic variation appeared inner population. And the average number of the gene flow was 1.085 3, which indicated that there were a certain degree of gene exchanges between each population ofP.massoniana. The result of AMOVA showed that the genetic differentiation index was 0.246 (P=0.001), which indicated that the genetic differentiation was evident among different populations although there were gene exchanges between them. The UPGMA clustering and Mantel test showed that every individual inner population was first gathered for a branch. And there was significant correlation between the genetic distance and geographical distance among these three populations(r=0.972,P=0.001). Therefore, it was concluded from above that theP.massonianawas at a higher level of genetic diversity in gymnosperms; and the majority of genetic variation distributed within population; and the genetic differentiation from different populations ofP.massonianawas hardly associated with the genetic drift but maybe caused by the difference from geographical and ecological environments. This study will provide theoretical reference and scientific basis for genetic improvement and plant introduction ofP.massoniana.
Pinusmassoniana, ISSR, genetic diversity, genetic structure, genetic distance
10.11931/guihaia.gxzw201507001
2015-07-07
2015-08-20
国家自然科学基金 (31260183);国家“863”项目 (2011AA10020301);贵州省重大专项(黔科合重大专项字 [2012]6001号)[Supported by the National Natural Science Foundation of China (31260183); National 863 Program (2011AA10020301); Special Key Program of Guizhou Province ([2012]6001)]。
杜明凤(1979-),女,贵州惠水人,博士研究生,副教授,主要从事森林培育、植物分子遗传与育种研究,(E-mail)dmf1979@126.com。
丁贵杰,教授,博士生导师,主要从事森林培育研究,(E-mail) gjdinggzu@ 126.com。
Q943;S718.3
A
1000-3142(2016)09-1068-08
杜明凤,丁贵杰. 不同种源马尾松ISSR遗传结构及影响因素分析 [J]. 广西植物, 2016, 36(9):1068-1075
DU MF, DING GJ. Analysis of genetic structure and its related factors ofPinusmassonianafrom different populations by ISSR marker [J]. Guihaia, 2016, 36(9):1068-1075
