基质辅助激光解析电离飞行时间质谱在革兰阴性杆菌对β内酰胺类抗生素耐药性检测中的应用进展
2016-09-26李媛睿
李媛睿, 俞 静, 刘 瑛
·综述·
基质辅助激光解析电离飞行时间质谱在革兰阴性杆菌对β内酰胺类抗生素耐药性检测中的应用进展
李媛睿, 俞 静, 刘 瑛
基质辅助激光解析电离飞行时间质谱; 革兰阴性杆菌; β内酰胺类抗生素; 耐药性
β内酰胺类抗生素是一类临床应用最为广泛的抗菌药物,它通过干扰细菌细胞壁肽聚糖的交联结构发挥杀菌作用。该类抗生素包括青霉素及其衍生物、头孢菌素、单酰胺环类和碳青霉烯类等,其中尤以碳青霉烯类抗生素的抗菌活性最强。2013年CHINET细菌耐药性监测数据显示:肠杆菌科细菌对头孢菌素的耐药率约为20%,对碳青霉烯类抗生素的耐药率接近7%;不发酵糖革兰阴性杆菌对头孢菌素和碳青霉烯类抗生素耐药率均接近50%[1]。耐药菌株不断出现给临床用药的选择带来很大困惑。针对上述细菌耐药性的严峻形势,需要应用新技术更加快速、准确地检测细菌及其对抗菌药物的耐药性,从而及时对患者进行合理的抗生素治疗,对医院内传播的耐药菌株采取有效的控制措施。
基质辅助激光解析电离飞行时间质谱(matrixassisted laser desorption ionization time of flight mass spectrometry,MALDI-TOF MS)是最新发展起来的一种快速检测生物大分子的软电离质谱技术,可用于细菌和真菌鉴定,具有简便、快速、准确和经济等优点[2]。近年来MALDI-TOF MS技术在微生物领域内的应用范围不断扩展,它不仅是细菌鉴定的有效工具,也为细菌耐药性检测研究提供了新方法。细菌对抗生素产生耐药性的原因包括:天然固有耐药、后天耐药基因突变和基因转移。革兰阴性杆菌对β内酰胺类抗生素产生的耐药机制主要有以下3种:产β内酰胺酶、外膜孔道蛋白结构改变和外排泵系统的过度表达等。本文从上述三方面综述了MALDI-TOF MS在革兰阴性杆菌β内酰胺类抗生素耐药性检测方面相关的研究进展,并展望该技术未来的应用前景。
1 应用MALDI-TOF MS检测β内酰胺酶
1.1 β内酰胺酶活性的检测
产β内酰胺酶是导致细菌对β内酰胺类抗生素耐药的主要原因,临床上重要的β内酰胺酶包括青霉素酶、超广谱β内酰胺酶(ESBL)、头孢菌素酶以及碳青霉烯酶。β内酰胺酶具有解开β内酰胺类抗生素的β内酰胺环的活性,应用MALDI-TOF MS检测抗生素及其降解产物变化,可以判断细菌是否具有β内酰胺酶活性。常见β内酰胺类抗生素及其降解产物的分子式和分子量见表1。

表1 β内酰胺类抗生素及其降解产物的分子式和分子量
应用MALDI-TOF MS检测抗生素等小分子样本的常用基质有2,5-二羟基苯甲酸(DHB)和α-氰基-4-羟基肉桂酸(HCCA)[3],分子量分别为154 u和190 u。由于基质的质谱峰可能会干扰待测抗生素的质谱峰,因此需要选择合适的基质,使基质峰和样品峰之间不存在重叠。相关研究显示:美罗培南[M+H]+(384 u)与HCCA二聚物[2M+H]+(380 u)的质谱峰有重叠,应选择DHB为检测美罗培南的基质[4];检测氨苄西林、哌拉西林、头孢噻肟、头孢他啶、亚胺培南和厄他培南等抗生素,均可选择HCCA为基质[5-7]。应用MALDI-TOF MS检测细菌的β内酰胺酶活性,可用Tris-HCl缓冲液和0.45% NaCl溶液作为细菌蛋白质的溶剂,其中加入SDS能有效降低上清液中细菌蛋白丰度,抑制基质信号,从而提高检测特异性[4,8]。目前市场上已有多种品牌及型号的MALDI-TOF MS仪和靶板:质谱仪通常配制MALDI离子源和线性TOF检测器,采用新型的反射式TOF检测器,能提高检测分辨率和质谱数据采集速度;AnchorChip质谱靶板表面疏水作用基团的中央具有亲水基团,溶剂蒸发时样品液滴收缩增加了靶板点样区的蛋白质丰度,相对常规靶板其检测灵敏度提高10~100倍[6]。
不同文献报道的应用MALDI-TOF MS检测β内酰胺酶活性的方法基本相同:将过夜培养的纯菌落配成菌悬液,加入一定浓度的β内酰胺类抗生素溶液,混匀后置于37 ℃孵育1~4 h,离心,取上清液和相应基质依次点至靶板,利用MALDITOF MS进行检测,根据抗生素峰是否缺失或峰强度降低及降解产物质谱峰是否出现,来判断待测菌株是否具有相应的酶活性。表2中显示,MALDI-TOF MS在1~4 h内可检测革兰阴性杆菌β内酰胺酶活性,除生物梅里埃公司的VITEK MS型质谱仪外,应用布鲁克公司的Ultraflex、Microflex和Autoflex型质谱仪检测β内酰胺酶活性的灵敏度和特异度达96%~100%,与传统方法相比具有操作简单直接,快速准确等优点,将来有可能成为临床微生物实验室的常规检测方法[9]。应用MALDI-TOF MS检测β内酰胺酶活性存在一定局限性:由于采用的基质和缓冲液不同,造成各实验室所得抗生素及降解产物质谱峰分子量存在差异,这就需要设置各自实验室的阳性和阴性对照,作为检测结果的判断依据[10];仅能够检测耐药株是否水解β内酰胺类抗生素,而不能判断菌株对抗生素的耐药程度[11]。
最近有研究报道,采用MALDI-TOF MS技术联合SepsityperTMkit试剂盒或分离胶促凝管,不但可直接检测阳性血培养中细菌,还可快速检测阳性血培养中细菌的β内酰胺酶活性,对临床调整抗生素治疗方案、控制血流感染有重要意义[12-13]。
1.2 β内酰胺酶的检测
β内酰胺酶是一类由细菌产生的酶蛋白质。将细菌蛋白质提取物用MALDI-TOF MS检测,可直接鉴定细菌是否产生β内酰胺酶特异的质谱峰,MALDI-TOF MS的检测结果可通过肽质量指纹谱(peptide mass fingerprinting,PMF) 分析得到验证。PMF分析是目前蛋白质组研究中鉴定蛋白质较为常用的方法:首先提取细菌蛋白质进行十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electropheresis,SDS-PAGE)或双向凝胶电泳(two-dimensional electrophoresis, 2-DE);再切取凝胶中β内酰胺酶蛋白条带,经胰蛋白酶消化后产生肽段,采用串联质谱(MALDI-TOF/TOF MS)或液相色谱质 谱(Liquid chromatography -mass spectrometry,LC-MS)鉴定出肽段序列;最后将肽段数据导入Mascot软件,搜索匹配蛋白质种类。Mascot是目前使用最广泛的蛋白质鉴定检索软件, 可以实现从质谱数据到蛋白质的鉴定。
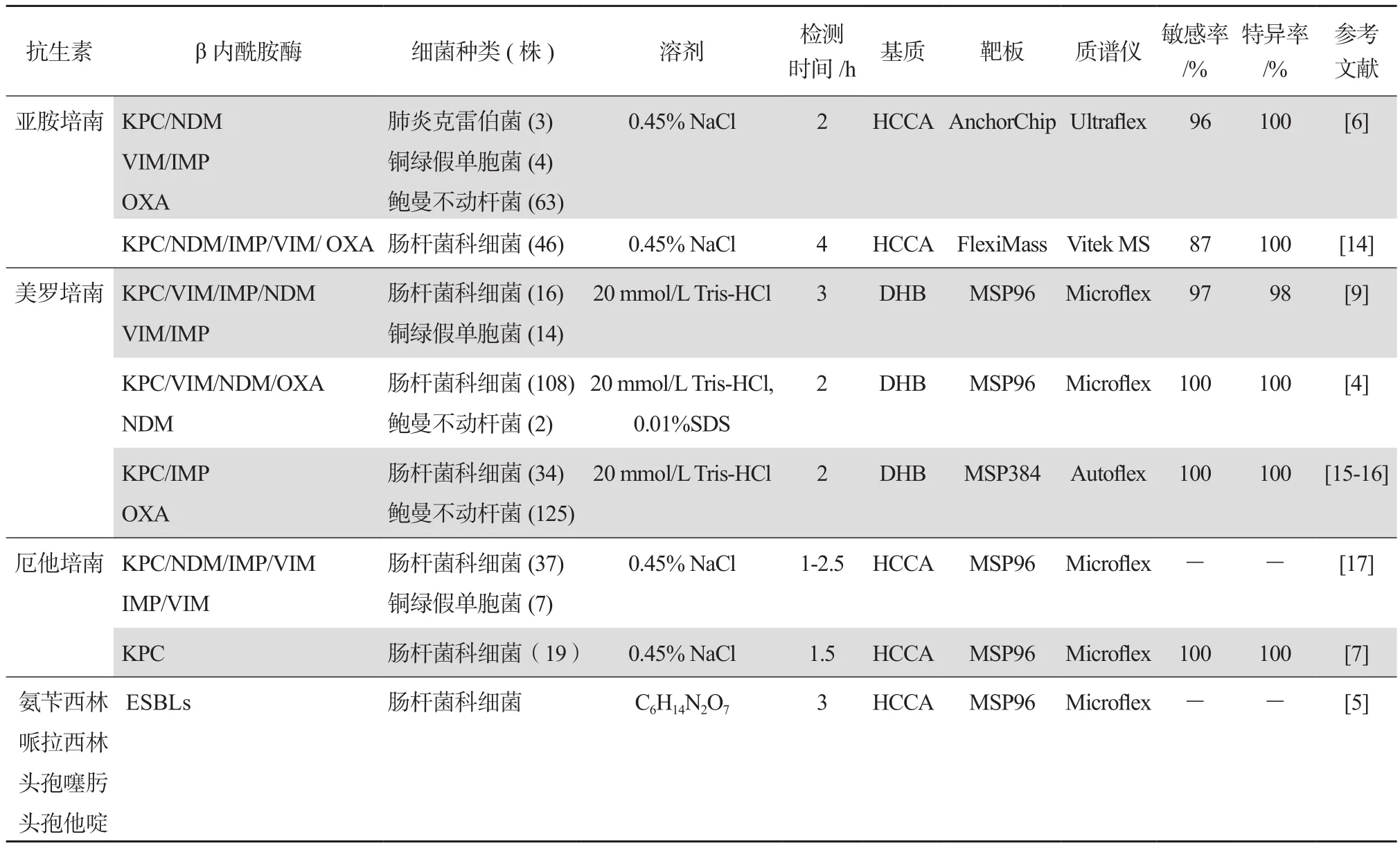
表2 应用MALDI-TOF MS检测革兰阴性杆菌β内酰胺酶活性
CAMARA等[18]在含氨苄西林的LB肉汤中培养耐药大肠埃希菌,通过甲酸和异丙醇裂解提取细菌蛋白质,将上清液点样至靶板,干燥后再点样基质芥子酸 (sinapic acid, SA),采用MALDI -TOF MS检测发现在29 ku处存在特征质谱峰,通过PMF分析验证该峰为β内酰胺酶。PAPAGIANNITSIS等[19]应用MALDI-TOF MS检测产头孢菌素酶弗氏枸橼酸杆菌的提取物,发现在39 850 u处有特异性质谱峰,经PMF分析确证为CMY-2型头孢菌素酶。LAU等[20]利用MALDI -TOF MS检测产碳青霉烯酶的肺炎克雷伯菌存在11 109 u特征质谱峰,经PMF分析鉴定为blaKPC型碳青霉烯酶,由于肺炎克雷伯菌pKpQIL质粒上的blaKPC基因与Tn4401转位子相连,可将blaKPC基因通过质粒转移接合至大肠埃希菌DH10B,采用MALDI -TOF MS和DNA序列分析均能证明发生转移结合的大肠埃希菌存在11 109 u特征性质谱峰和Tn4401转位子。
1.3 β内酰胺酶基因的SNP测序
PUSCH等[21]建 立 了 以MALDI-TOF MS为基础的单核苷酸多态性(single nucleotide polymorphism,SNP)基因分型方法:首先通过PCR扩增出一段含有SNP的DNA(SNP位点前后各50 bp左右),用SNP酶去除掉PCR体系中剩余的脱氧核糖核苷三磷酸(dNTP)和引物;再加入单碱基延伸引物,其3'末端碱基紧挨SNP位点,采用4种ddNTP替代dNTP;然后探针在SNP位点处仅延伸一个碱基,连接上的ddNTP与SNP位点的等位基因对应;最后应用MALDI-TOF MS检测延伸产物与未延伸引物间的分子量差异,确定该位点处碱基,能够推测DNA碱基序列的变化。
有相关研究报道[22-23],基于MALDI-TOF MS 的SNP测序技术能清楚区分细菌质粒携带的β内酰胺酶基因上位点突变的质谱峰差异,从而准确检测大肠埃希菌的SHV型和TEM型β内酰胺酶基因。基于MALDI-TOF MS的SNP测序是一种快速检测β内酰胺酶基因的新方法,其检测结果高度可靠、试剂成本低、适合于自动化操作且在一次实验中可检测多位点突变。
2 应用MALDI-TOF MS检测膜孔蛋白
革兰阴性细菌的外膜上存在多种外膜蛋白(outer membrane protein, Omp),其中膜孔蛋白(porin)对抗生素具有选择通透性。若膜孔蛋白结构发生改变或者缺失,外膜通透性减弱,运输至细菌壁膜间隙的β内酰胺类抗生素大量减少,使细菌对抗生素耐药。
PMF是一种直接、有效的蛋白质组鉴定技术,近年来质谱技术已经应用于细菌外膜蛋白质组研究[24]。通常采用SDS-PAGE或2-DE电泳分离细菌外膜蛋白,切取凝胶中目的蛋白条带,胰蛋白酶解蛋白质并纯化肽段,再进行质谱仪检测,最后在Mascot数据库中检索PMF代表的膜孔蛋白组分。应用质谱仪检测不同革兰阴性杆菌的膜孔蛋白的结果如表3所示[25-27]。最新一项研究建立了应用MALDI-TOF MS快速检测大肠埃希菌与肺炎克雷伯菌膜孔蛋白的方法,结果表明肺炎克雷伯菌质谱图中38 ku和36 ku处峰缺失,与SDS-PAGE上42 ku的OmpK36 ku和40 ku的OmpA条带缺失相对应;大肠埃希菌质谱图中38 ku、37 ku和35 ku处峰缺失,与SDS-PAGE上41 ku的OmpC、40 ku的OmpF和38 ku的OmpA条带缺失相对应[28]。该方法简化了操作流程,且分辨率较高,可用于细菌膜孔蛋白缺失的快速筛查。

表3 应用MALDI-TOF MS检测革兰阴性杆菌膜孔蛋白和外排泵相关蛋白质
3 MALDI-TOF MS检测外排泵系统
革兰阴性杆菌的外排泵系统通过主动外排多种药物,形成了低水平和非特异的耐药性。革兰阴性杆菌最主要的外排泵系统为耐药结节分化超家族(resistance nodulation cell division family,RND),它是一组跨越细菌内膜和外膜的蛋白质,由内膜转运蛋白、膜融合蛋白和外膜通道蛋白构成三聚体。应用质谱技术联合蛋白质分离方法和生物信息学工具,不仅能检测细菌外膜的膜孔蛋白,也能检测细菌外排泵系统中的蛋白质组分。
XU等[25]对提取的大肠埃希菌总蛋白进行2-DE电泳,提纯处理凝胶中蛋白质的肽段后,应用MALDI-TOF MS检测并检索,结果显示为外排泵系统中外膜通道蛋白TolC和YhiU,通过蛋白质印迹法(Western blotting)验证耐氨苄西林大肠埃希菌TolC和YhiU的表达量增加。在铜绿假单胞菌蛋白质组的研究中[26,30],应用MALDI-TOF MS能成功检测到外排泵系统中膜融合蛋白MexA和外膜通道蛋白OprM,经Western blotting证实MexA和OprM表达上调导致铜绿假单胞菌产生耐药性。应用MALDI-TOF MS检测不同革兰阴性杆菌的外排泵相关蛋白质结果如表3所示[25-26,29-30]。
4 展望
MALDI-TOF MS具有自动化、高通量、操作简便、样本用量少和检测准确度高等特点。它将微生物检测时间由以小时计算推进到以分钟计算,应用范围从微生物的快速鉴定扩大到耐药性检测。随着质谱技术的不断发展、操作流程的简化、微生物PMF和耐药相关蛋白质组数据库的丰富和扩展,我们相信临床微生物实验室未来也将具备检测分析细菌蛋白质组的能力。基于微生物PMF和蛋白质组图谱的MALDI-TOF MS技术是微生物领域的一场革命,这一技术未来可应用于细菌的分子流行病调查、细菌毒力因子和血药浓度检测,甚至可用来指导药物分子靶点的筛选和新药的设计,为患者提供个体化的治疗方案,它将全面推进微生物快速诊断与耐药性检测的发展。
[1]胡付品, 朱德妹, 汪复,等. 2013年中国CHINET细菌耐药性监测[J]. 中国感染与化疗杂志, 2014,( 5): 365-374.
[2]WIESER A, SCHNEIDER L, JUNG J, et al. MALDI-TOF MS in microbiological diagnostics-identification of microorganisms and beyond( mini review)[J]. Appl Microbiol Biotechnol,2012, 93(3): 965-974.
[3]RUELLE V, El MOUALIJ B, ZORZI W, et al. Rapid identification of environmental bacterial strains by matrixassisted laser desorption/ionization time-of-flight mass spectrometry[J]. Rapid Commun Mass Spectrom, 2004, 18 (18): 2013-2019.
[4]HRABAK J, STUDENTOVA V, WALKOVA R, et al. Detection of NDM-1, VIM-1, KPC, OXA-48, and OXA-162 carbapenemases by matrix-assisted laser desorption ionizationtime of flight mass spectrometry[J]. J Clin Microbiol, 2012, 50 (7): 2441-2443.
[5]SPARBIER K, SCHUBERT S, WELLER U, et al. Matrixassisted laser desorption ionization-time of flight mass spectrometry-based functional assay for rapid detection of resistance against beta-lactam antibiotics[J]. J Clin Microbiol,2012, 50(3): 927-937.
[6]KEMPF M, BAKOUR S, FLAUDROPS C, et al. Rapid detection of carbapenem resistance in Acinetobacter baumannii using matrix-assisted laser desorption ionization-time of flight mass spectrometry[J]. PLoS One, 2012, 7(2): e31676.
[7]胡燕燕, 孙谦, 蔡加昌,等. 基质辅助激光解析/电离飞行时间质谱仪检测KPC型碳青霉烯酶的研究[J]. 中华微生物学和免疫学杂志, 2012, 32(6): 561-565.
[8]GRANT DC, HELLEUR RJ. Surfactant-mediated matrixassisted laser desorption/ionization time-of-flight mass spectrometry of small molecules[J]. Rapid Commun Mass Spectrom, 2007, 21(6): 837-845.
[9]HRABAK J, WALKOVA R, STUDENTOVA V, et al. Carbapenemase activity detection by matrix-assisted laser desorption ionization-time of flight mass spectrometry[J]. J Clin Microbiol, 2011, 49(9): 3222-3227.
[10]LEE W, CHUNG HS, LEE Y, et al. Comparison of matrixassisted laser desorption ionization-time-of-flight mass spectrometry assay with conventional methods for detection of IMP-6, VIM-2, NDM-1, SIM-1, KPC-1, OXA-23, and OXA-51 carbapenemase-producing Acinetobacter spp., Pseudomonas aeruginosa, and Klebsiella pneumoniae[J]. Diagn Microbiol Infect Dis, 2013, 77(3): 227-230.
[11]HRABAK J, CHUDACKOVA E, WALKOVA R. Matrixassisted laser desorption ionization-time of flight( malditof) mass spectrometry for detection of antibiotic resistance mechanisms: from research to routine diagnosis[J]. Clin Microbiol Rev, 2013, 26(1): 103-114.
[12]CARVALHAES CG, CAYO R, VISCONDE MF, et al. Detection of carbapenemase activity directly from blood culture vials using MALDI-TOF MS: a quick answer for the right decision[J]. J Antimicrob Chemother, 2014, 69(8): 2132-2136.
[13]JUNG JS, POPP C, SPARBIER K, et al. Evaluation of MALDI-TOF MS for rapid detection of beta-lactam resistance in Enterobacteriaceae derived from blood cultures[J]. J Clin Microbiol, 2014,52(3):924-930.
[14]KNOX J, JADHAV S, SEVIOR D, et al. Phenotypic detection of carbapenemase-producing Enterobacteriaceae by use of matrix-assisted laser desorption ionization-time of flight mass spectrometry and the Carba NP Test[J]. J Clin Microbiol, 2014,52(11): 4075-4077.
[15]王利君, 范艳艳, 王玫,等. MALDI-TOF MS检测产碳青霉烯酶肠杆菌科细菌的应用价值[J]. 中华医学杂志, 2013, 93 (26): 2079-2081.
[16]王利君, 范艳艳, 王玫. 产OXA-23鲍曼不动杆菌的基质辅助激光解析电离飞行时间质谱检测研究[J]. 国际检验医学杂志,2013, 34(19): 2587-2589.
[17]BURCKHARDT I, ZIMMERMANN S. Using matrix-assisted laser desorption ionization-time of flight mass spectrometry to detect carbapenem resistance within 1 to 2.5 hours[J]. J Clin Microbiol, 2011, 49(9): 3321-3324.
[18]CAMARA JE, HAYS FA. Discrimination between wild-type and ampicillin-resistant Escherichia coli by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry[J]. Anal Bioanal Chem, 2007, 389(5): 1633-1638.
[19]PAPAGIANNITSIS CC, KOTSAKIS SD, TUMA Z, et al. Identification of CMY-2-type cephalosporinases in clinical isolates of Enterobacteriaceae by MALDI-TOF MS[J]. Antimicrob Agents Chemother, 2014, 58(5): 2952-2957.
[20]LAU AF, WANG H, WEINGARTEN RA, et al. A rapid matrix-assisted laser desorption ionization-time of flight mass spectrometry-based method for single-plasmid tracking in an outbreak of carbapenem-resistant Enterobacteriaceae[J]. J Clin Microbiol, 2014, 52(8): 2804-2812.
[21]PUSCH W, WURMBACH JH, THIELE H, et al. MALDITOF mass spectrometry-based SNP genotyping[J]. Pharmacogenomics, 2002, 3(4): 537-548.
[22]STURENBURG E, STORM N, SOBOTTKA I, et al. Detection and genotyping of SHV beta-lactamase variants by mass spectrometry after base-specific cleavage of in vitro-generated RNA transcripts[J]. J Clin Microbiol, 2006, 44(3): 909-915.
[23]IKRYANNIKOVA LN, SHITIKOV EA, ZHIVANKOVA DG,et al. A MALDI TOF MS-based minisequencing method for rapid detection of TEM-type extended-spectrum beta-lactamases in clinical strains of Enterobacteriaceae[J]. J Microbiol Methods,2008, 75(3): 385-391.
[24]HINES HB. Microbial proteomics using mass spectrometry[J]. Methods Mol Biol, 2012, 881: 159-186.
[25]XU C, LIN X, REN H, et al. Analysis of outer membraneproteome of Escherichia coli related to resistance to ampicillin and tetracycline[J]. Proteomics, 2006, 6(2): 462-473.
[26]PENG X, XU C, REN H, et al. Proteomic analysis of the sarcosine-insoluble outer membrane fraction of Pseudomonas aeruginosa responding to ampicilin, kanamycin, and tetracycline resistance[J]. J Proteome Res, 2005, 4(6): 2257-2265.
[27]DUPONT M, PAGES J M, LAFITTE D, et al. Identification of an OprD homologue in Acinetobacter baumannii[J]. J Proteome Res, 2005, 4(6): 2386-2390.
[28]胡燕燕. 耐碳青霉烯革兰阴性杆菌分子流行病学及MALDITOF MS在碳青霉烯耐药决定子快速检测中的应用研究[D].浙江大学, 2014.
[29]DOS SANTOS KV, DINIZ CG, VELOSO LDE C, et al. Proteomic analysis of Escherichia coli with experimentally induced resistance to piperacillin/tazobactam[J]. Res Microbiol,2010, 161(4): 268-275.
[30]章喻军, 王三英. 铜绿假单胞菌外膜亚蛋白质组图谱的建立[J]. 厦门大学学报(自然科学版), 2008,47(1): 111-115.
Matrix-assisted laser desorption ionization time of flight mass spectrometry for detection of β-lactam resistance in gram-negative bacilli
LI Yuanrui, YU Jing, LIU Ying. (Department of Laboratory Medicine, Xinhua Hospital, Shanghai Jiao Tong University School of Medicine, Shanghai 200092, China)
R378.2
A
1009-7708(2016)02-0229-06
10.16718/j.1009-7708.2016.02.019
2015-06-18
2015-07-15
上海交通大学医学院附属新华医院检验科临床微生物室,上海 200092。
李媛睿(1990—),女,硕士研究生,主要从事细菌耐药机制研究。
刘瑛,E-mail: lywjw0129@163.com。
