活性中心突变型胃蛋白酶原的酵母表达及高效激活研究❋
2016-08-13高晓梦张艳芳徐甲坤梁兴国
刘 宇, 郭 辉, 高晓梦, 张艳芳, 徐甲坤, 董 平❋❋, 梁兴国
(1. 中国海洋大学食品科学与工程学院, 山东 青岛 266003;2. 中国水产科学研究院黄海水产研究所, 山东 青岛 266071)
活性中心突变型胃蛋白酶原的酵母表达及高效激活研究❋
刘宇1, 郭辉1, 高晓梦1, 张艳芳1, 徐甲坤2, 董平1❋❋, 梁兴国1
(1. 中国海洋大学食品科学与工程学院, 山东 青岛 266003;2. 中国水产科学研究院黄海水产研究所, 山东 青岛 266071)
摘要:针对活性中心突变型胃蛋白酶原难以被激活且稳定性差的问题,本文研究了活性中心突变对胃蛋白酶原激活及其稳定性的影响。根据猪胃蛋白酶原A的基因序列设计引物,通过RT-PCR和重叠延伸PCR法分别获得野生型(rPG)和突变型(D32A和D215A)胃蛋白酶原基因片段,其中突变型胃蛋白酶原分别为活性中心关键氨基酸天冬氨酸(D)突变为丙氨酸(A)。将野生型和突变型胃蛋白酶原分别在酵母中成功表达,研究其激活、激活后的稳定性和活性情况。研究表明,可以成功地在酵母中表达并纯化野生型和突变型重组猪胃蛋白酶原rPG、D32A和D215A。在pH=2.0条件下,rPG在5 min内即可全部激活,且在37℃具有良好的稳定性,而D32A和D215A需24 h以上才可激活。通过在激活反应中加入野生型胃蛋白酶,D32A和D215A的激活时间可缩短至1 h,虽然激活过程有部分酶原发生降解,但激活后获得的突变型胃蛋白酶可稳定3 d,激活后的突变型胃蛋白酶无蛋白水解活性。研究结果表明,活性中心关键氨基酸突变后的胃蛋白酶原仍可激活,虽然在激活过程中胃蛋白酶原的稳定性差,但激活后产生的胃蛋白酶稳定性良好,这为进一步研究突变型胃蛋白酶提供了可能和保证。
关键词:猪胃蛋白酶原; 毕赤酵母表达; 突变; 酶原激活
引用格式:刘宇, 郭辉, 高晓梦, 等. 活性中心突变型胃蛋白酶原的酵母表达及高效激活研究 [J].中国海洋大学学报(自然科学版), 2016, 46(7): 54-61.
LIU Yu, GUO Hui, GAO Xiao-Meng, et al. Expression and activation of recombinant porcine pepsinogen with mutated active site [J].Periodical of Ocean University of China, 2016,46(7): 54-61.
胃蛋白酶是天冬氨酸蛋白酶家族的消化性蛋白酶[1],科学家对胃蛋白酶的关注超过了80年[2-3],这不仅是因为胃蛋白酶是重要的蛋白酶,更重要的是胃蛋白酶与生物体的消化代谢密切相关。目前,关于胃蛋白酶的研究主要集中在两方面:一方面为胃蛋白酶作为工具酶来制备多肽或胶原蛋白;另一方面为对胃蛋白酶系的完善,即不同物种中胃蛋白酶的提取纯化,探索其水解蛋白质的生化特性[4-6],以期找到丰富胃蛋白酶的工具酶性质。为了更好的利用胃蛋白酶,长久以来科学家们一直在努力阐明其作用机理[7-8]。
目前胃蛋白酶降解蛋白的活性中心关键氨基酸已经确定,为D32和D215,这一机理是通过X射线衍射分析酶和蛋白的结晶推测所得[9-10]。在酶活性机理的研究中,X射线衍射法无疑是最可靠和可信的方法之一,然而X射线衍射法在实际操作中困难重重,获得相应实验结果往往需要丰富的经验和漫长的探索时间。因此,近年来广受关注的蛋白质工程,成为研究酶必需基团和活性中心的又一重要方法[11-13]。这一技术主要是利用基因定点突变技术将酶中一个或几个氨基酸突变为其它氨基酸,再测定其活性来推测被置换的氨基酸是否为酶活性所必需。
使用蛋白质工程技术研究胃蛋白酶作用机理的过程中,需要首先获得酶原,这一方面是因为胃蛋白酶在生物体内其以酶原形式分泌,另一方面处于激活状态的酶稳定性大大弱于酶原,在表达纯化等过程中易于受到其他因素的影响,发生失活。因此,通常首先获取酶原,然后再对其进行激活处理[14-16]。胃蛋白酶原的激活是个自激活的过程,当pH<5.0时,酶原自动激活,转变为有生物活性的胃蛋白酶[17-18]。然而研究表明[19],胃蛋白酶原的激活需要D32和D215的共同参与,当D32变为A时,酶原不仅不能被激活,而且D32A酶原在酸性条件下不稳定,易变性降解。在这种情况下,关键氨基酸突变型酶原的激活成为解明胃蛋白酶作用机理的亟待解决的关键问题。
随着胃蛋白酶研究的持续深入,包括其可以水解糖类和核酸类物质[20-21]等新活性被陆续发现报道,对胃蛋白酶的结构和作用机理的研究也提出了越来越高的要求。因此,本文旨在系统研究猪胃蛋白酶原突变体D32A和D215A的激活可能性,借助酶促反应引发其高效激活,并对其激活后的酶的稳定性做系统评价,为早期的X射线衍射结果提供实验支持,同时也为解明胃蛋白酶的作用机理提供帮助。
1 材料与方法
1.1 实验材料及试剂
新鲜猪胃刮取胃底腺区。载体pPICZαA购自Invitrogen (San Diego, CA)。毕赤酵母(Pichiapastoris) X-33购自CICC (中国工业微生物菌种保藏管理中心)。E.coliTOP10、Trizol、TIANscript RT Kit、TIANpure Mini Plasmid Kit、质粒提取和连接试剂盒购自天根生化科技有限公司(北京)。胶回收试剂盒购自OMEGA公司。DNA聚合酶和限制性内切酶购自NEB有限公司 (USA)。荧光定量PCR试剂购自大连宝生物。DEAE Sepharose Fast Flow和Sephacryl S-200 HR购自GE Healthcare life sciences (USA)。引物购自英潍捷基(上海)贸易有限公司。
1.2 野生型猪胃蛋白酶原A及其突变体真核表达载体的构建
根据GeneBank中猪胃蛋白酶原A(PGA)的序列设计扩增其ORF(Open reading frame)的引物EX-F和EX-R(见表1),Trizol法提取猪胃组织总RNA,并利用天根TIANscript RT Kit试剂盒反转录生成第一链cDNA,以之为模板扩增PGA的ORF,具体反应体系及程序如下:20 μL反应体系包含5×buffer 4 μL,2mmol/L dNTP 2 μL,cDNA模板1 ng,上下游引物各1 μL,phusion DNA聚合酶为NEB公司产品。反应程序为98℃ 30 s;98℃ 10 s,63℃ 30 s,72℃ 45 s,30个循环;72℃ 5 min。
根据PGA基因序列,采用重叠延伸PCR法(见图1)[22]分别将其活性中心关键氨基酸D32和D215进行定点突变,将D的密码子GAT/C,突变为GCT/C,即丙氨酸A。具体引物序列见表1。
PCR产物经1.5%琼脂糖凝胶电泳后经OMEGA胶回收试剂盒回收纯化。克隆到pPICZαA载体上,热激法转化大肠杆菌TOP10。挑取阳性克隆送至华大基因测序。

图1 重叠延伸法定点突变原理示意图

引物名称Primername序列(5'-3')Sequence(5'-3')EX-FAGCTGAATTCAAGT-GGCTACTGCTGCTC(EcoRI)EX-RTATGCGGCCGCGGCCACGGGAGCCA(NotⅠ)32F1TGGAGATTGGGAGCCAGGAAAGAAC32R1GGTTGGAGGAGCCGGTGGC-AAAGATGACGGTGAAG32F2CTTCACCGTCATCTTTG-CCACCGGCTCCTCCAACC32R2GCGGGGATAGAACCAAGGCGGGAT215F1TGGAGATTGGGAGCCAGGAAAGAAC215R1GCAGAGAGGTGCCCG-TAGCCACAATGGCCTGGCAG215F2CTGCCAGGCCATTGTGGC-TACGGGCACCTCTCTGC215R2CACGATCCCAGAGGAAAAGCGATCAGART-FGTGGGTGCCCTCTGTCTACTGRT-RGAGGAGTCATCAGGGTTGAACTG
注:32F1,32R1,32F2和32R2为构建D32A突变体的引物;215F1,215R1,215F2和215R2为构建D215A突变体的引物;RT-F和RT-R为荧光定量引物。32F1,32R1,32F2和32R2 are primers for building mutant D32A. 215F1,215R1,215F2和215R2 are were primers for building mutant D215A. RT-F and RT-R are primers for RT-PCR.
1.3 重组质粒的转化、鉴定与筛选
提取上述3种重组质粒并应用PmeI使质粒线性化,电击法转化毕赤酵母X-33感受态细胞[27],30℃条件下分别经YPDS平板培养、YPD平板筛选单菌落和YPD液体增菌步骤后,提取重组酵母基因组,利用通用引物进行PCR鉴定,筛选阳性Mut+型转化子。
为得到大量目的蛋白,拟采用实时荧光定量RT-PCR来鉴定高表达的转化子。以含有目的基因的质粒为标准品,绘制标准曲线。采用TaKaRa的SYBR ® Premix Ex TaqTM(Tli RNaseH Plus)试剂盒,10 μL反应体系里包括5 μL 2×SYBR Premix Ex Taq,10 μmol/L上下游引物各0.4μL,3μL模板,1.6μL ddH2O。荧光定量PCR反应程序为:94℃ 2 min;94℃ 15 s;56℃ 15 s;72℃ 20 s,40个循环。60℃逐步升温到94℃进行溶解曲线分析。
分别随机选取7个上述鉴定为阳性的重组菌接种到10 mL YPD液体培养基中,参照李圣翔等[27]的方法进行0.5%甲醇诱导表达,于诱导表达4 d时提取菌液总RNA,反转录生成cDNA用于荧光定量分析。反应体系及程序同上,每个样品3个平行反应。
1.4 重组蛋白的表达纯化
将筛选出的高表达转化子按照1.3中的方法进行诱导表达,各表达1 L。表达4 d后,收集上清,60%饱和硫酸铵盐析,沉淀透析至25 mmol/L磷酸钾缓冲液(KPI,pH= 7.0,A液),上样于用A液平衡的 DEAE-Sepharose FF阴离子交换柱(2.5 cm×15 cm),A液(pH=7.0,含0.5mol/L NaCl)线性梯度洗脱,流速5 mL/min,分别收集洗脱峰,SDS-PAGE[23]电泳检测目的组分。含有目的组分的收集峰透析至A液并上样于A液平衡的Hiprep 16/60 sephacryl S-200 H凝胶柱,A液(pH=7.0,含0.15mol/L NaCl)洗脱,流速0.5 mL/min,收集洗脱峰,SDS-PAGE检测。
1.5 SDS-PAGE
参照Laemmli[23]的方法,采用不连续垂直平板电泳法对重组蛋白进行SDS-PAGE分析。其中分离胶浓度为12%,浓缩胶浓度为5%,考马斯亮蓝染色显示蛋白条带。
1.6 胃蛋白酶原的激活及其稳定性
所有操作皆在冰上进行。对于野生型胃蛋白酶原,在90 μL 0.5 mg/mL胃蛋白酶原溶液中加入10 μL 0.1 mol/L HCl,使溶液pH=2.0,25 ℃激活一定时间后,用1 mol/L NaAc(pH=9.6)终止反应,使溶液的pH=5.3。
对突变型胃蛋白酶原的激活采用如下2种方法:(1)参照上述野生型酶原的激活方法;(2)酶促激活方法:在90 μL胃蛋白酶原溶液中加入10 μL 1 mol/L HCl,使溶液的pH=2.0,同时加入一定量的重组猪胃蛋白酶(rP),25℃激活一定时间后,按上述方法终止反应。
激活后胃蛋白酶的稳定性采用如下方法进行分析:将按上述方法激活后的酶液的pH分别调至2.0、3.0、4.0和5.0,放置在37 ℃水浴锅中,分别于12、24、36和48 h取样,加入20 μL 0.1 mol/L NaOH和10 μL 4×蛋白电泳上样缓冲液终止反应,SDS-PAGE电泳检测。
1.7 胃蛋白酶活力测定
由于时间的限制,本文任然存在着不足之处:作者分析了40则语料,但是作为例子,呈现在论文中的只有6则;对于语料的分析基于作者自身的能力,可能存在分析不足的情况;对于广告语类型的划分,是根据作者自身的认知水平。
激活后胃蛋白酶活力的测定采用Anson法[24],酶活单位定义为在pH=2.0、37 ℃条件下,以血红蛋白为底物,每分钟使上清产生0.001吸光值所需要的酶量为一个酶活力单位。
2 结果与分析
2.1 猪胃蛋白酶原A野生型及其突变体的毕赤酵母表达纯化
本研究以猪胃组织总RNA反转录形成的cDNA为模板,通过RT-PCR及重叠延伸PCR法,分别获得了野生型猪胃蛋白酶原A(PGA)和突变体D32A和D215A基因片段,并分别克隆至pPICZαA载体,转化毕赤酵母X-33感受态细胞后,通过基因组PCR法,鉴定阳性转化子(结果未列出)。本研究选择的X-33为Mut+型分泌型表达菌[25],蛋白表达时需要定期加入甲醇做为诱导剂进行诱导表达,同时分泌型表达便于后续蛋白纯化。
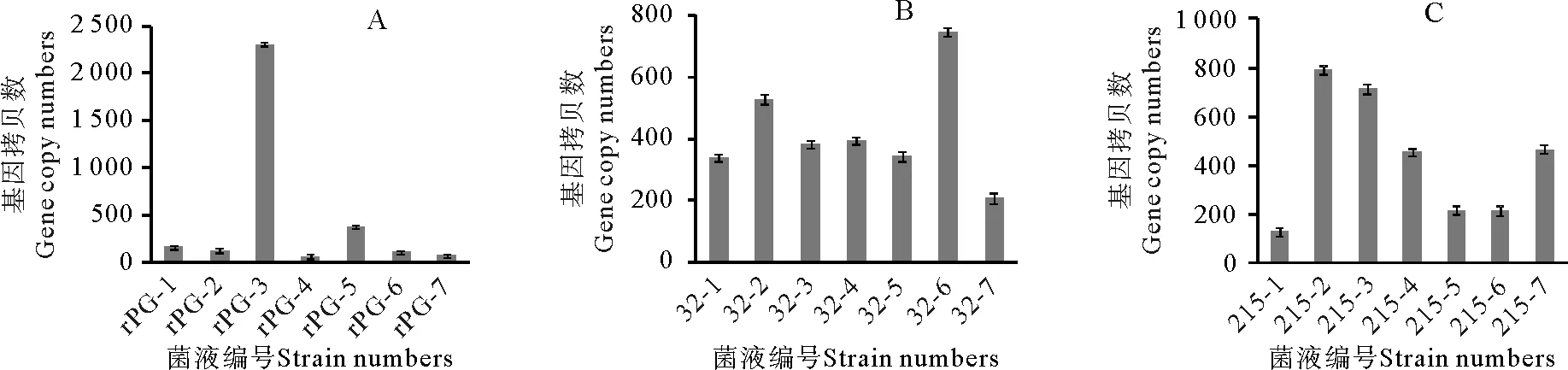
为得到大量的目的蛋白,本研究采用实时荧光定量RT-PCR法检测诱导表达4 d后转化子在RNA水平的表达情况,从阳性菌中筛选出高表达的转化子用于后续表达。根据荧光定量PCR结果,最终选择X-33-pPICZα A-P-3、X-33-pPICZα A-D32A-6和X-33-pPICZα A-D215A-2重组菌进行后续表达实验(见图2)。
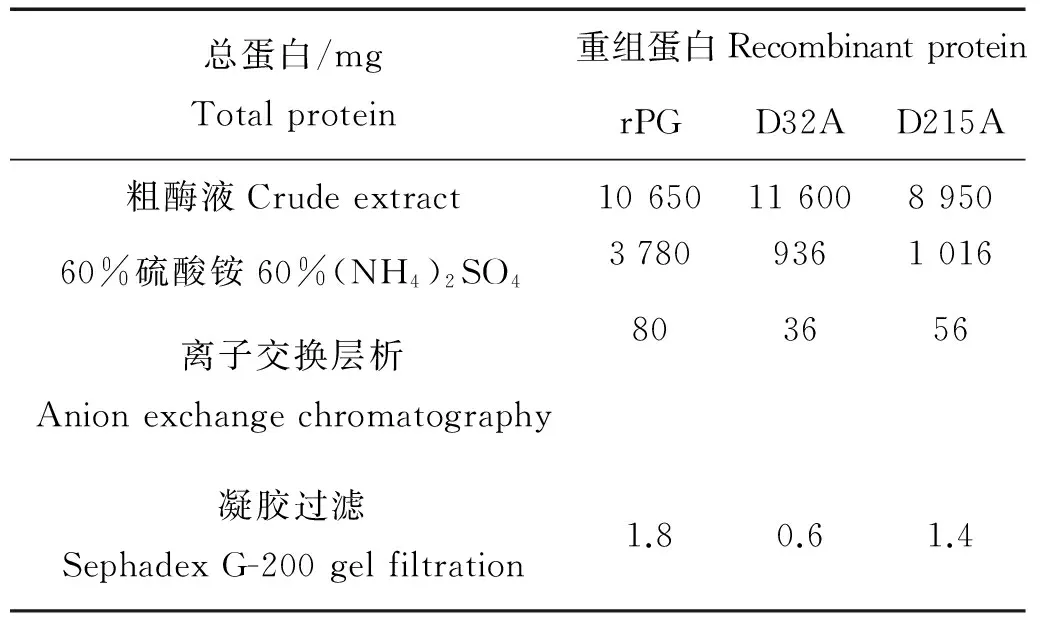
将甲醇诱导表达4 d后的上清液经硫酸铵盐析、DEAE-Sepharose FF阴离子交换柱和Hiprep 16/60 sephacryl S-200 H凝胶柱纯化后,得到重组蛋白rPG、mPG1(D32A)和mPG2(D215A)。每一步的纯化结果见表2和图3。经纯化后,在SDS-PAGE电泳图中可以看出,得到了较为单一的条带,分子量与预期分子量一致。实验结果表明,野生型猪胃蛋白酶原和突变型猪胃蛋白酶原在毕赤酵母X-33中成功地得到了表达。
2.2 重组猪胃蛋白酶原的激活
由图4可知,重组野生型rPG在pH=2.0条件下可迅速激活,5 min内即可全部激活,且可在37℃稳定存在4 d而不降解,且酶活无明显变化,与现有研究相符[26]。这一实验结果表明本研究中所采用的表达纯化方案可行,重组野生型猪胃蛋白酶原与天然猪胃蛋白酶原具有相同的激活稳定性,因此后续采用相同的方法表达纯化得到的突变体可用于激活稳定性研究。
(A.野生型;B.D32A突变型;C.D215A突变型。A. Wild-type; B. Mutant D32A; C. Mutant D215A.)
总蛋白/mgTotalprotein重组蛋白RecombinantproteinrPGD32AD215A粗酶液Crudeextract1065011600895060%硫酸铵60%(NH4)2SO437809361016离子交换层析Anionexchangechromatography803656凝胶过滤SephadexG-200gelfiltration1.80.61.4
采用1.6(1)的方法,即与野生型胃蛋白酶相同的激活方法,发现突变体D32A和D215A在pH=2.0条件下可以激活,但速度极慢,需24 h,且稳定性极差,激活后的酶的量不足酶原的1%(见图5A)。由于突变酶原在酸性条件下不稳定,为了提高激活效率,采用1.6(2)中阐述的改良的酶促激活方法,结果表明当在突变体D32A和D215A的激活反应体系中加入微量的野生型胃蛋白酶(rP)后(0.4 U rP,质量为突变体的0.3%),突变体D215A的激活速度显著加快,电泳分析表明在25oC条件下1 h内突变酶原的条带消失,被激活后酶的条带出现,即突变酶原完全被激活为酶(见图5B)。在突变型胃蛋白酶原的激活体系中加入不同含量的rP后发现,当rP的含量增加至1 U时,突变型胃蛋白酶原的稳定性下降,几乎看不到激活后的酶的条带;而将rP的含量减少至0.01 U时,酶促激活反应仍可在1 h内完成,且突变型胃蛋白酶的稳定性更好(见图6)。
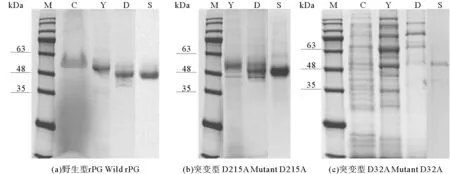
(M:蛋白分子量标准Protein marker;C:粗酶液Crude extract;Y:60%硫酸铵沉淀60% Ammonium sulfate precipitation;D:DEAE层析DEAE-FF;S:丙烯酰胺葡聚糖凝胶层析Sephacryl S-200 H.)
图3SDS-PAGE(12%)电泳检测纯化结果
Fig.3Analysis of purification results by 12% of SDS-PAGE
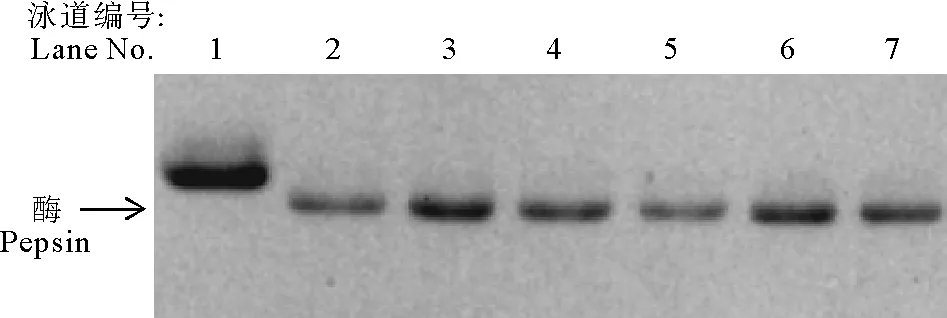
(1为未激活的rPG;2为激活5 min;3为激活30 min;4~7分别为激活后37℃放置1、2、3和4 d。1.rPG; 2. rPG activated for 5 min; 3. rPG activated for 30 min; 4-7.activated rP stay at 37℃ for 1 d, 2 d, 3 d and 4 d, respectively.)
图4重组rPG的激活
Fig.4Activation of rPG
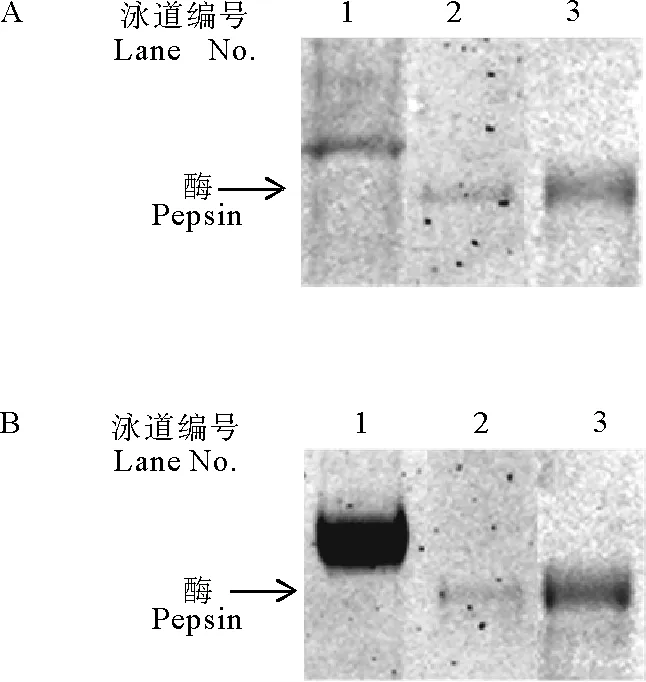
(A为突变体D32A在pH=2.0的条件下自然激活和酶促激活;B为突变体D215A在pH=2.0的条件下自然激活和酶促激活。其中1为未激活的酶原,2为自然激活,3为酶促激活。A. D32A activated at pH=2.0 under different conditions. B. D215A activated at pH=2.0 under different conditions. 1.Unactivated pepsinogen; 2. Natural activation; 3. Enzymatic activation.)
图5突变体D32A和D215A的激活
Fig.5Activation of mutant pepsinogen D32A and D215A

(1为未激活的D215A;2~4分别为D215A在1 U、0.1 U和0.01 U胃蛋白酶作用下的酶促激活结果;5为未激活的D32A;6~8分别为D32A在0.01 U、0.1 U和1 U胃蛋白酶作用下的酶促激活结果。1. Unactivated D215A; 2-4. D215A were enzymatic activated at 1 U、0.1 U和0.01 U rP conditions, respectively; 5. Unactivated D32A; 6-8. D32A were enzymatic activated at 1 U、0.1 U和0.01 U rP conditions, respectively.)
图6不同含量rP对突变体D32A和D215A的激活的影响
Fig.6The effect of rP concentration on activation of mutant pepsinogen D32A and D215A
2.3 突变型胃蛋白酶D32A和D215A的稳定性及其活性
分别将2.2中激活处理获得的突变型胃蛋白酶D32A和D215A放置于37℃水浴锅中,每隔24 h取样1次,SDS-PAGE分析其稳定性。实验结果(见图7)表明,突变型胃蛋白酶原D32A和D215A在激活过程中稳定性较差,与Lin[19]的研究相符,但激活后突变型胃蛋白酶D32A和D215A在3 d内可以保持相对稳定。
对激活后的突变型胃蛋白酶进行蛋白水解活性测定,发现突变型胃蛋白酶D32A和D215A皆无蛋白水解活性,将酶活测定反应时间延长至60 min,突变体仍无活性。因此激活获得的突变型胃蛋白酶D32A和D215A表现出不降解蛋白的性质,这符合早期的X射线衍射结果的推测结果[9-10]。同时,表明激活过程中加入的野生型胃蛋白酶的含量极低,不影响突变胃蛋白酶的利用和性质研究,即采用外加胃蛋白酶来激活突变型胃蛋白酶原是研究突变型胃蛋白酶的可行性方法。
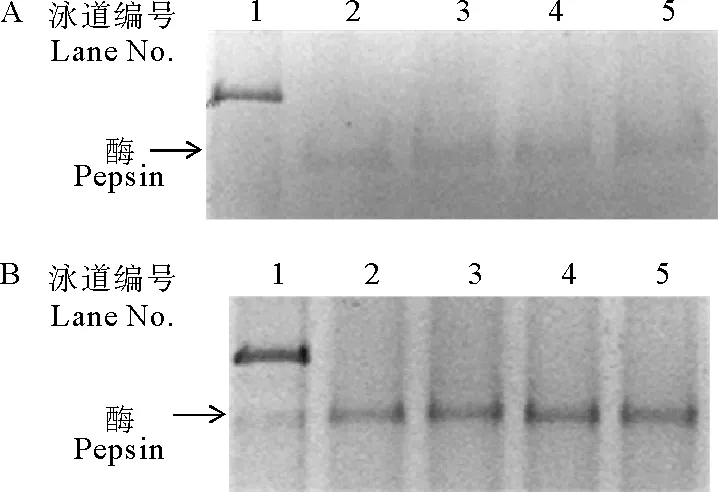
(A为D32A激活后稳定性;B为D215A激活后稳定性。其中1为酶原,2为激活8 h,3~5分别为激活后37℃放置1 d、2 d和3 d。A. The stability of D32A. B. The stability of D215A. 1. Unactivated pepsinogen; 2. Activated for 8 h; 3-5. activated mP stay at 37℃ for 1 d, 2 d and 3 d, respectively.)
图7突变体激活后稳定性
Fig.7Stability of mutant pepsin
3 讨论
本文通过定点突变技术将猪胃蛋白酶的活性中心关键氨基酸D32和D215突变为丙氨酸,并实现了突变型胃蛋白酶原D32A和D215A在毕赤酵母中表达。相比于大肠杆菌系统[14,19]胃蛋白酶的生物活性较低的缺点,酵母表达系统能够获得活性较好、结构完整的胃蛋白酶[16,25,27],虽然早在1989年即有对突变体D32A的研究[19],但当时利用的是大肠杆菌表达系统,受限于原核表达系统的表达后修饰体系的不完善,虽然表达后的D32A既不能激活,也没有蛋白水解活性,但不能完全排除表达体系对突变型胃蛋白酶原D32A性质和功能的影响。本研究实现了突变型胃蛋白酶原D32A和D215A在毕赤酵母中表达,并以野生型胃蛋白酶原的表达作为参照,既验证了实验方法的可行性,同时也首次在酵母中表达纯化得到突变型猪胃蛋白酶原D32A和D215A,为进一步研究D32和D215在胃蛋白酶催化中的作用提供了支持。
研究指出[19]胃蛋白酶原的激活需要D32和D215的共同参与,当D32突变后,酶原不能激活,甚至在酸性条件下酶原本身也不稳定。虽然,在本研究中发现,突变型胃蛋白酶原D32A和D215A在pH=2.0条件下可以激活为胃蛋白酶,得到了与之前报道略有不同的结果,但完全激活时间大于24 h,速度极慢。这种极低的效率和突变酶原在酸性条件下不稳定的性质,可能是前人认为其不能被激活的原因。
由于胃蛋白酶原的激活是自激活的过程[28-29],H+激活产生的胃蛋白酶可以进一步激活其他的胃蛋白酶原[30],因此本研究采用了酶促激活方式,当在突变型胃蛋白酶原D32A和D215A的激活过程中加入激活后的野生型胃蛋白酶时,突变型胃蛋白酶原的激活速率显著加快,1h即可全部激活。突变型胃蛋白酶原D32A和D215A在激活过程中的同样表现出了不稳定的特征,部分酶原在激活过程中有降解现象发生,这一现象与文献[19]中报道的一致,但激活后的突变型胃蛋白酶稳定性良好,在37℃可以稳定存在3d,即本实验首次实现了突变型猪胃蛋白酶原D32A和D215A的激活,这一结果使其具备了进一步用于酶学性质和作用机理研究的可能。
胃蛋白酶是体内重要的消化酶,对机体的健康有重要影响;同时胃蛋白酶也是重要的工具酶,在胶原蛋白及短肽的生产中有重要作用[31-33]。本研究一方面实现了胃蛋白酶活性中心关键氨基酸突变后的酶原高效激活,同时实现了激活后的突变型酶具有良好的稳定性。这一结果必将为胃蛋白酶的活性和结构研究提供重要支持,为阐明胃蛋白酶可降解多糖等新发现的机理研究打开新的方法之门。本研究中采用人工手段获得的突变型胃蛋白酶也将丰富胃蛋白酶家族酶系,为胃蛋白酶新活性的发现和胃蛋白酶的系统性研究提供帮助。
参考文献:
[1]Dunn B M. Structure and mechanism of the pepsin-like family of aspartic peptidases [J]. Chem Rev, 2002, 102(12): 4431-4458.
[2]Northrop J H. Crystalline Pepsin : I. Isolation and tests of purity [J]. J Gen Physiol, 1930, 13(6): 739-766.
[3]Bernal J D, Crowfoot D. X-ray photographs of crystalline pepsin [J]. Nature, 1934, 133(3369): 794-795.
[4]Bougatef A, Balti R, Zaied S B, et al. Pepsinogen and pepsin from the stomach of smooth hound (Mustelusmustelus): Purification, characterization and amino acid terminal sequences [J]. Food Chem, 2008, 107(2): 777-784.
[5]Nalinanon S, Benjakul S, Kishimura H. Purification and biochemical properties of pepsins from the stomach of skipjack tuna (Katsuwonuspelamis) [J]. Eur Food Res Technol, 2010, 231(2): 259-269.
[6]申锋, 杨莉莉, 熊善柏, 等. 胃蛋白酶水解草鱼鱼鳞制备胶原肽的工艺优化 [J]. 华中农业大学学报, 2010, 29(3): 387-391.
Shen F, Yang L L, Xiong S B, et al. Optimizing processing of collagen peptide extraction from scales of grass carp with pepsin [J]. Journal of Huazhong Agricultural University, 2010, 29(3): 387-391.
[7]Park Y N, Aikawa J, Nishiyama M, et al. Site-directed mutagenesis of conserved Trp39 in Rhizomucor pusillus pepsin: possible role of Trp39 in maintaining Tyr75 in the correct orientation for maximizing catalytic activity [J]. J Biochem, 1997, 121(1): 118-121.
[8]Kesavulu M M, Ramasubramanian S, Suguna K. Effect of dimethyl sulphoxide on the crystal structure of porcine pepsin [J]. Biochem Bioph Res Co, 2005, 331(4): 1510-1514.
[9]Abad-Zapatero C, Rydel T J, Erickson J. Revised 2.3 Å structure of porcine pepsin: evidence for a flexible subdomain [J]. Proteins, 1990, 8(1): 62-81.
[10]Cooper J B, Khan G, Taylor G, et al. X-ray analyses of aspartic proteinases: II. Three-dimensional structure of the hexagonal crystal form of porcine pepsin at 2. 3 Å resolution [J]. J Mol Biol, 1990, 214(1): 199-222.
[11]吴梧桐. 蛋白质工程技术与新型生物催化剂设计 [J]. 药物生物技术, 2004, 11(1): 1-6.
Wu W T. Protein engineering technique and the search for the new biocatalyst[J]. Design Pharmaceutical Biotechnology, 2004, 11(1): 1-6.
[12]樊晋宇, 崔宗强, 张先恩. 红色荧光蛋白的光谱多样性及体外分子进化 [J]. 生物化学与生物物理进展, 2008, 35(10): 1112-1120.
Fan J Y, Cui Z Q, Zhang X E. Optical spectra diversity and in vitro molecular evolution of red fluorescent proteins[J]. Prog Biochem Biophys, 2008, 35(10): 1112-1120.
[13]卢丽丽, 肖敏, 赵晗, 等. 糖苷合成酶-一类新型的寡糖高效合成工具 [J]. 生物化学与生物物理进展, 2006, 33(4): 310-320.
Lu L L, Xiao M, Zhao H, et al. Glycosynthases: a novel efficient synthetic tool for oligosaccharides[J]. Prog Biochem Biophys, 2006, 33(4): 310-320.
[14]Okoniewska M, Tanaka T, Yada R. The pepsin residue glycine-76 contributes to active-site loop flexibility and participates in catalysis [J]. Biochem J, 2000, 349: 169-177.
[15]Chen Z, Koelsch G, Han H P, et al. Recombinant rhizopuspepsinogen. Expression, purification, and activation properties of recombinant rhizopuspepsinogens [J]. J Biol Chem, 1991, 266(18): 11718-11725.
[16]Yoshimasu M A, Ahn J K, Tanaka T, et al. Soluble expression and purification of porcine pepsinogen from Pichia pastoris [J]. Protein Expres Purif, 2002, 25(2): 229-236.
[17]Kageyama T. Pepsinogens, progastricsins, and prochymosins: structure, function, evolution, and development [J]. Cell Mol Life Sci, 2002, 59(2): 288-306.
[18]Gildberg A. Aspartic proteinases in fishes and aquatic invertebrates [J]. Comp Biochem Phys B, 1988, 91(3): 425-35.
[19]Lin X L, Wong R N, Tang J. Synthesis, purification, and active site mutagenesis of recombinant porcine pepsinogen [J]. J Biol Chem, 1989, 264(8): 4482-4489.
[20]Shobha M S, Gowda L R, Tharanathan R N. A novel catalysis by porcine pepsin in debranching guar galactomannan [J]. Carbohyd Polym, 2014, 102: 615-621.
[21]Dong P, Zhang Y F, Liu Y, et al. Nucleic acids start Their digestion in stomach [C]. Kitakyushu, Japan: The 41st International Symposium on nucleic acids chemistry, 2014: 90-91.
[22]Bryksin A V, Matsumura I. Overlap extension PCR cloning: a simple and reliable way to create recombinant plasmids [J]. Biotechniques, 2010, 48(6): 463.
[23]Laemmli U K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4 [J]. Nature, 1970, 227(5259): 680-685.
[24]Anson M L. The estimation of pepsin, trypsin, papain, and cathepsin with hemoglobin [J]. J Gen Physiol, 1938, 22(1): 79-89.
[25]刘西梅, 安立国, 乔宪凤, 等. 毕赤酵母表达猪胃蛋白酶的研究 [J]. 华中师范大学学报(自然科学版), 2008, 42(3): 426-430.
Liu X M, An L G, Qiao X F, et al. Study of expressing pepsinogen in pichia pastoris [J]. Journal of Huazhong Normal University Natural Sciences, 2008, 42(3): 426-430.
[26]Nielsen F S, Foltmann B. Activation of porcine pepsinogen A [J]. Eur J Biochem, 1993, 217(1): 137-142.
[27]李圣翔. 猪胃蛋白酶原 A 基因克隆, 毕赤酵母表达及其酶活性分析 [D]. 四川: 四川农业大学, 2011.
Li S X. Cloning and Expression of Porcine Pepsinogen A in Pichia Pastoris and Detection of the Recombination Enzyme Activity[D]. Sichuan: Sichuan Agriculture University, 2011.
[28]Dee D R, Yada R Y. The prosegment catalyzes pepsin folding to a kinetically trapped native state [J]. Biochemistry, 2009, 49(2): 365-371.
[29]Varón1 R, Fuentes M E, García-Moreno M, et al. Contribution of the intra-and intermolecular routes in autocatalytic zymogen activation: application to pepsinogen activation [J]. Acta Biochim Pol, 2006, 53(2): 407-420.
[30]Andreeva N S, Rumsh L D. Analysis of crystal structures of aspartic proteinases: On the role of amino acid residues adjacent to the catalytic site of pepsin-like enzymes [J]. Protein Sci, 2001, 10(12): 2439-2450.
[31]王立晖, 袁永俊, 孙勇民, 等. 胃蛋白酶水解酪蛋白生成 β-酪啡肽-7的研究 [J]. 中国酿造, 2010, 29(4): 64-66.
Wang L H, Yuan Y J, Sun Y M, et al. Production of β-Casomorphin-7 by pepsin from casein [J]. China Brewing, 2010, 29(4): 64-66.
[32]Zeng S, Yin J, Yang S, et al. Structure and characteristics of acid and pepsin-solubilized collagens from the skin of cobia (Rachycentroncanadum)[J]. Food Chem, 2012, 135(3): 1975-1984.
[33]易翠平, 周素梅, 潘艳艳. 胃蛋白酶水解大米蛋白的研究 [J]. 食品科学, 2010, 31(10): 129-132.
Yi C P, Zhou S M, Pan Y Y. Pepsin hydrolysis of rice protein powder [J]. Food Science, 2010, 31(10): 129-132.
责任编辑朱宝象
基金项目:❋ 国家自然科学基金项目(31201327);国家教育部留学回国人员科研启动基金项目;山东省自然科学基金杰出青年基金项目(JQ201204);国家青年千人计划资助项目资助
收稿日期:2015-04-03;
修订日期:2015-12-03
作者简介:刘宇(1987-),女,博士生。E-mail:shishui87@163.com ❋❋通讯作者: E-mail: dongping@ouc.edu.cn
中图法分类号:Q556+.3
文献标志码:A
文章编号:1672-5174(2016)07-054-08
DOI:10.16441/j.cnki.hdxb.20150126
Expression and Activation of Recombinant Porcine Pepsinogen with Mutated Active Site
LIU Yu1, GUO Hui1, GAO Xiao-Meng1, ZHANG Yan-Fang1, XU Jia-Kun2,DONG Ping1, LIANG Xing-Guo1
(1.College of Food Science and Engineering, Ocean University of China, Qingdao 266003, China; 2.Yellow Sea Fisheries Research Institute Chinese Academy of Fishery Science, Qingdao 266071,China)
Abstract:The activation and stability of pepsinogen play an important role in studying pepsin activity. In this study, the activation and stability of mutated porcine pepsinogens of which the key amino acid at active site was mutated were investigated. According to the sequence in GenBank, the primers were designed to amplify the pepsinogen gene. RT-PCR and overlap extension PCR were used to obtain the cDNA that corresponds to the zymogenic form of porcine pepsin (EC 3.4.23.1), both the wild (rPG) and mutated types (D32A and D215A). The gene was cloned into the vector pPICZα A and transformed into Pichia pastoris X-33. The recombinant enzyme was purified through DEAE-cellulose anion exchange chromatography and Sephadex G-200 gelfiltration. The heterogeneous expression and activation property of wild and mutated pepsinogen were studied in details. The results showed that the recombinant pepsinogens (rPG, D32A and D215A) were successfully expressed and highly purified from P. pastoris. At pH 2.0, rPG could be activated in 5 min and stabilized in 37 ℃. And compared to commercial pepsin, the recombinant wild type pepsin had a similar activity. However, the activation of mutated pepsionogen D32A and D215A would need more than 24 h. In order to activate D32A and D215A, a roundabout enzyme-activated method was used. The D32A and D215A solution were adjusted to pH 2.0 with diluted HCl and added one unit of the wild-type pepsin. The mixture was incubated at 25 ℃ for 12 h. The results showed that D32A and D215A were activated in this way. Furthermore, the mutated pepsinogen could be activated in 1 h with this enzyme-activated method. Although the mutated pepsinogen would degrade during activation, the activated mutated pepsin has a relatively good stability. It had been reported that the active site amino acid D32 and D215 were necessary for the activation of pepsinogen and in acid solution D32A did not activate to pepsin but slowly denatured. However, in this study we first investigated the activate possibility of mutated pepsinogen of which active site key amino acid was mutated. And the result showed that mutated pepsinogen D32A and D215A could be activated with a roundabout enzyme-activated method. The activated mutated pepsin D32A and D215A could be stabled for at least 3 days. The results of our work will provide a possibility to study the property of mutated pepsin.
Key words:porcine pepsinogen; Pichia pastoris expression; mutation; activation
Supported by National Natural Science Foundation of China(31201327);Scientific Research Foundation for the Returned Overseas Chinese Scholars, State Education Ministry;Shandong Province Funds for Distinguished Young Scientists(JQ201204); National Youth Qianren Plan
