茶树两个Dof转录因子的分离及其在温度胁迫中的响应分析
2016-07-29李辉黄蔚刘志薇王永鑫吴致君庄静
李辉,黄蔚,刘志薇,王永鑫,吴致君,庄静*
茶树两个Dof转录因子的分离及其在温度胁迫中的响应分析
李辉1,3,黄蔚2,刘志薇1,3,王永鑫1,3,吴致君1,3,庄静1,3*
1. 南京农业大学园艺学院, 江苏 南京 210095;2. 南京农业大学作物遗传与种质创新国家重点实验室, 江苏 南京 210095;3. 南京农业大学茶叶科学研究所,江苏 南京 210095
摘要:茶树是一种重要的经济作物,温度胁迫对茶叶生产影响较大。本研究以茶树品种迎霜为实验材料,克隆获得2个编码Dof转录因子的基因CsDof1和CsDof2。序列分析显示,CsDof1含有1 389 bp,编码463个氨基酸;CsDof2含有1 458 bp,编码486个氨基酸。CsDof1和CsDof2转录因子都具有典型的锌指结构域,分别位于第133~195和124~186个氨基酸位点之间。对CsDof1和CsDof2转录因子理化性质、亲/疏水性、二级和三级结构分析显示,两者都是亲水性蛋白且显偏碱性。空间结构分析显示,CsDof1和 CsDof2均有 1个 α-螺旋,且Dof结构域的位点完全相同,均有4个半胱氨酸残基。实时定量PCR表明,CsDof1和CsDof2在不同茶树品种、不同温度胁迫下均能诱导表达,且表达呈现差异。
关键词:茶树;Dof转录因子;锌指结构;温度胁迫;基因表达
茶树[Camellia sinensis (L.) O. Kuntze]在我国主要分布于北纬18°~38°,东经94°~122°之间的地区,是我国一种重要的经济作物[1],富含茶多酚、植物碱、氨基酸、维生素、有机酸等有机化学成分[2]。茶树具有喜温怕寒的特性,高温和低温的交替变化会对茶树生长造成一定程度的伤害,从而影响茶叶的产量和品质。
当植物处于逆境胁迫时,转录因子能启动抗逆基因的表达,在逆境信号传递过程中发挥重要的作用[3]。Dof(DNA binding with one finger)转录因子属于锌指蛋白家族,在植物中能够参与逆境调控。Dof家族转录因子结构域中含有1个保守的CX2CX21CX2C基序,其中 4个Cys残基与1个Zn2+共价结合形成 1个单锌指C2C2结构域[4]。Dof转录因子通常包含2个主要的结构域,即位于N末端的保守DNA结构域和位于C末端的调控结构域。植物中第1个Dof转录因子于1993年从玉米中被鉴定出来[5],此后多种植物的Dof转录因子被分离出来,并进行了相应的基因功能研究和家族分类[6]。对拟南芥全基因组序列Dof成员进行系统分析的结果表明,拟南芥Dof家族包括36个转录因子,分为A、B、C、D 4个亚家族[7]。Moreno-Risueno等[8]将拟南芥、水稻、大麦、绿藻、苔藓、蕨类和裸子植物等七大代表物种的116个Dof成员进行系统分类后,得到A、B、C、D、E、F、G 7个亚家族。Dof转录因子在植物体内能调节种子发芽,参与植物的光形态建成,调节植物新陈代谢并响应逆境胁迫[9-12]。目前,对其他植物Dof转录因子研究较多,茶树Dof转录因子结构与功能的相关研究还鲜有报道。
本研究以茶树品种迎霜为材料,克隆得到2个编码茶树 Dof转录因子的基因CsDof1和CsDof2,并研究了其理化性质、进化树、亲水性/疏水性及三级结构。通过实时定量PCR方法,对茶树中CsDof1和CsDof2的组织特异性,以及温度胁迫下的诱导表达进行了分析,以期通过分子生物学技术手段,研究茶树Dof转录因子对温度胁迫的响应机制,挖掘与茶树抗逆有关的基因,为提高茶树温度适应能力奠定基础。
1 材料与方法
1.1 试验材料、菌株和质粒
供 试材 料 为 迎 霜 ( C. sinensis cv. Yingshuang)2年生扦插茶树幼苗,种植于南京农业大学茶叶研究所实验大棚。供试材料生长环境温度保持在25℃,土壤pH为5.6,空气湿度(70±10)%。取迎霜幼嫩叶片进行RNA的提取和cDNA的合成,并以迎霜幼嫩叶片的cDNA作为克隆基因的模板。
选取长势良好的云南十里香(C. sinensis cv. Yunnan Shilixiang)、安吉白茶(C. sinensis cv. Anji Baicha)、迎霜(C. sinensis cv. Yingshuang)茶苗,控制光照培养箱中湿度为75%,将茶苗进行温度处理(4℃低温和38℃高温)0、1、4、8、24 h。取不同处理的茶苗幼嫩叶片,进行RNA的提取和cDNA的合成,用于实时定量PCR。
大肠杆菌菌株 DH5α由南京农业大学茶叶科学研究所保存;质粒载体 pMD18-T vector、Ex Taq聚合酶、DL maker 2000、反转录试剂盒Prime Script RT Reagent Kit、实时定量试剂盒SYBR Premix Ex Taq Kit均购自大连TaKaRa公司;总RNA提取试剂盒Quick RNA Isolation Kit购自北京华越洋生物科技有限公司;引物合成和DNA测序由南京金斯瑞生物科技有限公司完成;DNA回收试剂盒购自杭州维特洁公司。
1.2 总RNA的提取及cDNA合成
按照Quick RNA Isolation Kit试剂盒说明书提取茶树云南十里香、安吉白茶、迎霜叶片的总 RNA,利用微量紫外检测仪 NanoDrop测定RNA浓度。用Prime Script RT Reagent Kit试剂盒将RNA反转录为cDNA。
1.3 茶树CsDof1和CsDof2基因的克隆
基于实验室茶树品种迎霜的转录组数据[13],利用软件Primer Premier 5.0设计两对引物,分别为CsDof1-F: 5'-ATGTCGGAATC AAAAGACCCT-3',CsDof1-R: 5'-TGAGC TTTCATGGAAGTCAAG-3'和CsDof2-F: 5'-ATGTCGGAATCAAAAGACCCT-3',CsDof2-R: 5'-TGAACTCTCATGGAAGTTGAA-3'。PCR扩增体系为20 μL:dd H2O 7 μL,Ex Taq酶10 μL,上游引物1 μL,下游引物1 μL,模板cDNA 1 μL。反应条件为:94℃预变性5 min;94℃变性30 s,52℃退火30 s,72℃延伸1 min,共35个循环;72℃延伸10 min。将迎霜叶片的cDNA作为克隆基因的模板。用12 g·L-1的琼脂糖凝胶电泳后分离条带,按照DNA回收试剂盒说明书回收 PCR产物,然后连接到pMD18-T载体上,转化至大肠杆菌DH5α。提取大肠杆菌菌液后送至南京金斯瑞公司测序。
1.4 序列分析
利用NCBI数据库(http://www.ncbi.nlm.nih.gov)对 Dof核苷酸和蛋白序列进行搜索和预测保守域[14];利用 MEGA5构建系统进化树[15];利用Origin 6.0进行荧光定量PCR数据分析及图表绘制;利用DNAMAN和ExPASy网站(http://www.expasy.org)对氨基酸亲水/疏水性进行分析以及多重序列比对;利用SOPMA网站(http://npsa-pbil.ibcp.fr/cgi-bin/npsa)进行二级结构预测[16];蛋白质三级结构模型[17]用Swiss-Model(http://www.Swissmodel.expasy.org)构建。
1.5 实时定量PCR反应
利用 iQ™5 software和 iQ™5Real- time PCR System,按照SYBR Premix Ex Taq Kit试剂盒说明书进行实时定量PCR(Quantitative real time RT-PCR)操作。相对定量参照基因的ΔΔCT法[18],表达差异等于2-ΔΔCT ,ΔCT=CT目标基因-CT actin,ΔΔCT=(CT目标基因-CT actin)处理组-(CT
目标基因-CT actin)对照组。茶树actin作为内参基因,与目标基因CsDof1和CsDof2一起扩增,它们的表达检测引物分别为CsDof1-R-F: 5'-TTCAAAGGATGATGTTGGTAG-3',CsDof1-R-R: 5'-GGAGGATAGAATGGCATAGGA-3'和CsDof2-R-F: 5'-CTCCGTGGTCGTACCCTTG-3', CsDof2-R-R: 5'-TACAACCCCAATAAGGCGG-3',及 ACTIN-F: 5'-CCATCACCAGAATCCAAGAC-3',ACTIN-R: 5'-GAACCCGAAGGCGAATAGG-3'。
2 结果与分析
2.1 茶树CsDof1和CsDof2基因的克隆
以迎霜叶片 cDNA为模板,分别以CsDof1-F、CsDof1-R和CsDof2-F、CsDof2-R两对引物经PCR扩增得到2个1 400 bp左右的片段(图1)。序列测定与分析表明,茶树CsDof1和 CsDof2基因片断分别为 1 389 bp 和1 458 bp,编码463和486个氨基酸(图2、图3)。
2.2 茶树CsDof1和CsDof2转录因子的进化分析
茶树 CsDof1和 CsDof2转录因子的进化分析参考 Moreno-Risueno等[8]的 Dof转录因子家族分类方法完成。在 Plant transcription factor database(http://planttfdb.cbi.pku.edu.cn)下载拟南芥、毛果杨、水稻等物种的 101个Dof成员,与CsDof1和CsDof2转录因子进行同源性比对并绘制进化树。结果(图4)表明,CsDof1和CsDof2转录因子同属于Dof转录因子家族中的A亚族。
2.3 茶树CsDof1和CsDof2转录因子氨基酸序
列比对及理化性质分析
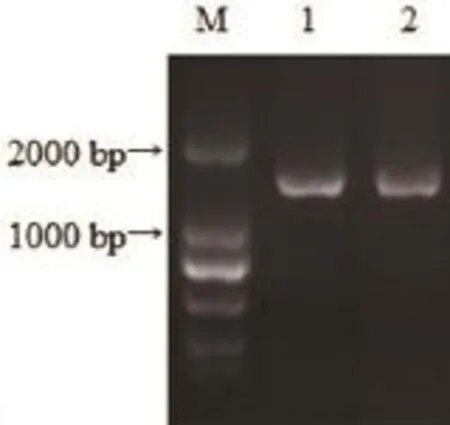
图1 克隆茶树CsDof1和CsDof2基因Fig. 1 CsDof1 and CsDof2 genes from C. sinensis by PCR amplification

图2 茶树CsDof1的核苷酸序列及其编码的氨基酸序列Fig. 2 cDNA and their deduced amino acid sequences of CsDof1 in C. sinensis
利用BLAST-conserved domains search对茶树CsDof1和 CsDof2转录因子进行保守域预测。结果如图5-A和5-B所示,CsDof1和CsDof2转录因子的结构域分别在第 133~195和第124~186个氨基酸位点之间,且均属于锌指蛋白。通过Blast同源检索与比对,将茶树CsDof1和CsDof2转录因子与多个物种的Dof转录因子的氨基酸序列进行多重序列比对,结果显示一致性为61.35%。在保守区域内9个赖氨酸、6个精氨酸、5个脯氨酸、4个甘氨酸、4个天冬氨酸、4个半胱氨酸、3个络氨酸、3个苏氨酸、2个甘氨酸、2个苯丙氨酸和1个丝氨酸残基完全保守。从图 5-C可以看出,CsDof1和CsDof2转录因子的zf-Dof结构域很相似,氨基酸序列都存在CX2CX21CX2C基序形成的单锌指结构,且含有Cys残基的保守结构域,CsDof1和CsDof2都含有Dof结构域的所有功能单元。
用ExPASy网站对CsDof1和CsDof2转录因子进行氨基酸组成成分及理化性质分析(表1)。结果表明,CsDof1和CsDof2蛋白分别含有463和486个氨基酸,理论等电点分别为5.97和 5.70,分子质量分别为 50.09 kDa和52.76 kDa。对其他植物 Dof转录因子进行氨基酸组成成分及理化性质分析,结果表明,不同植物中Dof转录因子理论等电点存在差异,但基本处于 5.41~6.97,且碱性氨基酸数目普遍高于酸性氨基酸。
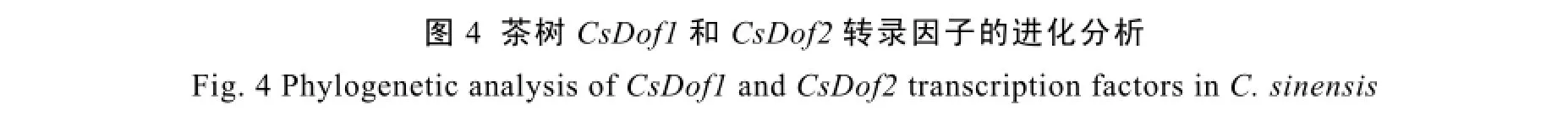
图4 茶树CsDof1和CsDof2转录因子的进化分析Fig. 4 Phylogenetic analysis of CsDof1 and CsDof2 transcription factors in C. sinensis
2.4 茶树CsDof1和CsDof2转录因子的亲/疏
水性分析
利用DNAMAN 6.0软件对茶树CsDof1 和CsDof2转录因子进行疏水性/亲水性分析。结果表明,茶树 CsDof1和 CsDof2转录因子亲水性最强的的位点分别是第 189位的赖氨酸(Lys)和第 180位的赖氨酸(Lys)(图6-A);疏水性最强的位点分别是第305位的缬氨酸(Val)和第236位的亮氨酸(Leu)(图6-B),CsDof1和CsDof2转录因子均属于亲水性蛋白。
2.5 茶树CsDof1和CsDof2转录因子的二级和
三级结构预测与分析
利用 SOPMA网站对茶树 CsDof1和CsDof2转录因子的二级结构进行预测。结果表明,CsDof1由 13.17%的 α-螺旋(Alpha helix)、5.62%的β-折叠(Beta turn)、11.02%的延伸主链(Extended strand)和 51.20%的随机卷曲(Random coil)组成;CsDof2由15.02%的α-螺旋(Alpha helix)、6.38%的β-折叠(Beta turn)、12.55%的延伸主链(Extended strand)和 66.05%的随机卷曲(Random coil)组成。因此,CsDof1和CsDof2主要组成部分为随意卷曲结构、α-螺旋、延伸主链和 β-折叠。通过 Swiss-Model网站对CsDof1和CsDof2进行三级结构预测(图7),CsDof1和 CsDof2具有相似的三级结构,均有1个α-螺旋,在Dof结构域中的位点完全相同,均有 4个半胱氨酸残基,表示了 Dof转录因子特有的锌指结构。
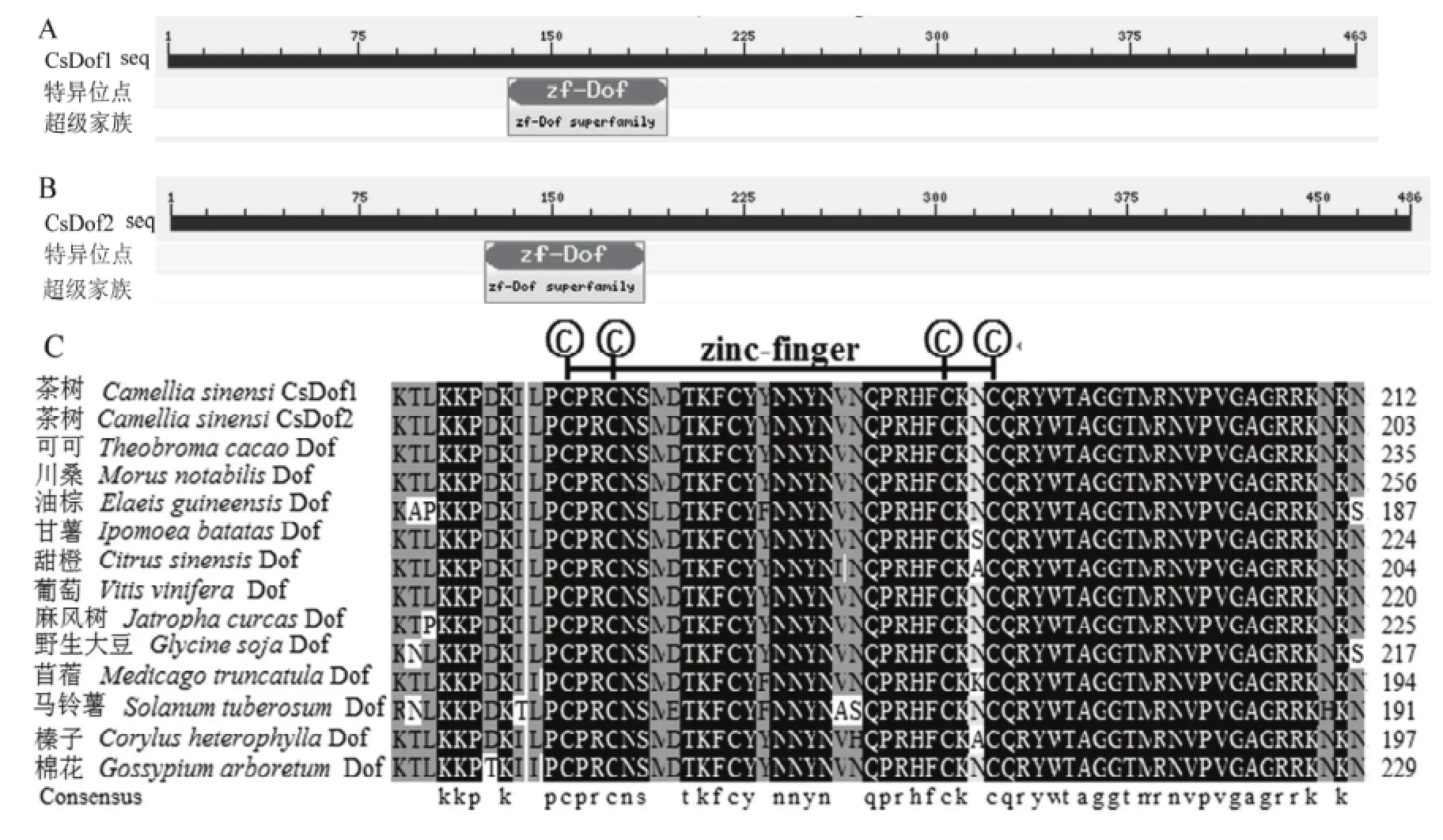
图5 茶树CsDof1(A)和CsDof2(B)转录因子的保守域及与其他物种氨基酸序列的多重比对(C)Fig. 5 The conserved domains of CsDof1 (A) and CsDof2 (B) and alignment of relative amino acid sequences in C. sinensis and other plants (C)
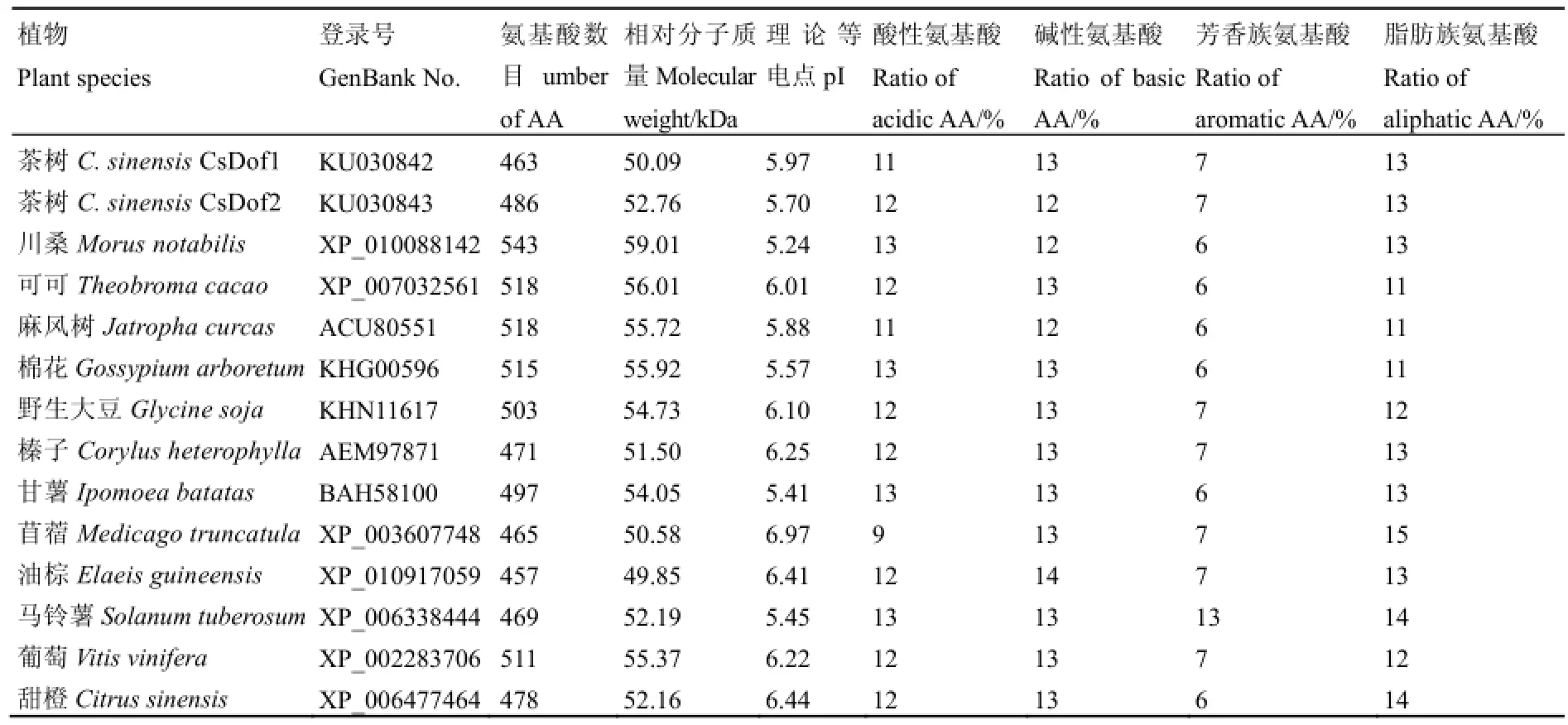
表1 不同植物中Dof转录因子氨基酸组成成分及理化性质分析Table 1 Comparison of amino acid sequences, physical and chemical characterizations of Dof transcription factors among different plants
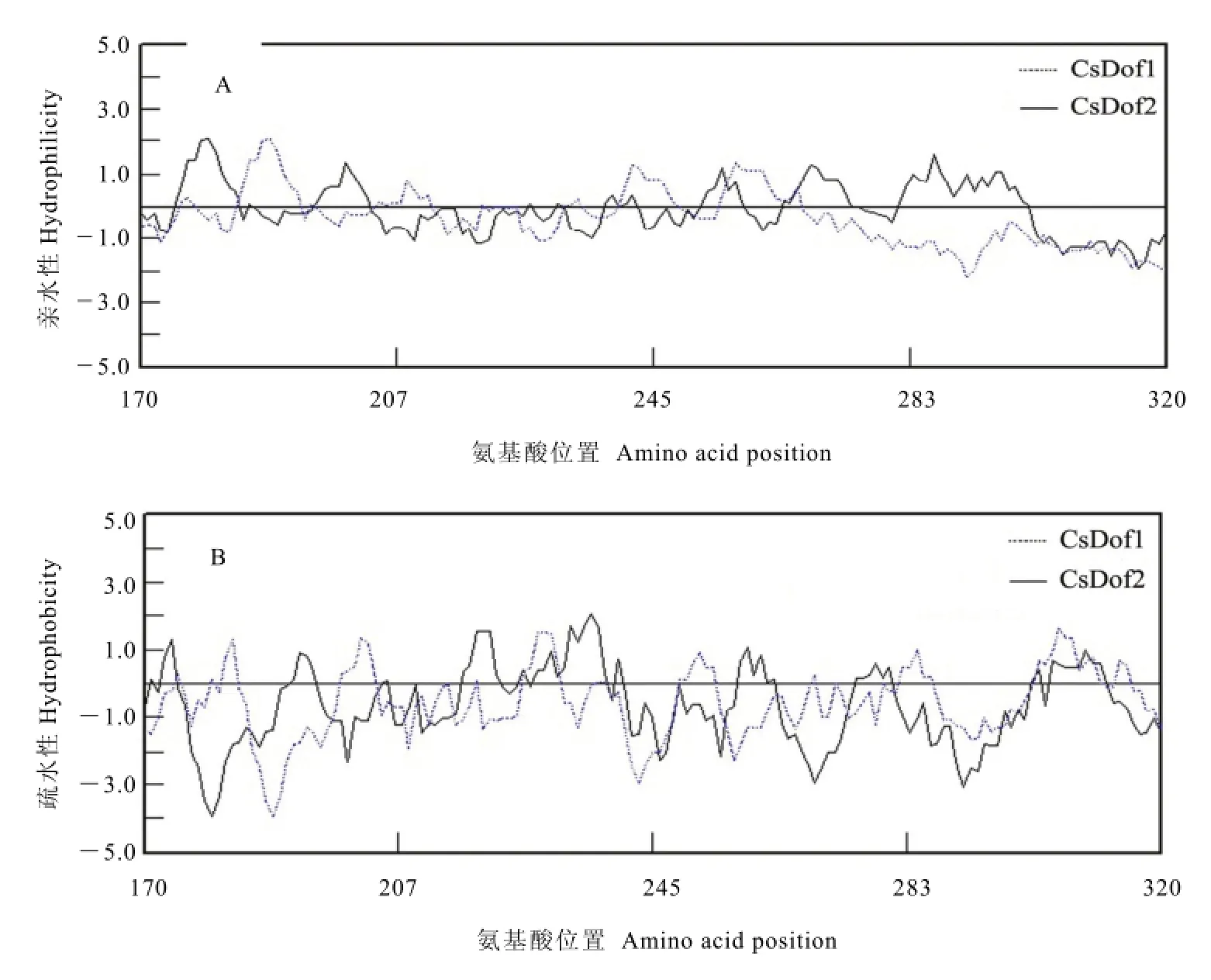
图6 茶树CsDof1和CsDof2氨基酸序列亲水(A)、疏水(B)性质分析Fig. 6 Analysis of hydrophilicity (A) and hydrophobicity (B) of the deduced amino acid sequences of CsDof1 and CsDof2 in C. sinensis
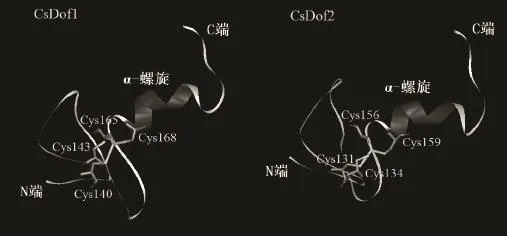
图7 茶树CsDof1和CsDof2转录因子的三级结构预测Fig. 7 The tertiary structures of CsDof1 and CsDof2 in C. sinensis
2.6 茶树CsDof1和CsDof2基因在高低温胁迫下的表达情况
通过实时定量 PCR分别检测 CsDof1和 CsDof2基因在经过高温(38℃)、低温(4℃)2种处理后的云南十里香、安吉白茶和迎霜中的表达情况(图8)。
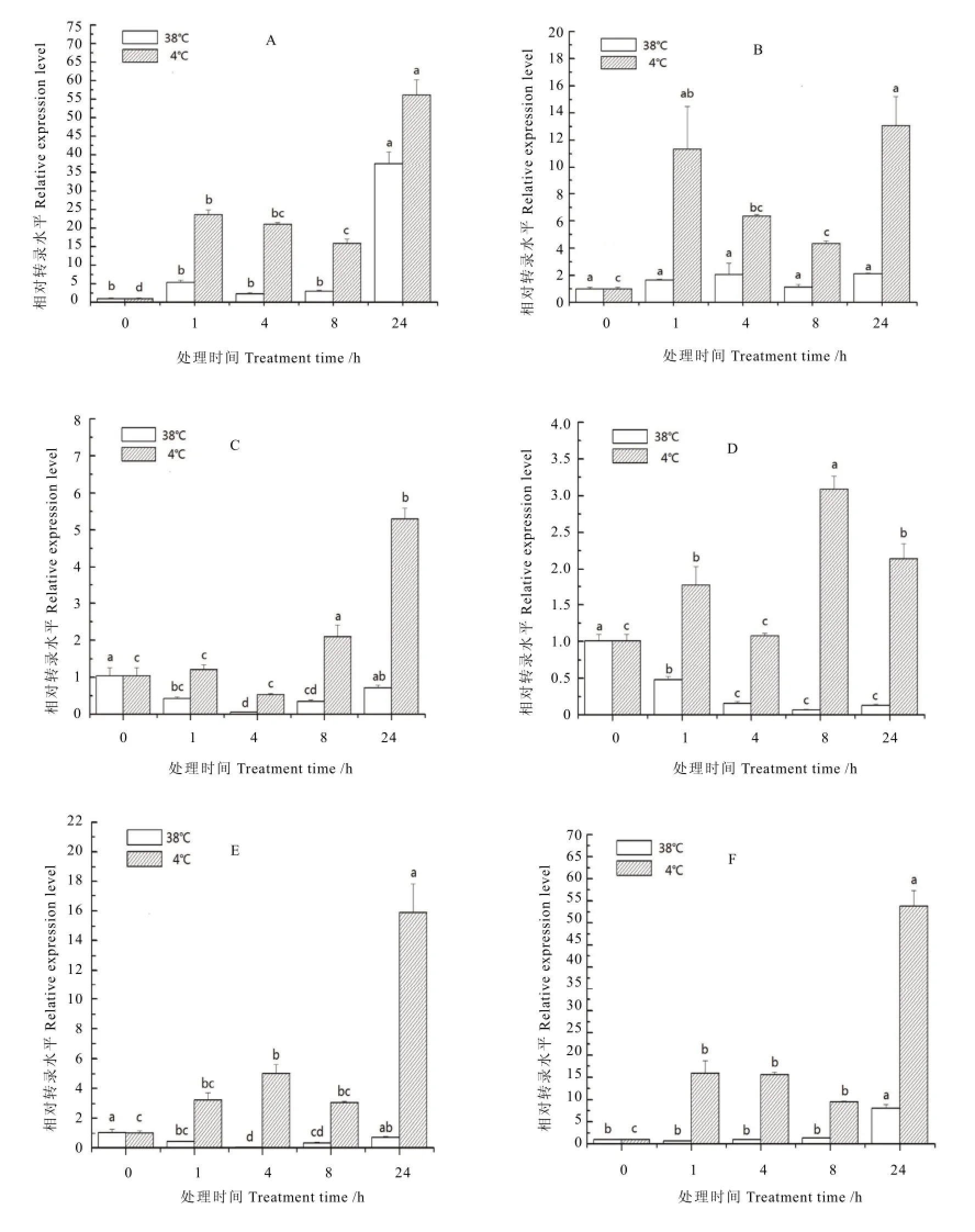
图8 3个茶树品种中CsDof1和CsDof2在高低温处理下的表达情况Fig. 8 Expression profiles of the CsDof1 and CsDof2 under high and low temperature stresses in three tea cultivars
从图中可以看出,CsDof1和CsDof2经不同处理后在 3个茶树品种中的基因表达情况存在差异。4℃处理下,茶树CsDof1和CsDof2在迎霜中的表达情况相似,均在处理 24 h后达到最大值,分别为对照组(0 h)的56倍和13倍;38℃处理下,CsDof1的表达量逐渐增加,在处理24 h后达到最大。在安吉白茶中,38℃处理下,CsDof1和CsDof2的表达量均表现出减少的趋势,CsDof2表达量减少的程度比CsDof1更明显;在4℃的处理下,CsDof1表达量先增多再降低再增多,CsDof1和CsDof2的表达量分别在处理24 h和 8 h后达到最大值,分别为对照组的5倍和3倍。当云南十里香受到高温胁迫时,CsDof2的表达量相对于对照组明显增加;低温胁迫下,CsDof1 和CsDof2的表达量均在处理24 h后达到最大值,与对照组相比变化明显,分别为对照组的16和54倍。
3 讨论
植物生长发育过程中会受到非生物逆境的影响,高低温胁迫是重要的非生物胁迫因子之一。为了适应非生物逆境胁迫,植物体内形成了复杂的调控机制,而转录因子在植物响应不同逆境方面具有重要作用,其表达调控也进一步对植物的生长发育产生影响。因此,分析与逆境胁迫相关的转录因子就显得尤为重要。
本试验克隆得到的CsDof1和CsDof2基因编码的 Dof转录因子都具有 Dof的典型特征,含有1个由52个氨基酸组成,高度保守的 zf-Dof结构域,在 Dof结构域中存在CX2CX21CX2C基序形成的单锌指结构,这与Umemura等[4]的研究结果相吻合。对 CsDof1 和CsDof2转录因子氨基酸组成成分、理化性质、亲水性/疏水性的分析结果显示,CsDof1 和CsDof2均属于亲水性蛋白,二级结构主要表现为随意卷曲和 α-螺旋结构。在三级结构中,两个转录因子Dof结构域中的位点完全相同,均有 1个 α-螺旋,都存在 4个半胱氨酸残基。系统进化树表明CsDof1和 CsDof2转录因子均属于Dof家族里的A亚族,与毛果杨A亚族亲缘关系最近。
安吉白茶、迎霜和云南十里香3个茶树品种的荧光定量PCR分析结果表明,高低温影响CsDof1和CsDof2的表达量,说明CsDof1 和CsDof2在茶树温度胁迫中具有调控作用。研究证明,在白菜中,4个 BraDof转录因子基因能够在低温下被诱导[19]。马静的研究也表明,白菜受高低温处理后,BraDof表达量迅速升高[20]。本试验中,3个不同茶树品种经过低温处理后,CsDof1和CsDof2表达量都高于对照组,Dof转录因子可能在茶树响应低温的过程中发挥着重要作用。
近年来的研究已表明,Dof转录因子家族参与调控植物体内多种生物学过程[8]。本文通过对CsDof1和CsDof2的分析,证明了它参与茶树对高低温的响应,且在不同茶树品种间表达量存在差异。为了进一步认识 CsDof1和CsDof2转录因子的调控机制,还需要对其调节途径和作用机理作进一步深入研究。本研究也说明,利用温度胁迫响应相关转录因子来提高茶树对温度胁迫的综合抗性,是一种有潜力的分子育种途径。
参考文献
[1]骆耀平. 茶树栽培学[M]. 北京: 中国农业出版社, 2008:
11-16.
[2]宛晓春. 茶叶生物化学[M]. 北京: 中国农业出版社, 2003: 9-50.
[3]刘欣, 李云. 转录因子与植物抗逆性研究进展[J]. 中国农学通报, 2006, 22(4): 61-65.
[4]Umemura Y, Ishiduka T, Yamamoto R, et al. The Dof domain,a zinc finger DNA-binding domain conserved only in higher plants, truly functions as a Cys2/Cys2 Zn finger domain [J]. The Plant Journal, 2004, 37(5): 741-749.
[5]Yanagisawa S, Izui K. Molecular cloning of two DNA-binding proteins of maize that are structurally different but interact with the same sequence motif [J]. Journal of Biological Chemistry, 1993, 268(21): 16028-16036.
[6]Yanagisawa S. The Dof family of plant transcription factors [J]. Trends in Plant Science, 2002, 7(12): 555-560.
[7]Lijavetzky D, Carbonero P, Vicente-Carbajosa J. Genome-wide comparative phylogenetic analysis of the rice and Arabidopsis Dof gene families [J]. BMC Evolutionary Biology, 2003, 3(1): 17.
[8]Moreno-Risueno M Á, Martínez M, Vicente-Carbajosa J, et al. The family of DOF transcription factors: from green unicellular algae to vascular plants [J]. Molecular Genetics and Genomics, 2007, 277(4): 379-390.
[9]Diaz I, Vicente-Carbajosa J, Abraham Z, et al. The GAMYB protein from barley interacts with the DOF transcription factor BPBF and activates endosperm-specific genes during seed development [J]. The Plant Journal, 2002, 29(4): 453-464.
[10]Mena M, Cejudo F J, Isabel-Lamoneda I, et al. A role for the DOF transcription factor BPBF in the regulation of gibberellin-responsive genes in barley aleurone [J]. Plant Physiology, 2002, 130(1): 111-119.
[11]Nakano T, Suzuki K, Ohtsuki N, et al. Identification of genes of the plant-specific transcription-factor families cooperatively regulated by ethylene and jasmonate in Arabidopsis thaliana [J]. Journal of Plant Research, 2006,119(4): 407-413.
[12]Yanagisawa S. Dof domain proteins: plant-specific transcription factors associated with diverse phenomena unique to plants [J]. Plant and Cell Physiology, 2004, 45(4): 386-391.
[13]Wu Z J, Li X H, Liu Z W, et al. De novo assembly and transcriptome characterization: novel insights into catechins biosynthesis in Camellia sinensis [J]. BMC Plant Biology,2014, 14(1): 277.
[14]Schäffer A A, Aravind L, Madden T L, et al. Improving the accuracy of PSI-BLAST protein database searches with composition-based statistics and other refinements [J]. Nucleic Acids Research, 2001, 29(14): 2994-3005.
[15]Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance, and maximum parsimony methods [J]. Molecular Biology and Evolution, 2011, 28(10): 2731-2739.
[16]Combet C, Blanchet C, Geourjon C, et al. NPS@: network protein sequence analysis [J]. Trends in Biochemical Sciences, 2000, 25(3): 147-150.
[17]Arnold K, Bordoli L, Kopp J, et al. The SWISS-MODEL workspace: a web-based environment for protein structure homology modelling [J]. Bioinformatics, 2006, 22(2): 195-201.
[18]Pfaffl M W. A new mathematical model for relative quantification in real-time RT-PCR [J]. Nucleic Acids Research, 2001, 29(9): 2002-2007.
[19]Jung H J, Dong X, Park J I, et al. Genome-wide
transcriptome analysis of two contrasting Brassica rapa doubled haploid lines under cold-stresses using Br135K Oligomeric Chip [J]. Plos One, 2013, 9(8): e106069.
[20]Ma J, Li M Y, Wang F, et al. Genome-wide analysis of Dof
family transcription factors and their responses to abiotic stresses in Chinese cabbage [J]. BMC Genomics, 2015, 16(1): 33.
中图分类号:S571.1;Q51;P423
文献标识码:A
文章编号:1000-369X(2016)03-312-11
收稿日期:2015-08-04
修订日期:2015-11-10
基金项目:国家自然科学基金(31200520、31570691)
作者简介:李辉,男,硕士研究生,主要从事茶树分子生物学研究。*通讯作者:zhuangjing@njau.edu.cn
Isolation and Expression Analysis of Two Temperature Responsive Dof Genes from Camellia sinensis
LI Hui1,3, HUANG Wei2, LIU Zhiwei1,3, WANG Yongxin1,3, WU Zhijun1,3, ZHUANG Jing1,3*
1. College of Horticulture, Nanjing Agricultural University, Nanjing 210095, China;2. State Key Laboratory of Crop Genetics and Germplasm Enhancement, Nanjing Agricultural University, Nanjing 210095, China;3. Tea Research Institute, Nanjing Agricultural University, Nanjing 210095, China
Abstract:Tea is an important economic crop. Tea production is affected by temperature factors. CsDof1 and CsDof2,which encode Dof transcription factors, were cloned from tea cultivar ‘Yingshuang’ [Camellia sinensis (L.) O. Kuntze]. The nucleotide and amino acid sequences, phylogenetic tree and molecular models of the CsDof1 and CsDof2 were analyzed. The results showed that the CsDof1 and CsDof2 are 1389 and 1458 nucleotides in length,which encode 463 and 486 amino acids respectively. Typical zinc finger domains are present in both CsDof1 and CsDof2, from coden 133 to 195 and 124 to 186 respectively. The CsDof1 and CsDof2 were hydrophilic proteins. The three-dimensional structures of CsDof1 and CsDof2 indicated the presence of a highly conserved Dof domain with four cysteine residues. Quantitative real-time PCR analysis showed that CsDof1 and CsDof2 were induced by different temperature stresses and the expression profiles were different among tea cultivars.
Keywords:Camellia sinensis, Dof transcription factor, zinc finger, temperature stress, gene expression
