贵州省安顺地区蛇源裂头蚴分离株的分子鉴定及种系发育关系分析
2016-07-27李金福刘艳丹裘学丽
李金福,刘艳丹,陈 艳,裘学丽
贵州省安顺地区蛇源裂头蚴分离株的分子鉴定及种系发育关系分析
李金福,刘艳丹,陈艳,裘学丽
贵州医科大学人体寄生虫学教研室,贵阳550004
摘要:目的对贵州省安顺地区蛇源裂头蚴分离株进行分子鉴定及种系发育关系的分析。方法采集贵州安顺地区4种常见野生蛇(王锦蛇、乌梢蛇、黑眉锦蛇、灰鼠蛇)来源的裂头蚴,分别提取各裂头蚴基因组DNA,PCR扩增ITS1和cox1基因,所得PCR扩增产物经纯化并T-A克隆后测序。运用ClustalX1.81生物分析软件,将所得序列与GenBank所录迭宫属绦虫和其它属绦虫基因序列进行多重序列比对分析,并应用软件MEGA 6.0构建系统发育树(邻位连接法)。结果成功扩增出8株裂头蚴的ITS1和cox1基因序列,ITS1大小为822~863 bp,cox1大小为404~415 bp,与预期的基因片段大小一致。基于ITS1和cox1基因序列构建的种系发育树与所有的猬迭宫绦虫均构成同一亚群,总分支的自展值高于50%,二者在不同的分离株之间呈现基因多态性。对8株裂头蚴ITS1和cox1序列的同源性进行比较,ITS1的同源性为70.27%~82.84%,而cox1的同源性为95.63%~99.50%,显示cox1的同源性高于ITS1。已向GenBank提交ITS1和cox1新序列各一条,登录号分别为KF990161和KJ418421。结论贵州安顺地区不同蛇源裂头蚴分离株之间存在基因多态性。从总分支的自展值来看,cox1比ITS1更适合用于种内遗传多态性研究,ITS1适合做定种的分子标记。
关键词:裂头蚴;第一内转录间隔区;细胞色素C氧化酶第一基因;种系发育分析
Supported by the Science and Technology Department of Guizhou Province (No. 2014-3024)
迄今,有关迭宫属绦虫种类的报告,全世界共计40余种之多,但多为从形态学、生物学及生物化学等方面鉴定的种类,因此,同物异名现象普遍[1]。猬迭宫绦虫是许多国家,包括我国的流行虫种,其同物异名也多达数十种。猬迭宫绦虫的中间宿主和终宿主种类较多,在不同的地理环境和宿主体内,虫体会出现基因遗传差异。开展贵州不同蛇源裂头蚴虫株的分子鉴定,分析不同蛇源裂头蚴分离株ITS1和cox1基因序列,比较其同源性,其目的是确定贵州省致病虫株的基因型,揭示贵州不同蛇源裂头蚴之间的种内关系,为进一步研究不同基因型蛇源裂头蚴的生物学特性、致病性差异以及药物敏感性等奠定基础[2-3]。
1材料与方法
1.1材料
1.1.1虫株来源从野生王锦蛇(Elaphecarinata)、乌梢蛇(Zaocysdhumnades)、黑眉锦蛇(Elaphetaeniura)及灰鼠蛇(Ptyaskorro)(捕获于贵州安顺地区)皮下检获裂头蚴后,先用生理盐水清洗虫体,然后对虫体进行形态鉴定,虫体保存于70%酒精溶液中。虫株编号:王锦蛇株1-2号(WJ1-2)、乌梢蛇株1-2号(WS1-2)、黑眉锦蛇株1-2(HM1-2)及灰鼠蛇株1-2(HS1-2)。
1.1.2主要试剂Universal Genomic DNA Extraction Kit Ver.3.0试剂盒、T-A克隆试剂盒购于大连宝生物公司;分子量标准DNA marker DL2000、2×Es Taq MasterMix(含染料)、 DNA凝胶回收试剂盒为北京康为生物有限公司产品;其它试剂为进口或国产分析纯。
1.1.3引物序列参考Jeon等[4]的文献设计扩增ITS1及cox1基因的两对引物。PL-cox1F:5′-TTTTTTGGGCATCCTGAGGTTTAT-3′、PL-cox1R:5′-TAAAGAAAGAACATAATGAAAATG-3′;PL-ITS1F:5′-AACAAGGTTTCCGTAGGTG-3′、PL-ITS1R:5′-AGCAGTCTGCGATTCACATT-3′。引物由北京鼎国生物股份有限公司合成。
1.2方法
1.2.1裂头蚴基因组DNA提取按Universal Genomic DNA Extraction Kit Ver.3.0试剂盒使用说明书提取裂头蚴基因组DNA,所获基因组DNA置于-20 ℃保存备用。
1.2.2PCR扩增ITS1序列和cox1基因的体系及条件50 μL反应体系:2×Es Taq MasterMix (含有EsTaqDNA Polymerase、2×Es Taq PCR Buffer,3 mmol MgCl2和400 μmol dNTP mix)25 μL、10 μmol/L 引物各2 μL、DNA 模板1 μL(<1 μg)和RNase-Free Water 20 μL。PCR反应条件:94 ℃预变性2 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 30 s,30个循环;72 ℃延伸2 min。
1.2.3PCR扩增基因片段测序PCR产物经胶回收纯化后做T-A克隆测序。序列测定交由上海美吉生物医药科技有限公司进行。
1.2.4序列数据分析测序所得序列先与NCBI数据库收录基因序列进行BLAST比对,鉴定所获虫株为何种裂头蚴,之后再与GenBank收录的所有迭宫属绦虫和其它属绦虫的ITS1基因序列和cox1基因序列进行种间和种内比对;用ClustalX 1.81进行序列同源性比较和完全比对。利用MEGA6.0软件对贵州安顺地区8株蛇源裂头蚴标本之间进行序列遗传多样性分析。
1.2.5分子系统发育树构建利用MEGA6.0软件构建系统发育树。采用贵州安顺地区分离的8株蛇源裂头蚴标本的cox1基因和ITS1序列,用GenBank已录其他属绦虫cox1基因和ITS1序列作外类群,以NJ法构建种间系统发育树;用GenBank已录猬迭宫绦虫cox1基因和ITS1序列作外类群,以NJ法构建种内系统发育树;以NJ法构建8株蛇源裂头蚴标本的株间系统发育树。种间、种内及株间的系统发育树自展值(Boot-strap values)重复1 000次,取多数的一致树。
2结果
2.1PCR扩增结果对贵州安顺地区8个裂头蚴标本的cox1和ITS1基因序列进行扩增,cox1大小约400 bp左右,与预期的cox1基因片段大小一致(图1)。ITS1大小约800 bp左右,与预期的ITS1基因片段大小一致(图2),8个标本cox1和ITS1基因扩增片段的大小及GC含量见表1。
2.2新序列提交用BankIt向NCBI提交了两个序列,cox1提交的是WS1标本,ITS1提交的是HM1标本,GenBank登陆号分别为KF990161和KJ418421。
2.3裂头蚴株之间同源性比较对贵州安顺地区8株蛇源裂头蚴的cox1基因和ITS1序列的同源性进行比较,结果显示cox1基因的同源性为95.63%~99.50%,而ITS1序列的同源性为70.27%~82.84%。
2.4安顺地区裂头蚴株与已知各属绦虫cox1基因和ITS1序列同源性比较用WS1标本的cox1基因、HM1标本的ITS1序列与GenBank已录各属绦虫cox1和ITS1序列做比较,发现贵州安顺地区裂头蚴株的cox1基因和ITS1序列与已录猬迭宫绦虫序列具有很高的同源性,与其它各属绦虫序列具有较低的同源性(表2)。
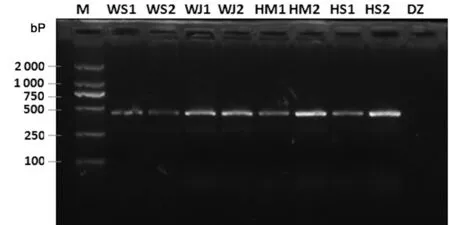
M:DL 2 000相对分子质量标准;WS1-2:为乌梢蛇1-2的cox1扩增片段;WJ1-2:为王锦蛇1-2的cox1扩增片段;HM1-2:为黑眉锦蛇1-2的cox1扩增片段;HS1-2:为灰鼠蛇1-2的cox1扩增片段;DZ:阴性对照。M: DL 2000 DNA Marker; WS1-2: PCR products of cox1 of samples 1-2 from Zaocys dhumnades; WJ1-2:PCR products of cox1 of samples 1-2 from Elaphe carinata; HM1-2: PCR products of cox1 of samples 1-2 from Elaphe taeniura; HS1-2: PCR products of cox1 of samples 1-2 from Ptyas korros; DZ: Negative control.图1 8株裂头蚴cox1基因PCR扩增产物的琼脂糖凝胶电泳分析Fig.1 Analysis of PCR amplified cox1 genes from 8 pleroceroid strains by agarose gel electrophoresis
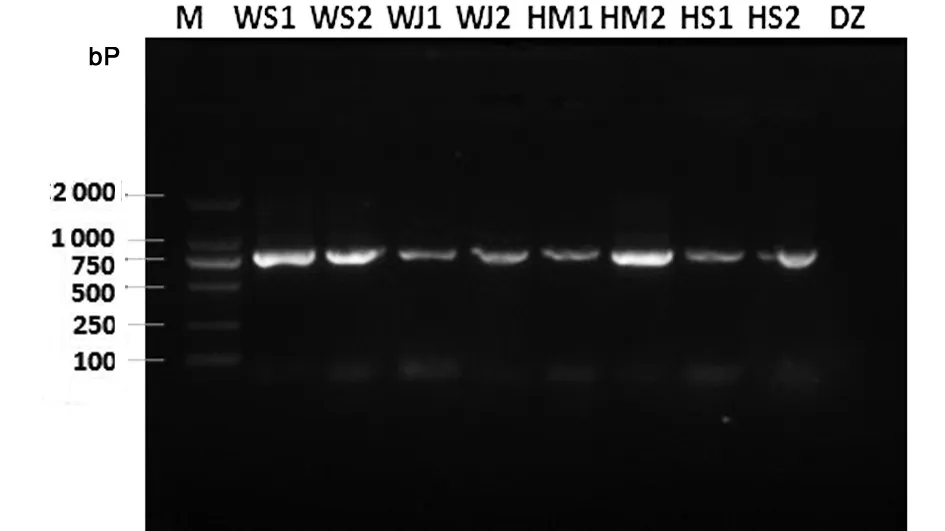
M:DL 2 000相对分子质量标准;WS1-2:为乌梢蛇1-2的ITS1扩增片段;WJ1-2:为王锦蛇1-2的ITS1扩增片段;HM1-2:为黑眉锦蛇1-2的ITS1扩增片段;HS1-2:为灰鼠蛇1-2的ITS1扩增片段;DZ:阴性对照。M: DL 2000 DNA Marker; WS1-2: PCR products of ITS1 of samples 1-2 from Zaocys dhumnades; WJ1-2: PCR products of ITS1 of samples 1-2 from Elaphe carinata; HM1-2: PCR products of ITS1 of samples 1-2 from Elaphe taeniura; HS1-2: PCR products of ITS1 of samples 1-2 from Ptyas korros; DZ: Negative control.图2 8株裂头蚴ITS1基因PCR扩增产物的琼脂糖凝胶电泳分析Fig.2 Analysis of PCR amplified ITS1 genes from 8 pleroceroid strains by agarose gel electrophoresis
表18株裂头蚴cox1及ITS1基因的长度及GC含量
Tab.1Guanine-cytosine (GC) content and length of the cox1 and ITS1 sequences of 8 pleroceroid strains
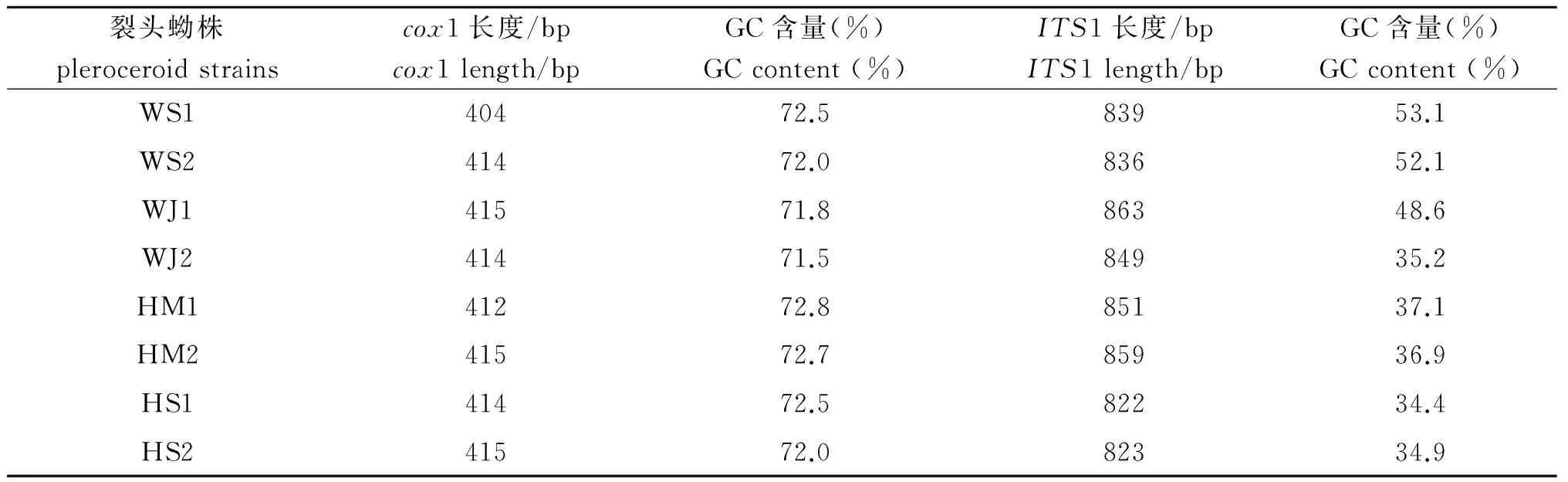
裂头蚴株cox1长度/bpGC含量(%)ITS1长度/bpGC含量(%)pleroceroidstrainscox1length/bpGCcontent(%)ITS1length/bpGCcontent(%)WS140472.583953.1WS241472.083652.1WJ141571.886348.6WJ241471.584935.2HM141272.885137.1HM241572.785936.9HS141472.582234.4HS241572.082334.9
表2裂头蚴与其他不同绦虫的cox1和ITS1序列同源性比对
Tab.2Homology comparison of cox1 and ITS1 sequences between plerocercoids and other different tapeworms
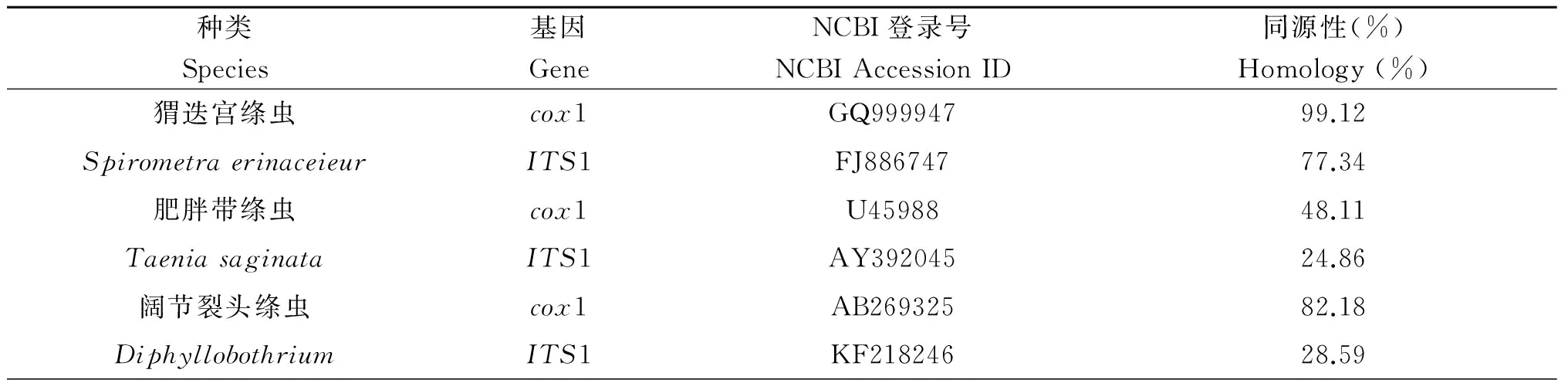
种类Species基因GeneNCBI登录号NCBIAccessionID同源性(%)Homology(%)猬迭宫绦虫cox1GQ99994799.12SpirometraerinaceieurITS1FJ88674777.34肥胖带绦虫cox1U4598848.11TaeniasaginataITS1AY39204524.86阔节裂头绦虫cox1AB26932582.18DiphyllobothriumITS1KF21824628.59
表2(续)
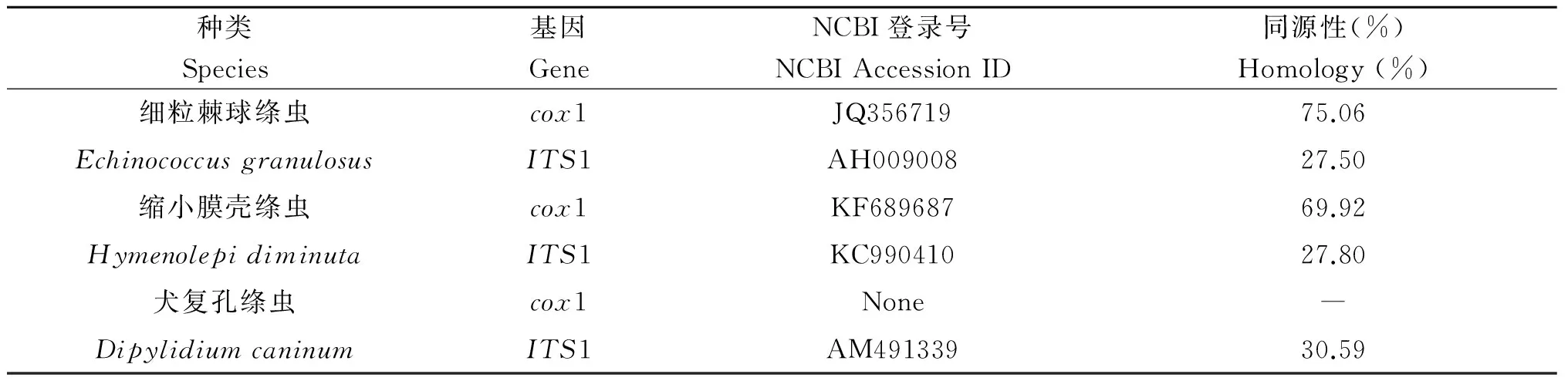
种类Species基因GeneNCBI登录号NCBIAccessionID同源性(%)Homology(%)细粒棘球绦虫cox1JQ35671975.06EchinococcusgranulosusITS1AH00900827.50缩小膜壳绦虫cox1KF68968769.92HymenolepidiminutaITS1KC99041027.80犬复孔绦虫cox1None―DipylidiumcaninumITS1AM49133930.59
2.58株裂头蚴差异分析根据同源性比对的结果,选取cox1基因,利用MEG6.0软件对贵州安顺地区8株蛇源裂头蚴标本之间进行序列多样性分析。根据完全比对结果,校对并除去cox1两端的不稳定序列,最终获得了397 bp的cox1基因片段,没有发现碱基的插人和缺失。对8个标本DNA序列进行遗传多样性分析,8个标本共有12个变异位点,其中有7个为简约信息位点。12个变异位点中,转换数为11个(3个为C/T转换,8个为A/G转换),仅有2个A/T颠换,转换/颠换率为5.5,转换明显多于颠换,表明8株蛇源裂头蚴标本的cox1基因序列变异还未达到饱和状态,可以将此基因应用于遗传变异和系统进化分析(表3)。
基于cox1基因,用MEGA6.0软件根据Kimura双参数模型计算了8株标本间的遗传距离。结果
表明,标本间的遗传距离差异不大,范围为0.005 07~0.003 120,平均遗传距离为0.018 28(表4)。
表38株裂头蚴cox1基因变异位点
Tab.3Mutation sites of cox1 sequences of 8 pleroceroid strains
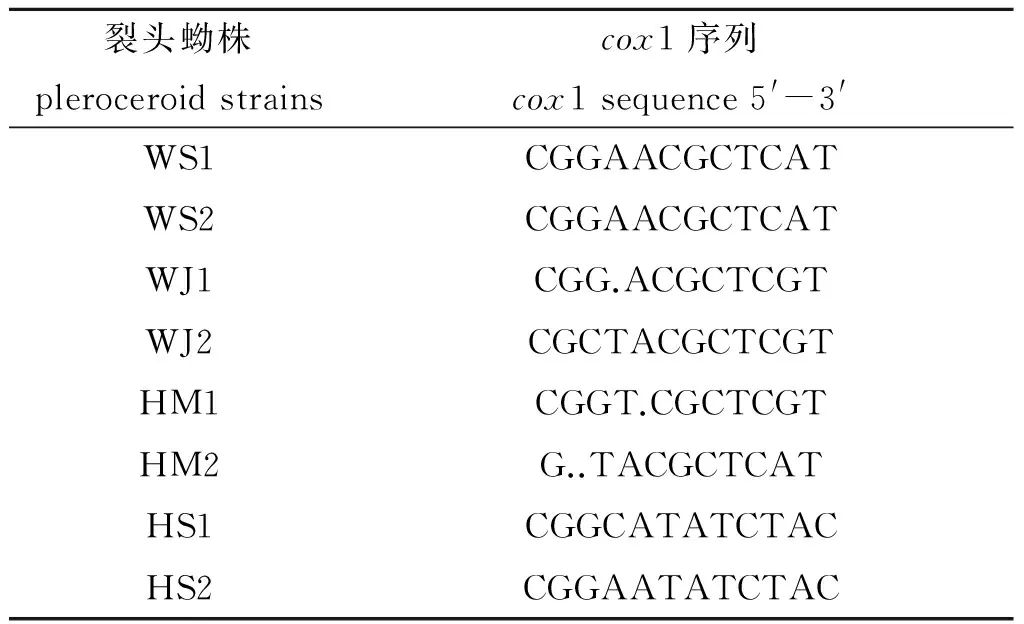
裂头蚴株cox1序列pleroceroidstrainscox1sequence5'-3'WS1CGGAACGCTCATWS2CGGAACGCTCATWJ1CGG.ACGCTCGTWJ2CGCTACGCTCGTHM1CGGT.CGCTCGTHM2G..TACGCTCATHS1CGGCATATCTACHS2CGGAATATCTAC
表48株裂头蚴cox1基因片段间的遗传距离
Tab.4Genetic distance of cox1 gene fragments of 8 pleroceroid strains
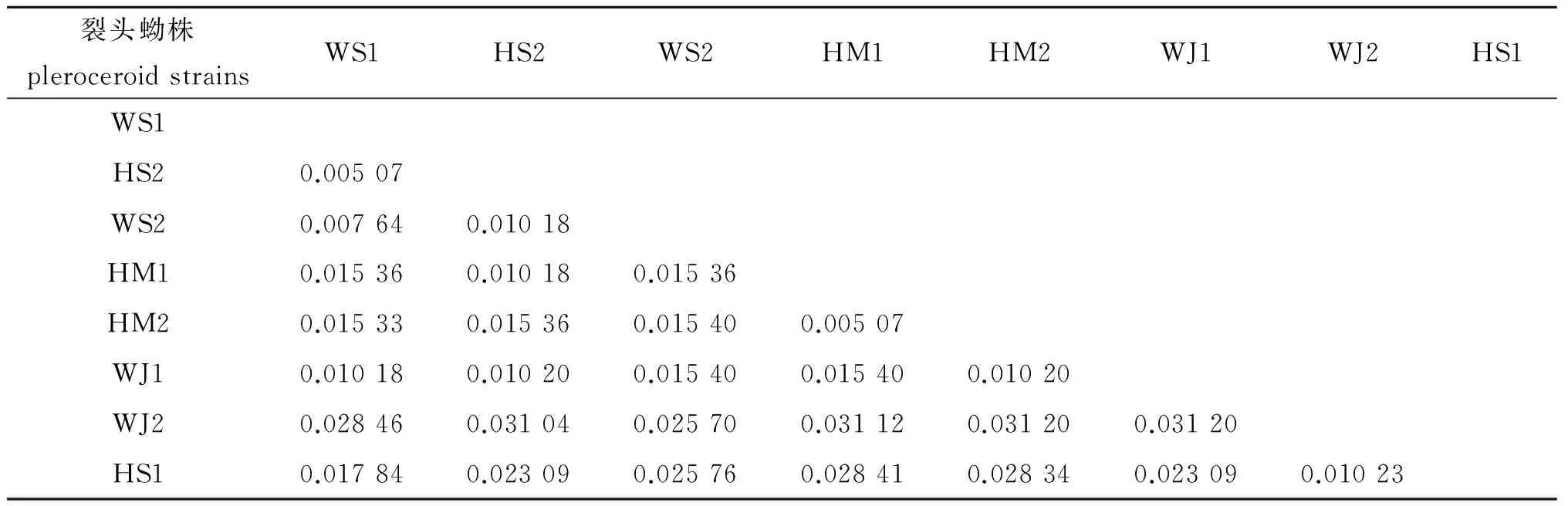
裂头蚴株pleroceroidstrainsWS1HS2WS2HM1HM2WJ1WJ2HS1WS1HS20.00507WS20.007640.01018HM10.015360.010180.01536HM20.015330.015360.015400.00507WJ10.010180.010200.015400.015400.01020WJ20.028460.031040.025700.031120.031200.03120HS10.017840.023090.025760.028410.028340.023090.01023
2.6应用NJ法构建系统发育树
2.6.1种间系统发育树构建采用WS1标本的cox1(KJ418421)基因和HM1标本的ITS1(KF990161)序列,用GenBank已录的其他属绦虫cox1基因(E.granulosusJQ356719,H.diminutaKF689687,D.latumAB269325,T.saginata U45988)和ITS1序列(D.caninumAM491339,E.granulosusAH009008,D.latumKF218246,T.saginataAY392045,H.diminutaKC990410)作外类群,以NJ法构建种间系统发育树。基于ITS1序列构建的多数种系发育树(图3)中,显示本次检测的裂头蚴株(HM1)与已知的其他属绦虫处于不同分支。亚群内出现3个主要拓扑分支,自展值高于40%。基于cox1基因构建的多数的种系发育树(图4)中,显示本次检测的裂头蚴(WS1)与所知的其他属绦虫处于不同分支,亚群内出现3个主要拓扑分支,自展值高于80%。

图3 基于ITS1基因序列以邻位置连接法构建的多数一致树(种间)Fig.3 Bootstrap cut-off consensus tree inferred from ITS1 gene given in text using the Neighbor-Joining method
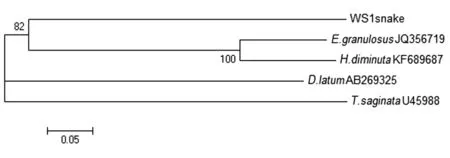
图4 基于cox1基因序列以邻位置连接法构建的多数一致树(种间)Fig.4 Bootstrap cut-off consensus tree inferred from cox1 gene given in text using the Neighbor-Joining method
2.6.2种内系统发育树构建采用WS1标本的cox1基因(KJ418421)和HM1标本的ITS1(KF990161)序列,用GenBank已录的猬迭宫绦虫cox1基因(S.erinaceieuropaeiAB278577,S.erinaceieuropaeiAB278576,S.erinaceieuropaeiAB278575,S.erinaceieuropaeiAB278574,S.erinaceieuropaeiAB278573,SpirometraGQ999947,)和ITS1序列(S.erinaceieuropaeiFJ886755,S.erinaceieuropaeiFJ886752,S.erinaceieuropaeiFJ886751,S.erinaceieuropaeiFJ886754,S.erinaceieuropaeiFJ886753,S.erinaceieuropaeiFJ886747)作外类群,以NJ法构建种内系统发育树。基于ITS1序列构建的60%种系发育树(图5)中,显示本次检测的裂头蚴株(HM1)与已知的猬迭宫绦虫均处于同一个亚群内。亚群内出现3个主要拓扑分支,自展值分别为72%,100%,和100%,HM1和FJ886747为一分支。基于cox1基因构建的60%的种系发育树(图6)中,显示本次检测的裂头蚴(WS1)与所知的猬迭宫绦虫形成同一个亚群,亚群内出现3个主要拓扑分支,自展值分别为57%,82%,和89%。WS1与GQ999947处于同一分支的亚支,自展值为100%。

图5 基于ITS1序列以邻位置连接法构建的60%多数一致树(种内) Fig.5 Bootstrap 60% cut-off consensus tree inferred from ITS1 gene given in text using the Neighbor-Joining method
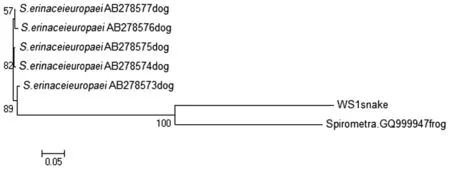
图6 基于cox1基因以邻位置连接法构建的60%多数一致树(种内)Fig.6 Bootstrap 60% cut-off consensus tree inferred from cox1 gene given in text using the Neighbor-Joining method
2.6.3株间系统发育树构建采用8株裂头蚴标本的cox1基因和ITS1序列以NJ法构建株间系统发育树。基于TIS1序列构建的60%株间发育树(图7)中,总共分有2个大分支,HS1和HS2为1分支的亚支,WS1和WS2为2分支的亚支,HM1和HM2是1分支的亚支,而WJ1和WJ2分属于2个分支,两大分支的自展值检测分别为68%和100%。基于cox1基因构建的60%的株间发育树(图8)中,总共分有3个大分支,WS1和HS2为1分支,WS2为2分支,WJ1,HM2和HM1号为3分支的亚支,HS1和WJ2为3分支的另一亚支。3大分支的自展值检测分别为81%和100%和68%。
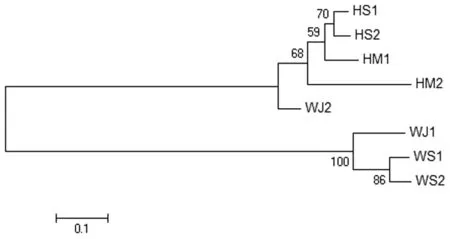
图7 用裂头蚴ITS1序列构建的系统发育树(邻位连接法)Fig.7 Phylogenetic construction based on plerocercoid ITS1 sequences(Neighbor Joining Method)
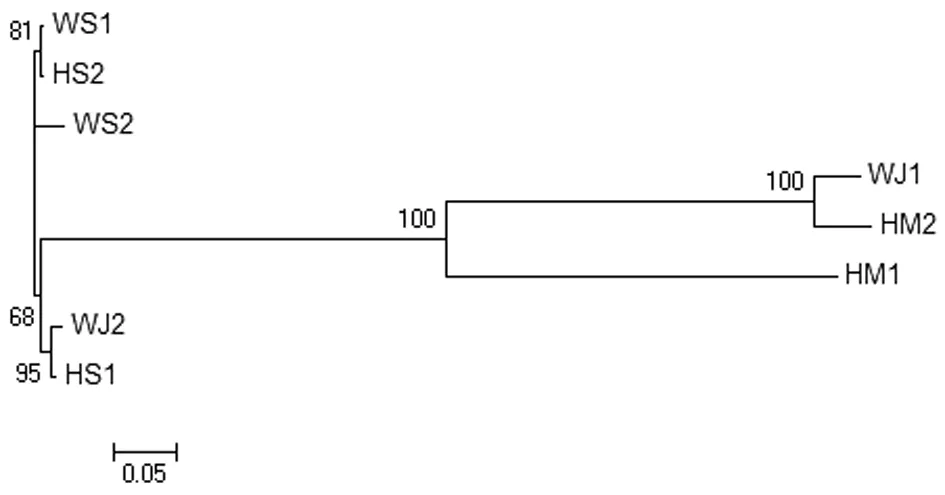
图8 用裂头蚴cox1基因构建的系统发育树(邻位连接法)Fig.8 Phylogenetic construction based on plerocercoid cox1 sequences(Neighbor Joining Method)
3讨论
在虫株的分子鉴定及种系发育关系的分析中,分子标记的选择十分重要,如果分子标记的变异太小,种群的差异不易发现,而变异太大,则可造成趋同进化的假象,混淆真正的亲缘关系[5]。ITS基因序列存在于核糖体DNA(rDNA)中,它的进化速度慢而且长度较小,相对保守,因而常被运用于分子发育种间及种内研究。cox基因位于线粒体DNA(MtDNA)中,线粒体基因组具有结构简单、母系遗传、缺少重组、进化速率快等特点,特别适合作为遗传学研究的标记,已被许多学者用来研究多种寄生虫的种间和种内遗传变异。因此,本文采用了这2个分子标记进行研究。
本研究测定了贵州安顺地区4种蛇源8个裂头蚴虫株的ITS1和cox1基因。ITS1大小为822~863 bp,各标本ITS1长度差异较大,而cox1大小为404~415 bp,各标本的差异较小。对8株裂头蚴ITS1和cox1序列的同源性进行比较,ITS1的同源性为70.27%~82.84%,而cox1的同源性为95.63%~99.50%,显示cox1的同源性高于ITS1。
取HM1标本的ITS1序列和WS1标本的cox1基因在GenBank里在线BLAST,发现ITS1有序列同源性达到77%,基因登录号是FJ886747,为Spirometraerinaceieuropaei的ITS1序列;发现cox1有序列同源性达到99%,基因登陆号是GQ999947,为Spirometraerinaceieuropaei的cox1基因。在种内系统发育树中,HM1标本的ITS1序列和FJ886747为同一分支,自展值100%,WS1标本的cox1基因和GQ999947为同一分支,自展值100%。通过ITS1及cox1两个基因的BLAST比对,种内系统发育树中的亲缘关系,可鉴定所得标本HM1及WS1均为猬迭宫绦虫。
另外,用HS1标本的cox1基因、WS1标本的ITS1序列和其他各属绦虫cox1和ITS1序列作序列对比,发现种内cox1基因序列差异(0.74%~4.37%)明显低于种间差异(17.82%~51.89%);种内ITS1序列差异(19.29%~29.73%)也明显低于种间差异(69.41%~75.14%),表明迭宫绦虫cox1和ITS1序列种内相对保守,种间差异较大,故均可作为种间遗传变异研究的分子标记,可用于迭宫绦虫的鉴定。
本研究运用NJ法,基于ITS1序列和cox1基因,分别构建了种间、种内系统发育树,以及8个蛇源裂头蚴标本之间的株间系统发育树,其中 Bootstrap method检测自展值1 000次,结果取多数发育树,大部分高于60,说明发育树比较可靠。
根据3种系统发育树,观察到基于cox1的发育树敏感度要比ITS1要高,树的进化距离值也比ITS1序列要低很多,由此可推断,在猬迭宫绦虫裂头蚴分子种系发育关系研究中,运用ITS基因序列有可能更适合于做定种的分子标记,而cox1更适合于做种内遗传多态性研究。
Zhu XQ等[6]基于cox1基因进行多态性分析的情况,他们发现在澳大利亚从不同宿主分离得到的S.erinaceieuropaei至少存在2种基因型。而Okamoto M[7]等的研究也同样支持了这个结论,他从不同国家分离得到的S.erinaceieuropaei,种内也存在不同基因型。本研究根据8个蛇源裂头蚴之间系统发育树的结果来看,同样支持这个结论,且进一步分析推测,不同蛇源裂头蚴基因型有差异,同种S.erinaceieuropaei,种内可能存在不同基因型。基于ITS1序列的系统发育树,8个裂头蚴标本可分3个基因型。基于cox1基因的系统发育树,8个裂头蚴标本可分2个基因型。
除了ITS序列与cox1基因可作为猬迭宫绦虫很好的分类鉴定的分子标记之外,线粒体nad基因也是绦虫分类鉴定理想的遗传标记之一。刘自逵等[8-9]的研究表明,猬迭宫绦虫nadl比cox1基因序列的种间与种内差异要大一些。伍慧兰等[10]在对猬裂头蚴nad4基因的分析中也发现日本株和湖南株的pnad4的序列存在一定的差异。今后在裂头蚴种群关系的研究中,可以结合这几种基因,为虫株作精确的鉴定和分类,为有效诊断和控制裂头蚴病奠定基础。
参考文献:
[1] Chen Y. Harm, prevention and cure of food-borne parasitic disease[M]. Guiyang: Guizhou science press, 2013: 15-50. (in Chinese)
陈艳.食源性裂头绦虫病的危害与防治[M].贵阳:贵州科技出版社,2013:15-50.
[2] Li WW, Li J, Li SQ, et al. Molecular identification and phylogenetic analysis of plerocercoids in snakes from Guilin city[J]. Prog Vet Med, 2011, 32(10): 28-30. (in Chinese)
李雯雯,李健,李树清,等. 桂林蛇源裂头蚴分离株的分子鉴定及种系发育关系分析[J]. 动物医学进展,2011,32(10):28-30.
[3] Wang ZR, Bao HE. Identification ofTaeniasaginataby mtCOI in four areas of Yunnan and Guizhou province[J].Chin J Parasitol Parasit Dis, 2003, 21(1): 20-23. (in Chinese)
王正蓉, 包怀恩. 用mtCOI技术测定云南及贵州四地区的牛带绦虫[J]. 中国寄生虫学与寄生虫病杂志, 2003, 21(1): 20-23.
[4] Jeon HK, Kim KH, Huh S, et al. Morphologic and genetic identification ofDiphyllobothriumnihonkaiensein Korea[J]. Korean J Parasitol, 2009, 47(4): 369-375. DOI: 10.3347/kjp.2009.47.4.369
[5] Li WP, Cui JY. Sequence and phylogenetic analysis of mitochondrialcox1 gene ofSpirometraerinaceieuropaei[J]. Chin
Livestock Poultry Breeding, 2012, 8(8): 60-61. (in Chinese)
李伟平,崔建杨.猬迭宫绦虫的线粒体cox1序列测定及种系发育关系分析[J].中国畜禽种业,2012,8(8):60-61.
[6] Zhu XQ, Beveridge I, Berger L, et al. Single-strand conformation polymorphism-based analysis reveals genetic variation withinSpirometraerinacei(Cestoda:Pseudophyllidea) from Australia[J]. Mol Cellular Probes, 2002, 16: 159-165. DOI: 10.1006/mcpr.2002.0406
[7] Okamoto M, Iseto C, Shibahara T, et al. Intraspecific variation ofSpirometraerinaceieuropaeiand phylogenetic relationship betweenSpirometraandDiphyllobothriuminferred from mitochondrial COI gene sequences[J]. Parasitol Int, 2007, 56: 235-238. DOI: 10.1016/j.parint.2007.03.003
[8] Liu ZK, Liu GH, Dai RS, et al. Sequence and phylogenetic analysis of mitochondrialcox1 andnad1 genes forSpirometraerinaceieuropaeiisolates from Hunan province[J]. Acta Veterinaria et Zootechnica Sinica, 2010, 41(4): 463-468. (in Chinese)
刘自逵,刘国华,戴荣四,等. 湖南省猬迭宫绦虫的线粒体cox1和nad1基因的序列测定及种系发育分析[J]. 畜牧兽医学报,2010,41(4):463-468.
[9] Liu DW, Zhu B, Wang XB, et al. Molecular cloning of two fragments cDNA containing repeat units inSpirometraerinaceieuropaei[J]. Chin J Dis Ctrl Prev, 2001, 5(4): 294-297. (in Chinese)
刘殿武,朱冰,王晓波,等. 猬迭宫绦虫幼虫含重复序列的cDNA 的分子克隆[J]. 疾病控制杂志,2001,5(4):294-297.
[10]Wu HL. Cloning and sequence analysis of mitochondrial NADH dehydrogenase subunit 4 ofSparganumerinaceieuropaeiisolates from Hunan Province[J]. Prog Vet Med, 2010, 31(12): 61-63. (in Chinese)
伍慧兰. 猬裂头蚴线粒体nad4基因的克隆及序列分析[J]. 动物医学进展,2010,31(12):61-63.
DOI:10.3969/j.issn.1002-2694.2016.04.012
通讯作者:陈艳,Email:chenyan757@sina.com
中图分类号:R383
文献标识码:A
文章编号:1002-2694(2016)04-0376-07
Corresponding author:Chen Yan, Email: chenyan757@sina.com
收稿日期:2015-09-24修回日期:2016-01-08
Molecular identification and phylogenetic analysis of plerocercoids in snakes from Anshun, Guizhou, China
LI Jin-fu, LIU Yan-dan, CHEN Yan, QIU Xue-li
(DepartmentofParasitology,GuizhouMedicalUniversity,Guiyang550004,China)
Abstract:In this study, we investigated the taxonomy position of plerocercoids from snakes in Anshun area, Guizhou Province, China. Plerocercoids were isolated from the wild snakes (Elaphe carinata, Zaocys dhumnades, Elaphe taeniura, Ptyas korros) in this area. Internal transcribed spacer 1 (ITS1) gene and the mitochondrial cytochrome oxidase 1 (cox1) gene of the plerocercoids were amplified by PCR, and the sequences were measured. The similarity of sequences was aligned by ClustalX 1.81, and the phylogenetic trees were constructed by MEGA 6.0 (Neighbor-Joining method, NJ). Results showed that the ITS1 and cox1 sequences of 8 pleroceroid strains were amplified successfully by PCR, and the homology of sequences was aligned by ClustalX 1.81. The ITS1 was 822-863 bp, while cox1 was 404-415 bp. The homology of ITS1 and cox1 was 70.27%~82.84% and 95.63%~99.50%, respectively. The phylogenetic trees were constructed by MEGA 6.0(NJ). We had submitted an ITS1 and a cox1 new sequences to GenBank. The accession ID were KF990161 and KJ418421 respectively. The study results suggest that gene polymorphism between different isolates of pleroceroids from snakes in cox1 and ITS1, cox1 is more suitable than ITS1 in studying intra-specific genetic polymorphism which considered from the bootstrap values, and ITS1 could be used as genetic market for the species identification.
Keywords:pleroceroid; ITS1; cox1; phylogenetic analysis
贵州省科技厅社会发展攻关项目资助[No.(2014)3024号]
