应用PCR-FINS技术鉴定金枪鱼罐头中金枪鱼种类
2016-07-21安丽艳孟镇仇凯钟其顶杨彤晖郭新光
安丽艳,孟镇,仇凯*,钟其顶,杨彤晖,郭新光
1(中国食品发酵工业研究院,北京,100015) 2(全国食品发酵标准化中心,北京,100015)
应用PCR-FINS技术鉴定金枪鱼罐头中金枪鱼种类
安丽艳1,2,孟镇1,2,仇凯1,2*,钟其顶1,2,杨彤晖1,2,郭新光1,2
1(中国食品发酵工业研究院,北京,100015) 2(全国食品发酵标准化中心,北京,100015)
摘要为考察金枪鱼罐头中DNA降解程度以及鉴定金枪鱼罐头中金枪鱼种类,文中根据鱼类线粒体基因组(m tDNA)细胞色素b(cyt b)基因和12S rDNA基因,利用3对通用引物分别扩增片段大小为 358 bp(cyt b)、224 bp(12S rDNA)、123bp(cyt b)的目的片段,同时,基于123 bp线粒体cytb基因片段,通过法医信息核苷酸测序(PCR-FINS)技术来鉴别金枪鱼罐头中金枪鱼的种类,并对17个市售金枪鱼罐头样品进行金枪鱼种类鉴定。结果表明:金枪鱼罐头中DNA降解严重,其片段大小在100~200 bp;该研究建立的PCR-FINS技术,可以实现金枪鱼罐头中金枪鱼种类的准确鉴别;17个市售金枪鱼罐头样品中,58.8%来源于价格低廉的鲣鱼,其次还包括长鳍金枪鱼、青干金枪鱼、扁鮀鲣、鲔鱼。
关键词PCR-FINS;金枪鱼罐头;种类鉴定
金枪鱼类是硬骨鱼纲、辐鳍亚纲、鲈形目、鲭科中金枪鱼属、狐鲣属、舵鲣属、鲣属和鲔属的统称,多达17个金枪鱼品种[1]。金枪鱼罐头因其营养美味、食用方便、货架期长、便于携带等特点,深受广大消费者的喜爱[2]。然而,金枪鱼罐头市场鱼目混杂,标示不清[3],市面上所销售的金枪鱼罐头,大多仅标示为金枪鱼罐头,未标注所用原料的具体种属信息。
水产加工品的原料品种鉴别已成为水产加工行业中面临的重要问题之一。2003年,欧盟建立了鱼类及其制品标签法,明确规定鱼类制品必须贴有标明商业名称、生产方法和捕获区域的标签[4]。美国食品药品管理局(FDA)列出一个可食水产品名单,规定水产品标示必须使用该名单中规定的名称[5]。我国国家标准GB7718—2011《食品安全国家标准预包装食品标签通则》也明确要求,食品名称应清晰的反映食品的真实属性[6]。
目前,国内对鱼类罐头鱼源性成分鉴别尚无相关报道。本研究采用PCR-FINS方法对金枪鱼罐头食品鱼源性成分进行鉴定,旨在建立高效、便捷的金枪鱼罐头中金枪鱼种类的PCR-FINS鉴定方法。
1材料和方法
1.1实验材料
金枪鱼罐头,购自当地的超市。黄鳍金枪鱼生鱼片作为阳性对照。
1.2主要试剂
CTAB裂解液[4% CTAB,100 mmol/L Tris-HCl(pH 8.0),1.4 mol/L NaCl,20 mmol/L EDTA(pH 8.0)];TE缓冲液(0.01 mol/L Tris,0.001 mol/L EDTA,pH 8.0);Taq DNA聚合酶、dNTPs、100 bp Marker,均购自北京全式金生物技术有限公司;琼脂糖、三氯甲烷/异戊醇体积比241、无水乙醇、异丙醇。
1.3主要仪器
冷冻离心机,美国Sigma公司;T gradient PCR扩增仪,德国Biometra公司;DYY-8C型高压电泳仪,北京六一仪器厂;CV-1000型凝胶成像系统,法国VIBER LOURMAT公司;压力蒸汽灭菌锅(LS-B50L),上海医用核子仪器厂。
1.4实验方法
1.4.1DNA提取
取100 mg金枪鱼罐头肌肉组织样品于2 mL离心管中,加入1 mLV(三氯甲烷)∶V(甲醇)∶V(水)=1∶2∶0.8,室温静置过夜,5 000 r/min离心3 min,弃上清液,无菌去离子水清洗样品,样品组织于-40 ℃保存备用。
参考UNSELD[7]的研究,并进行适当修改。取上述组织样品,加入1mL CTAB提取液[4% CTAB,100 mmol/L Tris-HCl(pH 8.0),1.4 mol/L NaCl,20 mmol/L EDTA(pH 8.0)],65℃温育1~2 h,期间每隔15 min颠倒混匀1次。温育后,离心取上清液,等体积的V(三氯甲烷)∶V(异戊醇)=24∶1抽提2次。加2/3体积预冷的异丙醇沉淀DNA,-20 ℃,1 h。离心弃上清液,沉淀用体积分数70%预冷的乙醇洗涤2次,干燥。沉淀用100 μL TE(0.01 mol/L Tris,0.001 mol/L EDTA,pH 8.0)缓冲液溶解,冰冻保存。
1.4.2基因扩增
为了考察金枪鱼罐头中DNA降解程度,以7种不同加工工艺的金枪鱼罐头为研究对象,1.4.1提取的线粒体基因组DNA为模板,参考文献[8-9],根据鱼类线粒体基因组(mt DNA)cytb基因和12Sr DNA 基因,选取3对通用引物(见表1)进行PCR反应。

表1 通用引物序列及扩增产物大小
PCR扩增体系为25μL:10×PCR buffer(Mg2+)2.5 μL,dNTP(2.5 mmol/L)2μL,20 pmol/μL引物各1 μL,模板DNA 2 μL,Taq DNA Polymerase 0.3 μL(1.5U),ddH2O 16.2 μL。PCR扩增条件为:94 ℃预变性300 s,94 ℃变性30 s,相应退火30 s,72 ℃延伸30 s,35个循环,最后在72 ℃延伸300 s。PCR扩增产物用2%的琼脂糖凝胶电泳检测。
1.4.3市售样品基因扩增
将市面上采集的17个金枪鱼罐头样品的线粒体基因组DNA利用通用引物59-3/59-5进行PCR扩增,扩增产物用2%的琼脂糖凝胶电泳检测并送往华大测序公司测序。
1.4.4数据处理
运用NCBI的BLAST程序,把所有样品的PCR产物测序结果与GeneBank数据库中已有金枪鱼线粒体cytb基因序列进行比较。
2结果与分析
2.1金枪鱼罐头DNA降解程度
由于金枪鱼罐头在加工过程中需经过高温高压处理,根据相关文献报道[10-11],DNA片段降解严重,为了考察金枪鱼罐头中DNA降解程度,同时保证获取足够数量和质量的DNA模板以适用于后续的分子生物学研究,使用表1中3对通用引物对提取的线粒体基因组DNA进行PCR扩增,扩增产物的琼脂糖凝胶电泳图见图1,汇总结果见表2。

M-100bp Mark; 1-阳性对照(黄鳍金枪鱼生鱼片);2-金枪鱼罐头(咖喱口味);3-块装金枪鱼(矿泉水浸罐头);4-油浸长鳍金枪鱼罐头;5-长鳍金枪鱼(水浸);6-金枪鱼罐头(原味);7-金枪鱼罐头(沙拉酱味);8-长鳍金枪鱼罐头(橄榄油浸渍);9-空白图1 3对通用引物(59-3/59-5、12S-F/12S-R、Cyt bL/ Cyt bH)PCR扩增结果Fig.1 PCR amplification results of three universal primers(59-3/59-5、12S-F/12S-R、Cyt bL/ Cyt bH)
提取的DNA质量用不同长度(123、224、358 bp)的目的片段来检测。从图1-A可以看出,所有样品均可扩增出123 bp目的片段,并且条带明亮清晰。从图1-B可以看出,3、4和8号罐头样品可以扩增出224 bp的目的片段,1、2、5、6和7号罐头样品不能扩增出224 bp目的片段。从图1-C可以看出,除阳性对照样品外,所有的金枪鱼罐头样品均不能扩增出358 bp的目的片段。

表2 3对通用引物PCR扩增结果汇总
注:“+”表示对应的PCR检测结果为阳性,“-”表示对应的PCR检测结果为阴性。
结果说明金枪鱼罐头中DNA降解严重,所获得片段大小在100~200 bp,与相关文献报道一致[10-11]。主要原因是:(1)高温加热引起DNA的降解;(2)辅料的添加对DNA造成一定程度的损伤,如DNA对酸碱环境十分敏感,7号罐头样品添加有沙拉酱,样品处于酸性环境中,使DNA降解程度更为严重。此外,罐头中某些添加剂可能会对PCR扩增造成一定的抑制作用。
2.2金枪鱼罐头样品123 bp目的片段PCR扩增及测序结果分析
以17个金枪鱼罐头样品的线粒体基因组DNA
为模板,利用通用引物59-3/59-5进行PCR扩增,目的片段长度为123 bp,扩增结果见图2。所有的样品均可扩增出清晰的条带。将上述PCR扩增产物进行测序,测序结果在NCBI上BLAST比对,比对结果见表3。不同种类的金枪鱼所占百分比见表4。

M:M-100bp Mark;1-阳性对照(黄鳍金枪鱼生鱼片);2-金枪鱼罐头A;3-金枪鱼罐头B;4-金枪鱼罐头C;5-橄榄油浸金枪鱼片;6-水浸金枪鱼块;7-水浸金枪鱼;8-葵花籽油浸金枪鱼;9-金枪鱼罐头(咖喱口味);10-金枪鱼罐头(水浸);11-金枪鱼罐头(植物油浸);12-块装金枪鱼(矿泉水浸罐头);13-橄榄油浸白肉金枪鱼罐头;14-金枪鱼(辣味油浸罐头);15-油浸长鳍金枪鱼罐头;16-长鳍金枪鱼(水浸);17-植物油浸金枪鱼;18-金枪鱼罐头(原味);19-空白图2 金枪鱼罐头样品PCR扩增结果Fig.2 PCR amplification results of canned tuna

编号商品名Cytb序列号相似度/%鉴定结果 标称原料2金枪鱼罐头AKF777811.1100Katsuwonuspelamis(鲣鱼)金枪鱼3金枪鱼罐头BKF777811.1100Katsuwonuspelamis(鲣鱼)金枪鱼4金枪鱼罐头CKF777811.1100Katsuwonuspelamis(鲣鱼)金枪鱼5橄榄油浸金枪鱼片KF777796.1100Thunnustonggol(青干金枪鱼)金枪鱼6水浸金枪鱼块JN007677.198Thunnusalalunga(长鳍金枪鱼)金枪鱼7水浸金枪鱼KF777811.198Katsuwonuspelamis(鲣鱼)金枪鱼8葵花籽油浸金枪鱼AB106835.1100Auxisthazard(扁鮀鲣)金枪鱼9金枪鱼罐头(咖喱口味)KM605252.1100Katsuwonuspelamis(鲣鱼)金枪鱼10金枪鱼罐头(水浸)JQ434036.198Katsuwonuspelamis(鲣鱼)金枪鱼11金枪鱼罐头(植物油浸)—100Euthynnusaffinis(巴鲣)或Euthynnuslineatus(黑鲔)金枪鱼12块装金枪鱼(矿泉水浸)KF777796.1100Katsuwonuspelamis(鲣鱼)金枪鱼13油浸白肉金枪鱼罐头KR023763.198Thunnusalalunga(长鳍金枪鱼)金枪鱼14金枪鱼(辣味油浸罐头)KF777811.1100Katsuwonuspelamis(鲣鱼)金枪鱼15油浸长鳍金枪鱼罐头KR023763.1100Thunnusalalunga(长鳍金枪鱼)长鳍金枪鱼16长鳍金枪鱼(水浸)KR023763.1100Thunnusalalunga(长鳍金枪鱼)长鳍金枪鱼17植物油浸金枪鱼KF777811.1100Katsuwonuspelamis(鲣鱼)金枪鱼18金枪鱼罐头(原味)KF777811.1100Katsuwonuspelamis(鲣鱼)金枪鱼
注:“—”:表示不能对应到具体的某一序列号。
本研究17个供试样品中,能够清晰的鉴定到原料物种水平的有16个样品,10号样品鉴定为巴鲣(E.affinis)或黑鲔(E.lineatus)。所有的样品中,其中2个样品能够使消费者清晰的了解该罐头的鱼源性成分,标示到具体的种类,其他15个样品均标示为金枪鱼,对于具体的鱼源性成分消费者并不清楚。
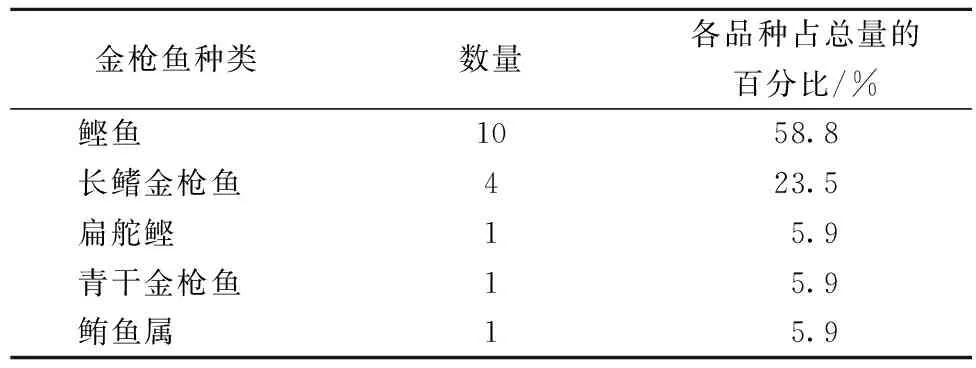
表4 通用引物FINS鉴定结果
食品法典委员会CAC(CODEX STAN 70—1981)标准规定,金枪鱼属、狐鲣属、鲣属和鲔属中的14种金枪鱼类均可加工成金枪鱼罐头[12],16个样品符合该规定,而我国国家标准GB/T24403—2009《金枪鱼罐头》中仅规定金枪鱼罐头原料为鲣鱼和鲔鱼类[13],11个样品符合该规定。8号样品鉴定为扁鮀鲣,不符合CAC标准规定和我国《金枪鱼罐头》国家标准规定。
根据鉴定结果,所采集金枪鱼罐头样品中有58.8%来源于价格低廉的鲣鱼。这说明市场上目前所销售的金枪鱼罐头加工原料大部分为价格低廉的鲣鱼,少数来自其他金枪鱼种类。基于11号样品仅仅能够鉴定到鲔鱼属,Genebank中关于黑鲔(E. lineatus)cytb基因序列相关信息较少,其中522 bp的黑鲔(E.lineatus)cytb基因序列与巴鲣(E.affinis)的线粒体cytb基因序列相比较,仅有3个碱基的差异,相似度高达99%,后续还需寻找其他区域cytb目标基因或线粒体其他区域(比如COI基因)的目标基因,将上述123 bp目标基因与其他目标基因结合起来进行金枪鱼种类的鉴定,在一定程度上弥补单一分子标记分析的不足。
3讨论
利用PCR测序法来进行物种鉴定的方法称为FINS法[14]。FINS方法对物种进行鉴别基于核苷酸序列变异,所以选取的目标基因要有足够的种间变异及较低的种内变异[15]。在近亲缘海洋动物种属鉴别中,线粒体DNA作为核外遗传物质,具有分子结构简单、严格的母系遗传、无重组、快速进化、多拷贝及稳定等优点[16],因此广泛的用于分析进化关系。在线粒体基因内,由于不同的区域进化速率存在差异,从而使得不同区域适于分析不同的分类单位。对于远缘物种间的分析可采用相对保守的基因序列进行比较,例如,rDNA基因相对比较保守,常用于种或科以上水平的研究[17]。对于一些亲缘关系较近的物种则需要依靠进化速率相对较快的区域进行分析,例如,cytb和D-loop基因进化较快,适合种群水平差异的检测[18],尤其是cytb的结构和功能较为清楚,现已被广泛地用于亲缘关系和系统进化的研究。
本研究结果表明,所采集的17个市售金枪鱼罐头中,58.8%来源于价格低廉的鲣鱼,其次还包括长鳍金枪鱼、青干金枪鱼、扁鮀鲣、鲔鱼。从分类学的角度讲,这几个品种都属于金枪鱼类,在营养价值和价格上存在较大的差异,但是它们亲缘关系比较近,外形和质地比较相似,特别是经过原料处理、蒸煮、杀菌等不同的加工工艺加工成罐头后,消费者很难判断其真实的鱼源性成分。此外,在某些种类的金枪鱼鱼体内,组氨酸含量比其他品种金枪鱼高很多,比如扁舵鲣极易腐败,肌肉中高含量的游离组氨酸在产组胺菌的作用下容易发生脱羧反应,生成大量组胺,为扁舵鲣的食用安全带来了隐患[19]。
本研究中采用的PCR-FINS方法能够准确、快速的实现金枪鱼罐头中鱼源性成分的鉴定,具有较强的实用性和应用价值,可为食品安监部门提供一定的借鉴和技术支持,对保护消费者合法权益、规范金枪鱼罐头市场、保障食品质量安全具有重要意义。该PCR-FINS技术的建立为我国《金枪鱼罐头》标准与国际相关标准接轨提供了技术依据,将进一步提升和规范我国的罐头工业市场。
参考文献
[1]邱凡. 西太平洋蓝鳍金枪鱼和黄鳍金枪鱼的遗传多样性及鲭科鱼类分子系统学研究[D]. 厦门:厦门大学, 2009:17-18.
[2]付才力, 李全宏, 黄尚荣,等. 世界主要鱼罐头市场概述[J]. 食品研究与开发, 2014,35(4):109-112.
[3]http://www.greenpeace.org/international/Global/international/publications/oceans/2010/azti_tinned_tuna_report_IA10GP1A%20FINAL
[4]MORETTI V M, TURCHINI G M, BELLAGAMBA F, et al. Traceability issues in fishery and aquaculture products[J]. Veterinary research communications, 2003, 27(1): 497-505.
[5]http://www.fda.gov/Food/GuidanceRegulation/Guidance Documents RegulatoryInformation/ucm113260.htm
[6]GB7718—2011.食品安全国家标准预包装食品标签通则[S].
[7]UNSELD M, BEYERMANN B, BRANDT P, et al. Identification of the species origin of highly processed meat products by mitochondrial DNA sequences[J]. Genome Research, 1995, 4(4): 241-243.
[8]张舒亚, 金丽琴, 蒋剑琼, 等. 食品中鱼源性成分 PCR 的检测方法[J]. 食品与发酵工业, 2010,36 (1): 142-145.
[9]REHBEIN H, KRESS G, SCHMIDT T. Application of PCR-SSCP to species identification of fishery products[J]. Journal of the Science of Food and Agriculture, 1997, 74(1): 35-41.
[10]RAM J L, RAM M L, BAIDOUN F F. Authentication of canned tuna and bonito by sequence and restriction site analysis of polymerase chain reaction products of mitochondrial DNA[J]. Journal of Agricultural and Food Chemistry, 1996, 44(8): 2 460-2 467.
[11]QUINTEIRO J, SOTELO C G, REHBEIN H, et al. Use of mtDNA direct polymerase chain reaction (PCR) sequencing and PCR-restriction fragment length polymorphism methodologies in species identification of canned tuna[J]. Journal of Agricultural and Food Chemistry, 1998, 46(4): 1 662-1 669.
[12]ALIMENTARIUS C. Codex Standard for Canned Tuna and Bonito[J]. Codex Stan, 1995: 1 970-1 981.
[13]GB/T 24403—2009. 金枪鱼罐头[S].
[14]姚琳, 江艳华, 李青娇,等. 基于DNA检测技术鉴定水产加工品原料的研究进展[J]. 中国渔业质量与标准, 2013, 3(1):33-39.
[15]黄文胜, 韩建勋, 董洁,等. FINS方法鉴定鱼翅和鲨鱼软骨的鲨鱼种类[J]. 食品科技, 2011,36(11):265-271.
[16]辛翠娜, 彭建军, 王莹,等.cytb分子标记技术在物种鉴定中的应用[J]. 野生动物学报, 2009, 30(4):217-221.
[17]MILINKOVITCH M C, ORTG, MEYER A, et al. Revised phylogeny of whales suggested by mitochondrial ribosomal DNA sequences[J]. Nature, 1993, 361(6 410):346-348.
[18]于旭蓉, 仇雪梅, 柳晓瑜,等. 线粒体DNA多态性在海洋动物群体遗传结构研究中的应用[J]. 生物技术通报, 2011, 10 (10):49-54.
[19]童铃. 不同贮藏温度下扁舵鲣组胺及产组胺菌的研究[D].杭州:浙江工商大学, 2015.
Development of a method for the identification of tuna species in canned tuna by FINS
AN Li-yan1,2,MENG Zhen1,2,QIU Kai1,2*,ZHONG Qi-ding1,2,YANG Tong-hui1,2, GUO Xin-guang1,2
1(China National Research Institute of Food and Fermentation Industries, Beijing 100015, China)2(National Standardization Center of Food and Fermentation, Beijing 100015, China)
ABSTRACTThis study aims to investigate the degree of DNA degradation and the species of tuna in the canned tuna products based on the analysis of fish mitochondrial genome including cytochrome b and 12S rDNA gene. A set of universal primers was used to amplify the targeted fragements with the length of 358 bp(cyt b),224 bp(12S rDNA),123 bp(cyt b)respectively. PCR-FINS technology was used to identify the tuna species in the canned tuna products by sequencing cytb gene of 123 bp, and 17 canned tuna products sourced from market were tested. Results showed that a very high degree of degradation in the canned tuna products for the DNA fragment sizes ranging from 100bp to 200bp. This study eastablished a accurate and reliable PCR-FINS technology with the ability to identify tuna species. Out of the 17 canned tuna samples, there were a variety of less valued species used; most popularly 58.8% of them were made from Katsuwonus, the remaining from Thunnus alalunga, Thunnus tonggol, Auxis thazard and Euthynnus.
Key wordsPCR-FINS;canned tuna;species identification
DOI:10.13995/j.cnki.11-1802/ts.201606028
收稿日期:2015-10-16,改回日期:2015-12-07
第一作者:硕士研究生(仇凯为通讯作者)。
