一株联苯基质筛选菌的生理及多氯联苯降解特性
2016-07-21杜丽婷白晓庆刘雪岚
胡 星,杜丽婷,王 斐,白晓庆,刘雪岚,杨 阳
(上海大学环境与化学工程学院,上海200444)
一株联苯基质筛选菌的生理及多氯联苯降解特性
胡星,杜丽婷,王斐,白晓庆,刘雪岚,杨阳
(上海大学环境与化学工程学院,上海200444)
摘要:以联苯为唯一的碳源和能源,从未被多氯联苯(polychlorinated biphenyls,PCBs)污染的土壤中筛选、分离出一株生长状况良好的菌株SYC01.分子生物学、生理鉴定结果表明:SYC01为革兰氏阴性、杆状、有鞭毛、能进行趋利避害运动的菌株,属于假单胞菌属;重金属对SYC01的生长多为不利影响,且影响排序为Cu2+?Cd3+/Cr3+/Pb2+>Fe3+;SYC01对PCB77和PCB52的去除以生物降解为主,降解率分别为34%和68%,是一株能降解高氯代PCBs、高抗毒、降解谱宽的高效降解菌,有原位修复PCBs污染土壤的应用潜力.
关键词:多氯联苯;生物降解;联苯;菌株SYC01
多氯联苯(polychlorinated biphenyls,PCBs)是一簇在联苯的双环上有1~10个取代氯原子的化合物的统称,理论上共有209个同类物和10种同分异构体.由于具有耐酸碱性、抗氧化性、无金属腐蚀性、耐热性、低可燃性、电绝缘性等优良的理化性质,PCBs曾作为一种理想的阻燃剂、添加剂、绝缘油、热载体等,被广泛应用于与人类生产生活紧密相关的塑料制品、油漆、复印纸、变压器、电容器、热交换器等商品之中[1].
然而,“油症”——1968年日本“米糠油事件”(20世纪世界八大公害事件之一)——首次将人们的关注点从PCBs的商业应用转向对人类健康安全的影响上[2].已有研究证明,PCBs是一种生物毒性物质,不仅会导致癌症发生[3],也会危害人体免疫、生殖、神经、内分泌等生命系统[2,4-5].因此,PCBs已作为首批危害严重的持久性污染物列入了《斯德哥尔摩公约》,在世界范围内停止生产.但经由生产事故、设备漏损,以及储存管理不当等原因进入人类生存环境中的PCBs[1],仍然威胁着人类健康和环境安全.例如,我国在“十·五”期间,对珠江三角洲、长江三角洲以及太湖流域等经济发达地区的土壤进行了全面评估,均发现了PCBs的累积现象,并检出了131种PCBs,其中15种PCBs的检出率为100%,最高累积浓度可超过200µg/L[6].这些能够沿食物链传递、在高级生物体内富集的PCBs,是人们面临的现实健康威胁.而对于这种大范围的面源型污染,目前仍然缺乏有效的治理手段.
作为自然界中的一员,微生物在环境物质循环中扮演着关键的角色.因此,以微生物为核心的降解技术是公认的成本低、无二次污染、环境友好的技术手段,且特别适合土壤及河底底泥中低浓度持久性有机污染物的原位修复[6].不过,现已获得的PCBs降解菌却存在着降解高氯代PCBs能力差、抗毒能力低下、降解谱窄等缺陷.因此,仍需发现新的菌株,以克服并解决上述缺陷.
本工作以联苯作为唯一的碳源和能源,从上海大学未被PCBs污染的绿化土壤中筛选、分离、纯化、挑选出一株生长状况良好的菌株,应用分子生物学和生理生化手段对其生命特性进行了研究,最后分析了菌株对PCBs的降解性能,以期实现该菌株的原位修复应用和实际推广.
1 材料与方法
1.1土壤样品
含有微生物的土壤样品取自上海大学宝山校区绿化区,其中取样点A生长的植物为三叶草,取样点B为长叶绿草,取样点C为小灌木.取样时先沿垂直方向将土壤剖开,然后获取适当的植物根系土壤.土壤样品的取样环境如图1所示.根据资料记载,此次采样环境历史上并未遭受过PCBs的污染.

图1土壤样品的取样环境Fig.1 Sampling environment of the soil samples
1.2主要试剂
PCB52和PCB77购自上海百灵威化学技术有限公司;13C-PCB141和13C-PCB208购自美国剑桥同位素实验室;联苯、正己烷、二氯甲烷、丙酮、无水硫酸钠、中性氧化铝(100~200目)购自上海国药集团化学试剂有限公司(其中正己烷、二氯甲烷、丙酮需经重蒸处理后使用);硅胶(80~100目)购自中国青岛海洋化工厂;纯水和去离子水由Milli-Q系统(Millipore公司)制取.
1.3培养基和缓冲溶液
液体合成培养基(liquid synthetic medium,SML)的配比为4.4 g/L K2HPO4+1.7 g/L KH2PO4+2.1 g/L NH4Cl+3.0 g/L NaCl+0.05 g/L酵母浸膏+10 mL基础盐溶液,其中基础盐溶液的配比为0.3 g/L CaCl·H2O+19.5 g/L MgSO4+5 g/L MnSO4·H2O+1 g/L FeSO4·H2O.为防止制备过程中产生沉淀,需待CaCl·H2O完全溶解后再加入其他物质.固体合成培养基(solid synthetic medium,SMS)和半固体合成培养基(semi solid synthetic medium,SMM)的配比是在SML的基础上分别增加15 g或5 g琼脂粉.
孟加拉红培养基(rose bengal medium,MM)的配比为10 g/L蛋白胨+1 g/L KH2PO4+ 0.5 g/L MgSO4+15 g/L琼脂粉+0.03 g/L孟加拉红+0.1 g/L氯霉素.LB液体培养基(LB liquid medium,LBL)的配比为10 g/L蛋白胨+5 g/L酵母浸膏+10 g/L NaCl.LB固体培养基(LB solid medium,LBS)的配比是在LBL的基础上增加15 g琼脂粉.
磷酸盐缓冲液(phosphate buffer saline,PBS)的配比为15.6 g/L K2HPO4+4.3 g/L KH2PO4,pH=7.2.
1.4菌株的筛选、分离、纯化及挑选
称取 0.5 g土壤样品放于 50 mL无菌水中,200 r/min震荡 30 min;吸取1 mL悬浊液于100 mL含0.1 mg/mL联苯固体的SML中(联苯为此培养基内唯一的碳源和能源),于28◦C,150 r/min培养7 d;之后,吸取1 mL菌悬液于新的100 mL含联苯的SML中,在相同条件下进行传代培养,并重复3次.
将最后一次培养的传代液梯度稀释,取10-5稀释倍数的溶液200µL涂布于LBS及MM平板上,于28◦C培养2 d以进行菌株的分离.挑取形态不同的单菌落至LBS平板上进行纯化.将3次纯化后的纯菌种保存在4◦C LBS斜面上备用.
分别将纯化后的菌株重悬于无菌水中,并涂布于含0.1 mg/mL联苯的SMS上,于28◦C培养2~3 d,从中挑选出利用联苯生长良好的菌株作为后续实验的菌种.
1.5菌株的生理分析
通过革兰氏染色研究菌株的染色反应及个体形态.在28◦C,50 r/min的条件下,每2 h吸取4 mL菌悬液测定其OD600吸光度,绘制菌株的生长曲线.
用SMM平板研究菌株的运动能力及重金属对其生长的影响.在平板中心分别加入200µL OD600=1的菌液:空白组只在右侧加入0.1 g联苯晶体,实验组则在联苯上再滴加20µL 0.05 mmol/L的重金属溶液(重金属溶液分别为CuSO4,K2Cr2O4,CdCl3,Pb(NO3)2和FeCl3).所有平板在28◦C下培养3 d.
1.6菌株的16S rRNA序列
提取菌株的基因组DNA,并进行PCR扩增反应.用于PCR扩增反应的正向引物为5’-AGAGTT TGATCCTG GCTCAG-3’(8~27 nt),反向引物为5’-AAGGAGGTGATCCAGCC-3’(1 541~1 557 nt).扩增体系总体积为20µL,包含2µL 10×buffer,2µL 25 mmol/L MgCl2,1.5µL 10 mmol/L dNTP,1µL Taq酶,1µL模板,30 pmol/L正向和反向引物各1µL,10.5µL ddH2O.扩增条件如下:首先95◦C,10 min;接着95◦C,1 min,55◦C,1 min,72◦C,1 min,共30个循环;最后72◦C,10 min,4◦C下保存.
选取约1 500 bp的条带,切胶、纯化、回收后送至上海生工生物工程公司测序.利用Blast软件将测得的序列与GenBank中已知的16S rDNA序列进行同源性比较,确定菌株的种属.利用BioEdite 3.0和MEGA 6.0软件,对相关菌株进行系统发育树构建.
1.7菌株的降解能力
在LBL中将菌株培养至2/3对数期,于4◦C下3 600 r/min离心5 min.弃上清,用pH=7.2的无菌PBS洗涤菌泥,再在上述条件下离心,重复2次.最后得到的菌泥再用无菌的PBS重悬,并调节OD600=1.
在10个玻璃瓶中均加入2 mg/L PCB的正己烷溶液.待正己烷挥发后,在4个活菌组中加入2 mL PBS菌悬液(OD600=1),在3个灭活组中则加入2 mL相同浓度的热杀菌液,在3个空白组中则加入2 mL无菌PBS溶液.PCB52和PCB77的最终浓度分别为0.1和0.5µg/mL.
PCBs的分析测定法参见文献[7].用内标法确定PCBs的浓度,每次测样前需绘制标准曲线.为保证数据可靠性,进行了回收率实验.结果表明,回收率为80%~110%,证明本方法可靠.
PCBs降解率η的计算方法如下:

式中,CC和CE分别为对照组和实验组液体中PCBs的浓度(µg/mL).
2 结果与讨论
2.1菌株的获得
2.1.1菌株的筛选
由于联苯是PCBs的基本骨架,因此以联苯作为唯一的碳源和能源来筛选能降解PCBs的菌株.
由图2可见,4次传代培养液均呈现出不同程度的混浊,因此从采样点A,B和C获得的土壤样品中,均能培养出以联苯为唯一基质生长的菌株.但是,传代培养液的颜色变化却显著不同,采样点B,C的规律不明显,而采样点A出现了“白色—浅棕色—暗棕色—深棕色”的明显变化规律.这说明随着传代培养的进行,采样点A中适应联苯环境的微生物大量存活,相应的代谢物累积于培养液中,使得其颜色逐步加深,同时也喻示着采样点A筛选出的菌株有别于其他两个采样点.

图2菌株筛选的传代培养Fig.2 Assage experiment of strain screening
2.1.2菌株的分离纯化及挑选
吸取采样点A最后一次传代培养的菌悬液,分别涂布于LBS和MM平板上,获得了多种不同菌落形态的菌株.
由图3可见,采样点A获得的传代菌液是不同种类菌株的混合物.在LBS平板上,存在浅黄、浅红和乳白色3种圆形菌落,即获得了至少3种类型的细菌;在MM平板上,存在玫红、浅红、浅红白心、浅红同心圆4种圆形菌落,即获得了至少4种类型的细菌.这些菌株或者是能够以联苯为唯一碳源和能源生长,或者是能够以其他菌株降解联苯后的代谢物生长的微生物.
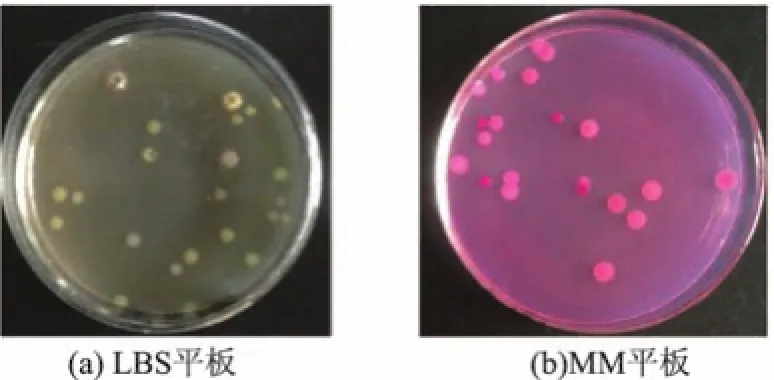
图3菌株的平板分离Fig.3 Plate experiment of strain isolating
当把纯菌种涂布于含联苯的SMS平板上后,发现了3株生长良好的菌株,表明这3株菌株拥有完整的联苯降解基因(bphABCDEFG)[1],能独立利用联苯作为唯一的生长基质.因此,选取其中一株生长迅速、菌落最大的细菌为进一步的研究对象,并命名为SYC01.
2.2菌株的分析
2.2.1菌株的生理
在LBS平板上,SYC01的菌落呈现扁平、未突起、边缘规则的圆形,整个菌落显得较为湿润,易被接种环挑起.革兰氏染色结果为红色,表明SYC01是一株革兰氏阴性菌.在显微镜下,SYC01的个体形态显示为杆状.
在LBL中,SYC01的适应期为6 h左右,6~30 h为对数生长期,在30 h后到达了稳定期.SYC01的2/3对数期的时间约为20 h,此时绝大多数菌株的生长最为旺盛、性能最为稳定,适用于降解实验.

图4 SYC01的运动及抗重金属影响的能力Fig.4 Moving ability and heavy metal resistance of SYC01
2.2.2菌株的运动及抗重金属影响
由图4可见,在空白组平板上,大范围的SMM培养基变得浑浊,且混浊程度较高,表明SYC01不仅能在以联苯为唯一基质的环境中生长良好,还能利用其鞭毛在半固体培养基中进行扩散.该运动能力有助于菌株在实际环境中趋利避害,成为生存竞争的“优胜者”.
与空白组相比,实验组中大多数重金属对SYC01的生长均有一定的负面影响.Cu2+严重抑制了SYC01的生长,在SMM中只观察到少许的浑浊;Cd3+,Cr3+和Pb2+对SYC01的生长也有抑制,但是抑制程度弱于前一组,在SMM中均有一定量的SYC01生长;Fe3+对SYC01的生长几乎没有影响,SMM中的浊度及扩散程度与空白组相似.因此,各重金属对SYC01生长的不利影响排序为Cu2+?Cd3+/Cr3+/Pb2+>Fe3+.这意味着当 SYC01应用于实际土壤中时,Cu2+的存在将严重削弱 SYC01的生长,并影响其降解PCBs的效果;但对于土壤中最常见的Cd3+,Cr3+和Pb2+,SYC01却具有较强的抗影响能力.
2.2.3菌株的16S rRNA序列
通过对SYC01基因组DNA的提取,以及PCR扩增反应,得到了SYC01的16S rDNA序列,Genbank登录号为KT266904.
在NCBI中利用Blast软件将SYC01的序列与GenBank中已知的16S rDNA序列进行了同源性比较.在查询覆盖率为100%的情况下,SYC01与十余种已登录的细菌的基因相似度为99%,且这些已登录的细菌均为假单胞菌属菌株.因此,可以推断SYC01是变形菌门、假单胞菌目、假单胞菌属中的细菌.
2.2.4菌株的系统发育树
以16S rDNA为基础,构建了SYC01与PCBs降解菌的发育树(见图5).可见,SYC01与ZIB-LLI的亲源最近.因此,SYC01很可能是一株恶臭假单胞菌,但这有待将来通过杂交分析进一步确定.然而,SYC01菌株与其他PCBs降解菌TMU56,CH07,KF707,LY402,M5和HC3在亲源上都有一些差别[6,8-10],尤其与W-02的差别巨大[11].这表明SYC01是一株有特殊进化途径的菌株.

图5通过16S rDNA构建的系统发育树Fig.5 Phylogenetic tree constructed by 16S rDNA
2.3菌株的PCBs降解性能
2.3.1菌株对PCBs的降解能力
通过对实验前后气相色谱(gas chromatography,GC)、回收率指示物13C-PCB141以及内标13C-PCB208响应值和相对响应值的分析计算,得到了各组PCBs的浓度(见图6).在实验过程中,3个空白组样品始终清澈透明,活菌组与灭活组在浊度上区别明显.
(1)菌株对PCB52的降解.
经计算发现,空白组中有19%损失率,说明在样品的挥发及GC预处理过程中造成了PCB52的损失.以空白组为对照,灭活组和活菌组的降解率分别为17%和68%,表明在SYC01的死细胞中,某些脂类和蛋白质具有降解或吸附PCB52的能力.而活菌组与灭活组的降解率差别巨大,表明SYC01活菌株确实具有对PCB52的降解能力.由此可知,SYC01在PCB52的降解过程中以生物降解为主.
唐伟[12]从被PCBs污染过的萧山和温岭土壤中获得了2株高效降解PCBs的菌株T29(Bacillus sp.)和W5(Corynebacterium sp.).它们对PCB52的降解率分别为38%和40%;而SYC01对PCB52的降解率为68%,表明SYC01也是高效的PCBs降解菌.
此外,在6~10氯代PCBs的厌氧降解中,间位氯的脱除最为容易,其次是对位,最后是邻位,因此容易形成邻位氯取代PCBs的大量富集[13].由于SYC01对邻位氯具有高效降解能力,表明其具有应用于PCBs厌氧还原/好氧氧化协同降解中复合菌团的潜力.

图6反应体系中PCB52和PCB77的浓度Fig.6 Concerntrations of PCB52 and PCB77 in the reaction systems
在1~6氯代PCBs的好氧降解中,大部分菌株仅局限于降解低于4氯取代的PCBs[14],且有两个氯原子在邻位(一个苯环上有两个邻位氯或两个苯环上各有一个邻位氯)的PCBs很难被降解[15].SYC01对PCB52的降解结果表明,该菌株具有降解高氯代和难降解PCBs的能力.
(2)菌株对PCB77的降解.
类似于PCB52的计算,本研究发现PCB77空白组中也有16%损失率,与PCB52差别不大,应是由相同原因造成的.以空白组为对照,灭活组及活菌组的降解率分别为9%和34%,表明在SYC01的死细胞中仍然存在与PCB77作用的物质,且在PCB77的降解过程中仍以生物降解为主.
De等[9]从印度东海岸海水中筛选出一株高效的PCBs降解菌CH07(Pseudomonas sp.).该菌株对PCB77的降解率为40%.在本研究中,SYC01对PCB77的降解率为34%,表明SYC01也是高效的PCBs降解菌.
由于PCB77是共平面结构分子,属于毒性最强的PCBs类型,而SYC01对其也有较强的降解能力,表明SYC01具有抗高毒性PCBs的能力.
2.3.2菌株降解PCBs的基因
目前,PCBs的好氧降解被认为是共代谢过程,因此联苯的降解基因和降解酶在菌株降解能力的表现中扮演了非常重要的角色.Furukawa等[16]从假单胞菌KF707中解读出了降解联苯和多氯联苯的基因簇bphA(A1A2A3A4),bphB,bphC和bphD.这些基因都线性地串联在一起,由同一操纵子ORF0进行调控.由这些基因编码的降解酶中,对菌株好氧生物降解性能影响最大的是降解途径中的第一个酶,即联苯双加氧酶(bphA),其α和β亚基(bphA1和bphA2)对底物的识别、结合和选择性都非常重要,是影响bphA降解性能的关键因素[1].
虽然SYC01和KF707同属假单胞菌属,具有很高的亲源性(见图5),但它们的降解能力相差悬殊,后者对PCB52的降解率只有9%,去除能力低下[17].Suenaga等[18]探究了这个现象,对KF707的bphA中335,376和377处的氨基酸进行了定点突变,将它们由异亮氨酸、苏氨酸和苯丙氨酸分别改变为亮氨酸、苯丙氨酸和天冬酰胺,从而提高了菌株降解PCB52的能力.该结果喻示了SYC01拥有特殊的基因,天然具备了高效降解PCB52的能力.
此外,尽管KF707的bphA是双对位氯取代PCB15的超级酶转化[19],但KF707却不能转化也有双对位氯取代的PCB77[17].对另一株假单胞菌TMU56(Pseudomonas aeruginosa)的研究表明,该菌株也不能降解PCB77,却能降解PCB52[8].由于SYC01既能降解PCB52,又能降解PCB77,可以表明由降解基因编码的bphA具有识别以及结合广泛的PCBs的能力,从而使得SYC01具有降解谱宽的特点.这对菌株未来的理论或应用研究均更具意义.
综上所述,SYC01是一株降解性能优良的菌株,对于将来实际应用于PCBs污染土壤原位修复颇具潜力.
3 结论
本研究从未经PCBs污染地区的土壤中,筛选出了一株有潜力应用于PCBs污染土壤原位修复的菌株SYC01,经研究得到如下结论.
(1)SYC01能以联苯为唯一的碳源和能源生长.
(2)SYC01属于假单胞菌属,为革兰氏阴性杆菌,有鞭毛,能运动.不同重金属对SYC01的影响程度不同,并且多为不利影响,影响排序为Cu2+?Cd3+/Cr3+/Pb2+>Fe3+.
(3)SYC01对于PCB52和PCB77的降解率分别为68%和34%,是一株能降解高氯代PCBs、高抗毒、降解谱宽的高效降解菌.
致谢感谢余应新教授、牛丽丽博士对本工作的大力支持!
参考文献:
[1]PIEpER D H.Aerobic degradation of polychlorinated biphenyls[J].Applied Microbiology and Biotechnology,2005,67:170-191.
[2]AOKI Y.Polychlorinated biphenyls,polychlorinated dibenzop-dioxins,and polychlorinated dibenzofurans as endocrine disrupters:what we have learned from Yusho disease[J].Environmental Research,2001,86:2-11.
[3]MAYEs B,MCCONNELL E,NEAL B,et al.Comparative carcinogenicity in Sprague-Dawley rats of the polychlorinated biphenyl mixtures Aroclors 1016,1242,1254,and 1260[J].Toxicology Sciences,1998,41:62-76.
[4]BORJA J,TALEON D M,AUREsENIA J,et al.Polychlorinated biphenyls and their biodegradation[J].Process Biochemistry,2005,40:1999-2013.
[5]FAROON O,JONEs D,DE ROsA C.Effects of polychlorinated biphenyls on the nervous system[J].Toxicology and Industrial Health,2001,16(7/8):305-333.
[6]贾凌云.多氯联苯降解菌的筛选及其降解性能研究[D].大连:大连理工大学,2008:135.
[7]李琛,余应新,张东平,等.上海室内外灰尘中多氯联苯及其人体暴露评估[J].中国环境科学,2010,30:433-441.
[8]AsHRAFOsADAT H Z,SEYED A S,EbRAHIM V F,et al.Extensive biodegradation of highly chlorinated biphenyl and Aroclor 1242 by Pseudomonas aeruginosa TMU56 isolated from contaminated soils[J].International Biodeterioration&Biodegradation,2009,63:788-794.
[9]DE J,RAMAIAH N,SARKAR A.Aerobic degradation of highly chlorinated polychlorobiphenyls by a marine bacterium,Pseudomonas CH07[J].World Journal of Microbiology and Biotechnology,2006,22:1321-1327.
[10]崔静岚.多氯联苯降解菌的筛选、降解特性研究及其应用[D].杭州:浙江大学,2013:67.
[11]任昱宗.多氯联苯降解菌的筛选、菌株性质研究及其活性酶性质分析[D].上海:东华大学,2007:83.
[12]唐伟.高效多氯联苯降解菌的筛选鉴定与降解性能研究[D].杭州:浙江大学,2013:48.
[13]MALTsEVA O V,TsOI T V,QUENsEN J F.Degradation of anaerobic reductive dechlorination produets of Aroclor 1242 by four anaerobic bacteria[J].Biodegradation,1999,10:363-371.
[14]王呈玉,孙玉成,曲迪,等.恶臭假单胞菌好氧降解高氯联苯的蛋白质组分析[J].环境科学学报,2012,32:2097-2103.
[15]ANYAsIL R O,ATAGANA H I.Biological remediation of polychlorinated biphenyls(PCB)in the environment by microorganisms and plants[J].African Journal of Biotechnology,2011,10:18916-18928.
[16]FURUKAwA K,MIYAZAKI T.Cloning of a gene cluster encoding biphenyl and chlorobiphenyl degradation in Pseudomonas pseudoalealigenes[J].Journal of Bacteriology,1986,166:392-398.
[17]GIbsON D T,CRUDEN D L,HADDOCK J D,et al.Oxidation of polychlorinated biphenyls by Pseudomonas sp.strain LB400 and Pseudomonas pseudoalcaligenes KF707[J].Journal of Bacteriology,1993,175:4561-4564.
[18]SUENAGA H,WATANAbE T,SATO M,et al.Alteration of regiospecificity in biphenyl dioxygenase by active-site engineering[J].Journal of Bacteriology,2002,184:3682-3688.
[19]ERICKsON B D,MONDELLO F J.Enhanced biodegradation of polychlorinated biphenyls after site-directed mutagenesis of a biphenyl dioxygenase gene[J].Applied and Environmental Microbiology,1993,59:3858-3862.
本文彩色版可登陆本刊网站查询:http://www.journal.shu.edu.cn
中图分类号:X 703
文献标志码:A
文章编号:1007-2861(2016)02-0188-09
DOI:10.3969/j.issn.1007-2861.2016.01.003
收稿日期:2016-01-16
基金项目:上海市大学生创新创业训练计划资助项目(CXSJ-15-122)
通信作者:胡星(1969—),女,副教授,博士,研究方向为环境污染物的微生物治理.E-mail:xhu@shu.edu.cn
Physiological and PCB-degrading properties of strain screening form biphenyl substance
HU Xing,DU Liting,WANG Fei,BAI Xiaoqing,LIU Xuelan,YANG Yang
(School of Environmental and Chemical Engineering,Shanghai University,Shanghai 200444,China)
Abstract:A strain,SYC01,was isolated from polychlorinated biphenyls(PCBs)-uncontaminated soil using biphenyl as sole carbon and energy source.Molecular and physiological assays indicated that SYC01 belonged to Pseudomonas genus,with gram-negative and rods appearance.It could move by its flagellumto avoid being harmed and seek being benefited.Heavy metals had negative impacts on the growth of SYC01 with the effect orders Cu2+?Cd3+/Cr3+/Pb2+>Fe3+.The biodegradation rate of PCB77 and PCB52 were 34%and 68%,respectively,suggesting that SYC01 was a high efficient degrader with high chlorinated PCBs degradation ability,high antitoxic capability and broad degradation spectrum.It can be used in in-situ bioremediation of PCBs-contaminated soils.
Key words:polychlorinated biphenyls(PCBs);biodegradation;biphenyl;strain SYC01
