梨遗传连锁图谱的构建与比较分析
2016-07-18薛华柏李秀根
王 磊,王 龙,薛华柏,李秀根,李 疆
(1新疆农业大学林学与园艺学院,乌鲁木齐 830052;2中国农业科学院郑州果树研究所,郑州 450009)
梨遗传连锁图谱的构建与比较分析
王磊1,2,王龙2,薛华柏2,李秀根2,李疆1
(1新疆农业大学林学与园艺学院,乌鲁木齐 830052;2中国农业科学院郑州果树研究所,郑州 450009)
摘要:【目的】利用已公开发表的梨和苹果的 SSR(Simple Sequence Repeat)引物以及从梨转录组开发的SSR引物构建本研究作图群体的遗传连锁图谱,为后期梨重要性状QTL定位和分子标记辅助选择等奠定基础。【方法】以西洋梨品种‘红茄’(Red Clapp Favorite)为母本,东方梨品种‘晚秀’(Mansoo)为父本,构建F1代作图群体。将所选用的SSR引物在亲本和4个子代个体进行PCR扩增,初步筛选出扩增结果符合JoinMap 4.0软件中“CP”作图模式要求的引物,随后在F1群体中检测,选用JoinMap 4.0软件对分离数据进行连锁分析,分别构建亲本的连锁图谱。以双亲图谱在各连锁群上的同源标记作为锚定位点,对双亲图谱进行整合。【结果】利用PCR技术对不同来源的共909对SSR引物(526对梨和283对苹果公开发表的SSR引物,从梨转录组开发的100对SSR引物)进行初步筛选后,发现来自苹果的SSR引物有效扩增片段的比例和多态性均较低,而来自梨和梨转录组开发的SSR引物相对较高。筛选出207对符合作图要求的SSR引物在群体中扩增,构建亲本的连锁图谱。母本图谱中的141个标记分布在17个连锁群上,总长度757.34 cM,标记间平均5.37 cM;父本图谱中的153个标记分布在19个连锁群上,总长度1 149.43 cM,标记间平均7.51 cM。【结论】对不同来源的SSR引物构建的双亲连锁图谱进行整合,最终得到一张由186个SSR标记,覆盖基因组长度1 125.33 cM的整合图谱。
关键词:梨;SSR;连锁图谱;整合图谱
联系方式:王磊,Tel:18538710151;E-mail:ewlei@163.com。通信作者李秀根,Tel:13653825516;E-mail:lixiugen@caas.cn。通信作者李疆,Tel:13899823969;E-mail:lijiangxj@163.com
0 引言
【研究意义】梨属于蔷薇科(Rosaceae)梨亚科(Pomoideae)梨属(Pyrus)植物。目前梨的栽培面积和产量在国内果树生产中均居第三位,在世界梨的栽培面积和产量中位居首位[1]。由于梨的童期较长,基因组杂合度较高,多数重要农艺性状是由多基因控制的数量性状,而常规育种方法效率低,需要大量的杂交群体,占用大面积的土地且耗费较多的人力和财力。分子标记辅助选择育种技术(molecular marker assisted selection,MAS)可在苗期对杂种群体进行提前筛选,选取具有优良性状的植株进行培育,这样能大量节省土地面积、人力和财力,从而提高育种效率,而高质量遗传图谱的构建又是分子标记辅助育种的基础。因此,构建梨遗传图谱具用重要意义。【前人研究进展】分子标记在梨的研究中起步相对较晚,但经过近些年来的努力,取得了很大进步。目前已有一些梨连锁图谱被构建,主要使用RAPD、AFLP、SSR和SNP等几种分子标记[1]。IKETANI等[2]利用RAPD标记,以‘Kinchaku’和‘幸水’杂交的82株F1群体为研究对象,分别构建了亲本的连锁图谱,‘Kinchaku’图谱的120个标记位点分布在18个连锁群,总长度为768 cM,‘幸水’图谱的78个标记位点分布在22个连锁群,总长度508 cM,这也是分子标记首次应用于梨的连锁图谱构建。YAMAMOTO等[3]从基因组文库开发SSR引物,参考蔷薇科不同属的SSR引物和AFLP标记结合,以欧洲的‘巴梨’为母本与日本的‘丰水’为父本杂交获得的63株F1为作图群体,在母本图谱中的226个标记位点(49个SSR标记)分布在18个连锁群,总长度949 cM;父本图谱的154个标记位点(42个SSR标记)分布在17个连锁群,总的遗传距离926 cM。YAMAMOTO等[4]对‘巴梨’和‘La France’图谱进行了加密整合,分别定位了447个和414个标记(SSR标记分别为118和134个),都分布在17个连锁群上,使其总长度达到了1 000 cM和1 156 cM,平均遗传距离2.3 cM和2.8 cM,使得连锁群的长度和平均遗传距离都得到了很大的提升。TERAKAMI等[5]用SSR和AFLP标记,以‘巴梨’与‘丰水’的63个F1代杂交群体来构建‘丰水’的连锁图谱,335个标记(105个SSR)分布在17个连锁群,总长度1 174 cM,平均3.5 cM。YAMAMOTO等[6]参考大量梨和苹果的公共SSR引物,用‘Akiakari’和‘Taihaku’杂交得到的93株F1为作图群体来构建连锁图谱,父本和母本两张图谱的SSR标记位点数分别为208个和275个,都分布在17个连锁群,总的遗传距离分别为799 cM和1 039 cM。TERAKAMI等[7]以‘巴梨’和‘丰水’杂交的63株F1代为作图群体,基于大规模的EST数据和基因组测序数据开发的SNP标记和SSR标记构建了‘丰水’的连锁图谱,951个标记位点(609个SNP,110个梨SSR,127个苹果SSR)分布在22个连锁群,遗传总长度1 341.9 cM,平均遗传距离 1.41 cM。中国在梨图谱构建研究起步相对较晚,但近年来也取得了不少的成绩。张丽等[8]以‘早美酥’和‘红香酥’杂交的129株F1代为作图群体,用AFLP分子标记构建了中国第一张梨的遗传连锁图谱,该图谱包括 20个连锁群,定位了 67个AFLP标记,遗传距离为785 cM,标记间平均间距为11.7 cM。孙文英等[9]用AFLP和SSR分子标记对‘鸭梨’和‘京白梨’杂交的145株F1群体构建了一张由368个AFLP标记和34个SSR标记分布在18个连锁群的连锁图谱,图谱总长度1 395.9 cM,平均图距为3.8 cM。韩明丽等[10]利用‘八月红’和‘砀山酥梨’杂交得到的91株F1为作图群体,构建了一个含有182个标记(61个苹果EST-SSR标记、68个苹果SSR标记、49个梨SSR标记)分属于19个连锁群的图谱,图谱总长度982 cM,各标记间平均5.4 cM。WU等[11]以‘八月红’和‘砀山酥梨’杂交的102个F1代为作图群体,利用‘砀山酥梨’的全基因组数据开发的SNP结合SSR标记,构建了一张包括3 241个标记(3 143 SNP,98 SSRs)分布于17个连锁群上,图谱总长2 243.4 cM,平均图距0.70 cM,这也是目前图谱长度最长,密度最高的梨的连锁图谱。CHEN等[12]以‘八月红’和‘砀山酥梨’杂交的56个F1代为作图群体,基于‘砀山酥梨’全基因组序列新开发的SSR引物及参考其他梨和苹果公共SSR引物来构建高密度连锁图谱,图谱共有734个SSR标记分布在17个连锁群上,覆盖基因组长度 1 661.4 cM,标记间平均间距 2.26 cM,这是迄今含有 SSR数量最多的梨分子遗传连锁图谱。【本研究切入点】SSR标记具有重复性好、多态性高、分布于整个基因组、共显性遗传等优点,在遗传连锁图谱的构建中发挥着重要的作用,也是近年来使用较多的分子标记之一。目前已构建的梨连锁图谱中大量使用SSR标记的相对较少,群体数量普遍偏小,这对梨的高质量图谱及公共图谱建立造成了一定影响。【拟解决的关键问题】将不同来源的SSR引物引入到本研究群体中,筛选出符合本群体作图要求的SSR引物,构建亲本标记类型全部由SSR引物组成的连锁图谱,为后期梨重要性状QTL定位和分子标记辅助选择等研究奠定基础。
1 材料与方法
本试验于2014年10月至2015年10月在中国农业科学院郑州果树研究所农业部果树育种技术重点实验室完成。
1.1 作图群体构建
以早熟红皮的西洋梨品种‘红茄’为母本,晚熟褐皮的东方梨品种‘晚秀’为父本杂交得到的165株F1代为作图群体(图1),该作图群体于2009年进行杂交和种子的收集,经沙藏处理,2010年播种,于2011年种植于中国农业科学院郑州果树研究所新乡基地,共计167株,长势较好。

图1 作图群体亲本果实Fig. 1 Parents fruit of mapping population
1.2 DNA提取
2015年4月上旬采集亲本与165株F1杂交群体的幼嫩叶片,利用改良的CTAB法提取植株总DNA[13],于1%琼脂糖凝胶电泳进行DNA质量检测,采用紫外分光光度计测定DNA浓度,稀释至50 ng·μL-1,储存于-20℃冰箱备用。
1.3 SSR引物选择与检测
本研究共选用909对SSR引物,其中来自苹果公开发表的283对[14-17],梨公开发表的526对[3,12,18-23],根据转录组开发的100对,均由英潍捷基(上海)贸易有限公司合成。
20 μL PCR反应体系:10×Buffer(Mg2+plus)2 μL,2.5 mmol·L-1dNTP 1.6 μL,10 mmol·L-1引物各0.6 μL,Taq DNA聚合酶为0.5 U,模板DNA 50 ng。PCR扩增在Biometra TProfessional Thermocycler上进行。PCR反应程序:94℃ 5 min;94℃ 30 S,Tm±2℃ 30 S,72℃ 45 S,30个循环;72℃ 10 min;4℃保存。SSR-PCR扩增产物采用 8%非变性聚丙烯酰胺凝胶(PAGE)电泳分离,硝酸银染色检测并拍照统计。
1.4 数据分析及图谱构建
基于HEMMAT等[24]和WEEDEN等[25]提出的“双假测交”理论,将 F1群体的各个标记分离类型按照JoinMap4.0软件中的“CP”作图模式(lm×ll、nn×np、hk×hk、ef×eg、ab×cd等5种分离类型)进行统计,无或扩增不清晰的标记记为“-”。将所有符合作图要求的标记数据导入到JoinMap4.0软件中,建立双亲图谱数据集,然后分别对双亲图谱数据集进行卡方检验,暂时先去除偏分离标记(P<0.05),选取 LOD=4.0 —10.0,最大重组率r=0.4,以Kosambi作图函数构建遗传连锁图谱,偏分离的标记逐一插入到图谱中,不改变其他标记的位置关系,否则去除,用MapChart 2.3软件绘制最终的连锁图谱。
1.5 双亲图谱的比较与整合
根据亲本各连锁群上的同源标记,利用MapChart2.3软件绘制双亲的比较图谱,对双亲图谱进行比较分析。以双亲图谱在各连锁群上的同源标记作为锚定位点,分别选择双亲图谱中同源标记所在的连锁群,通过JoinMap 4.0软件中“Combine Groups for Map Integration”功能,对双亲图谱含有相同标记的连锁群进行整合,最终构建一张整合图谱。
2 结果
2.1 SSR引物筛选
以双亲和随机4个F1单株对所选用的909对SSR引物(表 1)进行筛选,其中来自梨公开发表的 526 对 SSR引物中,469对(占此种来源 SSR引物的89.16%,下同)有扩增产物,378对(71.86%)引物在亲本间具有多态性;来自苹果公开发表的283对SSR引物中,166对(58.66%)有扩增产物,116对(40.99%)引物在亲本间具有多态性;来自转录组开发的100对SSR引物中,88对(88%)有扩增产物,59对(59%)引物在亲本间具有多态性。最终筛选出207对符合本研究群体作图要求的SSR引物,其中梨公共SSR引物133对(25.29%),苹果公共SSR引物43对(15.19%),转录组开发的SSR引物31对(31.00%)。从以上结果可以看出,来自梨的SSR引物有效扩增片段的比例均较高,从对亲本的多态性方面来看,来自梨公开发表的SSR引物相比从转录组开发的SSR引物多态性高;从来自苹果的SSR引物扩增结果可以看出,来源于苹果的SSR引物在梨的研究中有一定的通用性。
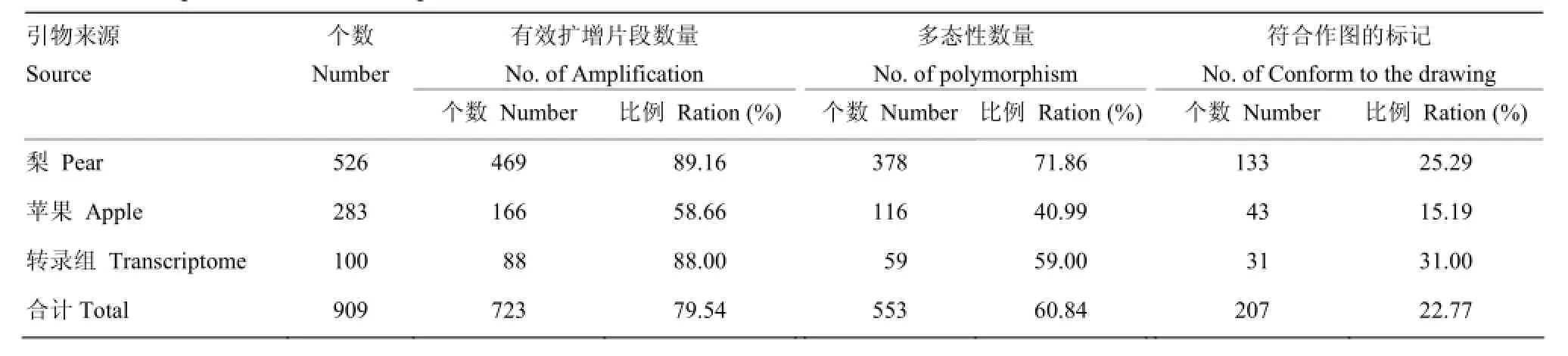
表1 引物扩增结果Table 1 Amplification results of primer
2.2 遗传图谱的构建
从909对SSR引物中筛选得到207对符合作图要求的SSR引物,在165株F1群体中进行验证(图2),用JoinMap4.0软件对群体数据进行连锁分析,构建双亲的连锁图谱:‘红茄’图谱的141个标记位点分布在17个连锁群,总长度757.34 cM,标记间平均5.37 cM,连锁群长度在18.80—63.29 cM,每个连锁群上有4—16个标记;‘晚秀’图谱的153个标记位点分布在19个连锁群,总长度1 149.43 cM,标记间平均7.51 cM,连锁群长度在5.67—119.41 cM,每个连锁群上有3—17个标记。对双亲连锁图谱参照CHEN等[12]所构建的梨连锁图谱的连锁群顺序进行调整,将母本和父本的各连锁群分别命名为R1—R17和M1—M17(图3)。
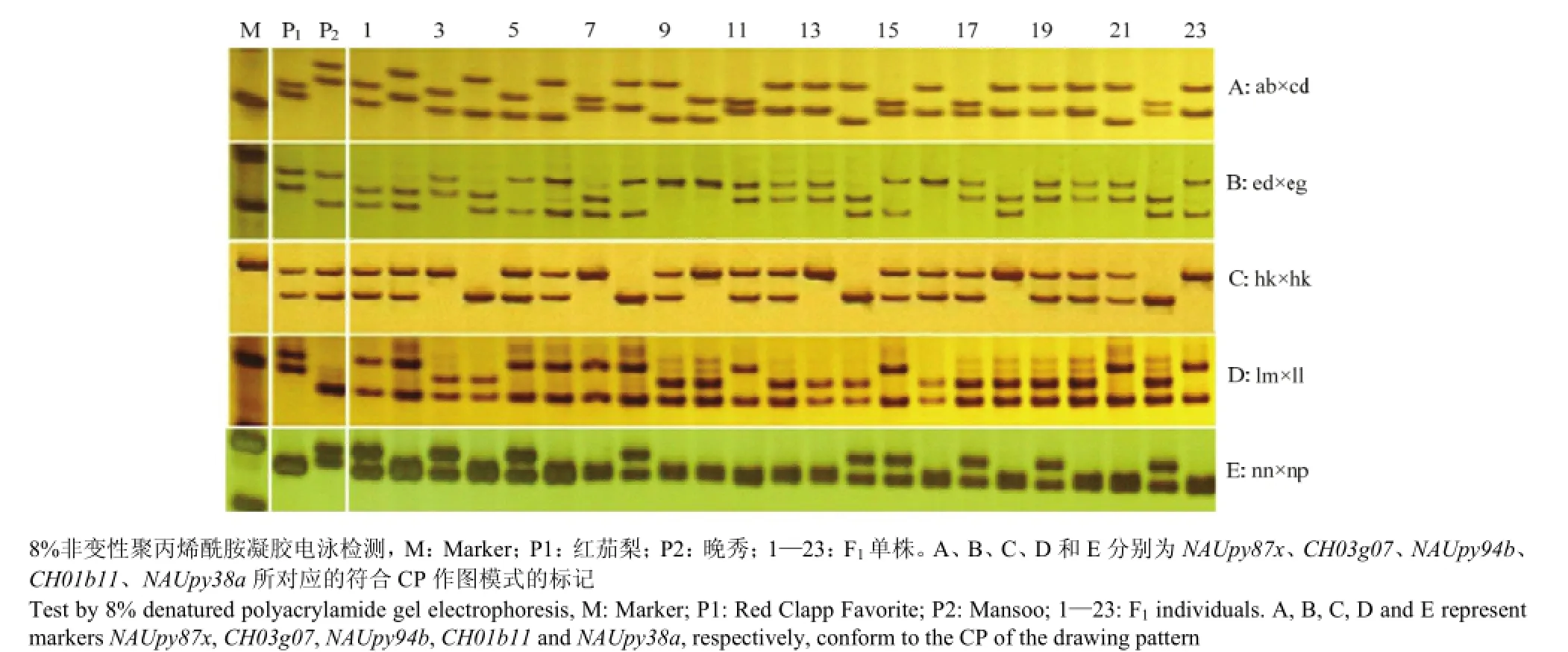
图2 不同类型SSR标记在F1部分群体扩增结果Fig. 2 Amplification results of different types of SSR marker in part F1group
研究中还发现有部分标记发生了偏分离(P<0.05),在母本图谱的141个标记中有4个标记(2.8%)发生了偏分离,父本图谱的153个标记中有10个发生了偏分离(6.5%)。从这些偏分离的标记分布位置来看,大多数分布较为集中,少量分散在不同的连锁群。


图3 双亲遗传连锁图谱及同源位点比较Fig. 3 Parental genetic maps and distribution of homologous loci
2.3 双亲图谱的比较分析
根据双亲图谱上98个同源标记位点,建立各连锁群间的同源关系(表2),父本的19个连锁群和母本的17个连锁群对应,父本的M4-1和M4-2和母本的R4对应,父本的M17-1和M17-2和母本的R17对应。在双亲图谱中,所有的连锁群都有对应的同源标记,位于第14连锁群的同源标记位点最多(13个),位于第11连锁群的同源标记位点最少(2个),平均每个连锁群上有5.8个同源标记。通过对两亲本连锁图谱的比较发现(表2、图3),父本图谱的标记数量比母本稍多,但其图谱长度约是母本图谱长度的1.5倍。从各连锁群的长度来看,父本图谱的大多数连锁群长度均比母本连锁群长,在第10、11、15连锁群父本的连锁群长度约是母本连锁群长度的2—3倍。通过同源标记的排列顺序来看,第7、8、10、13和14连锁群中有少量的同源标记有交叉现象,其他大部分的同源标记在连锁群上的排列顺序一致。

表2 图谱同源连锁群比较Table 2 An alignment of homologous linkage group of parental genetic maps
图4 整合图谱Fig. 4 Integrated of genetic map
2.4 图谱整合
利用同源标记作为锚定位点,对亲本的图谱数据进行整合,最终整合的图谱有17个连锁群(图4),与梨染色体数目一致(2n=34),共 186个标记,其中基于转录组新开发的30个SSR标记(表3)。来自母本图谱特有的标记41个,来自父本图谱特有的标记51个,来自双亲图谱共有的标记92个,整合图谱特有的标记2个。图谱覆盖基因组长度1 125.33 cM,相邻标记间最大间距34.78 cM,最小的在0.1 cM以内,标记间平均距离6.05 cM,连锁群长度在28.20—105.45 cM。

表3 新开发的SSR标记Table 3 Newly developed SSR loci
3 讨论
3.1 杂交亲本的选择
梨在全世界分布约 30多个种,大多数自交不亲和,种间亲缘关系较为复杂,梨的起源演化认为梨主要分为东方梨和西洋梨两大系统。近年来,曹玉芬等[26]和路娟等[27]通过 SSR分子标记技术对分别属于白梨、砂梨、秋子梨、新疆梨、西洋梨等41份梨品种和150份梨品种进行遗传多样性研究分析,表明西洋梨和东方梨的遗传距离较远。这与江先莆等[28]通过形态特征研究的结果一致。
从构建连锁图谱的角度来看,杂交亲本的选择至关重要,将直接影响到所构建图谱的难易程度和适用范围[29]。在考虑杂交后代可育的前提条件下,为了得到更多的多态性标记位点,一般选择亲缘关系远、表型差异较大的材料做杂交亲本。西洋梨与东方梨之间的亲缘关系较远,DNA水平差异较大,多态性丰富,更适合用于构建作图群体的亲本[30]。目前已构建的梨作图群体大都集中在西洋梨之间或者东方梨之间,而西洋梨和东方梨之间杂交的作图群体研究目前较少,主要有日本研究者所使用的‘巴梨’ב丰水’杂交群体[3-5,7],中国所使用的‘八月红’ב砀山酥梨’杂交群体[10-12]。‘八月红’是西洋梨‘早巴梨’和‘早酥’的后代,‘早酥’是东方梨‘苹果梨’和‘身不知’的后代,路娟等[27]通过对梨品种遗传多样性的研究认为‘早酥’属于东方梨,‘八月红’和‘身不知’都属于西洋梨。
本研究所选用的亲本组合是西洋梨‘红茄’×东方梨‘晚秀’,‘红茄’来源于美国,是茄梨的芽变品种,果型较小(平均单果重150 g),紫红色果皮,较早熟(七月上旬),肉质细腻,有香气,风味浓郁,后熟变软,具有典型西洋梨特性;父本‘晚秀’是韩国梨品种,果型较大(平均单果重500 g),褐皮,较晚成熟(十月中旬),具有典型的东方梨特性。这两个品种无论从地理分布还是果实的形态特征有着较大的差异,是一对较适合作图的亲本材料。同时以西洋梨与东方梨作为杂交亲本,对后期研究西洋梨和东方梨的起源和演化等方面也有着重要的意义。
3.2 不同来源SSR引物扩增差异
从前人研究的结果来看,来源于苹果的SSR引物在梨研究中也有着较好的应用。YAMAMOTO等[3]首次将来源于蔷薇科苹果、桃和樱桃的SSR标记引入梨的研究,发现苹果SSR引物在梨上的可转移性明显高于桃和樱桃。此后很多研究者都在梨的研究中引用苹果SSR引物[4,6,10,12]。本研究中,来源于苹果的SSR引物在亲本间有扩增产物与表现多态性的比例分别为58.66%和40.99%,这与陈慧等[31]获得的71.6%和40%的结果基本一致;刘金义等[32]通过对大量苹果SSR引物的筛选,发现约60%的引物在梨上都有扩增片段。苹果和梨都属于蔷薇科,亲缘关系较近,关玲等[33]将从苹果基因组开发的 SSR引物应用在梨的研究中,其结果也证明了SSR标记在近缘物种中具有良好的转移性。
近年来随着测序技术的发展及测序成本的降低,大量通过EST序列和转录组数据开发的SSR引物被应用到植物的遗传研究中。王西成等[34]利用NCBI收录的梨EST数据开发的48对SSR引物对4个梨品种进行扩增,64.6%具有扩增产物,56.3%具有多态性;崔海荣等[35]利用GenBank和GDR(genome database for rosaceae)中梨EST数据开发的25对SSR引物对杂交群体亲本进行遗传分析,其中56%有扩增片段,36%具有多态性;笔者课题组通过梨转录组开发的SSR引物,在亲本间有扩增产物与多态性的比例分别为88%和59%。可见,不同研究者所获得的结果差异较大,SSR引物的扩增产物比例差异可能来源于引物本身,无扩增产物的SSR引物可能是被内含子分割,导致其在基因组模板上没有结合位点;而多态性引物比例差异可能主要与各研究者所采用的试验材料遗传背景差异有关,不同来源的试验材料遗传背景不同,一般遗传背景越远,差异越大,多态性越高[36]。当然,以上差异还可能与研究者对数据处理分析的差异等有关。
在本研究的结果中,来自梨和苹果公开发表的SSR引物中符合作图要求的引物占多态性引物数量的1/3,来源于转录组的 SSR引物中符合作图要求的占其多态性引物数量的1/2,这比大多数研究者最终定位到图谱中的SSR标记占多态性引物数量70—80%[12,31]的比例偏低,这可能与双亲间纯合位点和零位点(null allele)的SSR引物占多态性引物比例较高有关。在引物的筛选时,为避免人为原因造成的无扩增片段而引起统计误差,本研究去除了在双亲中都含有零位点的标记[37];同时,部分引物在双亲间多态性较好,但都是纯合位点,其后代群体基因型完全相同,无法用于构图。此外,部分SSR引物的非特异性扩增片段较多,干扰带的存在增加了对相应标记基因型数据判读的准确性,为避免带型统计错误,对这部分SSR引物也予以舍弃,这也是符合作图标记数量偏少的原因之一。
3.3 标记的偏分离情况
偏分离在遗传连锁作图中是普遍存在的,苹果[38]、桃[39]、葡萄[40]、樱桃[41]等多种植物都存在一定比例的偏分离,一般偏分离通过影响标记间的重组距离而影响到连锁群上标记的排列顺序[42-43],也有研究者认为它可以促进生物进化[44]。
在本研究构建的双亲连锁图谱中,当P<0.05时,偏分离的标记在母本和父本图谱所占的比例分别为2.8%和6.5%,与目前已报道的研究中偏分离比率10% —30%[4,7,9]相比较低。从本研究的情况来看,造成偏分离比率较低可能与作图群体数量较大有关。理论上适当增大作图的群体数量,可以降低偏分离位点出现的概率,从而减少试验的误差。由于梨的作图群体构建难度较大,目前已构建的图谱中大多作图群体都小于150株,而适当的提高作图群体数量可以提高作图的精度,研究表明150—200株的作图群体更为适合[1]。偏分离的形成原因比较复杂,也可能与所选标记在染色体的位置或者作图亲本有关。
3.4 整合图谱的比较
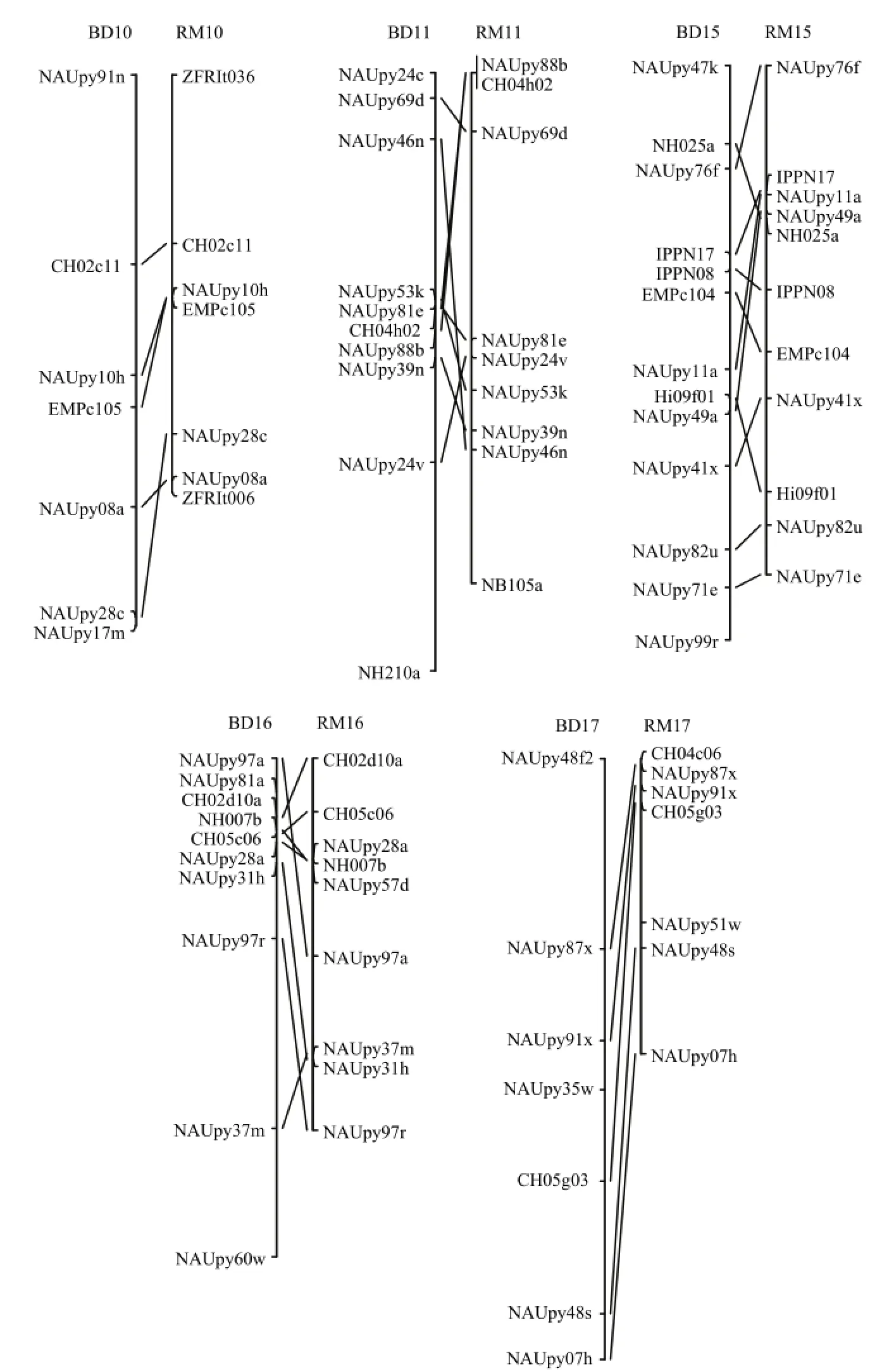
BD1—BD 17为CHEN等2014年构建的17个连锁群,RM1—RM17为本研究构建的双亲图谱的整合,其上的标记均为公开发表的SSR标记BD1 to BD17 are 17 linkage groups of Chen's genetic linkage map published in 2014. RM1—RM17 are 17 linkage groups of constructing integrated genetic linkage map in this research. All SSR markers on linkage groups have been published图5 双亲整合图谱与梨已发表图谱比较[12]Fig. 5 Comparison of integrated genetic map of pear and the map published by Chen et al.[12]
通过与CHEN等[12]构建的梨高密度SSR图谱进行比较(图5),两张连锁图谱有126个相同标记,其中121个SSR标记定位到相同的连锁群,部分标记在连锁群上排列顺序有所差异,存在交叉现象。这可能与标记偏分离现象有一定关系,LORIEUX等[42-43]通过对偏分离数据的模型分析证明了偏分离标记不仅影响标记间的重组距离,也影响到连锁群上标记的排列排列顺序。在本研究作图过程中,也发现有个别的偏分离标记(NAUpy59t和 ZFRIt051)能够改变其他标记的排列顺序。不同作图群体亲本遗传背景的不同也是导致标记顺序差异的重要原因之一,路娟等[27]通过25对SSR引物在150份梨品种进行遗传分析,发现来自东亚的种和品种与西方梨的相似系数较低,各自独立成组,而东亚的大部分品种之间相似系数较高,相互交错在一起。本研究所使用的母本材料是西洋梨‘红茄梨’,父本是东方梨‘晚秀’,双亲间遗传背景差异较大。而在CHEN等[12]构建的图谱所使用的母本是‘八月红’(‘早巴梨’和‘早酥梨’的后代)兼具西洋梨和东方梨的血统,父本是‘砀山酥梨’,双亲间的差异相对比本研究的偏小。此外,不同研究者所使用的作图软件和参数的设置等差异也可能导致标记在连锁群上的排列顺序发生变化。
大量来自梨或苹果的 SSR引物在研究中互相使用,为两者遗传图谱比较以及比较基因组学等研究奠定了基础。LU等[45]和PIERANTONI等[46]通过比较梨和苹果的遗传连锁图谱,均发现共有标记在两者的连锁群中所处的位置基本相同,呈现出较好的共线性。本研究最终整合的图谱中有38对SSR引物来自苹果,分布在除RM1和RM13外的其余15条连锁群上,每个连锁群1—7个标记位点,通过与苹果等图谱[15,47-49]比较,大部分标记位点均与上述的苹果连锁图谱一致,两个及以上同源标记所在连锁群的遗传距离差异较小,这也表明了梨和苹果作为2个不同的物种,基因组间的相似度较高。
通过图谱的比较还发现,部分标记所在的连锁群与前人研究结果不一致。NAUpy58f、NAUpy57d、NAUpy51w、NAUpy81a和NAUpy35w等5个SSR位点在CHEN等[12]构建的梨遗传图谱中分别位于BD2、BD3、BD14、BD16和BD17连锁群,而在本研究中这5个标记分别位于RM7、RM16、RM17、RM13和RM6连锁群。在KUNIHISA等[49]构建苹果遗传图谱的研究中,Hi03a03位点都被定位到双亲‘Orin’和‘Akane’遗传图谱的LG6连锁群,但本研究中其被定位在RM14连锁群。这可能是由于基因型统计误差造成的,也可能是生物在进化过程中染色体部分区域发生了交换或者易位等原因而导致[46]。
4 结论
利用不同来源的SSR引物分别构建了梨双亲的连锁图谱,标记数量分别为141和153个,图谱总长度分别为757.34 cM和1 149.43 cM,标记间的平均距离分别是5.37 cM和7.51 cM;将双亲的图谱进行整合,最终得到的整合图谱共有186个SSR标记分布在17个连锁群上,覆盖基因组长度1 125.33 cM,标记间平均距离6.05 cM。
References
[1] 殷豪, 吴俊, 张绍铃. 梨分子遗传连锁图构建及重要农艺性状定位.果树学报, 2012, 29(5): 918-928. YIN H, WU J, ZHANG S L. Construction of molecular genetic maps and location of important agronomic traits in pear. Journal of Fruit Science, 2012, 29(5): 918-928. (in Chinese)
[2] IKETANI H, ABE K, YAMAMOTO T, KOTOBUKI K, SATO Y, SAITO T, TERAI O, MATSUTA N, HAYASHI T. Mapping of Disease-Related Genes in Japanese pear using a molecular linkage map with RAPD markers. Breeding Science, 2001, 51(3): 179-184.
[3] YAMAMOTO T, KIMURA T, SHODA M, IMAI T, SAITO T, SAWAMURA Y, KOTOBUKI K, HAYASHI T, MATSUTA N. Genetic linkage maps constructed by using an interspecific cross between Japanese and European pears. Theoretical and Applied Genetics, 2002, 106(1): 9-18.
[4] YAMAMOTO T, KIMURA T, TERAKAMI S, NISHITANI C, SAWAMURA Y, SAITO T, KOTOBUKI K, HAYASHI T. Integrated reference genetic linkage maps of pear based on SSR and AFLP markers. Breeding Science, 2007, 57(4): 321-329.
[5] TERAKAMI S, KIMURA T, NISHITANI C, SAWAMURA Y, SAITO T, HIRABAYASHI T, YAMAMOTO T. Genetic linkage map of the Japanese pear 'Housui' identifying three homozygous genomic regions. Journal of the Japanese Society for Horticultural Science, 2009, 78(4):417-424.
[6] YAMAMOTO T, TERAKAMI S, TAKADA N, NISHIO S, ONOUE N, NISHITANI C, KUNIHISA M, INOUE E, IWATA H, HAYASHI T, ITAI A, SAITO T. Identification of QTLs controlling harvest time and fruit skin color in Japanese pear (Pyrus pyrifolia Nakai). Breeding Science, 2014, 64(4): 351-361.
[7] TERAKAMI S, NISHITANI C, KUNIHISA M, SHIRASAWA K, SATO S, TABATA S, KURITA K, KANAMORI H, KATAYOSE Y, TAKADA N, SAITO T, YAMAMOTO T. Transcriptome-based single nucleotide polymorphism markers for genome mapping in Japanese pear (Pyrus pyrifolia Nakai). Tree genetics & Genomes, 2014, 10(4):853-863.
[8] 张丽. 早美酥×红香酥 F1代群体分子遗传图谱的构建[D]. 保定:河北农业大学, 2006. ZHANG L. Construction of a molecular genetic map in Zaomeisu pear and Hongxiangsu pear F1hybrid population [D]. Baoding: Agricultural University of Hebei, 2006. (in Chinese)
[9] 孙文英, 张玉星, 张新忠, 乐文全, 张海娥. 梨分子遗传图谱构建及生长性状的QTL分析. 植物遗传资源学报, 2009, 10(2): 182-189. SUN W Y, ZHANG Y X, ZHANG X Z, LE W Q, ZHANG H E. Construction of a genetic linkage map and QTL analysis for some growth traits in pear. Journal of Plant Genetic Resources, 2009, 10(2):182-189. (in Chinese)
[10] 韩明丽, 刘永立, 郑小艳, 杨健, 王龙, 王苏珂, 李秀根, 滕元文.梨遗传连锁图谱的构建及部分果实性状 QTL的定位. 果树学报,2010, 27(4): 496-503. HAN M L, LIU Y L, ZHENG X Y, YANG J, WANG L, WANG S K, LI X G, TENG Y W. Construction of a genetic linkage map and QTL analysis for some fruit traits in pear. Journal of Fruit Science, 2010, 27(4): 496-503. (in Chinese)
[11] WU J, LI L T, LI M, KHAN M A, LI X G, CHEN H, YIN H, ZHANG S L. High-density genetic linkage map construction and identification of fruit-related QTLs in pear using SNP and SSR markers. Journal of Experimental Botany, 2014, 65(20): 5771-5781.
[12] CHEN H, SONG Y, LI L T, KHAN M A, LI X G, SCHUYLER S, KORBAN S S, WU J, ZHANG S L. Construction of a high-density simple sequence repeat consensus genetic map for pear (Pyrus spp.). Plant Molecular Biology Reporter, 2014, 33(2): 1-10.
[13] 陈昆松, 李方, 徐昌杰, 张上隆, 傅承新. 改良 CTAB法用于多年生植物组织基因组DNA的大量提取. 遗传, 2004, 26(4): 529-531. CHEN K S, LI F, XU C J, ZHANG S L, FU C X. An efficient macro-method of genomic DNA isolation from actinidia chinensis leaves. Hereditas, 2004, 26(4): 529-531. (in Chinese)
[14] GASIC K, HAN Y, KERTBUNDIT S, SHULAEV V, IEZZONI A, STOVER E, BELL R, WISNIEWSKI M, KORBAN S. Characteristics and transferability of new apple EST-derived SSRs to other rosaceae species. Molecular Breeding, 2009, 23(3): 397-411.
[15] SILFVERBERG-DILWORTH E, MATASCI C L, VAN DE WEG W E, VAN KAAUWEN M P W, WALSER M, KODDE L P, SOGLIO V, GIANFRANCESCHI L, DUREL C E, COSTA F, YAMAMOTO T, KOLLER B, GESSLER C, PATOCCHI A. Microsatellite markers spanning the apple (Malus×domestica Borkh.) genome. Tree Genetics & Genomes, 2006, 2(4): 202-224.
[16] GUILFORD P, PRAKASH S, ZHU J M, RIKKERINK E, GARDINER S, BASSETT H, FORSTER R. Microsatellites in Malus ×domestica (apple): Abundance, polymorphism and cultivar identification. Theoretical and Applied Genetics, 1997, 94(2): 249-254.
[17] LIEBHARDT R, GIANFRANCESCHI L, KOLLER B, RYDER C D, TARCHINI R, VAN DE WEG W E, GESSLER C. Development and characterisation of 140 new microsatellites in apple (Malus× domestica Borkh.). Molecular Breeding, 2002, 10(4): 217-241.
[18] FERNÁNDEZ-FERNÁNDEZ F, HARVEY N G, JAMES C M. Isolation and characterization of polymorphic microsatellite markers from European pear (Pyrus communis L.). Molecular Ecology Notes, 2006, 6(4): 1039-1041.
[19] INOUE E, MATSUKI Y, ANZAI H, EVANS K. Isolation and characterization of microsatellite markers in Japanese pear (Pyrus pyrifolia Nakai). Molecular Ecology Notes, 2007, 7(3): 445-447.
[20] YUE X Y, LIU G Q, ZONG Y, TENG Y W, CAI D Y. Development of genic SSR markers from transcriptome sequencing of pear buds. Zhejiang University Science B, 2014, 15(4): 303-312.
[21] YAMAMOTO T, KIMURA T, SHODA M, BAN Y, HAYASHI T, MATSUTA N. Development of microsatellite markers in the Japanese pear (Pyrus pyrifolia Nakai). Molecular Ecology Notes, 2002, 2(1):14-16.
[22] YAMAMOTO T, KIMURA T, SAWAMURA Y, MANABE T, KOTOBUKI K, HAYASHI T, BAN Y, MATSUTA N. Simple sequence repeats for genetic analysis in pear. Euphytica, 2002, 124(1):129-137.
[23] SAWAMURA Y, SAITO T, TAKADA N, YAMAMOTO T, KIMURA T, HAYASHI T, KOTOBUKI K. Identification of parentage of Japanese pear Housui. Journal of the Japanese Society for Horticultural Science, 2004, 73: 511-518.
[24] HEMMAT M, WEEDON N F, MANGANARIS A G, LAWSON D M. Molecular marker linkage map for apple. Journal of Heredity, 1994, 85(1): 4-11.
[25] WEEDEN N F, HEMMAT M, LAWSON D M, LODHI M, BELL R L, MANGANARIS A G, REISCH B I, BROWN S K, YE G N. Development and application of molecular marker linkage maps in woody fruit crops. Euphytica, 1994, 77(1/2): 71-75.
[26] 曹玉芬, 刘凤之, 高源, 姜立杰, 王昆, 马智勇, 张开春. 梨栽培品种SSR鉴定及遗传多样性. 园艺学报, 2007, 34(2): 305-310. CAO Y F, LIU F Z, GAO Y, JIANG L J, WANG K, MA Z Y, ZHANG K C. SSR analysis of genetic diversity of pear cultivars. Acta Horticulturae Sinica, 2007, 34(2): 305-310. (in Chinese)
[27] 路娟, 吴俊, 张绍铃, 吴华清, 张妤艳. 不同系统梨种质遗传多样性与分类关系的SSR分析. 南京农业大学学报, 2011, 34(2): 38-46. LU J, WU J, ZHANG S L, WU Q H, ZHANG Y Y. Genetic diversity and polygentic relationship among pears reveled by SSR analysis. Journal of Nanjing Agricultural University, 2011, 34(2): 38-46. (in Chinese)
[28] 江先莆, 初庆刚, 张长胜. 中国梨属植物的分类和演化. 莱阳农学院学报, 1992, 9(1): 18-21.JIANG X P, CHU Q G, ZHANG C S. Studies on the classification and evolution of the genus pyrus in china. Journal of Laiyang Agricultural College, 1992, 9(1): 18-21. (in Chinese)
[29] 蔡长福. 牡丹高密度遗传图谱构建及重要性状QTL分析[D]. 北京:北京林业大学, 2015. CAI C F. High-density genetic linkage map construction and QTLs analyses for phenotypic traits in tree peony [D]. Beijing: Beijing Forestry University, 2015. (in Chinese)
[30] 万怡震, 王跃进, 张今今, 孙马, 杨克强, 徐炎. 多年生果树植物分子遗传作图. 园艺学报, 2002. 29(S1): 629-634. WAN Y Z, WANG Y J, ZHANG J J, SUN M, YANG K Q , XU Y. Molecular genetic mapping in fruit crops. Acta Horticulturae Sinica, 2002, 29(S1): 629-634. (in Chinese)
[31] 陈慧, 宋跃, 李雷霆, 吴俊, 张绍铃. 梨遗传连锁图谱的构建及其与苹果图谱的比较. 西北植物学报, 2012, 32(7): 1343-1348. CHEN H, SONG Y, LI L T, WU J, ZHANG S L. Construction of a genetic linkage map in pear and compared the map with apples. Acta Botanica Boreali-Occidentalia Sinica, 2012, 32(7): 1343-1348. (in Chinese)
[32] 刘金义, 崔海荣, 王龙, 王新卫, 杨健, 章镇, 李秀根, 乔玉山. 梨果实酸/低酸性状的 SSR 分析. 果树学报, 2011, 28(3): 389-393. LIU J Y, CUI H R, WANG L, WANG X W, YANG J, ZHANG Z, LI X G, QIAO Y S. Analysis of pear fruit acid/low-acid trait by SSR marker. Journal of Fruit Science, 2011, 28(3): 389-393. (in Chinese)
[33] 关玲, 章镇, 王新卫, 薛华柏, 刘艳红, 王三红, 乔玉山. 苹果基因组SSR位点分析与应用. 中国农业科学, 2011, 44(21): 4415-4428. GUAN L, ZHANG Z, WANG X W, XUE H B, LIU Y H, WANG S H, QIAO Y S. Evaluation and application of the SSR loci in apple genome. Scientia Agricultura Sinica, 2011, 44(21): 4415-4428. (in Chinese)
[34] 王西成, 姜淑苓, 上官凌飞, 曹玉芬, 乔玉山, 章镇, 房经贵. 梨 EST-SSR标记的开发及其在梨品种遗传多样性分析中的应用评价.中国农业科学, 2010, 43(24): 5079-5087. WANG X C, JIANG S L, SHANGGUAN L F, CAO Y F, QIAO Y S, ZHANG Z, FANG J G. Development of EST-derived SSR markers for pear and evaluation of their application in pear genetic diversity analysis. Scientia Agricultura Sinica, 2010, 43(24): 5079-5087. (in Chinese)
[35] 崔海荣, 刘金义, 佟兆国, 王新卫, 章镇, 乔玉山. 砂梨 EST-SSR引物开发及其应用. 西北植物学报, 2010(8): 1551-1556. CUI H R, LIU J Y, TONG Z G, WANG X W, ZHANG Z, QIAO Y S. Development and application of EST-SSRs in sand pear. Acta Botanica Boreali-Occidentalia Sinica, 2010(8): 1551-1556. (in Chinese)
[36] STAUB J E, SERQUEN F C, GUPTA M. Genetic markers, map construction, and their application in plant breeding. HortScience, 1996, 31(5): 729-741.
[37] GAUDET M. Molecular approach to dissect adaptive traits in native European Populus nigra L.: Construction of a genetic linkage map based on AFLP, SSR and SNP markers [D]. Viterbo: Università degli Studi della Tuscia, 2006.
[38] FERNANDEZ-FERNANDEZ F, ANTANAVICIUTE L, VAN DYK M M, TOBUTT K R, EVANS K M, REES D J G, DUNWELL J M, SARGENT D J. A genetic linkage map of an apple rootstock progeny anchored to the Malus genome sequence. Tree genetics & genomes, 2012, 8(5): 991-1002.
[39] 高妍. 桃(Prunus persica (L.) Batsch) AFLP分子标记遗传连锁图谱的构建[D]. 陕西杨凌: 西北农林科技大学, 2008. GAO Y. construction of AFLP molecular markers linkage map in peach (Prunus persica (L.) Batsch) [D]. Yangling, Shaanxi: Northwest A&F University, 2008. (in Chinese)
[40] 刘镇东. 山葡萄高密度分子遗传图谱构建及抗寒性QTL定位研究[D]. 沈阳: 沈阳农业大学, 2012. LIU Z D. High density molecular genetic map construction in Virus amurensis Rupr. and QTL location for grape cold hardiness [D]. Shenyang: Shenyang Agricultural University, 2012. (in Chinese)
[41] 王晶, 闫国华, 张晓明, 周宇, 张开春. 甜樱桃高密度连锁图谱的构建. 果树学报, 2014, 31(s1): 29-35. WANG J, YAN G H, ZHANG X M, ZHOU Y, ZHANG K C. Construction of sweet cherry (Prunus avium) high density genetic linkage map. Journal of Fruit Science, 2014, 31(s1): 29-35. (in Chinese)
[42] LORIEUX M, GOFFINET B, PERRIER X, DELEON D G, LANAUD C. Maximum-likelihood models for mapping genetic markers showing segregation distortion. 1. Backcross populations. Theoretical and Applied Genetics, 1995, 90(1): 73-80.
[43] LORIEUX M, PERRIER X, GOFFINET B, LANAUD C, DELEON D G. Maximum-likelihood models for mapping genetic markers showing segregation distortion. 2. F2populations. Theoretical and Applied Genetics, 1995, 90(1): 81-89.
[44] CHARLESWORTH B. Driving genes and chromosomes. Nature, 1988, 332(6163): 394-395.
[45] LU M, TANG H, CHEN X, GAO J, CHEN Q, LIN L. Comparative genome mapping between apple and pear by apple mapped SSR markers. American-Eurasian Journal of Agricultural and Environmental Science, 2010, 9: 303-309.
[46] PIERANTONI L, CHO K H, SHIN I S, CHIODINI R, TARTARINI S,DONDINI L, KANG S J, SANSAVINI S. Characterisation and transferability of apple SSRs to two European pear F1populations. Theoretical and Applied Genetics, 2004, 109(7): 1519-1524.
[47] VELASCO R, ZHARKIKH A, AFFOURTIT J, DHINGRA A, CESTARO A, KALYANARAMAN A, FONTANA P, BHATNAGAR S K, TROGGIO M, PRUSS D, SALVI S, PINDO M, BALDI P, CASTELLETTI S, CAVAIUOLO M, COPPOLA G, COSTA F, COVA V, DAL RI A, GOREMYKIN V, KOMJANC M, LONGHI S, MAGNAGO P, MALACARNE G, MALNOY M, MICHELETTI D, MORETTO M, PERAZZOLLI M, SI-AMMOUR A, VEZZULLI S, ZINI E, ELDREDGE G, FITZGERALD L M, GUTIN N, LANCHBURY J, MACALMA T, MITCHELL J T, REID J, WARDELL B, KODIRA C, CHEN Z, DESANY B, NIAZI F, PALMER M, KOEPKE T, JIWAN D, SCHAEFFER S, KRISHNAN V, WU C, CHU V T, KING S T, VICK J, TAO Q, MRAZ A, STORMO A, STORMO K, BOGDEN R, EDERLE D, STELLA A, R N, GLEAVE A P, LAVEZZO E, FAWCETT J A, PROOST S, ROUZE P, STERCK L, TOPPO S, LAZZARI B, HELLENS R P, DUREL C E, GUTIN A, BUMGARNER R E, GARDINER S E, SKOLNICK M, EGHOLM M, VAN DE PEER Y, SALAMINI F, VIOLA R. The genome of the domesticated apple (Malus×domestica Borkh.). Nature Genetics, 2010, 42(10): 833-839.
[48] ANTANAVICIUTE L, FERNÁNDEZ-FERNÁNDEZ F, JANSEN J, BANCHI E, EVANS K M, VIOLA R, VELASCO R, DUNWELL J M, TROGGIO M, SARGENT D J. Development of a dense SNP-based linkage map of an apple rootstock progeny using the Malus Infinium whole genome genotyping array. BMC Genomics, 2012, 13(1): 203.
[49] KUNIHISA M, MORIYA S, ABE K, OKADA K, HAJI T, HAYASHI T, KIM H, NISHITANI C, TERAKAMI S, YAMAMOTO T. Identification of QTLs for fruit quality traits in Japanese apples: QTLs for early ripening are tightly related to preharvest fruit drop. Breeding Science, 2014, 64(3): 240-251. VECCHIETTI A, KATER M M, MASIERO S, LASSERRE P, LESPINASSE Y, ALLAN A C, BUS V, CHAGNE D, CROWHURST
(责任编辑 赵伶俐)
Construction of SSR Genetic Linkage Map and Comparison on Pears
WANG Lei1,2, WANG Long2, XUE Hua-bai2, LI Xiu-gen2, LI Jiang1
(1College of Horticulture & Forestry Sciences, Xinjiang Agricultural University, Urumqi 830052;2Zhengzhou Fruit Research Institute, Chinese Academy of Agricultural Sciences, Zhengzhou 450009)
Abstract:【Objective】The genetic linkage map was constructed based on the Simple Sequence Repeat primers of pears and apples that have been published and developed from the transcriptome of pears by our team, which will be the foundation for the important trait mapping of QTL and marker-assisted selection in the future.【Method】A F1mapping population was built on a cross between a western pear (Red Clapp Favorite) as the female parent, and an oriental pear (Mansoo) as the male parent. The SSR primers which matched the "CP" mode in the JoinMap 4.0 software in parents and four individuals by PCR application were selected and then tested in the F1group. The separation of data was analyzed by JoinMap 4.0 software, and the parent genetic map was constructed respectively. The genetic map of the parents was integrated based on anchor points which were homology marks in the parents' map on each linkages.【Result】909 pairs of SSR primers from different sources (526 pairs from pears and 283 pairs from apples that have been published, and 100 pairs were developed from the pear transcriptome by our team) was preliminarily screened through a PCR technique. The results showed that amplified fragments and polymorphism of SSR primers from apples were lower,but that the SSR primers from pears and developed through the transcriptome of pears were higher. The parents' genetic linkage mapwas constructed based on the 207 pairs of SSR primers which conformed to the drawing. A female genetic map was constructed by 141 markers which was distributed on 17 linkage groups and spanned 757.34 cM in genome with an average distance of 5.37 cM between markers; A male genetic map was constructed by 153 markers which was distributed on 19 linkage groups and spanned 1 149.43 cM in genome with an average distance of 7.51 cM between markers.【Conclusion】The integrated genetic linkage maps of parents were constructed by different sources of SSR molecular markers, which contained 186 SSR markers and finally covered the genome 1 125.33 cM.
Key words:pear; SSR; genetic linkage map; integrated the genetic map
收稿日期:2015-12-28;接受日期:2016-03-25
基金项目:国家“十二五”科技支撑计划(2013BAD02B01)、中国农业科学院创新工程(CAAS-ASTIP)
