作物分子身份证构建软件ID analysis的编制
2016-07-18胡振帮高运来齐照明蒋洪蔚刘春燕辛大伟胡国华潘校成陈庆山
胡振帮,高运来,齐照明,蒋洪蔚,刘春燕,辛大伟,胡国华,潘校成,陈庆山
(1东北农业大学农学院,哈尔滨150030;2黑龙江省农垦科研育种中心,哈尔滨150090;365301部队副业基地,黑龙江五大连池164100)
作物分子身份证构建软件ID analysis的编制
胡振帮1,高运来1,齐照明1,蒋洪蔚2,刘春燕2,辛大伟1,胡国华2,潘校成3,陈庆山1
(1东北农业大学农学院,哈尔滨150030;2黑龙江省农垦科研育种中心,哈尔滨150090;365301部队副业基地,黑龙江五大连池164100)
摘要:【目的】以作物品种或资源的SSR分子标记为基础数据,应用Visual Basic6.0开发作物分子身份证构建配套软件ID analysis,快速准确地筛选引物组合并高效鉴定品种。【方法】作物分子身份证理论由陈庆山提出,其原理是利用SSR标记在材料中的多态性特点,将多个标记进行排列组合,快速有效地区分品种资源的算法——逐步扩增法。通过对40份黑龙江省大豆品种的40对引物数据进行分子身份证构建,阐述该软件的详细操作过程。【结果】利用 Visual Basic6.0编程软件设计人机交互界面,通过编程实现核心算法,开发 ID analysis软件,软件集成全库构建、部分构建、ID判定、数据合并等功能。其中,全库构建是软件的核心功能,通过全库构建可以快速分析区分材料的最少标记;部分构建可以选择部分目标材料进行区分;ID判定可以在分子身份证数据库构建完成的前提下,对未知材料进行SSR分析,根据获得的分子身份证数据来确定材料是哪个品种或近似品种;数据合并功能可以将多次试验数据整合成一个数据集。利用软件全库构建功能对范例数据进行分析得到以下结果:①在40对引物对40个大豆品种的分子身份证构建中,共有13个引物由于缺失过多,不符合标准被剔除,剔除引物为:Sat_111、Sat_218、Satt231、Satt685、Satt514、Satt551、Satt077、Satt358、Satt424、Satt100、Satt838、Satt893和Satt891。②共有6个引物由于与其他引物相似系数过高,不符合标准被剔除,剔除引物为:Satt253、Satt192、Satt417、Sat_229、Satt127和Satt496。③在分析的40个品种中,共有5个品种具有7个特异等位基因,分别为引物Satt516在材料东农36中显示特异条带3;引物Satt253在材料东农36中显示特异条带1;引物Sat_229在材料嫩丰17中显示特异条带1;引物Satt192在材料东农42中显示特异条带3;引物Satt206在材料北丰19中显示特异条带1;引物Satt244在材料北丰19中显示特异条带4;引物Satt363在材料黑河14中显示特异条带1,因此,可以通过这些特异等位基因直接确定需要鉴定的品种。④仅需7对引物便可将40份大豆品种完全区分开,引物组合为:Satt398、Satt380、Satt453、Satt288、Satt244、Sat_092和Satt206。【结论】编制了用于作物分子身份证构建的软件ID analysis,软件界面友好、使用简便、高效、灵活。实现单个软件完成作物分子身份证的构建,达到了对资源品种鉴定的目的。随着技术的发展和毛细管电泳的应用,开发全自动分子身份证分析系统甚至开发基于分子身份证理论的快速资源鉴定仪将成为可能。
关键词:大豆;作物;SSR标记;分子身份证;软件开发
联系方式:胡振帮,Tel:0451-5519194;E-mail:zbhu@neau.edu.cn。通信作者陈庆山,E-mail:qshchen@sohu.com。通信作者潘校成,E-mail:soybean2007@126.com
0 引言
【研究意义】黑龙江省是中国优质大豆主产区,1986—2010年共审定大豆品种275份[1],2011—2015年审定大豆品种113份[2-9]。随着科研能力的提高,为满足人们生产、生活需求,越来越多的大豆新品种被育成,但市场监管能力不足,许多不良的品种充斥市场,给农民生产及品种权益保护带来严峻的考验,分子身份证软件的研发对相关研究开展、资源鉴定及品种权益保护具有重大意义。【前人研究进展】指纹图谱(fingerprinting)的形式多种多样,但主要分为化学指纹图谱和生物指纹图谱2种类型,其中,近红外透射光谱分析[10]、深度颜色特征[11]、高光谱图像处理[12]都属于化学指纹图谱,DNA条形码[13-15]、ISSR、SNP指纹图谱[16]都属于分子生物指纹图谱。分子指纹图谱具有数量多、分布广、多态性高、不受环境影响等优点[17]。在众多技术中SSR微卫星DNA又叫做简单重复序列,SSR标记属于共显性标记,并且具有良好的遗传多样性[18-19]。目前,整合的大豆SSR标记共有1 015对[20-22],丰富的SSR标记数量使基于SSR标记应用更加广泛[23-25]。在分子身份证研究方面,由陈庆山提出,高运来等[26]构建了大豆品种的分子身份证。近几年,张靖国等[27]以 20个梨栽培品种为例,利用17对SSR标记构建梨分子身份证体系。陆徐忠等[28]利用12对SSR标记对127份水稻品种构建了40位的身份证条码编号。徐雷锋等[29]以 96份百合种质资源为材料,使用20对SSR标记进行百合种质分子身份证构建试验。【本研究切入点】分子身份证构建方法分析样本材料数量有限,而使用的标记却很多,生成的字符串超长,记录繁琐,往往需要联合GeneMapper、MinimalMarket、Data Collection、Genemapper和条码生成器等多个软件[27-29],迄今为止,还未发现能够直接构建分子身份证的软件。利用SSR技术区分品种已成为主流,但SSR结果分析仍然停留在人工选择上,一直未找到合适的软件进行综合判读。【拟解决的关键问题】本研究拟开发一套适用于分析大量材料、具有较高效率的软件。实现单个软件完成分子身份证的构建。软件的研发有助于相关研究开展,特别是可以解决资源及品种权益保护中出现的身份鉴定问题。
1 材料与方法
1.1 数据获取
参试材料于 2008年播种于黑龙江省农垦科研育种中心基地,选择40对引物对40个大豆品种进行分析,参考陈庆山等[30-31]方法进行大豆种粒 DNA的提取、PCR扩增及电泳试验,并获得标记数据。标记试验在黑龙江省农垦科研育种中心实验室完成。
1.2 名词及符号定义
其中,行对应的是n个材料,列对应的是m个标记,其中,a11,a12…anm表示使用m个标记对n个材料的电泳条带码。
定义S1、S2…Sn,其中S1表示第1个材料,n为材料容量。
定义V1、V2…Vm,其中V1表示第1个标记,m为标记个数。
标记多态度:在分子标记中,单标记或标记组合的全部类型,叫作标记多态型,标记多态型的个数,叫作标记多态度,用d表示。
1.3 逐步扩增法
作物分子身份证理论及逐步扩增法的算法由组陈庆山[32]提出并应用于计算分子身份证,若标记的数量足够多,并且标记对材料的区分度好,这种方法就有可能找到分子身份证的可行格式。若有20个标记,每个标记有2个等位基因,则这些标记有220= 1 048 476>106种组合,即这些标记可区分百万个以上的不同材料。因此,对于一般的1 000以内的材料来说,一般20个标记足够了。
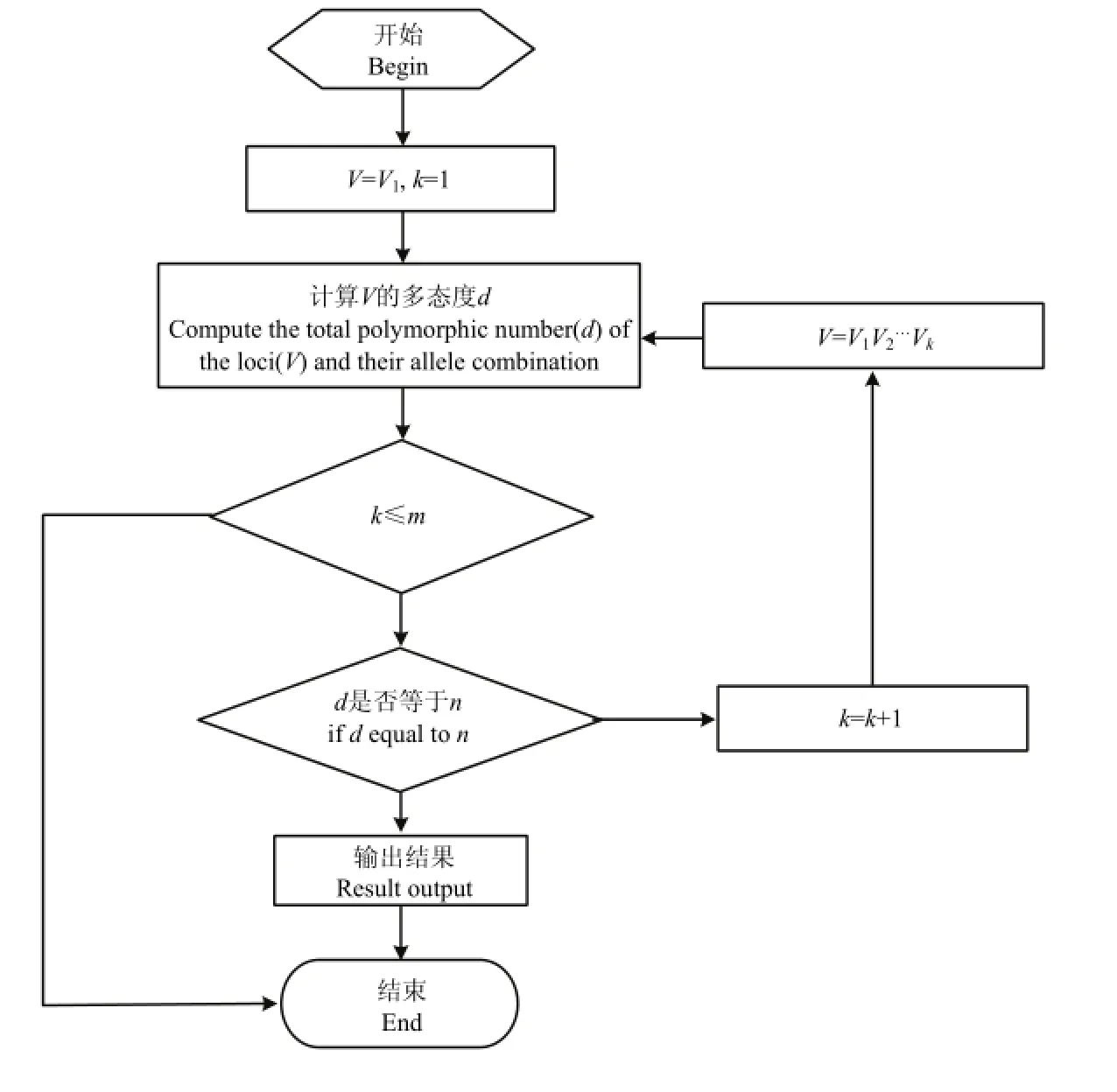
图1 逐步扩增法流程图Fig. 1 Program flow chart of step amplification method
逐步扩增法的具体过程(图 1):首先标记按照等位基因多少进行排序,计算相邻标记区分度相关系数,淘汰相关系数过高的引物。然后选择 V1,计算V1的多态度dv1看是否与材料容量n相等,若相等,表明V1就可以区分出所有的材料,V1即为分子身份证的一个可行格式;若不等,则说明V1不能区分出所有材料,算法继续引入V2构成V1V2标记组合,计算该标记组合的多态度dv1v2,看是否与材料容量n相等;若相等,表明V1V2可以区分出所有的材料,V1V2即为分子身份证的一个可行格式;若不等,算法继续引入新标记,直到将材料全部区分开为止,假设此时的标记组合为V1V2V3…Vm,则它即为该材料分子身份证的一个可行格式。
在实际的执行过程中,为了降低运算量,每引入一个标记,都要计算各材料对应标记组合的等位基因频率,若频率为 1,则对该材料从计算数组中剔除,加快了计算速度。
1.4 分子身份证构建的实现策略
作物分子身份证是针对作物种质资源或品种品系,基于作物分子标记的多态性检测手段,利用最简引物组合实现作物种质资源最大区分,并以类似于身份证的等位基因编码,作为标识和图形化的理论和技术。基于分子身份证的概念和构建算法,结合实际应用的需要,建立分子身份证的实现策略。策略共分 4个部分:全库构建、部分构建、选择分析和分子身份证判定(ID判定)。
1.4.1 全库构建 全库构建是分子身份证构建的基础,是基于数据库中的全部材料和标记信息,应用1.2、1.3算法,对全部材料进行分子身份证构建的策略。全库构建的基本步骤如下:
步骤 1:不符合标记的剔除。剔除标准首先标记的缺失太多(默认不超过 5%)其次是标记间相似系数太高(默认不高于0.8);
步骤 2:有效标记数量判别。若标记充足则转入步骤3,否则转入步骤4;
步骤 3:执行算法,计算出材料(品种)分子身份证,包括标出特异性条带;
步骤 4:以标记集能区分的材料数最多为依据,计算出材料的分子身份证。
1.4.2 部分构建 在全库构建的基础上,可选择性地对部分材料进行特异性引物条带的筛选和分子身份证的构建。部分构建以全库构建的材料、标记集为构建背景,选择部分材料以全部引物为标记利用算法进行分子身份证计算。部分构建的基本步骤如下:
步骤1:从全部材料中选择部分材料集;
步骤 2:不符合标记的剔除。剔除标准首先标记的缺失太多(默认不超过 5%)其次是标记间相似系数太高(默认不高于0.8);
步骤3:执行算法,计算出部分材料分子身份证,包括标出特异性条带;
步骤 4:以标记集能区分的材料数最多为依据,计算出部分材料的分子身份证。
1.4.3 选择分析 在全库构建的基础上,可选择部分标记对材料进行判别,主要用来考察部分标记(受关注的)在分子多态水平上区分材料的能力。由于选定了部分标记集,故算法上只需将供试材料的分子身份证编码标出即可。选择分析的结果可能会锁定唯一分子身份证的材料,也可能有多个共享一个分子身份证的材料,还可能由于缺失导致的具有不完全身份证的材料等几种可能。选择分析的基本步骤如下:
步骤1:从全部标记中选择部分标记集;
步骤2:对供试材料进行分子条带码标识;
步骤 3:将结果进行分类显示,唯一识别材料、分组识别材料和不确定材料。
1.4.4 分子身份证判定 在全库构建的基础上,选择几个标记,对待测材料进行基于选定标记的电泳试验,将电泳带型数字化,在全库构建的背景下,基于所选定标记计算该待测材料与其他材料间的相似度,判别该材料的类别归属,从而达到品种识别和品种鉴定的目的。分子身份证判定分析步骤如下:
步骤1:选定背景标记集;
步骤2:测定待测材料的带型;
步骤 3:在该标记集下,计算待测材料与数据库中全材料的相似度,以判别该材料的归属。
2 结果
2.1 分子身份证构建软件模块
分子身份证软件依据分子身份证的实现策略设计功能及界面(图 2),软件功能包括:数据库浏览及更新、全库构建、部分构建、输入构建、选择分析和ID判定等功能。
2.1.1 分子身份证软件简介 分子身份证软件应用Microsoft公司Visual Basic6.0 进行程序开发,软件在开发时充分考虑到使用的兼容性问题,软件可以在Windows9X/me/2000/XP/ win Vista/win7等大部份Windows的32位或64位操作系统下运行,软件的运行对计算机硬件环境要求不高,Intel奔腾CPU/512M内存/1G硬盘空间及以上机型都可运行。如果构建的标记及材料数量过多时,运算时间会相应增加,要想达到理想的运算效率,计算机的硬件配置不应过低。
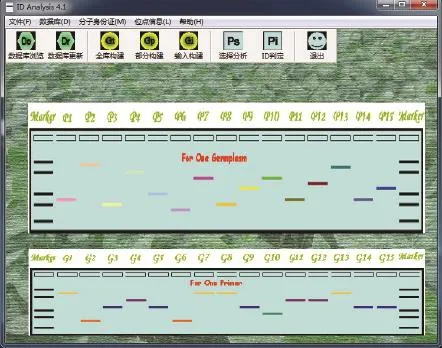
图2 分子身份证软件主界面Fig. 2 Software interface of ID analysis
分子身份证软件首发版本为ID Analysis 1.0,软件登记号:2007SR11870a,通过应用完善了软件的功能及操作界面,目前版本为ID Analysis 4.1,软件具有功能丰富、界面友好(图 3)、操作简单等优点,一步即可达到以往需要多个软件联合使用才能完成的任务。软件可以获得方式:发送索取软件的邮件给作者qshchen@126.com或访问“大豆设计网”站进行下载www.designsoybean.com。
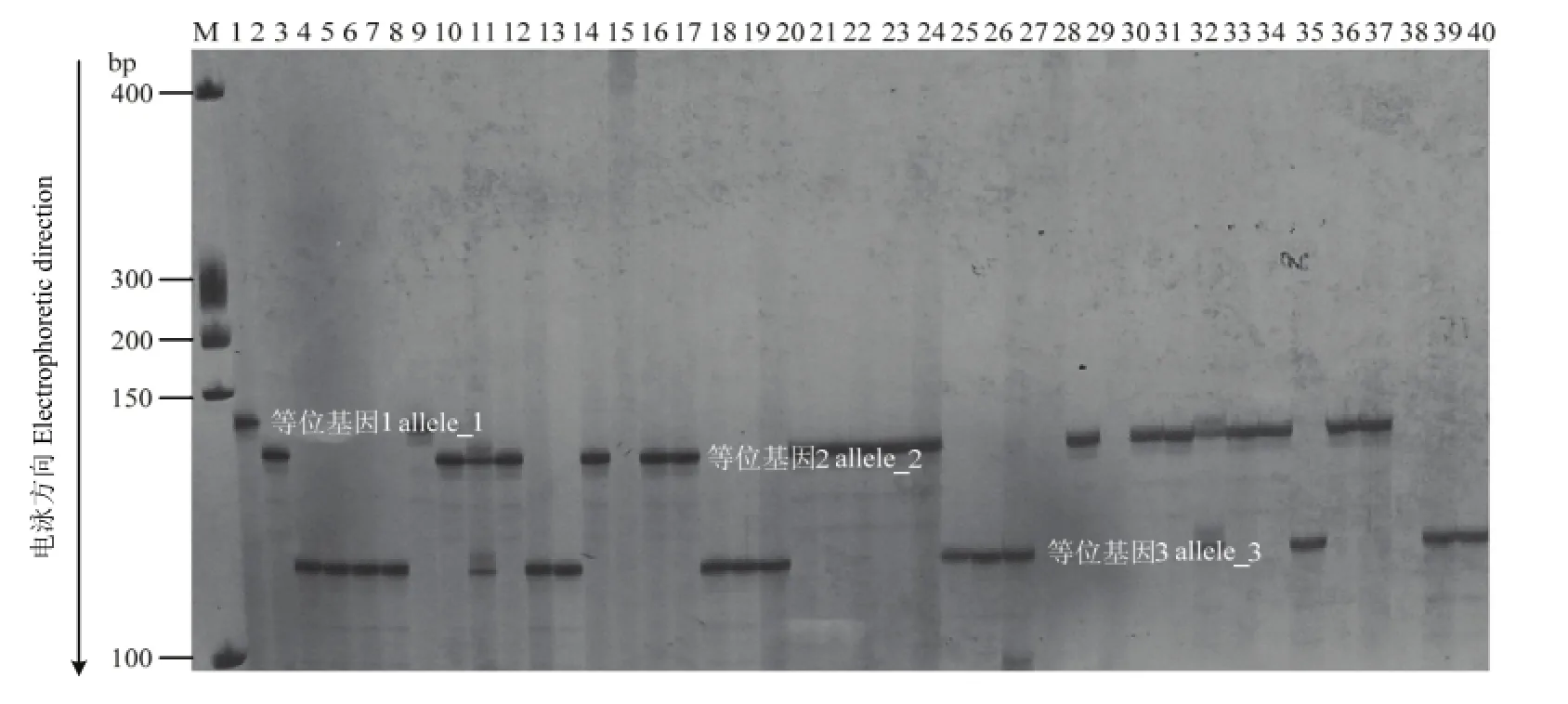
图3 Satt424等位基因特征图Fig. 3 Alleles feature of Satt424 primer
2.1.2 标记获取及数据文件准备 标记统计是将电泳胶图上的目标条带数字化的过程,具体原则是根据扩增片段的分子量由大到小依次按1、2、3、4 …… N的顺序进行记录。其中,0表示零等位基因(即该泳道由于基因片段丢失而无带),-1表示该品种数据由于试验操作造成缺失,-2表示该泳道出现杂合带型。图3为黑龙江省主栽大豆品种分子身份证构建试验中所获得的一张比较理想的电泳图,以此图为例阐明标记统计原则。
利用40对SSR引物对40份大豆品种进行电泳分析,共获得1 600个标记数据,将标记整理成软件可识别的文本文档(图4)。数据文本的第1行第1个位置表示数据矩阵大小,其中“40/40”表示该数据文本中的数据矩阵为40行40列,第一个40表示有40个材料,第二个40表示有40对引物。向右接着是引物信息,引物需要用加引号,矩阵大小及引物间加一半角空格,以换行符结尾。例如“40/40”“Satt516”“Satt338”“Satt573”。从第 2行开始每行表示1个材料,从左向右第1个位置表示材料名称,中英文皆可,但要加用引号,向右接着是该资源使用40对不同引物的电泳标记数据,资源名及带型标记间加一半角空格,以换行符结尾。例如:“合丰25” 1 1 3 3。
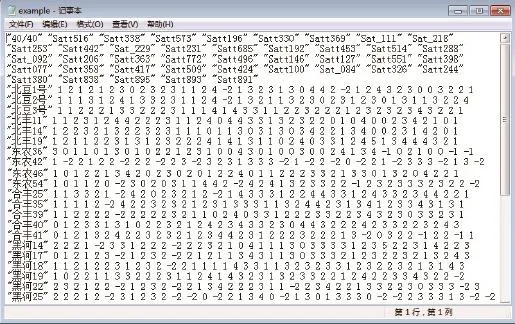
图4 软件可识别的数据文档Fig. 4 Data document for software input
2.1.3 数据集更新 分子身份证构建的基础是数据,数据是由引物和材料组成的二维标记矩阵集。由于数据缺失、引物更新和材料的变化而导致标记集数据的动态变化,而数据的改变进一步决定了分子身份证的构建也是动态可变的。因此软件设计开始时就考虑到由于对缺失数据的补充、新品种材料的更新,高多态性引物的加入等问题而导致数据集更新的麻烦。
为解决数据集更新的麻烦,软件开发了数据库合并功能(图 5)。可以根据引物和材料的列表对多个数据集进行整合,并可以对其发生改变的等位基因进行校验和提示,这样有利于整合最新的研究结果,开发全部材料最合适的分子身份证。
合并后的数据结果以文本形式输出(图 5),由结果文件可知,合并后的新数据集是由6份材料及4对引物组成,其中合并前二个数据集有1个差异数据,差异数据为“东农46,Satt516”,在a集中标记是1,在b集中标记是3,结果还显示了合并到新数据集中的材料、引物的数量及名称。
2.2 软件验证
2.2.1 全库构建 将40对引物对40份大豆品种的标记数据导入软件,具体如下:打开分子身份证软件,点击快捷工具栏的第三个图标“全库构建”,即可打开全库构建窗口(图 6)。点击文件下拉菜单-打开-浏览到数据文件-打开,导入数据文件,点击“ID analysis”按钮即可完成分子身份证构建。
构建完的数据会显示在窗口的数据显示区,点击窗口文件下拉菜单-输出-浏览文件保存位置-命名文件名-保存,结果文件以文本形式保存。文件内容共分4部分,第1部分指明分析时的参数;第2部分指明不符合引物信息;第3部分指明特异引物信息;第4部分给出引物组合及每个材料的分子身份证的编号(图6)。
由分子身份证构建结果可知,在40对引物对40个大豆品种的分子身份证构建中:共有13对引物由于缺失过多,不符合标准被剔除,剔除引物为Sat_111、Sat_218、Satt231、Satt685、Satt514、Satt551、Satt077、Satt358、Satt424、Satt100、Satt838、Satt893和Satt891。共有7对引物由于与其他引物相似系数过高,不符合标准被剔除,剔除引物为Satt253、Satt192、Satt417、Sat_229、Satt127和Satt496。在分析的40个品种中,共有5个品种具有7个特异等位基因,因此,可以通过这些特异等位基因直接确定需要鉴定的品种,通过计算仅需要7对引物即可区分40个大豆品种,引物组合为 Satt398、Satt380、Satt453、Satt288、Satt244、 Sat_092和Satt206,例如北豆3号在该引物组合下的分子身份证编号为2411343。
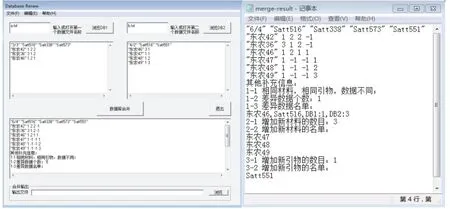
图5 数据合并窗口及结果Fig. 5 Data merge window and output result
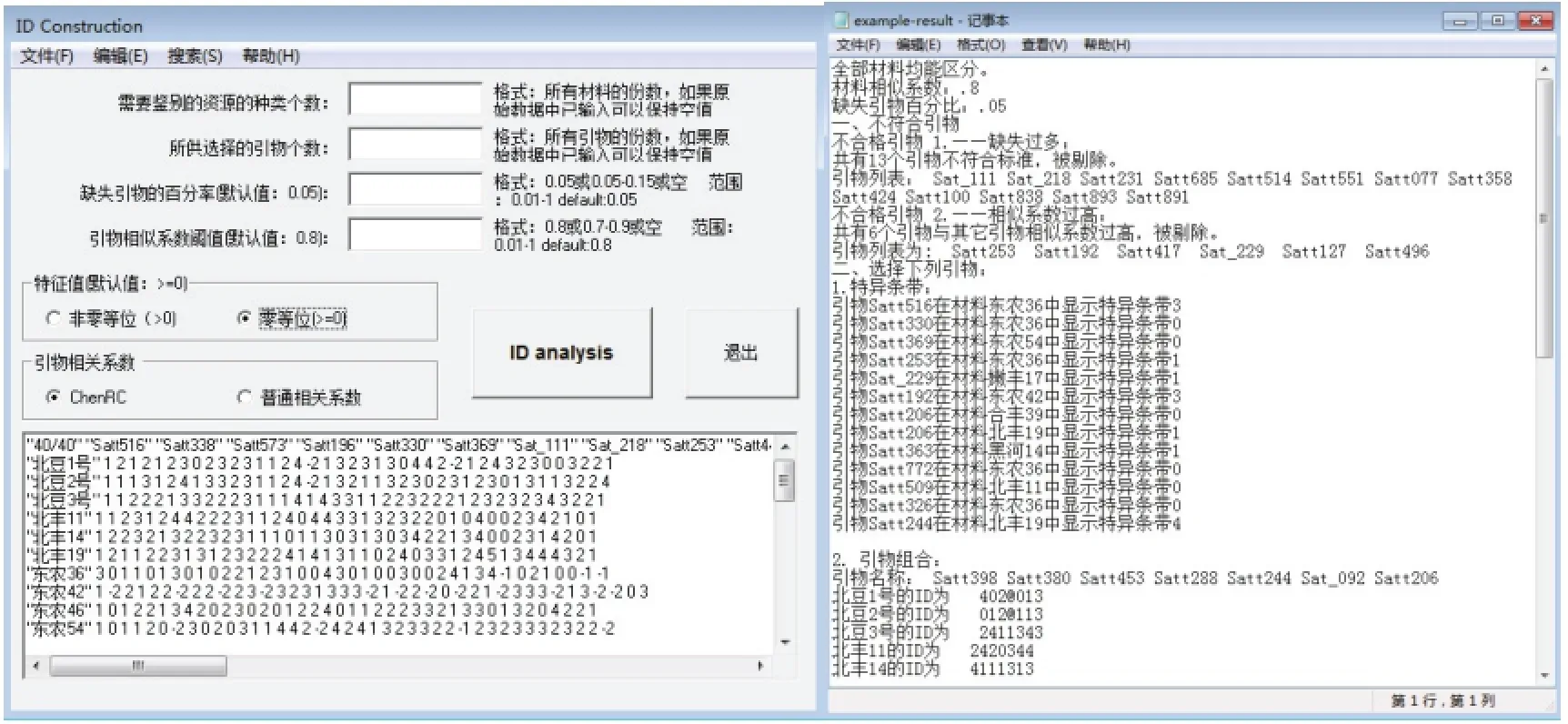
图6 全库构建窗口及结果Fig. 6 Full-library construction window and output result
2.2.2 部分构建 部分构建的具体操作如下:打开分子身份证软件,点击快捷工具栏的第四个图标“部分构建”,即可打开部分构建窗口(图 7)。点击文件下拉菜单-打开-浏览到数据文件-打开,导入数据文件。从材料栏里选择一些材料到目标材料栏内,点击“ID analysis”按钮即可完成部分材料的分子身份证构建。同时为了方便用户使用,软件还提供“输入构建”窗口,在该窗口中将材料的选择方式变为人工输入,其他功能相同。
构建完的结果文件内容共分3部分,第1部分指明不符合引物信息;第2部分指明特异引物信息;第3部分给出引物组合及被选择的部分材料的分子身份证的编号(图7)。
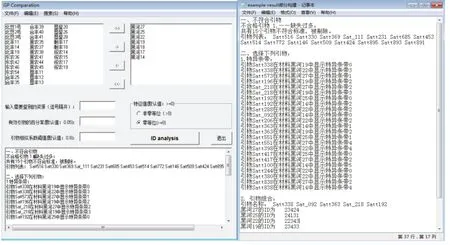
图7 部分构建窗口及结果Fig. 7 Partial-library construction window and output result
2.2.3 ID判定 ID判定的具体操作如下:打开分子身份证软件,点击快捷工具栏的第七个图标“ID判定”,即可打开ID判定窗口(图8)。点击文件下拉菜单-打开-浏览到数据文件-打开,导入数据文件。在“引物及ID”栏内输入引物名称及分子身份证编号,其格式为“Satt338,Satt369,Satt453:314”,点击“Possible GP”按钮即可计算出由引物组合以及身份证编号所确定的唯一材料名称。

图8 ID判定窗口Fig. 8 Molecular ID determination window and output result
有些时候可能需要考察部分受关注的引物在分子多态水平上区分材料的能力。此时可以使用“选择分析”功能,其结果可能是被唯一区分的材料,也可能是多个共享一个分子身份证的材料,或是由于缺失导致的具有不完全身份证的材料等几种可能。
3 讨论
3.1 软件算法改进
关于寻找最优引物组合,可以采用贪婪算法,穷举法等多种方法。贪婪算法[33-34]可提高效率,但较易错过最优解。穷举法[35]比较全面客观,但计算量大,耗时耗力。而逐步扩增法利用引物缺失率和引物相似系数对引物进行了有效的筛选、排序和删除,在运算过程中,逐步淘汰引物的等位基因组合频率为1的材料,大大提高了运算速率,实现了算法的优化改进。
关于标记多态度排序,在计算中可以看出,调整标记的顺序会直接影响结果,因此,可根据单个标记等位基因多态性大小进行排序,使区分能力较强的标记更早出现,这样就使标记组合的区分度迅速增加,从而加快算法搜索速度。
关于材料容量缩减,每次有新标记引入都会重新计算当前标记多态型下的每个材料条带码频数,而有部分材料在新标记入选前等位基因组合频率已经为1,达到了区分目的,没有重新计算的必要。因此,可以将被区分开的材料从计算的数据集中删除,逐步缩减材料容量,达到加快算法目的。
3.2 软件程序改进
到目前该软件已经在大豆、水稻、花生、玉米、高粱、真菌、木耳等多种作物上得到广泛应用[36-41],但仍然存在不足,由其是随着新材料的改良及新标记的发现[1,20-22]以及生物信息学的发展,在未来的分析中所要面对的数据量将更为庞大[35,42],甚至只能在Linux系统下才能分析。目前,ID analysis软件的核心算法在软件程序实现过程中仍然需要进一步优化,VB程序具有操作系统的局限性,以及执行效率低的缺点[43-45];另一方面开发在线的网络版分子身份证构建系统,对于方便更多研究人员使用,提高软件利用率等方面具有更大的意义。
相比VB程序语言,Java技术具有简单、完全面向对象、属于解释执行语言、安全性高、可移植性强、执行性能高、多线程以及动态性等优点[46-47]。利用Java技术实现分子身份证软件的核算法,即可打包成单机版软件,又可作为网络在线分析的服务端程序,并且可跨平台使用,解决了庞大标记数据的运算效率问题,达到一次开发多角度利用的目的。
3.3 软件引物的选取和图像的获得
分子身份证软件构建核心目标是为了利用最优引物对组合完成对目标材料群体的唯一性区分,如果具有较为完善的品种资源数据库系统,便可以解决资源的鉴定,育种材料的分析和候选审定材料的创新性判定等很多现实存在的棘手问题。
在软件数据库开发方面,可以基于研究对象特性开发的各类特殊分子标记来构建分子身份证。在标记开发方面,可以针对研究资源的特性,设计独特的分子身份证,利用在产量、品质、抗耐性上具有特殊性状的分子标记[48]对未知材料进行基因型分析和等位基因信息获取,可以直接完成材料对应性状的评价和分子辅助育种研究。
目前,软件只能对电泳胶图数字化后的数据进行分析,而由于传统电泳胶图分辨率低[49-50],在数字化过程中经常会出现识别偏差。为解决问题,软件应该具有图形分析功能,借助标识引物确定等位基因大小,对电泳胶图进行基于条带分子量大小的数字化识别。软件图形分析功能的开发必将提高资源、品种鉴定的准确性。从分析仪器发展水平来看,毛细管电泳将为分子身份证软件提供解决问题之道[51],在此基础上开发全自动分子身份证分析技术乃至基于分子身份证的快速资源鉴定仪都将成为可能。
4 结论
基于SSR标记在材料中的多态性特点,提出了快速有效地区分品种资源的算法——逐步扩增法。利用Visual Basic6.0编制了基于逐步扩增法的作物分子身份证构建软件——ID analysis。该软件可以构建作物分子身份证,实现了只需单个软件即可完成作物分子身份证构建的过程,达到了对资源品种鉴定的目的。
References
[1] 滕卫丽, 卢双勇, 高阳, 孙明明, 韩英鹏, 武小霞, 李文滨. 黑龙江省 1986-2010年大豆审定品种的品质性状分析. 作物杂志, 2011,25(2): 105-108. TENG W L, LU S Y, GAO Y, SUN M M, HAN Y P, WU X X, LI W B. Analysis of yield changes of soybean cultivars authorized in Heilongjiang province. Crops, 2011, 25(2): 105-108. (in Chinese)
[2] 孙明明. 2011年黑龙江省审定推广的大豆新品种. 大豆科学, 2011,30(4): 713-718. SUN M M. Examination and popularization of soybean varieties at Heilongjiang province in 2011. Soybean Science, 2011, 30(4):713-718. (in Chinese)
[3] 宋显军. 2012年黑龙江省审定推广的大豆新品种. 大豆科学, 2012,31(3): 504-510. SONG X J. Examination and popularization of soybean varieties at Heilongjiang province in 2012. Soybean Science, 2012, 31(3):504-510. (in Chinese)
[4] 王萍. 2013年黑龙江省审定推广的大豆新品种I. 大豆科学, 2013,32(3): 429-432. WANG P. Examination and popularization of soybean varieties at Heilongjiang province in 2013-I. Soybean Science, 2013, 32(3):429-432. (in Chinese)
[5] 王萍. 2013年黑龙江省审定推广的大豆新品种II. 大豆科学, 2013,32(4): 576-579. Wang P. Examination and popularization of soybean varieties at Heilongjiang province in 2013-II. Soybean Science, 2013, 32(4):576-579. (in Chinese)
[6] 孙明明, 王萍. 2014年黑龙江省审定推广的大豆品种I. 大豆科学,2014, 33(3): 463-466. SUN M M, WANG P. Examination and popularization of soybean varieties at Heilongjiang province in 2014-I. Soybean Science, 2014,33(3): 463-466. (in Chinese)
[7] 孙明明, 王萍. 2014年黑龙江省审定推广的大豆品种II. 大豆科学,2014, 33(4): 626-628. SUN M M, WANG P. Examination and popularization of soybean varieties at Heilongjiang province in 2014-II. Soybean Science, 2014,33(4): 626-628. (in Chinese)
[8] 孙明明, 王萍, 吕世翔. 2015年黑龙江省审定推广的大豆品种 I.大豆科学, 2015, 34(5): 918-920. SUN M M, WANG P, LÜ S X. Examination and popularization of soybean varieties at Heilongjiang province in 2015-I. Soybean Science,2015, 34(5): 918-920. (in Chinese)
[9] 孙明明, 王萍, 吕世翔. 2015年黑龙江省审定推广的大豆品种 II.大豆科学, 2015, 34(6): 1100-1102. SUN M M, WANG P, LÜ S X. Examination and popularization of soybean varieties at Heilongjiang province in 2015-II. Soybean Science, 2015, 34(6): 1100-1102. (in Chinese)
[10] 杨冬风, 朱洪德. 基于近红外透射光谱分析和BP神经网络的大豆品种识别. 大豆科学, 2013, 32(2): 249-253. YANG D F, ZHU H D. Recognition of soybean varieties based on near infrared transmittance spectroscopy and bp neural network. Soybean Science, 2013, 32(2): 249-253. (in Chinese)
[11] 张云丽, 韩宪忠, 王克俭. 基于深度颜色特征的灰度直方图玉米品种识别研究. 作物杂志, 2015(1): 156-159. ZANG Y L, HANG X Z, WANG K J. Study on corn variety identification based on depth and color features of gray histogram. Crops, 2015(1): 156-159. (in Chinese)
[12] TAN K Z, CHAI Y H, SONG W X, CAO X D. Identification of soybean seed varieties based on hyperspectral image. Transactions of the Chinese Society of Agricultural Engineering, 2014, 30(9):235-242.
[13] 任保青, 陈之端. 植物 DNA条形码技术. 植物学报, 2010, 45(1):1-12. REN B Q, CHEN Z R. DNA barcoding plant life. Chinese Bulletin of Botany, 2010, 45(1): 1-12. (in Chinese)
[14] SARWAT M, YAMDAGNI M M. DNA barcoding microarrays and next generation sequencing: Recent tools for genetic diversity estimation and authentication of medicinal plants. Critical Reviews in Biotechnology, 2016, 36(2): 191-203.
[15] VERE N D, RICH T C G, TRINDER S A, LONG C. DNA barcoding for plants. Methods in Molecular Biology, 2015, 1245: 101-118.
[16] 殷丽琴, 彭云强, 付绍红, 杨进, 陈涛, 黄敏, 余勤, 韦献雅, 牛应泽. 基于ISSR标记的彩色马铃薯遗传多样性分析及指纹图谱构建.西南农业学报, 2016, 29(1):20-25. YIN L Q, PENG Y Q, FU S H, YANG J, CHEN T, HUANG M, YU Q,WEI X Y, NIU Y Z. Genetic diversity and DNA fingerprint of pigmented potato (Solanum tuberosum L.) based on ISSR markers. Southwest China Journal of Agricultural Sciences, 2016, 29(1): 20-25. (in Chinese)
[17] SONG Q J, HYTEN D L, JIA G F, QUIGLEY C V, FICKUS E W,NELSON R L, CREGAN P B. Fingerprinting soybean germplasm and its utility in genomic research. Genes Genomes Genetics, 2015, 5(10):1999-2006.
[18] 黄丹娟, 马建强, 陈亮. 茶树 DNA分子指纹图谱研究进展. 茶叶科学, 2015, 35(6):513-519. HUANG D J, MA J Q, CHEN L. Research progress on DNA molecular fingerprinting of tea plant (Camellia sinensis). Journal of Tea Science, 2015, 35(6): 513-519. (in Chinese)
[19] TARGOŃSKA M, BOLIBOK-BRĄGOSZEWSKA H, RAKOCZYTROJANOWSKA M. Assessment of genetic diversity in secale cereale based on SSR markers. Plant Molecular Biology Reporter,2016, 34: 37-51.
[20] SONG Q J, MAREK L F, SHOEMAKER R C, LARK K G,CONCIBIDO V C, DELANNAY X, SPECHT J E, CREGAN P B. A new integrated genetic linkage map of the soybean. Theoretical and Applied Genetics, 2004, 109(1): 122-128.
[21] CHOI I Y, HYTEN D L, MATUKUMALLI L K, SONG Q, CHAKY JM, QUIGLEY C V, CHASE K, LARK K G, REITER R S, YOON M S, HWANG E Y, YI S I, YOUNG N D, SHOEMAKER R C,TASSELL C P, SPECHT J E, CREGAN P B. A soybean transcript map: gene distribution, haplotype and single-nucleotide polymorphism analysis. Genetics Society of America, 2007, 176: 685-696.
[22] 美国农业部大豆基因数据库[EB/OL]. [2008.12.23]. http://www. soybase.org/ dlpages. The USDA Soybean Genetic Database[EB/OL]. [2008.12.23]. http:// www.soybase. org/dlpages. (in Chinese)
[23] 郭数进, 杨凯敏, 霍瑾, 周永航, 王宏勇, 李贵全. 山西大豆自然群体产量及品质性状与SSR分子标记的关联分析. 山西农业科学,2015, 43(4): 374-377, 387. GUO S J, YANG K M, HUO J, ZHOU Y H, WANG H Y, LI G Q. Association analysis of yield,quality traits and SSR markers of natural soybean population in Shanxi. Journal of Shanxi Agricultural Sciences, 2015, 43(4): 374-377, 387. (in Chinese)
[24] 陈亮, 郑宇宏, 范旭红, 孟凡凡, 孙星邈, 张云峰, 王明亮, 王曙明.大豆SSR指纹图谱身份证的研究进展与展望. 大豆科技, 2015, 2:38-43. CHEN L, ZHENG Y H, FAN X H, MENG F F, SUN X M, ZHANG Y F, WANG M L, WANG S M. Progress and perspective on SSR fingerprint identification establishment in soybean. Soybean Science and Technology, 2015, 2: 38-43. (in Chinese)
[25] 王梓钰, 年海, 宋恩亮, 杨春明, 王新风, 马巍, 富健. 华南大豆重要农艺性状与SSR的关联分析. 贵州农业科学, 2015, 43(12): 6-8. WANG Z Y, NIAN H, SONG E L, YANG C M, WANG X F, MA W,FU J. Association analysis on important agronomic traits and SSR markers in South China. Guizhou Agricultural Sciences, 2015, 43(12):6-8. (in Chinese)
[26] 高运来, 朱荣胜, 刘春燕, 李文福, 蒋洪蔚, 李灿东, 姚丙晨, 胡国华, 陈庆山. 黑龙江部分大豆品种分子 ID 的构建. 作物学报,2009, 35(2): 211-218. GAO Y L, ZHU R S, LIU C Y, LI W F, JIANG H W, LI C D, YAO B C, HU G H, CHEN Q S. Establishment of molecular id in soybean varieties in Heilongjiang, China. Acta Agronomica Sinica, 2009, 35(2):211-218. (in Chinese)
[27] 张靖国, 田瑞, 陈启亮, 杨晓平, 胡红菊. 基于SSR标记的梨栽培品种分子身份证的构建. 华中农业大学学报, 2014, 33(1): 12-17. ZHANG J G, TIAN R, CHEN Q L, YANG X P, HU H J. Establishment of molecular id for pear cultirars based on SSR markers. Journal of Huazhong Agricultural University, 2014, 33(1): 12-17. (in Chinese)
[28] 陆徐忠, 倪金龙, 李莉, 汪秀峰, 马卉, 张小娟, 杨剑波. 利用SSR分子指纹和商品信息构建水稻品种身份证. 作物学报, 2014, 40(5):823-829. LU X Z, NI J L, LI L, WANG X F, MA H, ZHANG X J, YANG J P. Construction of rice variety indentity using SSR fingerprint and commodity information. Acta Agronomica Sinica, 2014, 40(5):823-829. (in Chinese)
[29] 徐雷锋, 葛亮, 袁素霞, 任君芳, 袁迎迎, 李雅男, 刘春, 明军. 利用荧光标记SSR构建百合种质资源分子身份证. 园艺学报, 2014,41(10): 2055-2064. XU L F, GE L, YUAN S X, REN J F, YUAN Y Y, LI Y N, LIU C,MING J. Using the fluorescent labeled SSR markers to establish molecular identity of lily germplasms. Acta Horticulturae Sinica,2014, 41(10): 2055-2064. (in Chinese)
[30] 陈庆山, 刘春燕, 吕东, 何建勋. 大豆 DNA提取基本原理的探讨.东北农业大学学报, 2004, 35(2): 129-134. CHEN Q S, LIU C Y, LÜ D, HE J X. The basic principle of DNA extraction from soybean. Journal of Northeast Agricultural University,2004, 35(2): 129-134. (in Chinese)
[31] 陈庆山, 刘春燕, 刘迎雪, 刘海燕, 陈立君, 付尧, 单继勋, 郭强,张丽娜. 核酸体外扩增技术. 中国生物工程, 2004, 24(5): 10-14. CHEN Q S, LIU C Y, LIU Y X, LIU H Y, CHEN L J, FU R, SHAN J X, GUO Q, ZHANG L N. Progress of nucleic acid amplification technologies. China Biotechnology, 2004, 24(5): 10-14. (in Chinese)
[32] 陈庆山. 作物分子身份证构建策略及其在大豆中的应用. 哈尔滨:黑龙江人民出版社, 2014. CHEN Q S. Crop Molecular Identity Building Strategy and the Application in the Soybean. Harbin: People Press Heilongjiang, 2014. (in Chinese)
[33] COWELL R G. A simple greedy algorithm for reconstructing pedigrees. Theoretical Population Biology, 2013, 83: 55-63.
[34] CHEN H, ZHOU Y C, TANG Y Y, LI L Q, PAN Z B. Convergence rate of the semi-supervised greedy algorithm. Neural Networks, 2013,44: 44-50.
[35] GUNVANT P, BABU V, RUPESH D, SILVAS P, BJORN N, ZHAO M Z, HUMIRA S, LI S, LI L, JUHI C, LIU Y, TRUPTI J, XU D,NGUYEN H T. Soybean (Glycine max) SWEET gene family: Insights through comparative genomics, transcriptome profiling and whole genome re-sequence analysis. BMC Genomics, 2015, 16: 520-536.
[36] 王黎明, 焦少杰, 姜艳喜, 严洪冬, 苏德峰, 孙广全. 142份甜高粱品种的分子身份证构建. 作物学报, 2011, 37(11): 1975-1983. WANG L M, JIAO S J, JIANG Y X, YAN H D, SU D F, SUN G Q. Establishment of molecular identity in 142 sweet sorghum varieties. Acta Agronomica Sinica, 2011, 37(11): 1975-1983. (in Chinese)
[37] 王蕾. 中国花生品种遗传多样性研究及分子 ID数据库的构建[D].青岛: 青岛科技大学, 2014: 51-56. WANG L. Analysis of genetic diversity and construction of molecular id in peanut varieties in China[D]. Qingdao: Qingdao University of Science Technology, 2014: 51-56. (in Chinese)
[38] 于潇, 许修宏, 刘华晶. 黑龙江部分野生黑木耳菌株的分子 ID构建. 中国农学通报, 2012, 28(13):171-175. YU X, XU X H, LIU H J. Establishment of molecular id in auricularia auricula in Heilongjiang. Chinese Agricultural Science Bulletin, 2012,28(13): 171-175. (in Chinese)
[39] 丁俊杰, 姜翠兰, 顾鑫, 杨晓贺, 赵海红, 申宏波, 仕相林, 刘春燕,胡国华, 陈庆山. 利用与大豆灰斑病抗性基因连锁的 SSR标记构建大豆品种(系)的分子身份证. 作物学报, 2012, 38(12): 2206-2216. DING J J, JIANG C L, GU X, YANG X H, ZHAO H H, SHEN H B,SHI X L, LIU C Y, HU G H, CHEN Q S. Establishment of molecular id of soybean varieties (Lines) using SSR markers linked to resistance genes against Cercospora sojina. Acta Agronomica Sinica, 2012,38(12): 2206-2216. (in Chinese)
[40] 李伟忠, 许崇香, 安英辉, 孙梅, 闵丽, 姜森, 陈庆山, 胡国华. 257份玉米自交系分子ID的构建. 玉米科学, 2013, 21(2): 24-30. LI W Z, XU C X, AN Y H, SUN M, MIN L, JIANG S, CHEN Q S,HU G H. Establishment of molecule id in 257 maize inbred lines. Journal of Maize Sciences, 2013, 21(2): 24-30. (in Chinese)
[41] 何琳, 何艳琴, 刘业丽, 邱强, 栾怀海, 韩雪, 胡国华. 2012年北方春大豆国家区试大豆品种纯度鉴定、分子ID构建及遗传多样性分析. 中国农学通报, 2014, 30(18): 277-282. HE L, HE Y Q, LIU Y L, QIU Q, LUAN H H, HAN X, HU G H. Purity identification, molecular ID establishment and genetic diversity analysis of soybeans attending national regional test of north spring soybean in 2012. Chinese Agricultural Science Bulletin, 2014, 30(18):277-282. (in Chinese)
[42] LEE Y G, JEONG N, KIM J H, LEE K, KIM K H, PIRANI A, HA B K, KANG S T, PARK B S, MOON J K, KIM N, JEONG S C. Development, validation and genetic analysis of a large soybean SNP genotyping array. The Plant Journal, 2015, 81(4): 625-636.
[43] 王良莹. 主流编程语言的特点与比较. 电脑编程技巧与维护,2009(6): 96-106. WANG L Y. The characteristics and comparisons of certain mainstream programming language wang liangying. Study of Computer Application in Education, 2009(6): 96-106. (in Chinese)
[44] 关琳琳. 试论 VB编程语言在软件开发中的应用. 河南科技,2013(1): 1-3, 44. GUAN L L. Discuss VB programming language application in software development. Journal of Henan Science and Technology,2013(1): 1-3, 44. (in Chinese)
[45] 李畅. 编程语言的特点与比较. 华中师范大学研究生学报, 2005,12(3): 145-148. LI C. The analysis of singing psychology in singing training and art performance. Huazhong Normal University Journal of Postgraduates,2005, 12(3): 145-148. (in Chinese)
[46] 马响. 基于java语言在web开发的知识探讨. 信息技术与信息化,2015(11): 169-171. MA X. Based on Java language knowledge in web development. Information. Technology and Information, 2015(11): 169-171. (in Chinese)
[47] 冀潇, 李杨. JavaScript 与Java在Web开发中的应用与区别. 通信技术, 2013, 46(6): 145-151. JI X, LI Y. Applications and differences of between Javascript and Java in web development. Communications Technology, 2013, 46(6):145-151. (in Chinese)
[48] 陈庆山. 大豆分子辅助育种体系构建、遗传—物理图整合与应用及转基因研究[R]. 黑龙江省农垦科研育种中心, 2009. CHEN Q S. Construction of Molecular Assisted Breeding System,Integration and Application of Genetic map and Physical map, and Transgenic Research in Soybean[R]. The crop research and breeding center of land-reclamation of Heilongjiang province, 2009. (in Chinese)
[49] 孟菲, 蔡小彦, 崔兴雷, 刘方, 王星星, 周忠丽, 王春英, 王玉红,彭仁海, 王坤波. 聚丙烯酰胺凝胶银染技术的优化. 中国棉花,2015, 42(4): 12-14. MENG F, CAI X Y, CUI X L, LIU F, WANG X X, ZHOU Z L,WANG C Y, WANG Y H, PENG R H, WANG K B. An optimized protocol for silver staining of polyacrylamide Gel. Journal Chinese Cotton, 2015, 42(4): 12-14. (in Chinese)
[50] 梁宏伟, 王长忠, 李忠, 罗相忠, 邹桂伟. 聚丙烯酰胺凝胶快速、高效银染方法的建立. 遗传, 2008, 30(10): 1379-1382. LIANG H W, WANG C Z, LI Z, LUO X Z, ZOU G W. Improvement of the silver-stained technique of polyacrylamide Gel electrophoresis. Hereditas, 2008, 30(10): 1379-1382. (in Chinese)
[51] SÁNCHEZ-PÉREZA R, BALLESTERB J, DICENTAA F, ARÚSB P,MARTÍNEZ-GÓMEZA P. Comparison of SSR polymorphisms using automated capillary sequencers, and polyacrylamide and agarose gel electrophoresis: Implications for the assessment of genetic diversity and relatedness in almond. Scientia Horticulturae, 2006, 108:310-316.
(责任编辑 李莉)
Software Development of -ID Analysis for Crop Molecular Identity Construction
HU Zhen-bang1, GAO Yun-lai1, QI Zhao-ming1, JIANG Hong-wei2, LIU Chun-yan2, XIN Da-wei1, HU Guo-hua2,PAN Xiao-cheng3, CHEN Qing-shan1
(1College of Agriculture in Northeast Agricultural University, Harbin 150030;2Land Reclamation Science & Research Breeding Center of Heilongjiang Province, Harbin 150090;3Sideline Base for 65301 Force, Wudalianchi 164100, Heilongjiang)
Abstract:【Objective】Based on the SSR molecular marker data of crops resources, software ID analysis was developed using visual Basic6.0 for crop molecular identity construction, which could screen primer combinations rapidly and accurately for efficient cultivar identification. 【Method】The crop molecular ID theory was proposed by Mr. Qingshan Chen. SSR markers in crop varieties showed high polymorphism characteristics, and a set of markers was permutated and combined to quickly and effectively divide varieties with step amplification method. Finally, 40 pairs of SSR data of 40 soybean varieties in Heilongjiang province were used for identity construction with the software. 【Result】 ID analysis software was developed with core algorithm by using Visual Basic 6.0 to design man-machine interactive interface. This software has integrated full-library construction, partial-library construction,molecular ID determination, and database merging function. The full-library construction was the core functions together which could quickly obtained the minimum SSR prime combination to distinguish all the varieties. Partial-library construction could be used for some target varieties identification. For an unknown materials, with the already existing molecular identity database and the SSR analysis data, molecular ID determination could be used to determine the variety names or similar varieties. Database merging could be used to integrate several experimental data into a data set. The following results were analyzed by full-library construction with case data. First, among the 40 pairs of SSR data of 40 soybean cultivars, a total of 13 primers were excluded because missing data were too much and did not meet the standards, they were Sat_111, Satt218, Satt231, Satt685, Satt514, Satt551, Satt077, Satt358,Satt424, Satt100, Satt838 ,Satt893, and Satt891. Second, 6 primers were excluded because they showed high similarity coefficient with other primers, and they were Satt253, Satt192, Satt417, Sat_229, Satt127, and Satt496. Third, 5 varieties showed 7 specific alleles among all 40 varieties. They were, allele 3 of Satt516 and allele 1 of Satt253 showed in Dongnong36, allele 1 of Sat_229 showed in Nenfeng 17, allele 3 of Satt192 showed in Dongnong42, allele 1 of Satt206 and allele 4 of Satt244 showed in Beifeng 19,and allele 1 of Satt363 showed in Heihe14. So these specific allelic genes could directly identify the varieties. Forth, only seven pairs of SSR primers could distinguish 40 soybean varieties completely. The primer combinations were Satt398, Satt380, Satt453, Satt288,Satt244, Sat_092, and Satt206. 【Conclusion】 In this research, the software analysis ID was developed to construct crop molecular identity. The software has a friendly interface and easy to be used, high efficiency and flexible. The construction of the crop molecular identity can be realized completely by using a single software, and thus achieving the purpose of variety identification. With the development of technology of the application of capillary electrophoresis and the molecular identity theory, the development of an automatic molecular identity analysis system and even a rapid resource identification system will become possible.
Key words:soybean; crops; SSR markers; molecular identity; software development
收稿日期:2016-02-22;接受日期:2016-04-18
基金项目:国家自然科学基金青年基金(31401465)、黑龙江省留学回国人员科技项目择优资助启动项目、哈尔滨市科技创新人才研究专项资金(杰出青年人才计划类)(RC2015JQ002004)、国家自然科学基金(31471516,31271747)
