牦牛星状病毒的RT-PCR检测方法的建立和变异分析
2016-07-14宋志刚
陈 曦,汤 承,张 斌,宋志刚,周 芳,岳 华
(西南民族大学生命科学与技术学院,成都 610041)
牦牛星状病毒的RT-PCR检测方法的建立和变异分析
陈曦,汤承,张斌,宋志刚,周芳,岳华*
(西南民族大学生命科学与技术学院,成都 610041)
近期作者实验室从腹泻牦牛粪便样本中鉴定出一种新型星状病毒(AstV)——牦牛AstV,本研究旨在建立检测牦牛AstV的RT-PCR方法。首先设计引物扩增牦牛AstV 630 bp的ORF2基因片段,进行序列分析;在此基础上设计检测引物,建立检测牦牛AstV的RT-PCR方法。结果显示,从20份牦牛腹泻粪便中扩增出13个牦牛AstVORF2基因片段,核苷酸相似性为99.3%~100%,而与牛肠源星状病毒(BAstV)ORF2基因的相似性仅为59.5%~80.8%;所建立的RT-PCR方法只扩增牦牛AstV的特异片段,对BAstV和其他无关病原无扩增;对病毒核酸的最低检测限为57.3 fg·μL-1;比较试验表明,所建立的RT-PCR方法对牦牛AstV的检出率明显优于现有检测BAstV的RT-PCR方法。其对牦牛腹泻与健康粪便样本中牦牛AstV的检出率分别为55.5%(76/137)和7.7%(2/26)(P<0.01)。试验结果表明,扩增的ORF2基因片段的核苷酸序列在牦牛AstV毒株间高度保守,但相对于BAstV变异大;基于该序列建立的检测牦牛AstV的RT-PCR方法特异性好,灵敏度高,稳定性好,为牦牛AstV的检测和流行病学调查提供了有力工具;同时,本试验结果也提示牦牛AstV与犊牦牛腹泻有联系。
牦牛;星状病毒;RT-PCR;ORF2基因;遗传变异
牛星状病毒(bovine astrovirus,BAstV)是于1978年从英国7日龄犊牛腹泻粪便中首次分离并命名的,由于该病毒感染SPF牛后未引起腹泻症状,被认为是无致病性的[1]。然而,1984年G.N.Woode等的研究发现BAstV分离株US1接种SPF牛后虽然不能引起临床症状,但能够引起牛回肠圆顶上皮细胞中的M细胞发生病变,并且粪便颜色由棕变黄;同时,该研究还表明BAstV单独存在时不能导致牛发生腹泻症状,但与其他病毒(如轮状病毒、冠状病毒等)混合感染时引起的临床症状更加严重[2]。近年的流行病学资料也表明,BAstV与犊牛腹泻有联系[3-5]。
最近,作者实验室采用宏病毒组技术首次从腹泻牦牛粪便样本中鉴定出AstV,并获得了一株牦牛AstV的基因组序列,与肠源BAstV的核苷酸序列相似性为46.4%~66.2%,其中ORF1a、ORF1b和ORF2与肠源BAstV的相应核苷酸序列相似性分别为61.7%~78.8%、74.4%~87.0%和40.6%~57.4%;而氨基酸序列的相似性分别为71.7%~91.6%、86.3%~96.4%和50.2%~64.2%[6]。该毒株的ORF2基因发生了基因重组现象,推导的重组位点位于核苷酸序列的804 bp处,序列分析显示1—804 bp片段与BAstV B76-HK的相似性最高,而805 bp以后的片段与鹿AstV CCAstV株的同源关系最近,是一种新型AstV[6]。本研究的目的是建立检测牦牛AstV的RT-PCR方法,并且对2014—2015年四川省阿坝州的137份腹泻犊牦牛粪便样本和26份健康犊牦牛粪便样本进行检测。
1 材料与方法
1.1病毒(菌)株与临床样本
牦牛AstV 核酸样本为宏病毒组测序证实的阳性样本;用于特异性验证的病毒(菌)株和核酸样本:牦牛源轮状病毒(bovine rotavirus,BRV.swun0105)、牦牛源牛病毒性腹泻-黏膜病病毒(bovine viral diarrhea virus,BVDV.swun0603)、牦牛源肠炎病毒(bovine enterovirus,BEV.swun0510);牦牛源K99大肠杆菌(swun4025)、牦牛源都柏林沙门菌(swun3736)、牦牛源产气荚膜梭菌(swun2930)、牦牛源空肠弯曲杆菌(swun1639);BAstV、牦牛源冠状病毒(bovine coronavirus,BCV)、牦牛源细小病毒、牦牛源Kobu病毒核酸样本以及牦牛源艾美尔球虫、牦牛源瑞氏隐孢子虫的DNA样本由本实验室保存。临床样本为2014年7月和2015年6月从四川省阿坝州采集的163份犊牦牛(≤3月龄)粪便样本,其中腹泻粪便样本137份,分别来自12个牧场;健康粪便样本26份,来自于同地区未发生腹泻性疾病的4个牧场,保存在-80 ℃。
1.2主要试剂及仪器
TRIzolTMReagent、PrimescriptTM、PMD19-T载体均购自TaKaRa公司;DNA Marker、大肠杆菌DH5α 感受态细胞、DNA凝胶回收试剂盒、质粒提取试剂盒均购自宝生物工程(大连)有限公司产品;凝胶成像系统Doc2000(Bio-Rad公司,美国);紫外分光光度计Cary50Probe(Vatian公司,美国);高速冷冻离心机(Eppendorf公司,德国)。
1.3引物设计与合成
本研究在对前期获得的宏病毒组数据和牦牛AstV S8株基因组序列(KM822593)分析的基础上[6],采用Primer 5.0引物设计软件设计引物(P1、P2):F:5′-TCTTTGGAGGGGAGGACC-3′,R:5′- ATCCCCCTTGGTGTTGGT-3′,扩增长度为630 bp的牦牛AstVORF2基因片段,该片段位于ORF2基因的1—630 bp处。从20份牦牛腹泻粪便样本中扩增目的片段并测序,应用MEGA5.1和MegAlign序列分析软件进行序列分析和遗传进化分析。
特异性检测牦牛AstV的RT-PCR的引物(P3、P4)设计根据上述序列分析的结果,选取保守序列设计特异性检测牦牛AstV的引物,引物信息见表1,扩增位于牦牛AstVORF2基因77—421 bp的长度为345 bp的目的片段,引物由大连宝生物工程有限公司合成。
表1三种RT-PCR方法的引物信息
Table 1Primer information for three kinds of RT-PCR assay
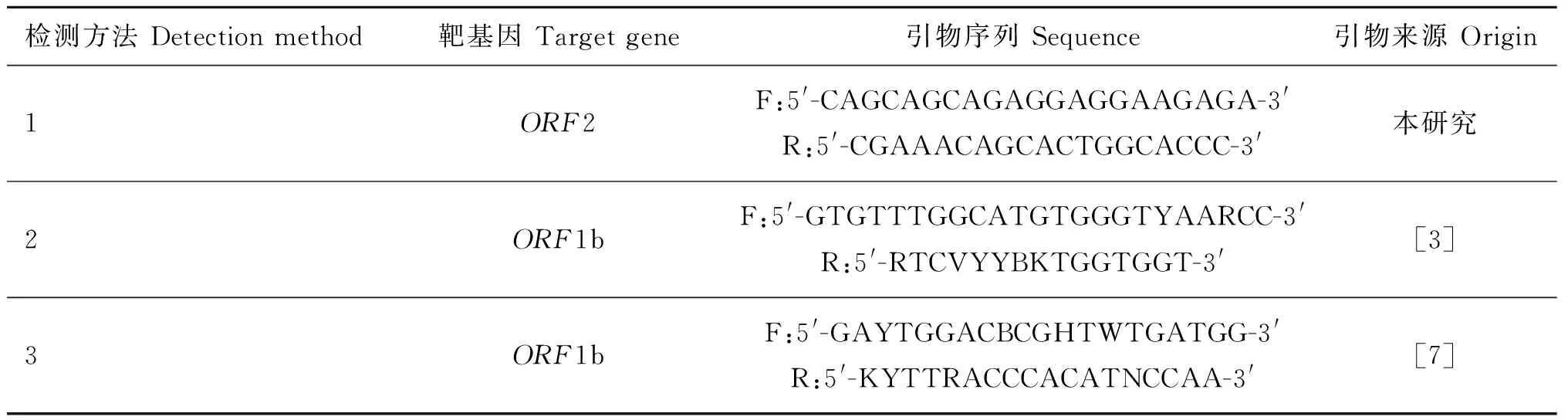
检测方法Detectionmethod靶基因Targetgene引物序列Sequence引物来源Origin1ORF2F:5'-CAGCAGCAGAGGAGGAAGAGA-3'R:5'-CGAAACAGCACTGGCACCC-3'本研究2ORF1bF:5'-GTGTTTGGCATGTGGGTYAARCC-3'R:5'-RTCVYYBKTGGTGGT-3'[3]3ORF1bF:5'-GAYTGGACBCGHTWTGATGG-3'R:5'-KYTTRACCCACATNCCAA-3'[7]
1.4核酸的提取与cDNA合成
将临床粪便样本与PBS(1∶5)充分重悬混匀,-80 ℃冰箱中反复冻融3次,3 000 r·min-1离心10 min,弃沉淀,再以12 000 r·min-1离心30 min,取上清,然后按照TRIzol Reagent说明书提取总RNA,并按照反转录试剂盒说明书合成cDNA,-20 ℃保存备用。细菌DNA采用酚—氯仿法提取,-20 ℃保存备用。
1.5牦牛AstV阳性标准品的制备
以牦牛AstV cDNA为模板,加入P3、P4引物扩增牦牛AstV的ORF2基因345 bp的片段,PCR产物经1.5% 琼脂糖凝胶电泳鉴定,用胶回收试剂盒回收目的片段,将其克隆至pMD19-T载体,并转化大肠杆菌DH5α感受态细胞,筛选出阳性克隆接入含氨苄青霉素的LB液体培养基中,37 ℃培养8 h,用质粒提取试剂盒提取重组质粒,送擎科生物有限公司测序。测序正确的阳性质粒作为AstV阳性标准品,核酸蛋白检测仪测定其浓度。
1.6反应体系及条件的优化
采用25 μL体系(预混酶12.5 μL,牦牛AstV阳性标准品2 μL,ddH2O补足至25 μL),对退火温度从50~60 ℃进行优化,再以优化的退火温度对引物浓度10 μmol·μL-1(0.1 ~1.0 μL)进行优化。
1.7敏感性测定
将牦牛AstV阳性标准品10倍递增稀释后作为模板(1×100、1×10-1~1×10-8),用建立的RT-PCR进行检测,确定其检测限。
1.8特异性评价
用建立的RT-PCR对“1.1”中特异性检测待检病原的核酸样本进行检测,并设立标准阳性样本和阴性对照,以评价该方法的特异性。
1.9稳定性评价
用建立的RT-PCR对3个阳性模板进行3次重复检测,以评价方法的稳定性。
1.10与现有检测BAstV的RT-PCR方法的比较
采用本试验中建立的RT-PCR方法和现有的2种检测BAstV的RT-PCR方法(表1)同时对54份牦牛腹泻粪便样本及80份肉牛腹泻样本进行检测,比较这三种方法的检出率。
1.11临床样本检测与统计学分析
采用建立的RT-PCR方法对上述137份腹泻粪便样本和26份健康粪便样本进行检测,随机选取10份阳性样本测序验证;分别统计,牦牛AstV在腹泻粪便样本和健康粪便样本的检出率,应用SPSS软件对检测结果产生的差异进行显著性分析。
2 结 果
从20份腹泻粪便样本中扩增出13个牦牛AstVORF2基因片段,长度为630 bp,毒株间的相似性为99.2%~100%,这一结果表明牦牛AstVORF2基因的1—630 bp区域高度保守,具备作为检测牦牛AstV 分子检测靶点的潜力。将扩增的牦牛AstVORF2基因片段与已报道的BAstV 毒株进行核苷酸同源性分析发现,牦牛AstVORF2与牛肠源BAstV毒株的相似性为59.5%~80.8%。牦牛AstV 与牛肠源和呼吸道源BAstV之间的遗传距离相对较近,但也存在明显的遗传距离,且所有牦牛AstV单独聚成一支(图1)。
2.2牦牛AstV检测引物的特异性
采用自行设计的引物对牦牛AstV核酸阳性样本进行PCR扩增,获得的目的片段大小与预期结果一致。测序结果证明目的片段长度为345 bp,为牦牛AstV 的特异性序列,与牦牛AstVORF2基因序列的相似性为100%。
2.3优化的RT-PCR反应体系及条件
优化的反应体系:预混酶12.5 μL,上下游引物P3、P4(10 μmol·L-1)各0.5 μL,cDNA 2 μL,ddH2O补足25 μL。反应条件:95 ℃预变性5 min;95 ℃变性30 s,56 ℃退火30 s,72 ℃延伸30 s,共35个循环;72 ℃延伸10 min。
2.4特异性
该方法能从牦牛AstV阳性核酸样本中检出目的基因片段,对BAstV阳性核酸样本无扩增,对其他无关病原不扩增。可见,本研究建立的RT-PCR只适合牦牛AstV的检测,而不能用于BAstV的检测。
我们在前面说过,实践标准的讨论具有启蒙的意义。我们可以从反思启蒙的角度来反思实践标准。在反思启蒙的理论中有两种启蒙辩证法值得我们重视。一个是黑格尔对于启蒙和信仰的分析,一个是霍克海默和阿多诺对于启蒙的分析。
2.5敏感性
牦牛AstV阳性标准品的浓度为57.3 ng·μL-1,敏感性检测结果显示,1×10-6稀释度阳性标准品仍可见目的条带,对AstV的核酸最低检测限为57.3 fg·μL-1。
2.6稳定性
对同一模板的3次重复检测结果一致,证明该方法具有良好的稳定性和重复性。
2.7三种RT-PCR检测方法的比较
三种检测方法的引物信息见表1。
检测方法1~3对54份牦牛腹泻粪便样本中牦牛AstV检测的阳性率分别为70.4%、0、29.6%,可见作者所建立的RT-PCR方法对牦牛AstV的检测效率明显优于现有的2种检测BAstV的RT-PCR方法。三种方法对80份肉牛腹泻粪便样本中BAstV检测的阳性率分别为0、0、8.7%,进一步证明本试验建立的RT-PCR方法只适用于牦牛AstV的检测。对方法2不能检出BAstV原因的分析发现,该文献报道的下游引物是错误的。
2.8临床样本检测
利用作者建立的RT-PCR方法从137份腹泻牦牛粪便和26份健康牦牛粪便中分别检出76份和2份阳性样本,对随机挑选10份阳性样本PCR产物测序结果证实为牦牛AstV特异片段。腹泻犊牦牛粪便样本中牦牛AstV的携带率为55.5%,显著高于健康犊牦牛粪便中的携带率7.7% (P<0.01),表明AstV感染可能与犊牦牛腹泻有联系。
3 讨 论
近年来,关于BAstV感染的流行病学及病原遗传变异的研究逐年增加[3,5,7-8]。作者最近利用二代测序技术首次从牦牛腹泻粪便样本中鉴定出AstV,是一种新型AstV[6],采用现有的两种检测BAstV的RT-PCR方法[3,7]对阳性样本却不能检出。因此,有必要建立检测牦牛AstV的RT-PCR方法。有资料表明,AstVORF2基因的前段区域在种内是高度保守的[9-11]。因此,为筛选分子检测靶点,作者对13株牦牛AstVORF2 1—630 bp的基因片段进行了克隆测序,结果显示该区域核苷酸序列在牦牛AstV种内也是高度保守的。基于该序列保守区域设计了检测引物,成功建立了检测牦牛AstV 的RT-PCR方法,具有特异性强、敏感度高、稳定性好的特点,为牦牛AstV的病原检测及病原流行病学调查提供了可靠手段。由于该方法是基于牦牛AstVORF2 基因保守序列建成的,而本试验获得的牦牛AstVORF2基因片段与BAstV 的核苷酸相似性仅为59.5%~80.8%,点突变很多。这可能是该方法不能对BAstV 阳性模板及牛腹泻粪便中BAstV阳性样本检出的原因。因此,本试验建立的RT-PCR方法只适用于牦牛AstV的检测。在后续研究中,可进一步研究建立检测牦牛AstV 和BAstV的双重RT-PCR及通用型RT-PCR,以满足不同研究的需要。
现有两种检测BAstV的RT-PCR方法对临床样本中牦牛AstV的检出率显著低于本研究建立的RT-PCR方法,分析其原因可能有两个:一是核苷酸序列变异。现有的检测BAstV的RT-PCR引物的靶基因都是ORF1b,而牦牛AstV的ORF1b的核苷酸序列与已知的BAstV的ORF1b的序列差异大,相似性仅为74.4%~87%,且核苷酸序列中存在很多点突变[6],这可能是主要原因;二是现有的两种PCR方法为解决BAstV的ORF1b核苷酸序列的点突变,均采用了简并引物[3,7],而简并引物会影响PCR检测的灵敏度[12]。实际上,作者用本研究建立的方法和文献[7]的方法检测均为阳性的样本,进行了灵敏度比较,结果前者的灵敏度较后者高100倍。
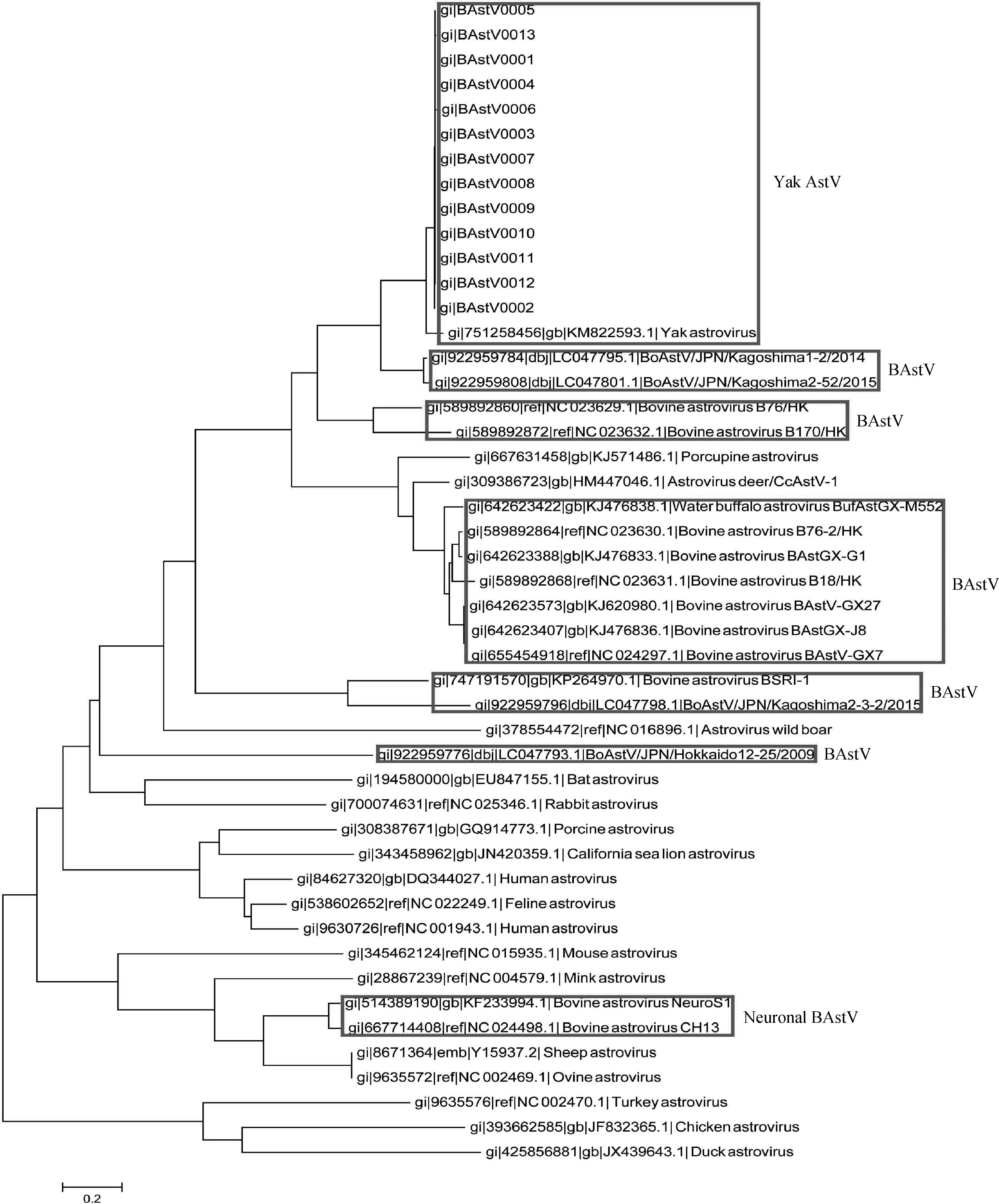
图1 牦牛AstV ORF2基因片段的邻近法进化树Fig.1 Neighbor-joining consensus tree for partial ORF2 gene fragment of yak AstV
J.K.Oem等和M.Candido等的流行病学调查表明,BAstV与犊牛腹泻有关[3,5],作者的检测结果也提示牦牛AstV可能与犊牦牛腹泻有联系。然而,C.P.Sharp等对新西兰牛的群体研究发现,BAstV在犊牛体内的检出率虽然高于成年牛,但认为BAstV并不是导致犊牛腹泻的主要病原[13]。鉴于牦牛AstV在犊牦牛腹泻粪便中有很高的检出率,有必要进一步确定其在牦牛腹泻的作用。
[1]WOODE G N,BRIDGER J C,MED J.Microbiol[M].1978(11):441-452.
[2]WOODE G N,POHLENZ J F,GOURLEY N E,et al.Astrovirus and Breda virus infections of dome cell epithelium of bovine ileum[J].JClinMicrobiol,1984,19(5):623-630.
[3]OEM J K,AN D J.Phylogenetic analysis of bovine astrovirus in Korean cattle[J].VirusGenes,2014,48(2):372-375.
[4]ALFRED N,LIU H,LI M L,et al.Molecular epidemiology and phylogenetic analysis of diverse bovine astroviruses associated with diarrhea in cattle and water buffalo calves in China[J].JVetMedSci,2015,77(6):643-651.
[5]CANDIDO M,ALENCAR A L,ALMEIDA-QUEIROZ S R,et al.Molecular detection and phylogenetic analysis of bovine astrovirus in Brazil[J].ArchVirol,2015,160(6):1519-1525.
[6]CHEN X,ZHANG B,YUE H,et al.A novel astrovirus species in the gut of yaks with diarrhea in the Qinghai Tibetan plateau,2013[J].JGenVirol,2015.doi:10.1099/jgv.0.000303.
[7]TSE H,CHAN W M,TSOI H W,et al.Rediscovery and genomic characterization of bovine astroviruses[J].JGenVirol,2011,92(Pt 8):1888-1898.
[8]NAGAI M,OMATSU T,AOKI H,et al.Full genome analysis of bovine astrovirus from fecal samples of cattle in Japan:identification of possible interspecies transmission of bovine astrovirus[J].ArchVirol,2015,160(10):2491-2501.
[9]JONASSEN C M,JONASSEN T O,SAIF Y M,et al.Comparison of capsid sequences from human and animal astroviruses[J].JGenVirol,2001,82(Pt 5):1061-1067.
[10]WANG Q H,KAKIZAWA J,WEN L Y,et al.Genetic analysis of the capsid region of astroviruses[J].JMedVirol,2001,64(3):245-255.
[11]WILLCOCKS M M,CARTER M J.Identification and sequence determination of the capsid protein gene of human astrovirus serotype 1[J].FEMSMicrobiolLett,1993,114(1):1-7.
[12]ISERTE J A,STEPHAN B I,GOI S E,et al.Family-specific degenerate primer design:A tool to design consensus degenerated oligonucleotides[J].BiotechnolResInt,2013,2013:383646.
[13]SHARP C P,GREGORY W F,MASON C,et al.High prevalence and diversity of bovine astroviruses in the faeces of healthy and diarrhoeic calves in South West Scotland[J].VetMicrobiol,2015,178(1-2):70-76.
(编辑白永平)
Development of an RT-PCR Assay and Variation Analysis for Yak Astrovirus
CHEN Xi,TANG Cheng,ZHANG Bin,SONG Zhi-gang,ZHOU Fang,YUE Hua*
(CollegeofLifeScienceandTechnology,SouthwestUniversityforNationalities,Chengdu610041,China)
Recently,our laboratory identified a novel astrovirus from fecal samples of diarrheic yak,the aim of this study was to develop an RT-PCR assay for detecting the novel yak astrovirus (yak AstV).Firstly,one pair primers were designed to amplifyORF2 gene fragments (630 bp in length) of yak AstV.By sequence analysis,one pair primers of RT-PCR assay were then designed for detecting yak AstV.We obtained 13ORF2 gene fragments from 20 fecal samples of diarrheic yak,which shared a homology of 99.3%-100% within fragments,but only a homology of 59.5%-80.8% with bovine astrovirus (BAstV).The RT-PCR assay developed in this study was specific to yak astrovirus,no amplifying for BAstV and other pathogens tested in this study.The detection limit of viral nucleic acid of the assay was 57.3 fg·μL-1.The assay was significantly better in detecting yak AstV compared to the other two reported assays for detecting bovine astrovirus.The detecting results of clinical samples by this assay showed that the positive rates of yak AstV were 55.5% (76/137) in diarrheic fecal samples and 7.7% (2/26) in healthy fecal samples.Compared to healthy samples,yak AstV was significantly more prevalent in diarrheic fecal samples (P<0.01).In conclusion,ORF2 gene sequences of yak AstV cloned in this study varied a lot compared with those of BAstV,but were highly conserved within yak AstV species.Thus,the RT-PCR for detecting yak AstV based on theORF2 sequences of yak AstV greatly improved the detection rate of yak AstV,providing a useful tool for detection of this novel virus and epidemiological investigation.Meanwhile,the results of this study indicated that yak AstV might be associated with diarrhea in yak calves.
yak;astrovirus;RT-PCR assay;ORF2 gene;genetic variation
10.11843/j.issn.0366-6964.2016.06.027
2015-10-30
“十二五”高技术研究发展计划(863计划)课题(2012AA101304);四川省科技计划项目青年基金(2014JQ0044);四川省教育厅创新团队(13TD0057);西南民族大学研究生创新型科研项目(CX2015SZ067)
陈曦(1990-),女,河南睢县人,硕士生,主要从事动物病原分子生物学研究,E-mail: 792875776@qq.com
岳华,博士,教授,Tel: +86-28-85528276, Fax: +86-28-85522727,E-mail: yhua900@163.com
S852.653
A
0366-6964(2016)06-1287-06
