新疆石榴种质资源遗传多样性的SRAP分析
2016-07-04王继莲丁晓丽马刘峰
陈 芸,王继莲,丁晓丽,马刘峰*
(1 喀什大学 生命与地理科学学院,新疆喀什844006;2 叶尔羌绿洲生态与生物资源研究自治区重点实验室,新疆喀什844006)
新疆石榴种质资源遗传多样性的SRAP分析
陈芸1,2,王继莲1,2,丁晓丽1,马刘峰1,2*
(1 喀什大学 生命与地理科学学院,新疆喀什844006;2 叶尔羌绿洲生态与生物资源研究自治区重点实验室,新疆喀什844006)
摘要:采用SRAP分子标记技术,对21份新疆石榴品种的遗传多样性与亲缘关系进行分析。从36对引物组合中筛选出16对扩增条带清晰、多态性高的引物组合分析供试材料,共检测出271个位点,其中194个为多态性位点,多态性比率达71.59% ,平均每对引物组合产生16.94个位点和12.13个多态性位点。21份石榴样品间的相似性系数为0.63~0.89,平均为0.77。UPGMA聚类结果显示,在相似性系数为0.776时,21份材料可被分为5个类群。新疆石榴品种间平均观察等位基因数(Na)、有效等位基因数(Ne)、Nei’s基因多样性指数(H)及Shannon’s 信息指数(I)分别为1.712 2、1.352 8、0.199 9及0.310 5。研究表明,新疆喀什地区石榴品种的遗传多样性最为丰富;SRAP聚类结果与石榴的表型特征存在一致性,且SRAP分子标记技术是研究石榴遗传多样性简单而有效的手段。
关键词:石榴;SRAP;遗传多样性
石榴(PunicagranatumL.)为石榴科(安石榴科Punicaceae)石榴属(PunicaL.)落叶灌木或小乔木,原产伊朗、阿富汗及印度西北部地区[1]。石榴是一种药、食两用的珍贵水果,营养价值极高。近年来,随着人们对石榴活性成分及药理作用研究的不断深入,石榴抗氧化、降血脂、降血糖、预防心脑血管疾病的功效广泛报道。石榴受到广大消费者的喜爱,市场需求日益增加。
新疆的南疆地区是石榴适生栽培区,这里很可能是中国石榴的原生或次生起源中心[2]。然而目前新疆石榴分类仍比较混乱,往往仅依据果实风味进行分类,存在同种异名、同名异种现象。国内应用分子标记技术分析石榴遗传多样性已有一些报道,而用分子标记技术研究新疆石榴遗传多样性的相关报道极少,仅涉及RAPD[3]和AFLP[4]2种标记技术,且只涉及新疆少数石榴品种。阐明新疆石榴基因型之间的亲缘关系和遗传多样性,是新疆石榴遗传育种及标准化栽培的基础,是十分重要和必要的。SRAP(sequence-related amplified polymorphism)即相关序列扩增多态性是一种基于PCR的分子标记技术[5],该标记具有多态性高、简便、快速、稳定、成本低等特点[6],已被广泛应用于红花[7]、柿子[8]、丹参[9]、花椒[10]等植物的遗传多样性分析。因此,采用分子标记技术SRAP分析新疆石榴地方品种的亲缘关系和遗传多样性,将为新疆石榴的分类、起源演化、种质资源引进和利用,分子标记辅助选择育种等提供科学依据。
1材料和方法
1.1材料
实验材料均为新疆石榴,包括了新疆石榴主栽品种和收集到的一些地方品种。具体采样地点、材料名称及编号见表1。从各石榴品种单株上采集健康的叶片数枚。近的采集地点,将新鲜叶片迅速放入自封袋中,置于车载冰箱内,带回实验室,储存于- 70℃冰箱备用。远的采集地点,将叶片放入提前装有2/3左右变色硅胶的自封袋中,并保存少量空气,密封。每天注意观察变色硅胶的颜色,及时更换变色硅胶,带回实验室,常温保存,备用。
1.2方法
1.2.1样品基因组DNA提取与SRAP-PCR扩增基因组DNA提取参考改良CTAB法[11],因石榴成熟叶片杂质多,略加改进。改进处如下:液氮磨样前加入4%的PVP和0.02 g抗坏血酸;以硅胶干燥叶片为材料时,提取缓冲液体积与材料质量之比增至原来的2倍;异丙醇沉淀后,增加1次酚:氯仿:异戊醇抽提。提取后的基因组DNA采用1%琼脂糖凝胶电泳检测,将各样本基因组DNA用双蒸水稀释至约50 ng/μL,-20 ℃保存。SRAP扩增引物参照Li等[5]、Mohammad等[12]发表的引物序列(表2),由上海生工合成。Taq酶和dNTP购自北京天根生物技术公司。扩增体系优化后确定为:反应体系总体积25 μL,含DNA 75~100 ng, Mg2+2 mmol/L,dNTPs 0.5 mmol/L,上、下游引物各0.4 μmol/L,Tap DNA聚合酶2U,10×PCR Buffer 2.5 μL,补足超纯水至25 μL。参照Li等[5]的扩增程序:94 ℃预变性5 min;94 ℃变性1 min;35 ℃复性1 min,72 ℃延伸1 min,5个循环;94 ℃变性1 min,50 ℃复性1 min,72 ℃延伸1 min,35个循环;72 ℃延伸10 min。4 ℃保存。
1.2.2SRAP扩增产物检测取8 μL扩增产物与2 μL载样缓冲液混匀,用8%非变性聚丙烯酰胺凝胶电泳检测,电泳缓冲液为1×TBE,220 V恒压电泳2~3 h。电泳结束后,银染法染色,数码相机拍照保存。
1.2.3数据分析电泳图谱上同一位置有清晰条带的记为“1”,无条带记为“0”,个别扩增效果不理想导致条带不够清晰或缺失记为“2”,形成数据矩阵。采用NTSYS-pc2.10e软件进行聚类分析。对数据矩阵用SimQual程序求得Jaccard相似系数,获得相似系数矩阵。继而采用SHAN程序中的UPGMA (unweighted pair-group method using arithmetic averages)方法聚类分析,通过Treeplot模块生成聚类图。用PopGen Version 1.3l软件计算Nei’s genetic identity、genetic distance(GD)、观察等位基因数(Na)、有效等位基因数(Ne)、Nei’s基因多样性指数(H)及Shannon’s 信息指数(I)。

表1 21个石榴品种的名称和来源
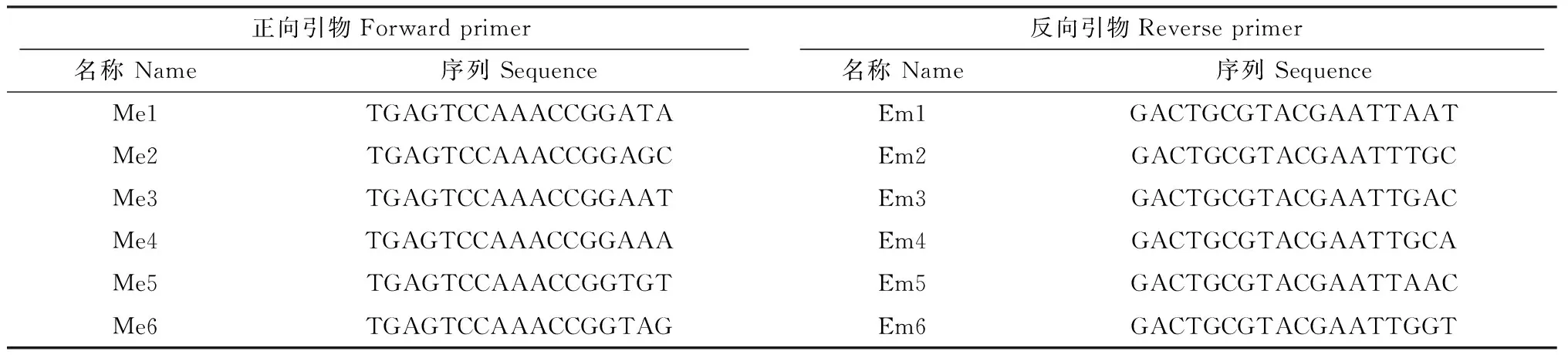
表2 采用的SRAP标记的引物序列
2结果与分析
2.1SRAP扩增结果
以甜石榴(Ks1)、赛柠檬(Cl2)和小叶观赏石榴(Hm3)3个地理位置相差较远、表型特征差异较大品种的基因组DNA为模板,采用36对引物组合进行PCR扩增。筛选出16对扩增条带丰富、多态性高的引物组合,对所有石榴样本进行遗传多样性分析。扩增结果(表3)显示,16对引物组合在21个样本中共扩增得到271个位点,其中194个位点为多态性位点,多态比率为71.59%。平均每对引物可扩增得到16.94个位点, 其中12.13个位点有多态性。SRAP引物在石榴样本中扩增得到的DNA 片段多数集中于100~2 000 bp之间。代表性图谱如图1。扩增结果表明SRAP标记能检测出较多的遗传位点,能获得多态性较好的PCR结果。
2.2遗传相似性分析
根据扩增所得电泳条带,用NTSYS-pc2.10e软件对21份石榴品种进行遗传相似性分析,通过SimQual程序求得Jaccard相似系数,得到各样品间遗传相似系数(GS)矩阵。结果表明,21份石榴样品间的相似系数为0.63~0.89,平均为0.77,变化幅度不大。其中,来自和田皮山县皮亚勒玛乡的皮亚曼二号与来自喀什市阿瓦提乡的甜石榴间的相似系数最小为0.6347,说明二者的亲缘关系最远;来自喀什市阿瓦提乡的甜石榴与来自喀什市佰什克然木乡的酸甜石榴之间的相似系数最大达到0.8856,说明二者的亲缘关系最近。

表3 SRAP引物组合扩增石榴基因组DNA的多态位点数

M.DL2000;1~21依次为21份石榴样品,顺序同表1图1 引物组合me6me6的扩增效果M.DL2000; 1-21. Pomegranate samples, the order is same as Table 1Fig. 1 PCR amplified patterns by primer me6em6
2.3新疆石榴种质资源的聚类分析
根据SRAP标记电泳图谱,用NTSYS-pc2.10e软件对21份石榴材料进行聚类分析,结果如图2。21个石榴品种首先分成了两大类,Ps2和Ps3单独聚在了一起,其他品种聚在了一起,说明Ps2和Ps3与其它品种间亲缘关系较远。此2种石榴均为广受消费者青睐的鲜食品种,表型也很相似,都具有茎刺多、花色红、单瓣花、果皮红、籽粒大而深红色等特点,果实的萼片形态也相似。只是在同样的栽培条件下,相同树龄的石榴树上Ps2的果实更大,平均单果重也更高。Ps2和Ps3聚在一起证实了二者在分子水平上具有相似的遗传基础。在遗传相似系数为0.776时,21个石榴品种可分为Ⅴ个类群。第Ⅰ类群为来自和田皮山县皮亚勒玛乡的Ps2;第Ⅱ类群为Ps3;第Ⅲ类群共12种,包括了喀什地区的大部分品种:来自喀什市的Ks2、Ks3、Ks4、Ks5,来自喀什地区叶城县的Yc1、Yc2、Yc3、Yc4、Yc5。另外,来自和田地区的Ps4、Cl2和来自哈密的Hm1也聚在了此类群中。这一类群的石榴均为红花、单瓣、红皮、硬籽儿的鲜食石榴。第Ⅳ类群共2种,来自和田地区的Ps1、Cl1聚在了一起。这2个品种也是红花单瓣的鲜食品种,但具有区别于其他品种的共同特点:果皮色泽暗红并且无茎刺。第Ⅴ类群共5种,样品中所有的观赏石榴(Ats1、Ks6、Hm2、Hm3)均聚在了此类群,来自喀什的甜石榴品种Ks1与这几种观赏石榴聚在了一起。从聚类图看, Ats1、Ks6与Ks1的亲缘关系较近。Ats1和Ks6是红花、单瓣的赏食兼用石榴,Ks1是红花、单瓣、红皮鲜食石榴。Hm2和Hm3为重瓣观赏石榴,亲缘关系较近,聚在一起。

样品编号同表1图2 基于SRAP标记产生的石榴聚类图Number of samples are the same as Table 1Fig. 2 Dendrogram of 21 pomegranate accessions based on SRAP
2.4新疆不同地区石榴SRAP遗传多样性
依据各石榴样品所分布的地理位置,将21份材料分为3组。其中来自喀什地区的11个品种和阿图什市的1个品种合并为第一组;来自和田地区的6个品种编为第二组;来自哈密三道岭的3个品种编为第三组;用PopGen Version 1.3l软件计算各生态区域石榴的遗传多样性指数,结果如表4。由表4数据可知,3个地区石榴的观察等位基因数(Na)、Nei’s基因多样性指数(h)、Shannon's 信息指数(I)都以喀什地区为最高;和田地区其次;哈密三道岭地区最低。说明参试品种中新疆喀什石榴遗传多样性最为丰富。这可能也与新疆石榴主要分布于南疆,北疆地区很少,故而哈密地区采集到的样本数有限有关。

表4 不同地区石榴SRAP标记的遗传多样性指数
3讨论
3.1SRAP标记遗传多样性适用性分析
许多学者研究表明,SRAP标记是植物遗传多样性研究的理想工具。该标记能将遗传关系很近的品种区分开来[13],与AFLP标记相比,它所提供的信息更接近于农艺性状的差异和历史演变结果[14]。本试验结果表明,SRAP标记稳定性和重复性好,扩增条带清晰、丰富,可以在石榴中产生多态性。实验中采用的16对引物共扩增得到271个位点,其中194个位点为多态性位点,多态比率为71.59%。平均每对引物可扩增得到16.94个位点, 其中12.13个位点有多态性。单对引物扩增位点数高于前人用SRAP标记对柿属[15]、葱[16]、青稞[17]的研究,但低于该标记对国兰[18]和玫瑰[19]的研究。其原因可能与参试的石榴品种数量有限有关。本研究中,新疆的21个品种多态性位点百分率以及平均每对引物扩增位点数均高于热娜·卡司木等[3]用RAPD标记对新疆石榴亲缘关系的研究所得的结果。多态性位点百分率也高于赵丽华等[20]用AFLP对42份川滇石榴品种遗传多样性的研究结果(65.66%)。这一结果充分表明了SRAP标记扩增效果理想、多态性高,是遗传多样性分析的理想工具。同时,本研究多态性位点百分率低于马丽等[21]采用ISSR标记对82个石榴品种遗传多样性的分析结果以及苑兆和等[4]利用荧光标记AFLP对中国石榴群体遗传多样分析的结果。表明石榴在新疆虽然经过了多年的人工选育及基因突变的积累,产生了较丰富的变异,形成了稳定而丰富的资源库。然而,新疆石榴毕竟品种有限,且多为红皮石榴,表型比较相似,与中国其他区域及国外丰富的石榴遗传资源相比,遗传多样性偏低。近年来,新疆石榴产业化发展,一些具有市场竞争力的石榴品种(籽粒大的、甜的、皮色鲜艳)规模化种植,而许多流传下来零星分布的地方品种消失了,丢失了许多珍贵的石榴资源。为了进一步丰富新疆石榴资源基因库,建议一方面建立新疆种植资源圃,保护现有的石榴资源;另一方面可以从中国其他地区和国外引进一些品质好的石榴品种。
3.2新疆石榴聚类分析
本研究中,SRAP标记对21份石榴样品的聚类结果表现出如下几个特点:第一,聚类结果与石榴的部分表型特征具有一致性。果皮色泽暗红并且无茎刺的Ps1、Cl1聚在一起。供试样品中4个观赏石榴品种(Ats1、Ks6、Hm2、Hm3)聚在一组,单瓣的Ats1、Ks6与Ks1聚在一起,重瓣的Hm2和Hm3聚在一起。证实果皮颜色、花瓣轮数、茎刺有无等特征可作为石榴的分类依据。第二,聚类结果与样品的地理位置分布有相关性。地理位置近的品种往往聚在一起。如喀什地区的样品,除了观赏石榴(Ks6)和甜石榴(Ks1)外,其余9种均聚在第Ⅲ类群,来自和田地区的Ps1、Cl1聚在了一起构成了第Ⅳ类群。第三,聚类结果与样品果实风味分类结果不一致。酸石榴和甜石榴聚在一起。这与赵丽华等[20]的研究结果一致。证明石榴果实风味不是石榴分类的主要依据。由于石榴属于小杂果,长期以来并未得到足够重视,分类较混乱,还没有建立统一的石榴品种分类方法和分类体系。新疆石榴往往仅依据果实风味结合地名进行命名,如叶城酸石榴、喀什甜石榴。汪小飞等[22]对石榴分类的研究表明,在石榴形态特征中,花瓣、花色、结实性、果色、果实大小及果重是品种分类的主要依据,而籽粒软硬、萼筒形状、新叶颜色、茎刺有无等特征可作为次级分类依据。上述分类特征中,并未提及果实风味。
根据本研究结果,在石榴育种实践中可选用地理位置较远、亲缘关系也较远的品种杂交,以实现新疆石榴品质的遗传改良。
参考文献:
[1]刘文江.新疆石榴资源及其开发利用[J].干旱区研究,2007,24(2):219-221.
LIU W J. Resources ofPunicagranatumLinn .and its exploitation and utilization in Xinjiang[J].AridZoneResearch, 2007,24(2):219-221.
[2]尹燕雷,任建辉,何天明,等.新疆的石榴栽培状况[J].落叶果树,2006,6:15-16.
YIN Y L, REN J H, HE T M,etal. Cultivated situation of pomegranate in Xinjiang[J].DeciduousFruits, 2006,6:15-16.
[3]热娜·卡司木, 帕丽达·阿不力孜,朱焱.新疆石榴品种的RAPD分析[J].西北植物学报,2008,28(12):2 447-2 450.
RENA K, PALIDA A, ZHU Y. RAPD analysis ofPunicagranatumL.cultivars in Xinjiang[J].ActaBot.Boreal.-Occident.Sin., 2008,28(12):2 447-2 450.
[4]苑兆和,尹燕雷,曲健禄,等.中国石榴栽培群体遗传多样性的荧光AFLP分析[J].遗传学报,2007,34(12):1 061-1 071.
YUAN Z H, YIN Y L, QU J L,etal. Population genetic diversity in Chinese pomegranate(PunicagranatumL.)cultivars revealed by fluorescent—AFLP markers[J].JournalofGeneticsandGenomics, 2007,34(12):1 061-1 071.
[5]LI G, QUIROS C F. Sequence-related amplified polymorphism(SRAP),a new marker system based on a simple PCR reaction:its application to mapping and gene tagging inBrassica[J].TheoreticalandAppliedGenetics, 2001,103:455-461.
[6]林忠旭,张献龙,聂以春.新型标记SRAP在棉花F2分离群体及遗传多样性评价中的适用性分析[J].遗传学报,2004,31(6): 622-626.
LIN Z X, ZHANG X L, NIE Y C. Evaluation of application of a new molecular marker SRAP on analysis of F2segregation and genetic diversity in China[J].ActaGeneticaSinica, 2004,31(6):622-626.
[7]MOKHTARI N, RAHIMMALEK M, TALEBI M,etal. Assessment of genetic diversity among and withinCarthamusspecies using sequence-related amplified polymorphism (SRAP) markers[J].PlantSystematicsandEvolution, 2013, 299:1 285-1 294.
[8]JING Z B, RUAN X F, WANG R,etal. Genetic diversity and relationships between and within persimmon (DiospyrosL.) wild species and cultivated varieties by SRAP markers[J].PlantSystematicsandEvolution, 2013, 299:1 485-1 492.
[9]SONG Z Q, LI X F, WANG H G,etal. Genetic diversity and population structure ofSalviamiltiorrhizaBge in China revealed by ISSR and SRAP[J].Genetica, 2010, 138:241-249.
[10]FENG S J, YANG T X, LIU Z S,etal. Genetic diversity and relationships of wild and cultivatedZanthoxylumgermplasms based on sequence-related amplified polymorphism (SRAP) markers[J].GeneticResourcesandCropEvolution, 2015, 62:1 193-1 204.
[11]郭凌飞,邹明宏,曾辉.利用改良CTAB法提取澳洲坚果成熟叶片高质量DNA[J].分子植物育种,2007,5(6):187-190.
GUO L F ,ZOU M H, ZENG H,etal. High quality DNA extraction from mature leaves of macadamia by modifled CTAB protocols[J].MolecularPlantBreeding, 2007, 5(6):187-190.
[12]BADRALDIN EBRAHIM SAYED-TABATABAEI MM. Use of SRAP markers to assess genetic diversity and population structure of wild, cultivated, and ornamental pomegranates(PunicagranatumL.) in different regions of Iran[J].PlantSystematicsandEvolution, 2012, 298:1 141-1 149.
[13]AHMAD R,POTTER D,SOUTHWICK S M.Genotyping of peach and nectarine cultivars with SSR and SRAP molecular marker[J].JournalofAmericanSocietyforHorticulturalScience, 2004, 129(2):204-210.
[15]韩振诚,潘学军,安华明,等.贵州柿属植物种质资源遗传多样性的SRAP分析[J].果树学报,2015,32(5):751-762.
HAN Z C, PAN X J, AN H M,etal. Genetic diversity ofDiospyrosLinn. in Guizhou based on SRAP[J].JournalofFruitScience, 2015, 32(5): 751-762.
[16]李慧芝,尹燕枰,张春庆,等.SRAP在葱栽培品种遗传多样性研究中的适用性分析[J].园艺学报,2007,34(4):929-934.
LI H Z, YIN Y P, ZHANG C Q,etal. Evaluation of Application of SRAP on analysis of genetic diversity in cultivars ofAlliumfistulosumL.[J].ActaHorticuhuraeSinica, 2007, 34(4):929-934.
[17]杨平,刘仙俊,刘新春,等.利用SRAP标记研究四川高原青稞育成品种的遗传多样性[J].遗传, 2008, 30(1):115-122.
YANG P, LIU X J, LIU X C,etal. Genetic diversity analysis of the developed qingke (hulless barley) varieties from the plateau regions of Sichuan Province in China revealed by SRAP markers[J].Hereditas(Beijing), 2008, 30(1):115-122.
[18]唐源江,曹雯静,吴坤林.基于SRAP 标记的国兰种质资源遗传多样性分析及分子身份证构建[J].中国农业科学,2015,48(9):1 795-1 806.
TANG Y J, CAO W J, WU K L. Genetic diversity analysis and molecular identification card construction of ChineseCymbidiumgermplasms based on SRAP Markers[J].ScientiaAgriculturaSinica, 2015, 48(9):1 795-1 806.
[19]徐宗大,赵兰勇,张玲,杨志莹.玫瑰SRAP 遗传多样性分析与品种指纹图谱构建[J].中国农业科学, 2011,44(8):1 662-1 669.
XU Z D, ZHAO L Y, ZHANG L,etal. Analysis of genetic diversity and construction of fingerprint ofRosarugosaby SRAP[J].ScientiaAgriculturaSinica, 2011, 44(8):1 662-1 669.
[20]赵丽华,李名扬,王先磊.川滇石榴品种遗传多样性及亲缘关系的AFLP分析[J].林业科学, 2010, 46(11):168-173.
ZHAO L H, LI M Y, WANG X L. Genetic diversity and genetic relationship of pomegranate(Punicagranatum)in Sichuan and Yunnan evaluated by AFLP markers[J].ScientiaSilvaeSinicae, 2010, 46(11):168-173.
[21]马丽,侯乐峰,郝兆祥,等.82个石榴品种遗传多样性的ISSR分析[J].果树学报,2015,32(5):741-750.
MA L, HOU L F, HAO Z X,etal. Genetic diversity analysis of 82 pomegranate(PunicagranatumL.) cultivars by ISSR markers[J].JournalofFruitScience, 2015, 32(5):741-750.
[22]汪小飞,周耘峰,黄埔,等.石榴品种数量分类研究[J].中国农业科学,2010,43(5):1 093-1 098.
WANG X F, ZHOU Y F, HUANG P,etal. Study on the classification on pomegranate cultivars by numerical taxonomy[J].ScientiaAgriculturaSinica, 2010, 43(5):1 093-1 098.
(编辑:宋亚珍)
Genetic Diversity of Germplasm Collection of Pomegranate in Xinjiang Using SRAP Markers
CHEN Yun1,2, WANG Jilian1,2, DING Xiaoli1, MA Liufeng1,2*
(1 College of Biology and Geography Science, Kashgar University, Kashgar, Xinjiang, 844006,China ;2 The Key Laboratory of Ecology and Biological Resource in Yarkant Oasis at Colleges & Universities under the Department of Education of Xinjiang Uygur Autonomous Region, Kashgar, Xinjiang, 844006, China)
Abstract:Sequence-related amplified polymorphism technique was adopted to analyze 21 pomegranate accessions. Sixteen primer combinations with clear band patterns and polymorphism were selected from thirty-six primer combinations. 271 loci were detected by sixteen pairs of SRAP primers. Among them 194 were polymorphic, the polymorphic rate was 71.59%, an average of 16.94 loci and 12.13 polymorphic loci were amplified by each primer pair. The genetic similarity coefficient of 21 pomegranate accessions ranged from 0.63 to 0.89, with an average of 0.77. An unweighted pair group method with arithmetic mean (UPGMA) tree established from Jaccard similarity coefficients suggested that 21 pomegranate accessions were clustered into five subgroups. The average value of observed number of alleles, effective number of alleles, Nei’s gene diversity index, Shannon’s information index are 1.712 2, 1.352 8, 0.199 9 and 0.310 5, respectively. There was rich genetic diversity among the pomegranate accessions in Kashi Prefecture. The clustering results using SRAP markers were consistent with the phenotypic characteristics of pomegranate. Sequence-related amplified polymorphism (SRAP) is a simple and effective method to research on genetic diversity of pomegranate.
Key words:Punica granatum L.; SRAP; genetic diversity
文章编号:1000-4025(2016)05-0916-07
doi:10.7606/j.issn.1000-4025.2016.05.0916
收稿日期:2016-02-12;修改稿收到日期:2016-04-21
基金项目:新疆维吾尔自治区高校科研计划(XJEDU2013S35)
作者简介:陈芸(1980-),硕士,讲师,主要从事植物分子遗传研究。E-mail:chenyun8111@126.com。 *通信作者:马刘峰,博士,副教授,主要从事植物基因工程研究。E-mail:maliufeng@126.com。
中图分类号:Q346+.5;Q789
文献标志码:A
