福氏志贺菌2a型外膜蛋白A的生物信息学分析与重组表位疫苗分子的设计
2016-06-29刘祥
刘 祥
(陕西理工学院 生物科学与工程学院, 汉中 723001)
福氏志贺菌2a型外膜蛋白A的生物信息学分析与重组表位疫苗分子的设计
刘 祥
(陕西理工学院 生物科学与工程学院, 汉中 723001)
摘要福氏志贺菌外膜蛋白OmpA(outer membrane protein A)具有较强的免疫原性,在疫苗上有应用前景。采用MEGA(molecular evolutionary genetics analysis)软件对OmpA的系统发生分析显示,福氏志贺菌与肠道细菌间亲缘关系较其它菌属更近。利用在线软件预测OmpA为亲水的分泌型蛋白,存在多个酶切位点;采用TMHMM(transmembrane hidden markov models)Server v.2.0程序预测OmpA无跨膜结构并定位于细胞膜外;SOPMA(self-optimized prediction method with alignment)服务器预测OmpA二级结构中含无规则卷曲41.09 %,α-螺旋26.44 %,β-转角10.06%,β-片层22.41 %。通过SignalP 4.1软件分析显示,OmpA的1~21位氨基酸为信号肽序列。采用Swiss-Model程序预测的三维模型显示OmpA为桶装结构。利用ABCpred(artificial neural network based B-cell epitope prediction)和BepiPred(B-cell epitopes prediction)方案,预测OmpA存在3个B细胞表位。运用神经网络与量化矩阵法预测OmpA具有1个CTL表位。使用MHC-Ⅱ类分子结合肽在线程序预测显示OmpA具有1个Th表位。设计获得抗原性较好的OmpA蛋白重组表位多肽。为OmpA多表位串联疫苗的研究奠定基础。
关键词福氏志贺菌;OmpA蛋白;细胞表位;蛋白结构
福氏志贺菌(Shigellaflexneri)为革兰阴性菌,可引起人类的细菌性痢疾,是一种高危传染性肠道致病菌,给人类健康带来影响[1,2]。该菌导致的疾病在发展中国家流行,并以儿童为主要患病群体[3]。然而随着抗生素的广泛使用,福氏志贺菌耐药性菌株不断产生[4],给该菌的治疗感染带来一定的难度。需要研发一种安全、无耐药性的新型疫苗。外膜蛋白A(Outer membrane protein A, OmpA)为革兰阴性菌主要外膜蛋白[5],是福氏志贺菌主要毒力蛋白之一,可增强福氏志贺菌对宿主的粘附与感染作用[6]。小鼠的免疫保护试验发现,OmpA蛋白可激活小鼠体内的细胞免疫和体液免疫,实现对福氏志贺菌感染的有效抵抗[7,8]。因而,OmpA是一种很好的蛋白亚单位候选疫苗[9]。
我们前期已经对OmpA蛋白进行了原核表达与发酵条件研究[6, 10]。而对OmpA蛋白性质缺乏了解,给疫苗的研制带来一些困难。本研究借助于生物信息学方法,采用多参数预测的方式,对OmpA蛋白的系统进化关系、理化性质、信号肽、跨膜结构、高级结构,以及优势的B/T细胞表位进行预测,并设计OmpA蛋白的重组表位疫苗分子。为OmpA蛋白疫苗的研制,尤其是多表位串联疫苗的研究奠定基础。
1材料与方法
1.1材料
依据GenBank公布的福氏志贺菌2a型外膜蛋白OmpA序列,登录号为NP_706879.2,全长共348个氨基酸。序列如下:
MKKTAIAIAVALAGFATVAQAAPKDNTWYTGAKLGWSQYHDTGFIPNNGPTHENQLGAGAFGGYQVNPYVGFEMGYDWLGRMPYKGDNINGAYKAQGVQLTAKLGYPITDDLDIYTRLGGMVWRADTKANVPGGASFKDHDTGVSPVFAGGVEYAITPEIATRLEYQWTNNIGDANTIGTRPDNGLLSLGVSYRFGQGEAAPVVAPAPAPEVQTKHFTLKSDVLFNFNKATLKPEGQAALDQLYSQLSNLDPKDGSVVVLGYTDRIGSDAYNQGLSERRAQSVVDYLISKGIPADKISARGMGESNPVTGNTCDNVKQRAALIDCLAPDRRVEIEVKGIKDVVTQPQA。
1.2方法
1.2.1OmpA生物信息学分析
利用MEGA软件对NCBI已公布的OmpA蛋白序列氨基酸进行系统发生分析。通过生物信息学相关预测网站和软件,分析OmpA蛋白的主要理化性质。OmpA蛋白生化指标预测网站:http://web.expasy.org/cgi-bin/protparam/protparam,酶切位点预测网站:http://web.expasy.org/peptide_cutter/, 亲水性预测网站:http://web.expasy.org/protscale/,软件Signal P4.1预测信号肽,软件TMHMM Serverv.2.0预测蛋白跨膜结构,SOPMA服务器在线预测二级结构,三维结构采用Swiss-Model预测方法,B细胞抗原表位预测采用在线服务ABCpred与BepiPred方法,细胞毒性T细胞(CTL)抗原表位预测通过CTLpred在线服务,辅助T细胞(Th)抗原表位预测网站:http://www.imtech.res.in/raghava/propred/。
1.2.2OmpA重组表位疫苗的设计
将获得的B/T细胞表位,通过拼接,每个表位之间以甘氨酸-甘氨酸-甘氨酸-甘氨酸(GGGG)短肽连接。采用DNAStar Protean软件,分析多表位的各种排列方式,以各表位相对独立且具有较好抗原性参数的连接方式,作为多表位疫苗的氨基酸设计序列,再翻译为核酸序列,最终获得重组表位疫苗基因序列。
2结果与分析
2.1OmpA蛋白序列系统发生分析
通过MEGA软件对NCBI已公布的OmpA蛋白序列进行系统发生分析。结果(图1)显示:同一菌属的亲缘关系更近;衣原体与其它细菌的亲缘关系较远;福氏志贺菌与肠道细菌间亲缘关系较其它菌属更近,表明OmpA蛋白对不同种的肠道致病菌可能存在交叉免疫保护作用。
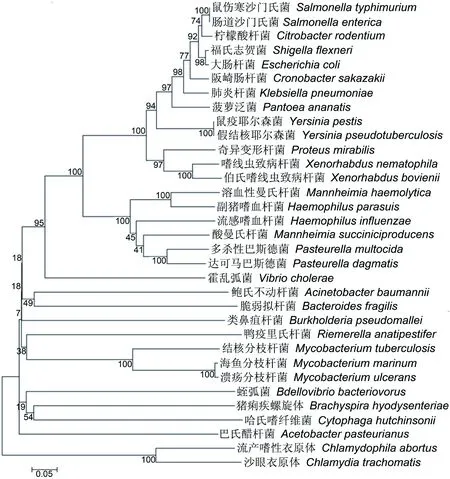
图1 MEGA软件构建的OmpA氨基酸序列进化树
2.2OmpA蛋白生化指标预测
将OmpA蛋白序列提交至网站http://web.expasy.org/protparam/,结果显示,总平均疏水性指数 (Grand average of hydropathicity, GRAVY) 为-0.318,表明OmpA蛋白为亲水性蛋白。OmpA分子量为37.283 ku,等电点为5.65,脂溶性指数(Aliphatic index)为78.53。利用在线网站: http://www.expasy.org/protscale/预测OmpA的氨基酸中疏水性最强的分值为2.8,最弱为-2.56(图2)。将OmpA氨基酸序列提交至网站:http://web.expasy.org/peptide_cutter/分析其酶切位点,结果显示OmpA蛋白存在多个酶切位点,其中蛋白酶 K(Proteinase K)有171个,嗜热菌蛋白酶(Thermolysin)97个,胃蛋白酶(Pepsin)(pH值>2)92个,低特异性糜蛋白酶(Chymotrypsin-low specificity)60个。
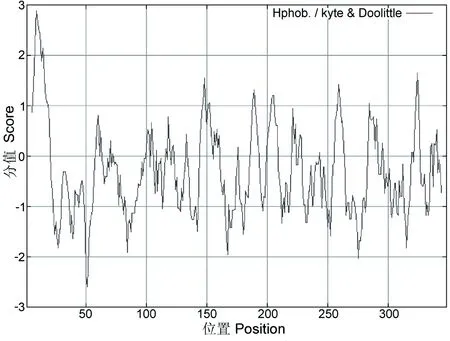
图2 福氏志贺菌OmpA蛋白疏水性分析
2.3OmpA蛋白信号肽与跨膜结构预测
将OmpA氨基酸序列利用软件Signalp 4.1分析,结果显示OmpA蛋白在1~21位氨基酸可能为信号肽序列(图3)。根据在线服务软件 TMHMM Serverv.2.0程序预测显示,OmpA蛋白在1~22位氨基酸可能为跨膜结构,但可信度仅有0.45(图4);而且1~21位氨基酸预测为信号肽,蛋白成熟时该多肽被切除。可见,OmpA蛋白没有跨膜结构,位于细菌的膜外。

图3 福氏志贺菌OmpA蛋白信号肽预测
2.4OmpA蛋白二级结构与三级结构预测
利用SOPMA方法预测OmpA蛋白二级结构,结果显示无规则卷曲氨基酸数占总氨基酸数的41.09 %,α-螺旋为26.44 %,β-转角为10.06 %,β-片层为22.41 %(图5)。采用SWISS-MODEL程序,预测OmpA蛋白的三维空间结构,模版蛋白选用2k0l.1.A,序列相似度为82.86 %,结果显示OmpA为桶状结构,如图6所示。
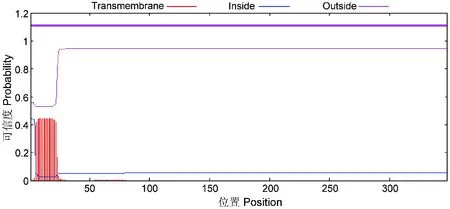
图4 福氏志贺菌OmpA蛋白跨膜区预测

图5 福氏志贺菌OmpA蛋白二级结构预测
h:α-螺旋;t:β-转角;c:无规则卷曲;e:β-片层

图6 福氏志贺菌OmpA蛋白三维结构预测
2.5OmpA蛋白B细胞表位预测
采用ABCpred软件,通过人工神经网络方法,预测线性表位,获得OmpA可能的B细胞表位区段,见表1所示。通过BepiPred 1.0 Server软件预测OmpA优势B细胞表位,该方法依据氨基酸的性质(可及性、亲水性、柔韧性、极性、转角) 和隐形马尔可夫模型,结果如表2所示。综合BepiPred和ABCpred两种方案的共同多肽序列,获得OmpA可能的优势B细胞表位区段为:37~50,83~92,292~300。
2.6OmpA蛋白的T细胞表位预测
2.6.1OmpA蛋白的CTL表位预测 CTL(细胞毒性T淋巴细胞)表位预测采用nHLAPred软件,该方法运用神经网络与量化矩阵法(ANN+QM),分别预测HLA-A2、HLA-A*0201、HLA-A*0202、HLA-A*0203、HLA-A*0205的分子结合肽,阈值设为0.5,结果如表3所示。综合分析获得OmpA的CTL抗原表位为:239~247位的ALDQLYSQL。

表1 ABCpred方案预测福氏志贺菌OmpA蛋白B细胞表位的肽段位置

表3 福氏志贺菌OmpA蛋白CTL抗原表位预测
2.6.2OmpA蛋白的Th表位预测Th(T辅助细胞)使用ProPred在线程序,对表位进行预测,预测类型选择DRB1-0101、DRB1-0102和DRB1-0301结合肽,结果见表4所示。综合分析获得OmpA的优势Th抗原表位为:286~294位的YLISKGIPA。
2.7OmpA蛋白重组抗原表位的设计
依据OmpA蛋白的B、T细胞表位预测获得的5个表位结果,将这5个表位以GGGG氨基酸接头连接。采用DNAStar Protean软件,分析线性表位的各种排列方式,结果发现:epitope3- epitope5- epitope1- epitope4- epitope2排列的重组多表位间相对独立,抗原性参数较好(图7)。设计的表位疫苗多肽序列为:IPADKISARGGGGYLISKGIPAGGGGSQYHDTGFIPNNGP
GGGGALDQLYSQLGGGGPYKGDNINGA(下划线为柔性肽序列)。翻译为核酸序列,即获得的OmpA蛋白重组表位为:ATCCCGGCAGACAAGATCTCCGCACGTGGTGG
CGGTGGCTACCTGATCTCCAAAGGTATCCCGGCAGGTG
GCGGTGGCTCCCAGTACCATGACACTGGTTTTATTCCT
AACAATGGTCCGGGTGGCGGTGGCGCTCTGGATCAGC
TGTACAGCCAGCTGGGTGGCGGTGGCCCGTACAAAGG
CGACAACATCAACGGCGCA(下划线为柔性肽的核酸序列)。
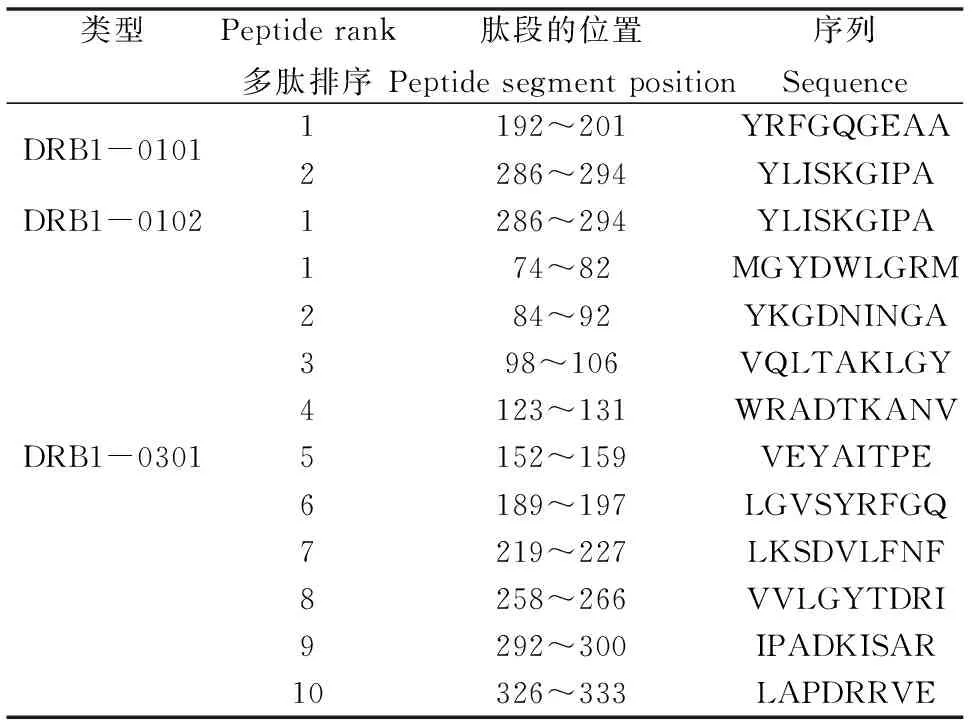
表4 福氏志贺菌OmpA蛋白Th抗原表位预测

图7 重组多表位分子的抗原性分析
Fig 7 Antigenic analysis of recombinant multi-epitopes molecule
3讨论
通过生物信息学分析,可很好地预测物种间的亲缘关系、蛋白的理化性质、高级结构;尤其是蛋白抗原表位的预测,可减少实验的盲目性,实现人工针对性地合成多肽表位疫苗,在疫苗研制和免疫诊断上应用广泛[11,12]。
本研究通过计算机辅助技术,对OmpA氨基酸序列进行系统发生分析,结果显示福氏志贺菌与肠道细菌亲缘关系更近,表明OmpA蛋白对不同种的肠道致病菌可能存在交叉免疫保护作用,这为研制广谱抑菌的OmpA免疫制剂提供依据。此外,对福氏志贺菌OmpA蛋白进行理化指标与高级结构预测。结果发现OmpA为亲水性蛋白,具有信号肽序列。因而,OmpA可能是通过信号肽指导作用分泌到细胞外,OmpA是一种分泌性蛋白。二级结构分析显示OmpA主要形成无规则卷曲。无规则卷曲易于产生盘旋、扭曲,暴露于蛋白外层,常含有优势的抗原表位[13],使得OmpA具有较强的免疫激活作用。OmpA三级结构呈现桶装,这与大肠杆菌OmpA结构相似[5],可能与物质的选择性通过有关。
B细胞表位预测常用的代表方案为BepiPred和ABCpred。BepiPred是利用氨基酸的性质,如亲水性、柔韧性、可及性、极性、暴露表面、转角等,以及隐形马尔可夫模型预测线性表位[14];ABCpred基于人工神经网络算法,准确率可高达65.9%[15]。本研究结合BepiPred和ABCpred两种软件进行B细胞表位预测,发现OmpA的B细胞抗原表位区段为:37~50,83~92,292~300。
CTL抗原表位是与MHC-Ⅱ类分子结合,经抗原呈递作用,最终诱发CTL免疫应答的短肽,对CTL特异性细胞毒作用具有决定性[16-18]。CTL表位预测的方法较多,有人利用软件BIMAS、SYFPEITHI和RANKPEP对CTL表位进行预测[12, 19]。王光祥等利用在线软件,采用人工神经网络法结合量化矩阵法成功预测羊口疮病毒F1L蛋白CTL表位[16]。然而,关于福氏志贺菌OmpA的CTL表位预测尚未开展。本研究利用人工神经网络法与量化矩阵法,进行OmpA蛋白的CTL表位预测。获得OmpA的CTL表位为:239~247位的ALDQLYSQL。
Th表位是与MHC-Ⅱ类分子结合的抗原表位,经过抗原呈递作用,最终可被相应的CD4+T细胞识别结合,激活Th细胞活性,以辅助B细胞产生抗体[20]。Th表位预测方法较为成熟,病毒蛋白[16]、细菌蛋白[18]均实现很好的Th表位预测。本研究预测获得了OmpA蛋白的Th表位为286~294位的YLISKGIPA。
多肽疫苗接头的选择上,要求构建的蛋白多肽具有一定的分子刚性,各表位间相对独立,并且抗原参数要趋于完美[21]。常用的接头氨基酸序列有GGGG[22]、GGGGS[23]、KK[24]、AAY[21]等。本研究采用GGGG氨基酸接头对OmpA预测获得的B细胞、CTL细胞,以及Th细胞表位进行不同组合的串联,最终获得抗原性较好的重组多肽。然而,新合成的重组多肽需要进一步的试验,验证其免疫学功能。
参考文献:
[1]GEORGE D T, BEHM C A, HALL D H, et al.Shigellaflexneriinfection inCaenorhabditiselegans: cytopathological examination and identification of host responses [J]. PLoS One, 2014, 9(9):106085.
[2]朱阵, 王婧, 张继瑜, 等. 福氏志贺菌2型多重PCR检测方法的建立 [J]. 中国畜牧兽医, 2014, 41(8):34-39.
[3]DRAGOI A M, AGAISSE H. The serine/threonine kinase STK11 promotesShigellaflexneridissemination through establishment of cell-cell contacts competent for tyrosine kinase signaling [J]. Infect Immun, 2014, 82(11):4447-4457.
[4]王俊, 祁伟, 王淑香, 等. 天津地区福氏志贺菌整合子携带及耐药性研究 [J]. 中国病原生物学杂志, 2014, 9(2):146-149.
[5]陈春琳,俱雄,万健, 等. 革兰阴性菌外膜蛋白A研究进展 [J]. 生物技术, 2014, 24(2):98-103.
[6]刘祥. 福氏志贺菌2型外膜蛋白A的原核表达及多克隆抗体的制备 [J]. 动物医学进展, 2015, 36(5):6-10.
[7]PORE D, MAHATA N, PAL A, et al. Outer membrane protein A (OmpA) ofShigellaflexneri2a, induces protective immune response in a mouse model [J]. PLoS One, 2011, 6(7):22663.
[8]BHOWMICK R, PORE D, CHAKRABARTI M K. Outer membrane protein A (OmpA) ofShigellaflexneri2a induces TLR2-mediated activation of B cells: involvement of protein tyrosine kinase, ERK and NF-κB [J]. PLoS One, 2014, 9(10):109107.
[9]PORE D, CHOWDHURY P, MAHATA N, et al. Purification and characterization of an immunogenic outer membrane protein ofShigellaflexneri2a [J]. Vaccine, 2009, 27(42):5855-5864.
[10]刘祥, 李惠. 大肠埃希菌外膜蛋白OmpA表达质粒构建和诱导条件优化 [J]. 生物技术, 2013, 23(4):32-36.
[11]AMAT-UR-RASOOL H, SAGHIR A, IDREES M. Computational prediction and analysis of envelop glycoprotein epitopes of DENV-2 and DENV-3 pakistani isolates: a first step towards dengue vaccine development [J]. PLoS One, 2015, 10(3):0119854.
[12]胡晓波, 朱乃硕. 狂犬病毒MHC限制性CTL与Th表位的预测与鉴定 [J]. 中国免疫学杂志, 2013, 29(7):736-740.
[13]MANZANO-ROMN R, DAZ-MARTN V, OLEAGA A, et al. Identification of protective linear B-cell epitopes on the subolesin/akirin orthologues ofOrnithodorosspp. soft ticks [J]. Vaccine, 2015, 33(8):1046-1055.
[14]CHEN H, YANG H W, WEI J F, et al. In silico prediction of the T-cell and IgE-binding epitopes of Pera 6 and Blag 6 allergens in cockroaches [J]. Mol Med Rep, 2014, 10(4):2130-2136.
[15]SAHA S, RAGHAVA G P. Prediction of continuous B-cell epitopes in an antigen using recurrent neural network [J]. Proteins, 2006, 65(1):40-48.
[16]王光祥, 尚佑军, 吕占禄, 等. 羊口疮病毒F1L蛋白二级结构分析与表位预测 [J]. 中国人兽共患病学报, 2012, 28(12):1185-1190.
[17]LI J, BAI J, GU L, et al. Prediction and identification of HLA-A*0201-restricted epitopes from leukemia-associated protein MLAA-22 which elicit cytotoxic T lymphocytes [J]. Med Oncol, 2014, 31(12):293.
[18]白雪娟, 赵亚静, 梁 艳, 等. 结核潜伏感染蛋白 Rv1737c B 细胞、CTL 及 Th 表位预测与分析 [J]. 实用临床医药杂志, 2015, 19(3):5-9.
[19]李章球,胡海燕,刘顺会, 等. 生物信息学预测GPⅡb/Ⅲa抗体CTL、Th的混合表位 [J]. 中国组织工程研究与临床康复, 2009, 13(4):709-712.
[20]LEROUX L P, DASANAYAKE D, ROMMEREIM L M, et al. Secreted toxoplasma gondii molecules interfere with expression of MHC-II in interferon gamma-activated macrophages [J]. Int J Parasitol, 2015, 45(5):319-332.
[21]季晶晶, 李槿年. 拟态弧菌 OmpU抗原表位的预测与多表位疫苗分子的设计 [J]. 水生态学杂志, 2008(5):75-79.
[22]石晓妮, 窦永喜, 张海瑞, 等. 小反刍兽疫病毒H 和F 蛋白多抗原表位的表达及其反应原性 [J]. 中国兽医科学, 2011, 41(5):474-478.
[23]秦培兰, 李艳, 米荣升, 等. 微小隐孢子虫三个基因主要抗原表位区的串联表达及其抗原性分析 [J]. 中国动物传染病学报, 2012, 20(3):36-43.
[24]董林, 王艳萍, 沈志强, 等. 猪圆环病毒2型多表位串联体诱导表达及其免疫活性 [J]. 中国兽医学报, 2014, 34(11):1716-1720.
Bioinformatics analysis of Shigella flexneri 2a outer membrane protein A and molecular designing of recombinant epitope vaccine
LIU Xiang
(College of Biological Sciences and Engineering, Shaanxi University of Technology, Hanzhong 723001, China)
AbstractShigella flexneri outer membrane protein A (OmpA) has good immunogenicity for application prospect in vaccine development. In this paper, phylogenetic analysis found that Shigella flexneri and enteric bacterium relationship was higher than others by MEGA. By online software, the prediction results showed that OmpA protein is a hydrophilic and secreted protein, exists multiple restriction enzyme sites. Using TMHMM Server v.2.0 software predicted that OmpA has not transmembrane structure and locats outside cell membrane. OmpA secondary structure contains random coil helix 41.09 %, alpha helix 26.44 %, beta turn 10.06 %, extended strand 22.41 % by SOPMA method, respectively. Using the SignalP 4.1 software analysis showed that the 1-21 amino acids of OmpA are signal peptide sequence. The 3D structure prediction found that OmpA protein is barrel structure by Swiss-Model program. Using ABCpred and BepiPred prediction program, OmpA has 3 B cell epitopes. By the prediction method of quantitative matrix and artificinal neural network, OmpA protein has 1 CTL epitopes. OmpA protein has 1 Th epitopes using an online server prediction for MHC class Ⅱ peptide binding affinity. OmpA recombinant epitope peptide is designed with better immunogenicity. This paper would lay a foundation for the study of OmpA multiepitope tandem vaccine.
Key wordsShigella flexneri; OmpA protein; cell epitopes; protein structure
收稿日期:2015-07-29;修回日期:2015-08-10
基金项目:陕西省农业科技创新与攻关(2016NY-088)
作者简介:刘 祥,博士,讲师,主要从事蛋白质组学与免疫学研究,E-mail:liuxiang888525@163.com。
中图分类号Q816
文献标识码B
文章编号2095-1736(2016)03-0099-05
doi∶10.3969/j.issn.2095-1736.2016.03.099
