蛇床子三螺旋转录因子基因的克隆与表达分析
2016-06-29王希强尹登科吴家文
王希强, 尹登科, 储 俊, 吴家文
(1. 安徽中医药大学 科研实验中心 新安医学教育部重点实验室, 合肥 230038;2. 安徽中医药大学 药学院, 合肥 230038)
蛇床子三螺旋转录因子基因的克隆与表达分析
王希强1, 2, 尹登科2, 储俊1, 吴家文1
(1. 安徽中医药大学 科研实验中心 新安医学教育部重点实验室, 合肥 230038;2. 安徽中医药大学 药学院, 合肥 230038)
摘要研究的目的是克隆中药蛇床子[Cnidium monnieri (L.) Cuss.]三螺旋转录因子(Trihelix transcription factor,ttf)基因,诱导、鉴定其蛋白体外表达,并利用生物信息学方法分析该蛋白的结构和性质。主要采用RT-PCR方法扩增目的基因,连接到pMDTM19(Simple)-T克隆载体中,核酸序列鉴定正确后再将目的基因克隆到pET-22b(+)表达载体上,将获得的重组质粒转化到大肠杆菌E.coli BL21细胞中,以不同浓度的诱导剂诱导其表达,进一步利用聚丙烯酰胺凝胶电泳和免疫印迹方法检测目的蛋白的表达情况,并利用各种生物学软件对目的蛋白进行生物信息学分析。结果获得了ttf,构建了该基因的克隆和表达重组质粒,该基因在大肠杆菌BL21中能够被诱导表达,Western Blot结果表明,在预计的分子质量21 ku位置得到了目的蛋白;生物信息学分析表明,目的蛋白为可溶性蛋白,可能定位于细胞核和线粒体中;部分区域极有可能形成卷曲螺旋。研究首次成功体外克隆并在原核生物中表达了蛇床子三螺旋转录因子,并对其进行了系统的生物信息学分析,为进一步研究其结构和功能奠定了基础。
关键词蛇床子;三螺旋转录因子;基因克隆;蛋白质表达;生物信息学
转录因子是一类能与基因启动子区域中的顺式作用原件发生特异性结合的核蛋白,通常也称为反式作用原件,一般由寡聚化位点、核定位信号域、DNA结合域及基因调控域等功能区域组成[1,2],其中DNA结合域特异性结合基因中的顺式作用原件,从而精确调控基因的转录与翻译;基因调控域激活或者抑制基因表达;富含精氨酸和赖氨酸的核定位信号域主要调控转录因子出入核的过程[3];寡聚化位点通常与DNA结合域协同作用。这些区域通过与特异DNA序列或蛋白分子伴侣结合调控基因的转录过程从而表现出特定的生物学功能。植物中已经被鉴定出超过60种的转录因子,根据其DNA结合域的不同将转录因子主要分为B3家族、AP2/EREBP家族、bZIP家族、MYB家族以及三螺旋家族[4-8]。
三螺旋转录因子家族基因由于其DNA结合域含有3个串联的螺旋结构域而得名,其保守蛋白结构域就是由3个螺旋状的蛋白结构域两两形成环状或折角的超二级结构,功能主要表现在光应答、对生物胁迫和非生物胁迫具有应答反应以及对植物特定组织的发育进行调控等[5-7]。
三螺旋转录因子能与基因序列上的光应答元件GT发生特异性结合,故该基因家族也被称为GT家族。三螺旋转录因子的亚家族分为:GT-1、GT-2、GT-3a、GT-3b、GT-4、GT-γ、ASIL1、ASIL2、SH4及SIP1等,这些亚家族至少含有一个三螺旋结构域[6, 7]。其中GT-1和GT-2在拟南芥和水稻等模式植物中已经被体外克隆[3]。
本实验室采用Illumina HiSeq 2 500高通量测序获得了没有参考基因的中药蛇床子的转录组数据。我们提取了蛇床子叶的总RNA,采用RT-PCR方法以特定引物扩增目的基因ttf,并将此基因定向克隆到pMDTM19(Simple)-T载体,并进一步克隆到表达载体pET-22b (+),利用IPTG(Isopropyl β-D-1-Thiogalactopyranoside)进行诱导表达,摸索其最佳表达条件,为该基因大量表达和纯化以得到高浓度的目的蛋白从而进一步研究其结构和功能提供了很好的基础。
1材料与方法
1.1实验材料
1.1.1植物材料和试剂
野生蛇床子[Cnidiummonnieri(L.)Cuss.]植株采自安徽省桐城市,由安徽中医药大学王德群教授鉴定,根、茎、叶、花等新鲜样品由灭菌的双蒸水清洗后液氮快速冷冻,然后立即置于-80℃冰箱备用。总RNA提取试剂盒、逆转录试剂盒、DNA凝胶回收试剂盒、质粒抽提试剂盒均购自合肥欣乐生物科技公司,高保真KOD-Plus DNA聚合酶购自日本TOYOBO公司,DNA连接酶、限制性内切酶XhoⅠ和NdeⅠ均购自TaKaRa公司,His-tag抗体中的鼠抗(AM1010a)购自ABGENT公司,二抗(W402B)购自Promega公司,设计的引物由苏州金唯智生物科技有限公司合成,其余试剂均为进口或国产分析纯。
1.1.2实验菌株和载体质粒
分子克隆用的大肠杆菌E.coli菌株DH5α、表达菌株BL21、载体质粒pET-22b (+)均由中国科学技术大学生命科学学院施蕴渝院士实验室惠赠;实验所用的pMDTM19(Simple)-T载体质粒购自TaKaRa生物公司。
1.1.3实验仪器
实验用的主要仪器有凝胶成像系统(BIO-RAD),DNA扩增仪(德国耶拿分析仪器股份公司),核酸电泳仪(Amersham公司),蛋白电泳仪,恒温恒速摇床,超净操作台,恒温水浴锅,超精度电子天平,紫外-可见分光光度计均为国产产品。
1.2实验方法
1.2.1蛇床子总RNA的提取及cDNA合成
采用OMEGA公司的Plant RNA Kit提取蛇床子的总RNA,分别在260 nm和280 nm处测其紫外吸收值并计算D260 nm/D280 nm,根据测定的结果将总RNA浓度调至200 ng/μL,再使用逆转录试剂盒进行逆转录合成cDNA,凝胶电泳鉴定总RNA提取和cDNA合成的结果。
1.2.2蛇床子ttf引物的设计
基于目的基因蛇床子ttf的cDNA序列,利用Primer 5.0软件设计一对特异性扩增引物:ttf-S:5′ CGAATCATATGATGAAGCAACAGTTTCCATATTAC 3′(35 bp),ttf-A:5′ CGACTCGAGATTAGATTTATCTTCCCTTCTAAGC 3′(34 bp),在上下游引物中分别插入了NdeⅠ和XhoⅠ的酶切位点,分别为5′ CATATG 3′和5′ CTCGAG 3′。
1.2.3蛇床子ttf的克隆
以逆转录产物cDNA为模板进行RT-PCR。50 μL PCR扩增体系中含10 μL的5×easypfubuffer缓冲液、1 μL 10 mmol/L dNTP mix、3 μL 4μmol/μL上下游引物、3 μL cDNA、0.4 μL easypfu高保真DNA聚合酶、29.5 μL ddH2O。PCR 扩增程序为94℃预变性3 min;94℃变性30 s,40℃退火30 s,72℃延伸75 s,共10个循环;随后94℃变性30 s,55℃退火30 s,72℃延伸75 s,共25个循环。胶回收RT-PCR产物,并再次以回收RT-PCR产物为模板,用Taq酶代替pfu高保真DNA聚合酶进行PCR,体系不变。
1.2.4蛇床子ttf克隆重组质粒构建
用DNA凝胶回收试剂盒纯化回收Taq酶PCR后的目的基因条带,然后与克隆载体pMDTM19(Simple)-T 连接,采用pMDTM19(Simple)-T载体1 μL,回收的ttf7 μL,T4DNA连接酶1 μL,10×T4DNA连接酶缓冲液1 μL进行重组,16℃连接4 h。连接产物转化大肠杆菌DH5α感受态细胞,次日挑单克隆菌落进行扩大培养。首先利用目的基因ttf的上下游引物对菌落进行PCR初步鉴定,然后纯化质粒再次进行PCR 鉴定和NdeI /XhoI双酶切鉴定,挑选鉴定正确的阳性重组子送至华大基因测序。
1.2.5蛇床子ttf原核表达重组质粒构建
将测序正确的重组pMD19-T-ttf质粒和表达载体pET-22b(+)分别用NdeI 和XhoI双酶切,胶回收ttf条带与线性化的pET-22b(+)载体,T4DNA连接酶于16℃连接过夜,将连接产物转化到大肠杆菌DH5α感受态细胞,过夜培养后进行菌落PCR初筛,质粒PCR鉴定和NdeI/XhoI双酶切进一步鉴定,再次将阳性重组子pET22b-ttf送样测序。
1.2.6重组质粒pET22b-ttf在大肠杆菌内诱导表达
将重组质粒pET22b-ttf转化进感受态细胞BL21中,次日挑单克隆到10 mL LB培养基中扩大培养,在37℃的恒温培养箱中,以250 r/min转速培养至D600 nm值为0.6~0.8时,加IPTG诱导表达蛋白(IPTG浓度分别为0、0.1、0.5和1 mmol/L),在25℃或37℃、150 r/min条件下诱导其表达6 h。停止诱导表达后,以转速8 000 r/min离心5 min收集菌体,因为目的蛋白的等电点(pI)为7.85,所以选择pH 6.5的PBS缓冲液(含500 mmol/L NaCl)2 mL重悬菌体,超声破碎菌体(频率750 Hz,超声1 s,停3 s,超声60次), 13 000 r/min高速离心后将上清和沉淀分离。进一步利用SDS-PAGE蛋白电泳分析和Western Blot方法检测蛋白在上清或沉淀中表达情况。
1.2.7TTF蛋白Western Blot检测
重组质粒PET22b-ttf表达的目的蛋白C末端有His-tag(6个组氨酸标签),利用His-tag特异性抗体对样品进行Western Blot检测。将诱导表达的菌体破碎后离心,得到上清蛋白和沉淀蛋白分别进行聚丙烯酰胺凝胶电泳1.5 h,200 mA恒定电流转硝酸纤维素膜2 h,全脱脂奶粉(5 g/10 mL PBST)封闭3h,PBST洗3次(10 min/T),4℃一抗孵育过夜,次日PBST洗3次(10 min/T),室温避光二抗孵育2 h,PBST洗3次(10 min/T),暗室曝光。
1.2.8TTF蛋白生物信息学分析
通过序列翻译软件将蛇床子中的ttf开放阅读框翻译成氨基酸序列进行比对;通过Protparam在线分析软件分析目的基因翻译出的蛋白中氨基酸的带电性、各氨基酸所含百分数,以及蛋白的亲水-疏水性;通过InterProScan软件分析ttf编码的目的蛋白含有的各种基序及其结构域;通过ExPASy在线软件Coils预测目的蛋白卷曲螺旋结构;通过SmartBlast在线软件进行目的蛋白氨基酸序列比对和进化分析、二级结构的预测和溶剂可接近性的分析。
2结果与分析
2.1蛇床子ttf克隆和鉴定
2.1.1蛇床子总RNA的提取及鉴定
利用植物RNA提取试剂盒提取蛇床子总RNA,然后立即做琼脂糖凝胶电泳鉴定,如图1显示,两个提取的总RNA样品中RNA的28s和18s和5s 3个亚基的条带均清晰可见,样品的紫外吸光度值D260 nm和D280 nm的比值为1.83,说明RNA提取纯度较高且基本没有降解。

图1 总RNA琼脂糖凝胶电泳图
2.1.2蛇床子ttf的克隆
用提取的mRNA为模板逆转录合成cDNA,再对其进行RT-PCR,为了使克隆的目的基因末端带上A碱基,以便更好地和T-vector连接,再次以纯化的RT-PCR的基因产物为模板用Taq酶进行了PCR,2次均得到了比较清晰的目的条带(约540 bp处,如图2 所示)。

图2 ttf的克隆
A:RT-PCR结果图;B:Taq酶再次PCR结果图。箭头所指的是目的基因位置
2.1.3重组质粒pMD19-ttf的构建
纯化Taq酶PCR产物中的目的基因条带,然后与pMDTM19(Simple)-T载体连接,将连接产物转化到DH5α感受态细胞,单克隆扩大培养后抽提质粒,琼脂糖凝胶电泳检测,结果如图3A所示,质粒条带位置正确。以重组质粒为模板进行PCR鉴定,得到的目的条带在750 ku和500 ku之间,符合目的条带540 bp大小(图3B)。再用NdeI /XhoI两种酶将质粒样品进行双酶切鉴定,结果如图3C所示,目的基因条带以箭头示意,位置正确,而250 bp位置也有条带是因为pMDTM19(Simple)-T载体中有NdeI酶切位点。送pMD19-ttf重组质粒到华大基因测序,结果与转录组测序得到的cDNA序列完全一致。
2.1.4重组质粒pET22b-ttf的构建
用NdeI /XhoI双酶切重组的质粒pMD19-ttf,纯化目的基因条带,然后与用同样双酶切的表达载体pET22b(+)连接,转化到DH5α感受态细胞,单克隆扩大培养后抽提质粒,琼脂糖凝胶电泳检测,结果如图4A所示质粒条带位置正确。同样进行PCR和双酶切鉴定,结果如图4B和4C所示,目的条带位置正确。再次将阳性重组子pET22b-ttf送样测序,测序结果证明重组质粒正确无误。
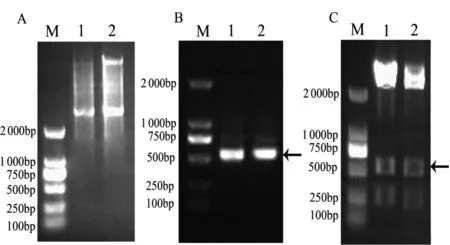
图3 重组质粒pMD19-ttf的纯化和鉴定
A:重组质粒电泳结果图;B:目的基因PCR鉴定图;C:重组质粒酶切结果图。箭头所指的是目的基因位置
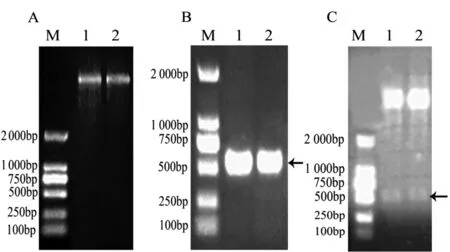
图4 重组质粒pET22b -ttf的纯化和鉴定
A:重组质粒电泳结果图;B:目的基因PCR鉴定结果图;C:重组质粒酶切结果图。箭头所指的是目的基因位置
2.2重组质粒pET22b-ttf的诱导表达
将测序正确的重组质粒pET22b-ttf转化到表达菌体BL21中,以不同的IPTG浓度分别在37℃和25℃进行诱导表达,然后进行SDS-PAGE电泳及Western Blot鉴定。目的蛋白条带在大约21ku的位置,与TTF蛋白理论分子量相符,说明重组质粒在大肠杆菌BL21中成功表达(图5),且从图中可以明显看出,当IPTG的浓度为0.1 mmol/L时,无论在25℃还是37℃目的蛋白的表达量均多于其它条件;37℃诱导表达的蛋白量比25℃多;蛋白在沉淀中表达明显多于上清。
2.3生物信息学分析结果
2.3.1ttf序列分析
重组质粒测序结果表明插入序列与ttf的ORF完全一致,cDNA经序列翻译工具(http://www.bio-soft.net/sms/index.html)翻译出蛋白质序列,两者比对结果如图6所示, cDNA序列含540 bp, 其编码的蛋白质含179个氨基酸。
2.3.2ttf编码蛋白理化特性分析
Protparam (http://web.expasy.org/cgi-bin/protparam/protparam)序列分析表明ttf编码的蛋白中谷氨酸 (E) 有29个、含量最高,占16.2%;蛋白理论分子质量为21 254.7 u,等电点为7.85,含有36个负电荷氨基酸(Asp + Glu),37个正电荷氨基酸(Arg + Lys),这与它的等电点偏碱性是一致的。 此蛋白在280 nm处的摩尔消光系数为27 960 M-1cm-1,0.1%浓度(1 g/L)的Abs为1.315。蛋白质在溶液中的不稳定指数为57.87,高于阈值40,属于不稳定蛋白。脂肪指数为39.89,总平均亲水性(GRAVY)为-1.498,蛋白质总体亲水性较高。
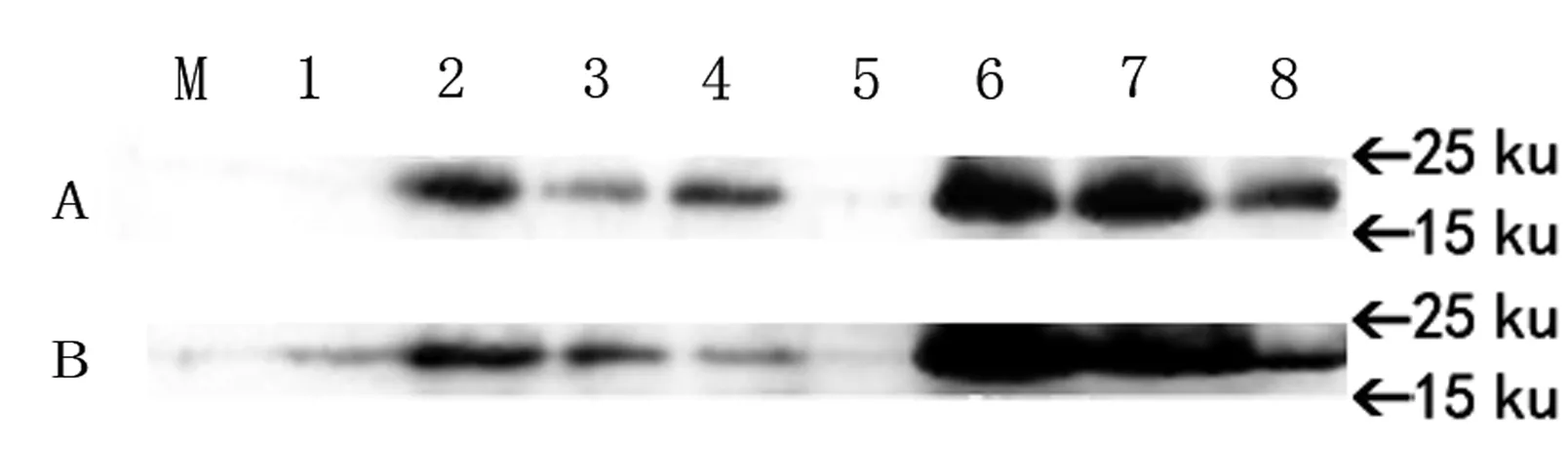
图5 不同温度和不同浓度IPTG对TTF蛋白表达的影响
A:25℃蛋白表达的结果;B:37℃蛋白表达的结果。其中1和5分别为空白对照的上清和沉淀中目的蛋白;2和6分别为0.1 mmol/L IPTG诱导表达的上清和沉淀中目的蛋白;3和7分别为0.5 mmol/L IPTG诱导表达的上清和沉淀中目的蛋白;4和8分别为1 mmol/L IPTG诱导表达的上清和沉淀中目的蛋白

图6 ttf cDNA序列和氨基酸序列
2.3.3TTF蛋白基元、结构域和定位分析
Motif Scan(http://hits.isb-sib.ch/cgi-bin/motif_scan/)分析显示ttf编码蛋白含有各种motif(基元),包括一个酰胺化位点、1个N- 糖基化位点、2个cAMP和cGMP依赖的蛋白激酶磷酸化位点,3个酪蛋白激酶II磷酸化位点,5个蛋白激酶C磷酸化位点、3个N-蛋白质豆蔻酰化位点;一个富含精氨酸区域, 一个富含谷氨酸区域,一个双侧核定位信号(NLS)序列,一个Ras结合区域,一个死亡结构域 (表1)[9]。
PSORT (http://psort.hgc.jp/form.html) 预测TTF定位于核中的可能性为93.3%,定位于线粒体基质空间的可能性为100%,与其转录因子的功能相一致[10]。
2.3.4TTF蛋白亲水-疏水性分析
根据ProtScale Sever在线分析软件,利用Hphob/Kyte&Doolittle的方法,对TTF蛋白的亲水-疏水性进行分析。正值越大,疏水性越强;负值越小,亲水性越强,如图7所示最大正值为1.178,最小负值为-3.656,绝大多数氨基酸显示亲水性负值,提示该蛋白为亲水性很强的蛋白。

表1 TTF蛋白基元及结构域分析

图7 TTF蛋白亲水-疏水性分析
Fig 7 Hydrophilicity and hydrophobicity analysis of TTF
2.3.5TTF蛋白卷曲螺旋结构预测与分析
利用http://www.expasy.org/tools/中的Coils分析工具,对TTF蛋白序列进行卷曲螺旋的倾向性预测,按照几率大于50%就可能形成螺旋的规则,以window=14为试验参数,Y111-R142,R144-A163很可能是卷曲螺旋,以window=21为试验参数,E116-A167很可能是卷曲螺旋,而以window=28为试验参数, M108-E175很可能是卷曲螺旋;三种试验参数几率都大于50%的区域 E116-R142(24AA),R144-A163(20AA),这两段区域形成卷曲螺旋的可能性极大 (图8)。这与SMART(http://smart.embl-heidelberg.de)网站预测的115~167(53AA)为Coiled-coil区域结果基本一致。

图8 TTF蛋白卷曲螺旋结构分析
2.3.6TTF同源蛋白序列比对和进化分析
利用NCBI(http://www.ncbi.nlm.nih.gov/)中的SmartBlast对ttf编码蛋白进行序列比对[11],根据最好的3个比对的结果以及两个目前研究最清楚的两个物种得到5个同源性较高的蛋白质,这些蛋白质在NCBI中的登记号,根据功能的归类,来源的物种以及与ttf比对的分析见表2。这些同源蛋白的氨基酸序列比对结果见图9。根据系统发育关系分析(http://www.ncbi.nlm.nih.gov/tools/cobalt/cobalt.cgi),ttf与芝麻的三螺旋转录因子聚在一起,表明其与芝麻的亲缘关系最近(图 10)。

表2 TTF同源蛋白比对结果分析
2.3.7TTF二级结构预测和溶剂可接近性分析
利用ExPASy中的Predict Protein(http://www.predictprotein.org/)对TTF蛋白进行二级结构预测,结果表明1、138为蛋白质结合区域;21、32~38、62~68、67、80、82~84为DNA结合区域;8~28、52~56、63~72、85~176为螺旋区。蛋白溶剂可接近性分析表明82.68%的氨基酸暴露在溶剂中,13.41%的氨基酸被埋藏在内部,可以推测目的蛋白是可溶性的[12-17](图11)。

图9 TTF同源蛋白的氨基酸序列比对
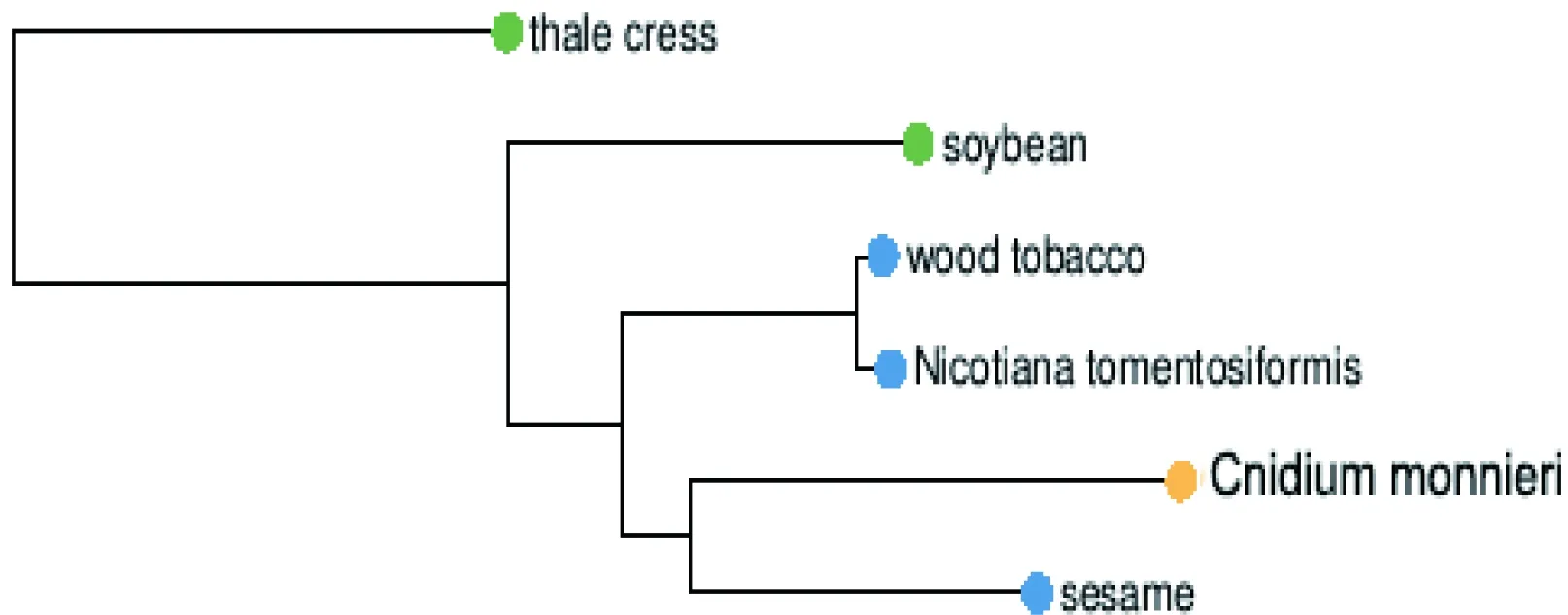
图10 ttf的进化树分析

图11 TTF蛋白二级结构和溶剂可接近性分析
3结论与展望
植物的抗逆性是多基因控制的,寒冷、旱涝、高盐等胁迫因素是影响植物生长发育以及形态的重要因素。三螺旋转录因子家族基因的功能主要是对生物胁迫和非生物胁迫具有应答反应,也对植物特定组织的发育进行调控等。本研究首次成功地将蛇床子三螺旋转录因子基因克隆到原核表达载体pET-22b (+)上,然后将重组质粒pET-22b-ttf转化到E.coliBL21细胞中,利用IPTG进行诱导,目的基因得到了成功表达。Western Blot结果表明,0.1 mmol/L IPTG诱导表达蛋白效果最好,37℃条件下表达量较多。利用Protparam软件分析了蛇床子ttf表达的蛋白质的氨基酸组成、分子量、等电点、稳定性等特性,TTF蛋白含有179个氨基酸残基、理论分子质量为21 254.7u,等电点为7.85;TTF蛋白为可溶性蛋白,但在溶液中不稳定。利用Motif Scan分析了该蛋白质的各种基元,包括各种活性位点和结构域;PSORT预测了该蛋白质主要定位于细胞核和线粒体中;Predict Protein二级结构预测该蛋白质中规则的二级结构主要是卷曲螺旋,Coils软件预测E116-R142、 R144-A163两段区域形成卷曲螺旋的可能性极大;根据NCBI的比对结果进行进化分析表明ttf与芝麻的亲缘关系最近。生物信息学分析表明,TTF应该属于GT-3类,其中GT-3a特异性地结合DNA核心序列5′-GTTAC-3′,亲缘关系很近的GT-3a和GT-3b能形成同源或异源二聚体,且主要在植物的花蕾和根中表达。本研究为进一步研究蛇床子TTF蛋白的结构、生物学功能以及蛇床子中草药的抗逆性奠定了理论和实验基础。
参考文献:
[1]刘强, 张贵友, 陈受宜. 植物转录因子的结构与调控作用[J]. 科学通报, 2000, 45(10):1465-1468.
[2]罗军玲, 赵娜, 卢长明. 植物Thrihelix转录因子家族研究进展[J]. 遗传, 2012, 34(12):1561-1560.
[3]关秋玲, 陈焕新, 张毅, 等. 植物GT元件和GT因子的研究进展[J]. 遗传, 2009, 31(2):123-130.
[4]陈清, 汤浩茹, 董晓莉, 等. 植物Myb转录因子的研究进展[J]. 基因组学与应用生物学, 2009, 28(2):365-372.
[5]宿福园. 参与柿单宁代谢的MYB、bHLH及WD40转录因子基因的克隆及分析[D]. 武汉:华中农业大学, 2012, 6:50-80.
[6]李月. 棉花逆境胁迫应答Trihelix转录因子的鉴定及功能的分析[D]. 石河子: 石河子大学, 2013:2-30.
[7]赵娜. 甘蓝型油菜Trihelix家族SIP1亚家族基因的克隆与功能研究[D]. 北京: 中国农业科学院, 2012: 2-15.
[8]李红霞, 汪妤, 张战凤, 等. 植物转录因子与作物抗逆胁迫关系的研究进展[J]. 麦类作物学报, 2013, 33(3):613-618.
[9]SIGRIST C J, CERUTTI L, DE CASTRO E, et al. PROSITE, a protein domain database for functional characterization and annotation[J]. Nucleic Acids Res, 2010, 38(Database issue):161-166.
[10]COKOL M, NAIR R, ROST B. Finding nuclear localization signals[J]. EMBO REPORTS, 2000, 1(5):411-415.
[11]ALTSCHUL S F, MADDEN T L, SCHFFER A A, et al. Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J]. Nucleic Acids Res, 1997(25):3389-3402.
[12]OFRAN Y, ROST B. ISIS: interaction sites identified from sequence[J]. Bioinformatics, 2007(23):13-16.
[13]YANG X X, DENG Z L, LIU R. RBRDetector: Improved prediction residues on RNA-binding protenin structures using complementary feature-and template-based strategies[J]. Proteins: Structure, Function and Bioinformatics, 2014, 82(10):2455-2471.
[14]SCHLESSINGER A, PUNTA M, YACHDAV G, et al. Improved disorder prediction by combination of orthogonal approaches[J]. PLoS ONE, 2009, (4):4433-4433.
[15]HAMP T, KASSNER R, SEEMAYER S, et al. Homology-based inference sets the bar high for protein function prediction[J]. BMC Bioinformatics, 2013, 14(3):1-10.
[16]YUAN Y, YU J, JIANG C, et al. Functional diversity of genes for the biosynthesis of paeoniflorin and its derivatives in Paeonia[J]. Int J Mol Sci, 2013(14):18502-18519.
[17]ROST B, YACHDAV G, LIU J. The predict protein server[J]. Nucleic Acid Res, 2004, 32(Web Server issue): 321-326.
Cloning and expression analysis of trihelix transcription factor gene in Cnidium monnieri
WANG Xi-qiang1, 2, YIN Deng-ke2, CHU Jun1, WU Jia-wen1
(1. Key Laboratory of Xin′an Medicine, Ministry of Education;2. School of Pharmacy, Anhui University of Traditional Chinese Medicine, Hefei 230038, China)
AbstractThe aim of this study is to clone the Trihelix transcription factor (ttf) gene in a Chinese medicinal plant Cnidium monnieri(L.) Cuss. and express it in E.coli cells, followed by bioinformatic analysis. The ttf cDNA was amplified by RT-PCR from Cnidium monnieri(L.)Cuss, and plasmid pMD19-T-ttf was constructed by TA cloning. The cDNA fragment was subcloned to pET-22b(+), and recombinant plasmid pET-22b-ttf was verified by restriction endonuclease analysis and DNA sequencing. The recombinant plasmid was transformed into E.coli BL21, and pET-22b-ttf fusion protein was expressed with induction of IPTG at different concentrations, and then illustrated by sodium dodecyl sulfatepolyacrylamide gel electrophoresis (SDS-PAGE). Western-Blot assay revealed that fusion protein could be identified by His-tag antibody. The fusion protein was about 21 ku as expected. The molecular characteristics such as physiochemical properties, conserved domain, and sub-cellular localization of the TTF protein were determined using a series of bioinformatic tools. ttf was cloned and recombinant plasmid pET-22b-ttf was constructed, and then the fusion protein was expressed successfully, which will lead to the further study of the structure and function of ttf.
Key wordsCnidium monnieri (L.) Cuss.; Trihelix transcription factor; gene clone; protein expression; bioinformatic analysis
收稿日期:2015-12-22;修回日期:2016-01-13
基金项目:安徽省自然科学基金项目(1608085MH177);安徽省高校优秀青年人才支持计划重点项目(gxyqZD2016139);安徽省2015年度留学人员科技活动择优资助项目;安徽中医药大学自然科学研究基金项目(2015zr017, 2015zr018);安徽中医药大学人才引进科研项目(2015RC002)
作者简介:王希强,硕士,研究方向为中药分子生物学,E-mail:xqwang547@163.com; 通信作者:吴家文,教授,博士,研究方向为中药分子生物学,E-mail:wujiawen@ahtcm.edu.cn。
中图分类号Q71;Q344+.13
文献标识码A
文章编号2095-1736(2016)03-0024-06
doi∶10.3969/j.issn.2095-1736.2016.03.024
