家蚕淀粉酶基因的克隆、表达与分析
2016-06-18吕言娜陈晓妍邵丽萍李轶女胡小元张志芳易咏竹
吕言娜, 陈晓妍, 邵丽萍, 李轶女, 胡小元, 张志芳, 易咏竹
1.江苏科技大学, 江苏镇江 212018;2.中国农业科学院生物技术研究所, 北京 100081
家蚕淀粉酶基因的克隆、表达与分析
吕言娜1,2,陈晓妍1,2,邵丽萍1,2,李轶女2,胡小元2,张志芳2,易咏竹1*
1.江苏科技大学, 江苏镇江 212018;2.中国农业科学院生物技术研究所, 北京 100081
摘要:淀粉酶是家蚕体内比较重要的一种消化酶。利用real-time PCR技术对Bmamy5和Bmamy7基因在家蚕5龄3 d不同组织的mRNA转录水平进行了分析,结果显示Bmamy7只在家蚕5龄3 d中肠和马氏管组织中有转录,在中肠组织的转录高达3×109个拷贝/μg总RNA,在马氏管组织中的转录丰度较低,只有1.9×106拷贝/μg总RNA;Bmamy5只在中肠组织中有转录,转录水平为1.5×109个拷贝/μg总RNA。根据在家蚕转录组数据库中预测的编码家蚕淀粉酶的cDNA序列设计引物,利用RT-PCR技术克隆到两个家蚕淀粉酶基因Bmamy5和Bmamy7,Bmamy7基因长1 608 bp,ORF长1 512 bp,编码503个氨基酸;氨基酸序列分析表明Bmamy7具有典型的α-淀粉酶结构域。Bmamy5全长1 196bp,ORF长738 bp,编码245个氨基酸。对Bmamy5和Bmamy7进行了原核表达,Bmamy7重组蛋活性较弱,但未检测到Bmamy5重组蛋白的目的条带。研究结果为家蚕淀粉酶基因的研究与应用提供了参考。
关键词:家蚕;淀粉酶基因;组织表达;实时定量PCR
家蚕取食桑叶中的淀粉、糊精,首先被中肠壁细胞分泌的淀粉酶分解为麦芽糖,再被麦芽糖酶分解为葡萄糖,进入到血液中,作为家蚕生命活动的能源和自身的物质基础,参与重要的生理生化代谢[1]。因此,淀粉酶是家蚕中比较重要的一种消化酶,在家蚕体内碳水化合物的代谢中具有重要作用[2]。根据作用方式的不同,淀粉酶可分为α-淀粉酶(EC3.2.1.1)和β-淀粉酶(EC3.2.1.2) 两类,前者广泛分布于动物、植物和微生物体内,后者主要存在于植物中[2~4]。α-淀粉酶是内切型淀粉酶, 随机水解淀粉α-1,4 糖苷键;β-淀粉酶是外切型淀粉酶,从非还原性末端水解淀粉α-1,4 糖苷键[1]。
家蚕的血液和消化液中都含有淀粉酶[5],但是对于家蚕体内淀粉酶的研究却不多,继2000年家蚕α-淀粉酶基因全长序列(GenBank序列号:U07847)提交之后,直到2008年,杜周和等[6]克隆了另外一个α-淀粉酶基因(Bmamy2,家蚕基因组ID:BGIBMGA003057-TA),获得其全长序列为1 752 bp,编码583个氨基酸,并对其组织表达特异性、基因结构和序列特征进行了分析,表明Bmamy2在家蚕中肠和脂肪体中都有表达,且在中肠的表达丰度较高。家蚕淀粉酶是一个多基因家族,至少包含11个以上不同基因拷贝,1997年Janecek[7]报道了在研究过的所有生物中,α-淀粉酶蛋白质的三级结构和几个氨基酸基序(amino acid motifs)均十分保守,而富于变化的是内含子的数目和插入位点。黄健华等[8,9]和杜周和等[10]发现家蚕的α-淀粉酶基因在不同家蚕品种间具有多态现象,认为家蚕淀粉酶是一个与家蚕的起源和进化密切相关的分子标记基因,值得更深入的研究和探讨[9]。
以深入了解家蚕淀粉酶的基因结构和表达水平为目的,本实验根据在家蚕转录组数据库中搜索的结果,预测到含有编码家蚕淀粉酶完整ORF的cDNA序列,克隆并测序验证了家蚕两个α淀粉酶基因,并对其基因结构及序列进行分析,利用实时荧光定量PCR技术检测这2个基因在家蚕幼虫5龄3 d不同组织中的表达水平,为进一步研究家蚕淀粉酶基因的多态性提供基础信息。
1材料与方法
1.1材料与主要试剂
PMD 18-T载体购自TaKaRa公司。E.coli菌株TOP10、BL21(DE3)菌株、pET22b原核表达载体为中国农业科学院生物技术研究所保存,家蚕供试品种JY1由江苏科技大学蚕业研究所提供。
Trizol试剂购自Invitrogen公司;逆转录酶M-MLV、限制性内切酶BamHⅠ、SalⅠ、T4 DNA连接酶购自Promega公司;LATaq酶、DEPC购自TaKaRa公司,DNS、无水CaCl2、AP、Tris碱、IPTG、Ampicillin购自Sigma公司,TEMED(N,N,N’,N’-四甲基乙二胺)为瑞典LKB Bromma公司产品,其他化学试剂均为国产分析纯。
1.2仪器
离心机、冷冻离心机购自Sigma公司;超净工作台购自苏州净化设备有限公司;核酸电泳系统、凝胶成像仪购自Bio-Rad公司;SDS-PAGE电泳系统、iQ5荧光定量PCR仪购自Bio-Rad公司。
1.3实验方法
1.3.1Bmamy5和Bmamy7基因的获得在家蚕转录组数据库Silk Trans DB(http://124.17.27.136/gbrowse2/)[11]中用家蚕淀粉酶氨基酸保守序列进行TBlastn,根据比对结果在Silk Trans DB的Genome Browser中查询含有编码家蚕淀粉酶的基因序列,将外显子拼接,初步预测到含有编码家蚕淀粉酶完整ORF的核苷酸序列。
1.3.2Bmamy5和Bmamy7基因序列及编码蛋白质序列的生物信息学分析运用DNAStar软件分析目的基因核苷酸序列的分子质量、碱基组成和碱基分布。SignalP 4.1 Server分析蛋白质是否存在信号肽,运用PBIL LYON-GERLAND信息库对蛋白质序列进行二级结构预测,主要是Hopfield神经网络(HNN)预测,SMART分析蛋白质的结构域。将目的基因序列用Blast(http://blast.ncbi.nlm.nih.gov/Blast.cgi)程序进行检索,并下载具有代表性的淀粉酶氨基酸序列,利用Clustal X和GeneDoc软件将这些序列进行同源性比对。
1.3.3荧光定量PCR检测Bmamy5和Bmamy7在家蚕不同组织中的表达取5龄3 d家蚕幼虫中肠、脂肪体、马氏管、血细胞、丝腺和头部组织,用Trizol法提取家蚕各组织的总RNA,cDNA第1链以Oligo d(T)为引物,在反转录酶M-MLV作用下合成cDNA。以家蚕各组织cDNA为模板,在家蚕淀粉酶基因Bmamy5和Bmamy7基因CDS序列附近设计特异性引物Bmamy5F1、Bmamy5R1、Bmamy7F1、Bmamy7R1(引物序列见表1)进行PCR,并以BmActinA3为对照验证反转录效果。
扩增产物以1%的琼脂糖凝胶检测。初步检测Bmamy5和Bmamy7基因在各组织中的转录情况,并根据目的基因序列,设计荧光定量特异性引物Bmamy5F2、Bmamy5R2、Bmamy7F2、Bmamy7R2(引物序列见表1),检测目的基因在各组织中的转录水平。

表1 本研究所用引物
测定含有目的基因的质粒浓度,按1010~104个拷贝/μL进行梯度稀释作为标准样,每一个样品设3个重复,进行荧光定量PCR。以所得Ct值对相应的拷贝数的对数作图,即得该基因扩增的标准曲线及其线性方程y=ax+b。取1 μL家蚕各组织的cDNA做模板,进行荧光定量PCR,在BioRad iQ5 real-time PCR检测系统上进行操作。PCR扩增程序为:95℃预变性3 min;95℃ 15 s,58℃ 15 s,72℃ 20 s,40个循环;最后以每10 s上升0.5℃的速度从60℃到95℃记录溶解曲线;对荧光定量PCR结果进行分析,根据标准曲线方程计算x值,则待测基因的拷贝数Y=10x。
1.3.4RT-PCR克隆家蚕淀粉酶基因以家蚕中肠组织cDNA为模板,以家蚕淀粉酶基因Bmamy5和Bmamy7基因序列设计特异性引物Bmamy5F3、Bmamy5R3、Bmamy7F3、Bmamy7R3(引物序列见表1)进行反转录PCR,扩增产物以1%的琼脂糖凝胶检测。
1.3.5Bmamy5和Bmamy7基因的克隆及表达根据预测的编码家蚕淀粉酶的CDS序列设计包含SalⅠ和BamHⅠ酶切位点的特异性上、下游引物Bmamy5F4、Bmamy5R4、Bmamy7F4、Bmamy7R4(引物序列见表1)以家蚕5龄3 d中肠cDNA为模板进行PCR扩增,PCR反应体系(25 μL):10×PCR缓冲液2.5 μL,5 mmol/L dNTP 0.5 μL,20 μmol/L上、下游引物各0.5 μL,cDNA 0.5 μL,LATaqDNA聚合酶0.5 μL,加水补足25 μL。反应程序:95℃预变性5 min;95℃ 30 s,57℃ 40 s,72℃ 2 min,30个循环;72℃延伸10 min。PCR扩增产物用玻璃奶法回收,将回收产物与PMD18T连接进行TA克隆,转化大肠杆菌感受态TOP10,提取质粒,酶切鉴定筛选阳性克隆,质粒送擎科生物技术公司测序验证。将验证正确的质粒进行SalⅠ和BamHⅠ双酶切,用T4 DNA连接酶连接到同样经过SalⅠ和BamHⅠ双酶切的pET22b载体上,构建重组表达质粒pET22b-Bmamy5和pET22b-Bmamy7,转化大肠杆菌BL21表达菌株,当菌液OD值达到0.8左右时,加入终浓度为0.5 mmol/L的IPTG诱导表达,继续培养5 h之后,将诱导表达Bmamy5和Bmamy7淀粉酶的大肠杆菌菌液进行超声波破碎,分离上清和沉淀,与未诱导大肠杆菌菌液的上清和沉淀一起进行SDS-PAGE电泳。
设计Bmamy7基因的含BamHⅠ和EcoRⅠ酶切位点的特异性上下游引物Bmamy7F5、Bmamy7R5(引物序列见表1)。将Bmamy7基因连接到pVL1393载体中,测序确证后将含有目的基因片段的pVL1393与线性化的杆状病毒共转染BmN细胞,收集共转染液并纯化,感染5龄家蚕幼虫,待家蚕发病后取蚕血淋巴样品进行淀粉酶活性的检测。
2结果与分析
2.1家蚕淀粉酶基因序列的获取及分析
用NCBI上已知的家蚕淀粉酶蛋白质序列的保守结构域在家蚕转录组数据库中进行TBlastn,根据比对结果在SilkTransDB的Genome Browser中查询基因完整的核苷酸序列,得到9个编码家蚕淀粉酶的基因,家蚕基因组ID分别为BGIBMGA006066、BGIBMGA006067、BGIBMGA005068、BGIBMGA003055、BGIBMGA003056、BGIBMGA-003057、BGIBMGA005240、BGIBMGA005239、BGIBMGA001876,用由这些基因推导的氨基酸序列在NCBI和SMART上进行结构域预测,9个基因都具有α-淀粉酶功能域,但其中有7个不具有淀粉酶C端结构,因此,选取具有C端结构的两个基因BGIBMGA005240、BGIBMGA001876(即Bmamy5与Bmamy7)进行研究分析。
2.2Bmamy5和Bmamy7基因的生物信息学分析结果
用SMART在线分析表明,淀粉酶由30~403位的氨基酸残基构成淀粉酶典型的Domain A、Domain B和Domain C 3个结构域,共同构成催化中心。Domain A由8个α-螺旋和8个β-折叠组成,是催化位点。Domain B由70个氨基酸组成,位于Domain A的第3个α螺旋β折叠之间,是钙离子结合位点,钙是该酶必需的辅助因子。Domain C由β折叠组成一个希腊钥匙结构[3,11],蛋白质的C端包含于Domain C,与α淀粉酶的酶活性有关[12]。
SMART在线预测及Blast比对检测到Bmamy7基因编码的蛋白质序列具有典型的α-淀粉酶结构域,如图1所示,119位的天冬酰胺和182位的天冬氨酸是钙离子结合部位,212位和314位的天冬氨酸以及249位的谷氨酸是该蛋白的催化位点。
利用SignalP 4.1 Server分析Bmamy5和Bmamy7蛋白,结果表明Bmamy5和Bmamy7蛋白都存在信号肽,信号肽剪切位点分别在第17位和第19位氨基酸。 利用Protscale程序分析可知Bmamy5和Bmamy7蛋白序列大部分为亲水区域,少数部分为疏水区域。

图1 Bmamy5和Bmamy7基因编码蛋白质功能结构域Fig.1 Domain structure of protein coded by Bmamy7 and Bmamy5 gene. A(1):Bmamy5蛋白的SMART结构预测;A(2):Bmamy7蛋白的SMART结构预测;B(1):Bmamy5蛋白的保守结构分析;B(2):Bmamy7蛋白的保守结构分析。
下载与Bmamy5和Bmamy7相似性较高的淀粉酶氨基酸序列,用MAGA6.0构建进化树,从图2中可以看出,Bmamy5与柑橘凤蝶(Papilioxuthus)和蓓带夜蛾(Mamestraconfigurata)的亲缘关系较近,而Bmamy7 与小蔗杆草螟(Diatraeasaccharalis)的亲缘关系较近。
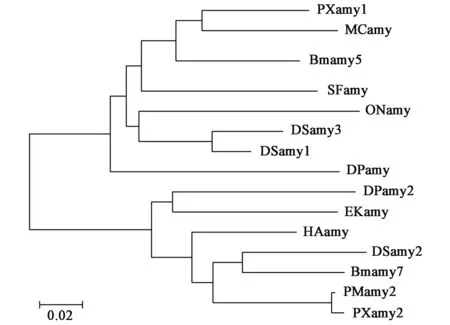
图2 Bmamy5、Bmamy7和其他物种淀粉酶的进化分析图Fig.2 Evolutionary tree of Bmamy5, Bmamy7 and amylase in other species.DS: 小蔗杆草螟(Diatraea saccharalis);DP:大红斑蝶(Danaus plexippus);PX:柑橘凤蝶(Papilio xuthus);MC:蓓带夜蛾(Mamestra configurata);SF:草地贪夜蛾(Spodoptera frugiperda);EK:地中海粉螟(Ephestia kuehniella);PM:金凤蝶(Papilio machaon);ON:玉米螟(Ostrinia nubilalis);HA:棉铃虫(Helicoverpa armigera);Bm:家蚕(Bombyx mori)
2.3荧光定量PCR检测Bmamy5与Bmamy7基因在各组织中的转录情况
根据1.3.3设计的引物,以家蚕5龄3 d幼虫的马氏管、头、中肠、血细胞、脂肪体、气管丝、丝腺7个组织cDNA为模板进行RT-PCR分析,检测目的基因在家蚕各组织中的转录情况,用ActinA3基因为对照检测各组织反转录产物。结果如图3A所示,Bmamy5只在中肠组织中转录,Bmamy7在中肠组织中高量转录,在马氏管中也有转录,但转录水平较低。
荧光定量PCR所得Bmamy5与Bmamy7基因的标准曲线为分别为:Y=-2.972 1X+39.732,R2=0.993 3和Y=-3.834 6X+41.844,R2=0.996 5。根据标准曲线计算的各组织目的基因的拷贝数如图3B所示,家蚕淀粉酶基因Bmamy7在马氏管和中肠组织中有转录,在中肠的转录可达3×109个拷贝/μg总RNA以上,而在马氏管只有1.9×106个拷贝/μg总RNA,Bmamy5只在中肠中有转录,有1.5×109个拷贝/μg总RNA,而在其他组织中难以检测到。
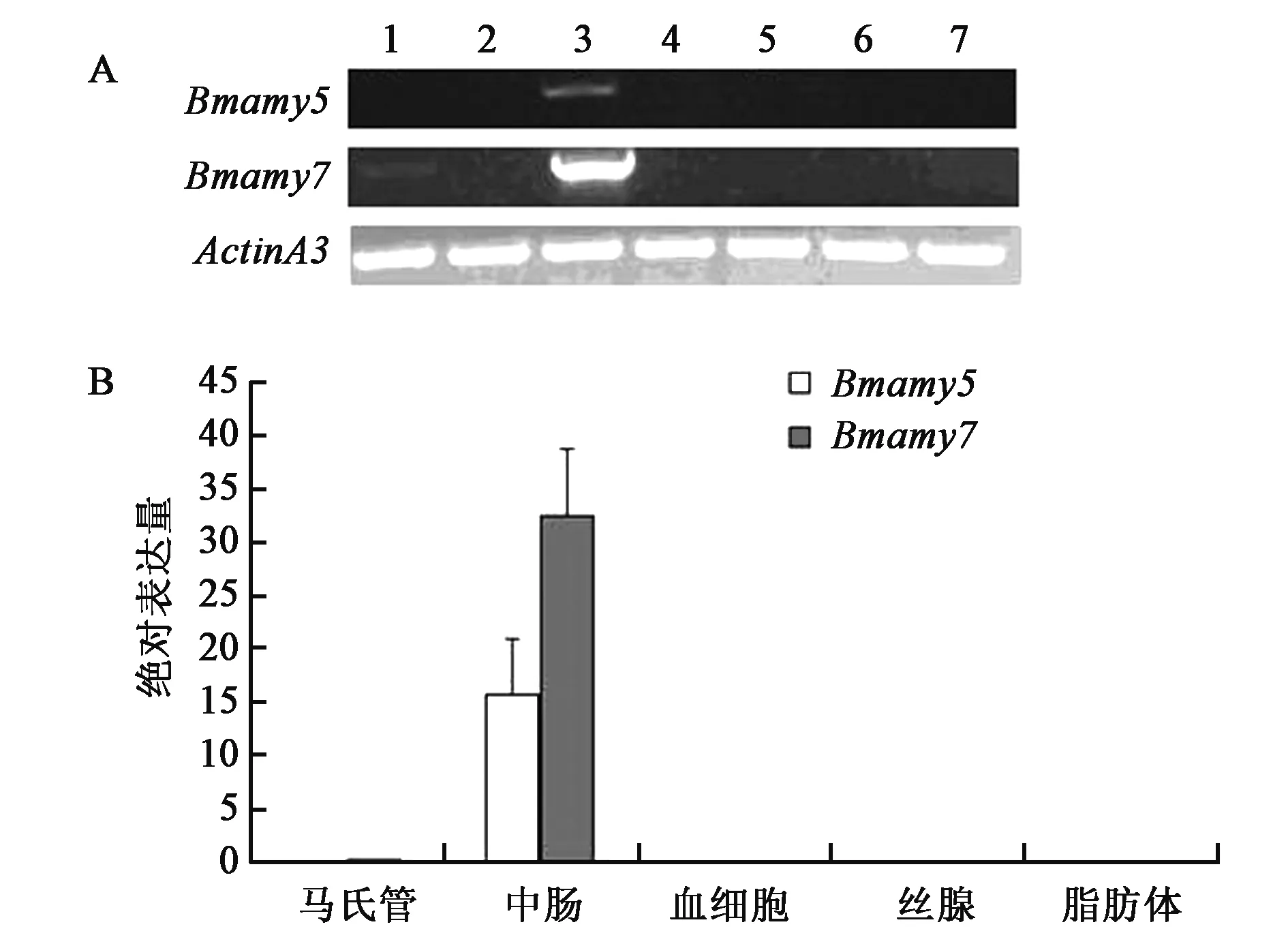
图3 家蚕5龄3天幼虫不同组织中Bmamy7和Bmamy5的转录Fig.3 The transcription of Bmamy7 and Bmamy5 in different tissues of Bombyx mori.A:反转录PCR鉴定Bmamy7和Bmamy5在家蚕各组织中的转录。1:马氏管;2:头部;3:中肠;4:血细胞;5:脂肪体;6:气管丝;7:丝腺。B:荧光定量PCR定量分析Bmamy7和Bmamy5在家蚕各组织中的转录水平。
2.4Bmamy5与Bmamy7基因的克隆
根据1.3.4设计的引物,以家蚕5龄3 d幼虫中肠组织cDNA为模板进行PCR扩增Bmamy5与Bmamy7基因,Bmamy5基因长1 196 bp,Bmamy7基因片段长1 608 bp,产物经1%的琼脂糖凝胶电泳,结果如图4所示,PCR产物大小与预期大小一致。
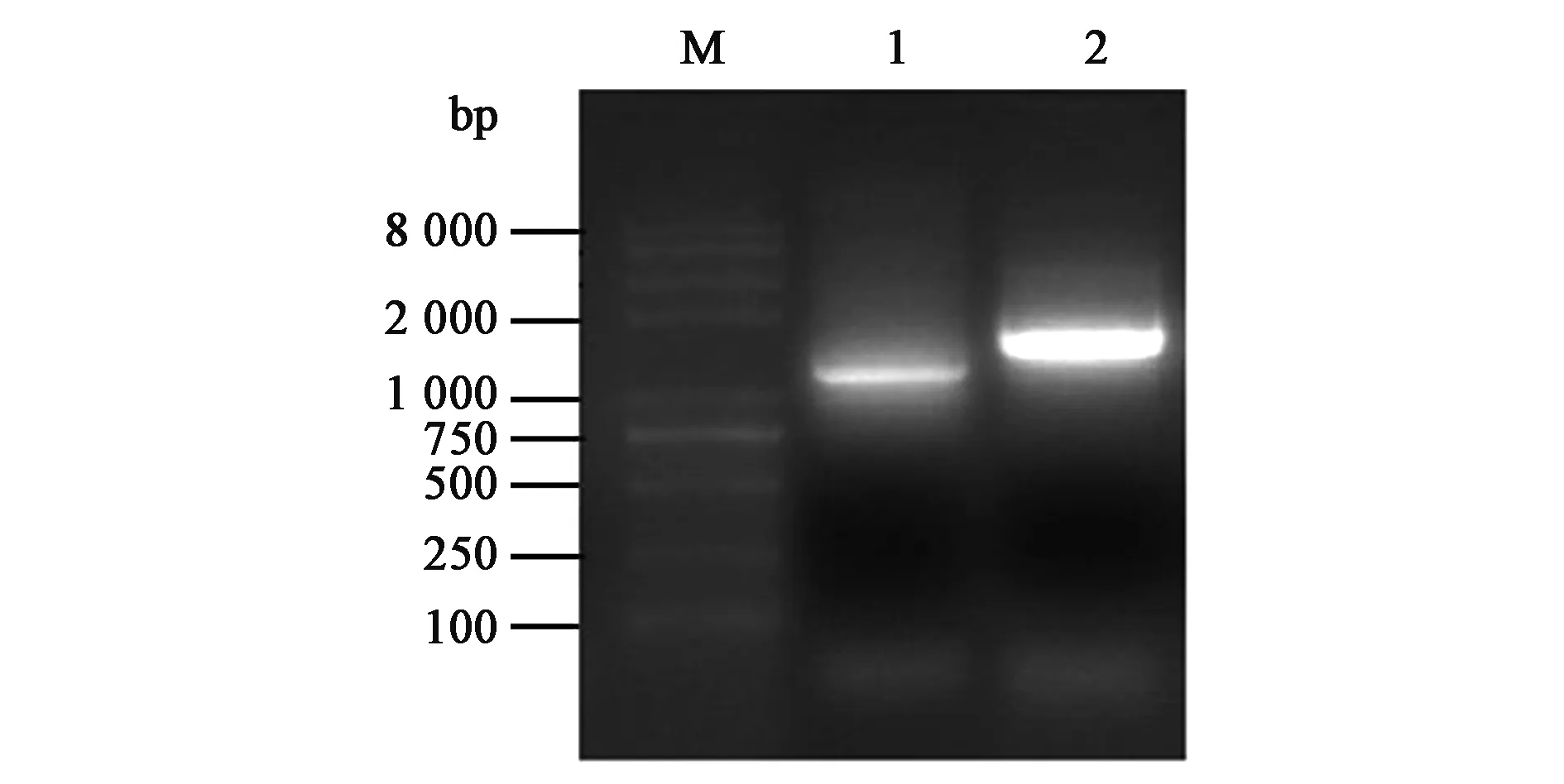
图4 RT-PCR扩增目的片段电泳图Fig.4 The electropherogram of amplified products of purpose fragments by RT-PCR.M:DNA分子质量标准;1:家蚕淀粉酶基因Bmamy5;2:家蚕淀粉酶基因Bmamy7
2.5Bmamy5与Bmamy7基因的克隆和表达载体鉴定
如图5所示,Bmamy7在大肠杆菌中表达的产物存在于包涵体沉淀中,采用低温诱导得到的Bmamy7重组蛋白仍然是以包涵体的形式存在,且在48~63 kDa之间有一条明显的条带,与预测的Bmamy7表达的蛋白质分子量61.69 kDa大小相符,但未能检测到表达产物具有明显的淀粉酶活性。然而Bmamy5未能检测出明显的表达条带,经过各种优化诱导条件,依然没有明显目的条带出现。
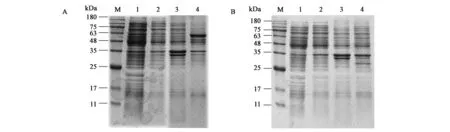
图5 大肠杆菌表达家蚕淀粉酶Bmamy7和Bmamy5的SDS-PAGE检测Fig.5 SDS-PAGE detection of amylase Bmamy7 and Bmamy5 expressed in E.coliA: SDS-PAGE检测Bmamy7原核表达;M:蛋白质分子质量标准;1:未诱导大肠杆菌菌液上清;2:0.5 mmol/L IPTG诱导Bmamy7大肠杆菌菌液上清;3:未诱导大肠杆菌菌液沉淀;4:0.5 mmol/L IPTG诱导Bmamy7大肠杆菌菌液沉淀;B:SDS-PAGE检测Bmamy5原核表达;1:未诱导大肠杆菌菌液上清;2:0.5 mmol/L IPTG诱导Bmamy5大肠杆菌菌液上清;3:未诱导大肠杆菌菌液沉淀;4:0.5 mmol/L IPTG诱导Bmamy5大肠杆菌菌液沉淀
根据1.3.5的方法将Bmamy7基因在家蚕体内进行了表达,但是表达的蚕血淋巴样品中淀粉酶的活性与对照组相比并没有明显差别,具体原因有待于进一步研究。
3讨论
通过荧光定量PCR对家蚕淀粉酶基因Bmamy5和Bmamy7在家蚕幼虫不同组织中的分布进行了研究,结果表明家蚕淀粉酶基因存在组织特异性,家蚕淀粉酶基因Bmamy7在马氏管和中肠组织中有转录,中肠中的转录丰度较高,而Bmamy5只在中肠中有转录。因为中肠是家蚕的主要消化器官,桑叶中淀粉的水解和利用主要发生在中肠中,所以淀粉酶基因在中肠中的转录水平会比较高,而马氏管紧贴于中肠,推测有可能会对淀粉有部分的水解作用,但是相对于中肠而言,马氏管消化淀粉的作用是十分微弱的。
通过生物信息学初步分析了Bmamy5和Bmamy7基因序列及编码蛋白质的结构和部分性质,信号肽分析表明基因Bmamy5编码的蛋白在N端前17个氨基酸区域有可剪切的信号序列,而Bmamy7编码的蛋白序列前19个氨基酸区域内有可剪切的信号序列;二级结构预测显示Bmamy5和Bmamy7蛋白主要由α-螺旋、β-折叠和无规则卷曲构成;通过Bmamy5、Bmamy7和其他物种淀粉酶的进化关系分析,表明Bmamy5与柑橘凤蝶和蓓带夜蛾的亲缘关系较近,而Bmamy7与小蔗杆草螟的亲缘关系较近。
目前,对于昆虫体内淀粉酶的研究并不多,而且研究多从淀粉酶基因的高度保守性进行遗传进化分析,或从同源性方面来推断其功能。真正通过异源表达来确证其酶学功能和特性的研究还很少,因此,本实验试图将家蚕淀粉酶基因Bmamy5和Bmamy7克隆到原核表达载体pET22b,通过IPTG诱导表达,期望表达出有活性的家蚕淀粉酶。然而,没有检测到Bmamy5的表达产物,Bmamy7表达产物也仅存在于包涵体沉淀中,经过多次的变性和复性实验,用DNS法也未能检测到明显的淀粉酶活性。推测可能是因为该酶来源于真核生物中,只有通过较复杂的修饰过程才能形成正确的空间结构,形成具有生物学活性的蛋白质。对此,我们利用杆状病毒表达系统,在家蚕中表达了Bmamy7,但是结果并不理想,推测这可能与家蚕淀粉酶本身的活性比较弱有关[13],并且家蚕淀粉酶基因可能较难进行外源表达,具体原因还有待于进一步探索研究。
参考文献
[1]廖 芳, 李关荣, 鲁 成,等. 家蚕淀粉酶研究进展[J]. 蚕学通讯, 2002, 22(3): 30-35.
[2]Vihinen M, Mantsala P. Microbial amylolytic enzymes[J]. Crit. Rev. Biochem. Mol. Biol., 1989,24: 329-418.
[3]Pujadas G, Palau J. Evolution of alpha-amylases: architectural features and key residues in the stabilization of the (beta/alpha)(8) scaffold[J]. Mol. Biol. Evol., 2001,18: 38-54.
[4]Nielsen J E, Borchert T V. Protein engineering of bacterial alpha-amylases[J]. Biochim. Biophys. Acta, 2000,1543: 253-274.
[5]Chatterjee S N, Rao C G. Chatterjee G K. Genetic variability of amylase activity in the mulberry silkworm Bombyx mori and its significence[J]. Sericologia, 1992, 32:671-683.
[6]杜周和, 董占鹏, 鲁 成,等. 家蚕淀粉酶基因(Bmamy2)的分子克隆及表达特征和序列分析[J]. 蚕业科学, 2008, 34(4): 642-649.
[7]Janecek S. Alpha-amylase family: molecular biology and evolution[J]. Prog. Biophys. Mol. Biol., 1997, 67: 67-97.
[8]黄健华, 苗雪霞, 李木旺,等. 家蚕基因特异性CAPs标记获得及其分子系统学应用[J]. 遗传, 2005, 27(4): 584-588.
[9]黄健华, 贾世海, 张 勇,等. 家蚕α淀粉酶基因的多态性研究[J]. 中国农业科学, 2006, 39(11): 2390-2394.
[10]杜周和, 刘俊凤, 陈义安,等. 家蚕淀粉酶基因系统发育及遗传多样性[J]. 西南农业学报, 2014, 27(1): 442-449.
Li Y, Wang G, Tian J,etal.. Transcriptome analysis of the silkworm (Bombyxmori) by high-throughput RNA sequencing[J]. PLoS ONE, 2012,7(8), e43713.
[11]Matsui I, Svensson B. Improved activity and modulated action pattern obtained by random mutagenesis at the fourth beta-alpha loop involved in substrate binding to the catalytic (beta/alpha)8-barrel domain of barley alpha-amylase 1[J]. J. Biol. Chem., 1997,272:22456-22463.
[12]Vihinen M, Peltonen T, Mantsala P,etal.. C-terminal truncations of a thermostable Bacillus stearothermophilus alpha-amylase[J]. Protein Engin., 1994,7: 1255-1259.
[13]向仲怀. 家蚕遗传育种学[M]. 北京:中国农业出版社,1994, 73-77.
Cloning, Expression and Sequence Analysis of Amylase Gene in the Silkworm,Bombyxmori
LV Yan-na1,2, CHEN Xiao-yan1,2, SHAO Li-ping1,2, LI Yi-nv2, HU Xiao-yuan2, ZHANG Zhi-fang2, YI Yong-zhu1*
1.JiangsuUniversityofScienceandTechnology,JiangsuZhenjiang212018,China;2.BiotechnologyResearchInstitute,ChineseAcademyofAgriculturalSciences,Beijing100081,China
Abstract:Amylase is one of the important digestive enzymes in silkworm and known for hydrolyzing starch and glycogen. The mRNA expression level of the Bmamy7 and Bmamy5 in different tissues of 5th3d larva was analyzed by real-time PCR. Absolute quantitative analysis results indicated, Bmamy7 expressed in midgut was up to 3×109 copies/μg total RNA, its expression level in the malpighian was low, only 1.9×106 copies/μg total RNA; however, Bmamy5 was only expressed in midgut and the mRNA expression level was 1.5×109 copies/μg total RNA. The specific primers were designed according to the cDNA sequence predicted encoding amylase in silkworm transcript database, two amylase genes were cloned by RT-PCR. The complete sequence of Bmamy7 is 1 608 bp, ORF consists 748 bp, encodes 503aa. Amino acid sequence analysis showed that Bmamy7 had a typical structure domain of α-amylase. The complete sequence of Bmamy5 is 1 196 bp,ORF consists 738 bp and encodes 245aa. Bmamy5 and Bmamy7 gene was expressed in prokaryotic expression respectively. The activity of recombinant Bmamy7 protein was very weak. The expression product of Bmamy5 was not detected by SDS-PAGE. The results provided references for research and application of amylase gene in the silkworm.
Key words:Bombyx mori; amylase gene; tissue-specific expression; real-time quantitative PCR
收稿日期:2016-01-18; 接受日期:2016-03-03
基金项目:国家863计划项目(2011AA100603);国家973计划项目(2012CB114600)资助。
作者简介:吕言娜,硕士研究生,研究方向为家蚕分子生物学。E-mail:lvyannall@163.com。*通信作者:易咏竹,副研究员,研究方向为昆虫表达系统和病毒基因工程。E-mail:yiyongzhu@126.com
DOI:10.3969/j.issn.2095-2341.2016.03.08
