与甘蔗抗黑穗病性相关的14—3—3基因的克隆和表达分析
2016-05-30肖新换阙万才黄宁刘峰苏炜华吴期滨苏亚春
肖新换 阙万才 黄宁 刘峰 苏炜华 吴期滨 苏亚春


摘 要 為了解14-3-3基因在甘蔗黑穗病抗性中的作用,本研究以课题组前期构建的甘蔗抗黑穗病抑制消减杂交文库中筛选出的1个与14-3-3基因同源的EST序列作为探针,利用电子克隆和RT-PCR方法从甘蔗ROC22品种中分离到1个Sc14-3-3基因(GenBank Accession Number:KJ577592),并对其序列信息和基因表达情况进行分析。结果显示,该基因ORF长771 bp,编码256个氨基酸残基;Sc14-3-3是定位于细胞质的亲水蛋白,无跨膜螺旋区及信号肽,有10个Ser、3个Thr和7个Tyr潜在的磷酸化位点,并在N-端有4个蛋白结合区和1个多核苷酸结合区,对翻译和能量新陈代谢起着重要作用;该蛋白属非ε类群,其N-端和中间区域在不同物种间相对保守,C-端差异相对较大。基因表达模式分析显示,Sc14-3-3基因可能参与甘蔗对生物逆境的细胞免疫响应,其对MeJA介导的信号途径应答较SA和ABA早;黑穗病菌胁迫下,Sc14-3-3基因在甘蔗抗感品种中的表达模式存在差异,其在抗黑穗病品种YC05-179中上调表达且表达量变化明显,揭示了该基因参与甘蔗对黑穗病的应答反应。以上结果为甘蔗Sc14-3-3基因的功能研究积累了一定的基础资料,并为今后甘蔗的抗病分子育种提供后备基因资源。
关键词 甘蔗;14-3-3基因;生物信息学;荧光定量PCR
中图分类号 S566.1 文献标识码 A
Abstract In order to understand the function of 14-3-3 gene in sugarcane smut resistance, based on an EST highly similar to 14-3-3 gene selected from the suppression subtractive hybridization library corresponding to sugarcane smut resistance, we cloned a Sc14-3-3 gene(GenBank Accession Number: KJ577592)from sugarcane ROC22, by electronic cloning using this EST as the probe to search the dbEST couple with RT-PCR amplification method. Sequence information and expression patterns of Sc14-3-3 gene were analyzed. The results showed that the Sc14-3-3 gene contained a complete ORF(771 bp)encoding 256 amino acid residues. Sc14-3-3 was a hydrophilic protein without signal peptides and transmembrane helix localized in the cytoplasm. The protein contained 10 serine, 3 threonine and 7 tyrosine potential phosphorylation sites and had four protein binding regions and a polynucleotide binding domain in its N-end, which played an important role in the translation and energy metabolism. Amino acid homology analysis revealed that the protein belonged to non-ε groups, and its N-terminal and middle regions were relatively conservative in different species, C-terminal relatively larger difference. The expression analysis showed that the Sc14-3-3 gene might be involved in the immune response which was caused by biotic stress. The quantitative results suggested an earlier reply to MeJA signaling pathway than in SA and ABA stresses. Under the smut pathogen stress, the expression patterns of Sc14-3-3 were different between sugarcane susceptible cultivar ROC22 and resistant cultivar YC05-179. Compared with ROC22, Sc14-3-3 gene was up-regulated and had significant expression change in YC05-179. The phenomenon indicated that the Sc14-3-3 gene involved in the smut resistance of sugarcane cultivars. This study will accumulate the basic information for its functions to further identified, and provide back-up genetic resources for future molecular disease-resistant breeding in sugarcane.
Key words Sugarcane; 14-3-3 gene; Bioinformatics; Real-time PCR
doi 10.3969/j.issn.1000-2561.2016.01.019
1967年,Mooore等[1]在牛脑细胞中首次发现一种大小在25~32 ku之间的异源二聚体酸性可溶蛋白,根据二乙氨乙基纤维素柱(DEAE cellulose)层析分离出片段数及其在淀粉凝胶电泳上的迁移率,命名为14-3-3蛋白。在之后的很长一段时间里,14-3-3蛋白被认为是一种脑特异蛋白,直到它首次在4种不同的植物中得到报道,即大麦(Hordeum vulgare)[2]、拟南芥(Arabidopsis thaliana)[3]、菠菜(Spinacia oleracea)和月见草(Oenothera biennis)[4]。随后的研究显示,14-3-3蛋白存在于几乎所有的真核生物中[5]。迄今,已在多种植物中分离克隆到该蛋白基因,如大麦[2]、菠菜和月见草[3]、水稻(Oryza sativa)[6]、玉米(Zea mays)[7]、橡胶树(Hevea brasiliensis)[8]、拟南芥[3,9]等。研究表明,14-3-3蛋白的分布具有组织特异性,主要分布于细胞质中[10],同时也分布于质膜[11]、类囊体膜[12]、线粒体基质[13]、叶绿体基质[14]以及细胞核[15]上。Sehnke等[16-17]研究发现14-3-3蛋白在亚细胞中的定位与其蛋白结构上的不同异构型有关。此外,研究还揭示14-3-3蛋白序列高度保守,不同家族成员在结构上都表现出极大的相似性[18]。动植物14-3-3蛋白同工型具有70%的相似性,然而却不存在进化的同源性[19]。
前人研究表明,14-3-3蛋白属于生物细胞中重要的调节蛋白,通常以同源或者异源二聚体的形式存在,通过识别特异的磷酸化序列,与多种靶标信号蛋白结合,调控基因的表达[20]。其中,磷酸化的丝氨酸和苏氨酸残基的靶蛋白较易与14-3-3蛋白结合,调节植物的基础代谢、物质运输及植物病害和胁迫应答反应等[21]。Seehaus等[22]利用DDRT-PCR(differential display reverse transcription PCR,差异显示反转录PCR)技术筛选感染假单孢菌(Pseudomonas syringae pv. glycinea)大豆(Glycine max)的超敏反应中的差异表达基因,获得了一个表达量增加的14-3-3基因。Hill等[23]在所构建的棉花(Gossypium spp.)根部感染枯萎病菌(Fusarium oxysporum f. sp. vasinfectum)24 h的文库中,检测到了14-3-3基因的转录积累。Finnie等[24]在无毒白粉病菌(Blumeria graminis)感染后的大麦表皮细胞层中,发现14-3-3基因和H+-ATP酶基因的表达量增加,同时14-3-3蛋白和H+-ATP酶复合物同步增加。蔡英卿等[25]以龙眼(Dimocarpus longan)为研究材料发现,龙眼体胚发生过程中,在与转录因子相互作用下,14-3-3蛋白参与激素信号转导途径,调控胚性相关基因的表达;同时,该蛋白在结合质膜H+-ATP酶和钾离子通道蛋白后,参与龙眼体胚发生过程胚性维持或形态建成。Yan等[26]以拟南芥为实验材料,使用酵母双杂技术证实,锚蛋白重复序列和14-3-3蛋白家族可以相互作用并参与介导防卫应答的PR1表达负调控。罗炼芳等[27]研究发现,甘蔗(Saccharum spp.)14-3-3基因(GenBank Accession Number:AY222859)在不同组织中均有表达,且在甘蔗的茎中,14-3-3基因有着较高的表达水平,推测14-3-3基因可能参与了多种信号通路,并且与甘蔗的糖代谢相关。
甘蔗是世界上最重要的糖料作物,同时,甘蔗作为C4作物,具有较强的光合速度和干物质积累能力,是最具有发展潜力的可再生能源作物。然而,在生产过程中,甘蔗病害和不良农艺性状严重影响甘蔗的生长及产量。因此,抗性和優良农艺性状的研究非常紧迫,而挖掘与此相关的基因是首要基础。植物14-3-3蛋白与其靶蛋白相互作用,调控细胞信号转导途径、参与生物和非生物胁迫及病原防卫应答[28]。目前,有关14-3-3基因的研究已有不少相关报道,但甘蔗中仅见有关14-3-3基因的组织特异性表达及其与糖代谢和矿物元素吸收之间关系的初步探讨[27],尚未见14-3-3基因在甘蔗抗病性中功能及其作用途径的研究报道。
SSH(suppression subtractive hybridization,抑制消减杂交)技术以抑制PCR扩增为基础,能够高效、快速的筛选2个不同生物材料间的差异表达基因,尤其适合于甘蔗这种基因组尚不完全清晰的物种[29]。前期,本课题组利用SSH技术研究甘蔗抗黑穗病性的分子机制,获得了一些与甘蔗黑穗病抗性相关的基因或ESTs。为此,本研究以黑穗病菌胁迫下,甘蔗ROC22品种SSH文库中筛选出的与14-3-3基因同源的EST序列为探针,应用电子克隆及RT-PCR技术从甘蔗中分离获得Sc14-3-3蛋白基因,并对其序列特征与基因表达情况进行分析,以期获得该基因功能的基础资料,为研究甘蔗抗黑穗病分子机制奠定一定的理论基础,及为利用转基因技术培育抗黑穗病甘蔗品种提供基因资源。
1 材料与方法
1.1 材料
甘蔗品种ROC22(感黑穗病品种)和崖城05-179 (YC05-179,抗黑穗病品种)由福建农林大学农业部福建甘蔗生物学与遗传育种重点实验室提供。具体研究材料如下:(1)取ROC22植株的侧芽用于RT-PCR扩增验证;(2)水培法处理ROC22和YC05-179的蔗茎,并在蔗芽生长点部位针刺接种5×106个/mL的黑穗病菌孢子,对照组接种无菌水。分别取0、24、48、72、120 h的蔗芽,用于生物逆境胁迫表达分析研究;(3)沙培法培育YC05-179的蔗茎至其长出第四或第五叶,选取长势一致的蔗苗洗净沙子,于大烧杯中复水1周,分别用水杨酸(salicylic acid,SA,5 mmol/L)、茉莉酸甲酯(methyl jasmonate,MeJA,100 μmol/L)和脱落酸(abscisic acid,ABA,100 μmol/L)进行喷射叶面处理,并取处理6、12、24 h的蔗叶用于非生物逆境胁迫表达分析研究,同时以0 h未处理的蔗叶作为对照。以上所有取材均立即用液氮速冻,然后保存于-80 ℃冰箱,供实时荧光定量PCR(real time quantitative PCR,RT-qPCR)检测用。
1.2 方法
1.2.1 总RNA的提取和cDNA的合成 采用Trizol法提取甘蔗材料的总RNA,经检测合格后再利用TaKaRa公司的PrimeScriptTM RT-PCR Kit反转录试剂盒合成第一链cDNA,-20 ℃保存备用。
1.2.2 甘蔗Sc14-3-3基因的电子克隆及RT-PCR扩增验证 以黑穗病菌胁迫下,甘蔗ROC22品种SSH文库中筛选出的与14-3-3基因同源的EST序列为探针,在甘蔗EST数据库中进行tblastn搜索,找到与此探针序列同源性较高的EST序列,并使用DNAStar软件中的SeqMan对相关的Unigene进行拼接,得到全长cDNA序列,然后运用NCBI中的Conserved Domains(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)分析该序列的保守结构域,并用ORF Finder在线程序(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)分析其开放阅读框。
使用Primer 5.0对拼接获得的电子克隆序列进行RT-PCR扩增引物设计,引物信息如表1中的Sc14-3-3a。参考TaKaRa公司的ExTaq酶说明书,以ROC22蔗芽cDNA为模板进行RT-PCR扩增。其扩增程序为:94 ℃ 4 min;94 ℃ 30 s,52 ℃ 30 s,72 ℃ 1 min,35个循环;72 ℃ 10 min。以1.0%琼脂糖凝胶电泳进行扩增产物的检测,当产物片段大小与电子克隆预测大小相同时,使用Gel Extraction Kit纯化回收该PCR扩增产物,进行TA克隆并送测序。
1.2.3 生物信息学分析 使用多种软件及在线分析工具进行生物信息学分析,所用的主要数据库及工具列于表2。
1.2.4 甘蔗Sc14-3-3基因的RT-qPCR引物设计及筛选 根据RT-qPCR引物设计原则,并以该目的基因的保守序列为参考,用AlleleID7.6软件设计荧光定量PCR引物,引物序列见表1中的Sc14-3-3b。将逆转录模板按照2倍稀释法稀释,反应体系依SYBR Green PCR Master Mix试剂盒(Roche)说明书进行配置,按照程序(50 ℃ 2 min;95 ℃ 10 min;95 ℃ 15 s,60 ℃ 1 min,45个循环)在ABI 7500荧光定量PCR仪上进行实验。实验时每个样品一式3份,反应后进行溶解曲线分析和标准曲线制定,以鉴定引物的特异性及扩增效率是否良好。
1.2.5 甘蔗Sc14-3-3基因的表达量测定及分析
分别以(1)ROC22和YC05-179接种黑穗病菌后0、24、48、72、120 h的蔗芽cDNA,(2)YC05-179经SA、MeJA、ABA诱导0、6、12、24 h的叶片cDNA为模板进行甘蔗Sc14-3-3基因的表达量测定。根据RT-qPCR引物的筛选结果,参考前人的研究选择GAPDH(glyceraldehyde-3-phosphate dehydrogenase)作为内参基因[30](引物序列见表1中的GAPDH),反应时每个样品设置3次重复,按照上述反应体系和反应程序进行扩增。待扩增程序结束后,在ABI PRISM7500 real-time PCR system软件上对数据进行初步筛选分析后,导出定量数据文件,采用2-△△CT算法分析各基因的表达模式。数据分析结果由Excel表格计算得出。
2 结果与分析
2.1 Sc14-3-3基因的电子克隆及RT-PCR验证
以黑穗病菌胁迫下,甘蔗ROC22品种SSH文库中筛选出的与14-3-3基因同源的EST序列为探针,电子克隆到1条拼接序列,其具有完整的开放读码框,编码256个氨基酸,且包含14-3-3基因的保守序列,命名为Sc14-3-3基因(GenBank Accession Number:KJ577592)。实验以ROC22蔗芽的cDNA为模板进行的RT-PCR验证结果如图1所示。PCR产物连接载体、转化、测序后,经NCBI中的blastp比对可知,该基因的氨基酸序列与罗炼芳等[27]报道的甘蔗Sc14-3-3蛋白相同,但编码氨基酸的碱基及该基因的3′端却有所差异(2.66%)(图2),说明甘蔗的不同品种之间对14-3-3基因有碱基偏好性,同时也暗示甘蔗Sc14-3-3家族基因中不同成员之间的序列差异可能主要体现在其3′位置。
2.2 Sc14-3-3基因编码蛋白的氨基酸序列分析
Sc14-3-3基因推导编码氨基酸序列的疏水性/亲水性分析结果显示,整条多肽链没有明显的疏水区,GRAVY值为-0.501,推测甘蔗Sc14-3-3蛋白是一种亲水蛋白。由跨膜结构预测可知,Sc14-3-3蛋白没有跨膜螺旋区,不是跨膜蛋白。信号肽预测结果表明,Sc14-3-3蛋白不含信号肽。根据PredictProtein和NetPhos 2.0 Server在线软件预测可知,该蛋白定位于细胞质(图3);存在20个潜在磷酸化位点,即10个Ser磷酸化位点、3个Thr磷酸化位点和7个Tyr磷酸化位点(图4);结合位点区域位于N-端,包括4个蛋白结合区和1个多核苷酸结合区,以氨基酸残基位点表示分别为第1、15、34、40和58位(图5)。使用Profun 2.2 Server对甘蔗Sc14-3-3蛋白进行功能预测,结果显示,该蛋白的主要功能为翻译和能量代谢。
2.3 Sc14-3-3基因编码蛋白的同源比对及进化分析
以甘蔗Sc14-3-3氨基酸序列为探针,使用NCBI中的BlastP程序,筛选出多个14-3-3蛋白。从中选取的谷子(Setaria italica_gb|XP_004968972.1|)、玉米(Zea mays_gb|NP_001151592.1|)、短花药野生稻(Oryza brachyantha_gb|XP_006659421.1|)、高粱(Sorghum bicolor_gb|XP_002445779.1|)、二穗短柄草(Brachypodium distachyon_gb|XP_003574509.1|)和田野贝母(Fritillaria agrestis_gb|AAC04811.1|)与甘蔗Sc14-3-3蛋白的氨基酸序列相似性分别为99%、99%、98%、98%、95%和91%。使用DNAMAN软件中的多重比对功能,对甘蔗14-3-3蛋白的氨基酸序列与以上6个物种的氨基酸序列进行多序列比对(图6),結果显示,不同物种间,该蛋白的N-端和中间区域相对保守,C-端差异相对较大,推测该基因的结构是甘蔗为适应环境或生长发育所致。
为进一步研究Sc14-3-3蛋白的分类,从NCBI中选取了拟南芥和水稻的8个14-3-3蛋白,并用MEGA5.0软件构建了系统进化树(图7)。结果表明,甘蔗Sc14-3-3蛋白与拟南芥GF14 φ、GF14 ψ、GF14 κ和水稻GF14 f聚在同一分支,拟南芥GF14 ε、GF12 ρ、GF14 μ和水稻GF14 g聚在另一分支。研究显示,植物14-3-3蛋白主要分为2个类群:ε类群和非ε类群[19]。拟南芥GF14 φ、GF14 ψ、GF14 κ和水稻GF14 f为非ε类群,拟南芥GF14 ε、GF12 ρ、GF14 μ和水稻GF14 g为ε类群[6,31],其中,14-3-3基因的ε类群不同于非ε类群的基因之处在于ε类群都有一个相同的外显子结构[31],故推测该Sc14-3-3基因属于非ε类群,无此外显子结构。
2.4 Sc14-3-3基因的RT-qPCR分析
2.4.1 Sc14-3-3基因引物的评估 采用2倍稀释法稀释ROC22蔗芽组织的cDNA模板,其浓度梯度分别为2-1,2-2,2-3,2-4,2-5。图8为Sc14-3-3基因RT-qPCR产物的溶解曲线图,由图8可见,该基因扩增产物的溶解曲线均为单峰,表明该引物(Sc14-3-3b)的特异性较高。根据CT值制定用于扩增Sc14-3-3基因的标准曲线(图9),其相关系数R2=0.995 8∈(0.99,1),表明该曲线的线性关系成立。引物的扩增效率与曲线斜率相关,即扩增效率(E)=10(1/斜率)-1,由此算出引物Sc14-3-3b的E=0.987∈(0.95,1.05),表明扩增效率较高,可用于基因的定量表达分析研究。
2.4.2 甘蔗Sc14-3-3基因在SA、MeJA和ABA胁迫下的表达特性分析 SA[32]、MeJA[33]和ABA[34]等3种激素是良好的生物胁迫模拟剂。在SA、MeJA和ABA胁迫下,甘蔗Sc14-3-3基因的表达特性分析如图10所示。具体地,SA、MeJA、ABA胁迫下,YC05-179中Sc14-3-3基因均上调表达,但表达情况却有所不同:胁迫6~24 h的YC05-179材料中,MeJA处理下Sc14-3-3基因的相对表达量在6 h时急剧增加,约为0 h的4.21倍,之后表达量虽仍在增加,但增加的幅度逐渐降低;SA和ABA胁迫下Sc14-3-3基因的相对表达量均随着处理时间的增加而增加,但在各时间点的相对表达量均较MeJA胁迫下的要低。此外,在12 h时,SA和ABA胁迫对Sc14-3-3基因的表达量的影响效果相似,均约为对照组的2.00倍,但6 h时,SA胁迫下该基因的相对表达量大于ABA胁迫,24 h时却与此相反。从以上研究结果可以推测,甘蔗Sc14-3-3基因與其响应生物胁迫(比如黑穗病菌)的机制有关。
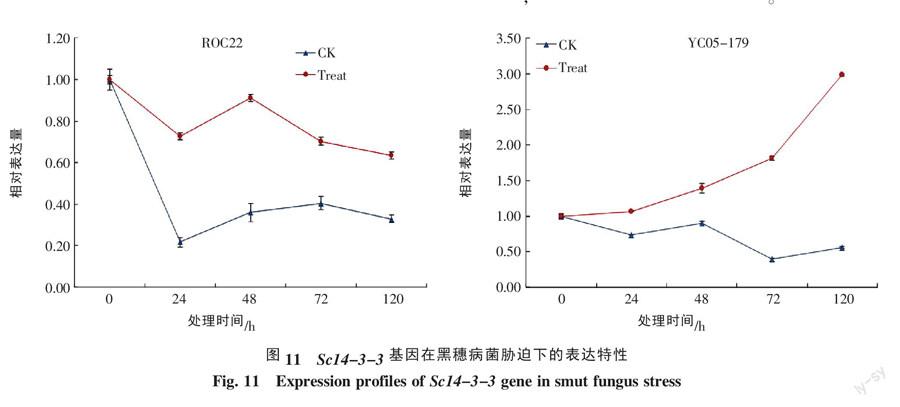
2.4.3 甘蔗Sc14-3-3基因在黑穗病菌胁迫下的表达特性分析 黑穗病菌胁迫下,甘蔗Sc14-3-3基因在ROC22和YC05-179材料中的表达特性分析如图11所示。在ROC22材料中,无论是对照组还是处理组,甘蔗Sc14-3-3基因的表达都受到抑制,且随处理时间的增加,其表达量的变化不明显,但处理组的相对表达量普遍比对照组高,其中,24 h和48 h时表现最为显著;YC05-179材料中,对照组的Sc14-3-3基因的表达受到抑制,且各处理时间点的表达量没有明显差异,而处理组中Sc14-3-3基因的表达量随着处理时间的延长不断增加,120 h时,处理组和对照组中该基因的相对表达量相差最大。以上研究证明,本研究所克隆的Sc14-3-3基因在甘蔗抗性材料中积极应答黑穗病菌胁迫,可能与甘蔗抗黑穗病性有关。
3 讨论与结论
在细胞信号转导中,信号分子通常使靶分子磷酸化,进而激活或者抑制靶分子的生物活性[28]。然而,有研究表明,仅磷酸化并不足以完成信号调节全过程,还需要和14-3-3蛋白结合来完成靶蛋白的转化从而调控其活性[35-37]。14-3-3蛋白在真核生物中广泛存在且高度保守,调节生命活动的许多过程[5,18]。活性形式的14-3-3蛋白以二聚体形式,通过接头或支架作用与发生磷酸化的目标靶蛋白相应结构域结合,并发生相互作用,调节细胞信号转导过程[38-39]。14-3-3蛋白对不同靶蛋白的调节效应差别很大,如可以导致靶蛋白激活、灭活、降解、稳定性增高或者改变靶蛋白胞内定位[40-42],尽管如此,14-3-3蛋白与磷酸化靶蛋白结合从而调控信号转导过程依然是广泛存在的[35]。
本研究基于构建的甘蔗SSH文库,结合电子克隆技术和RT-PCR扩增技术,从甘蔗品种ROC22中克隆到了一个甘蔗14-3-3蛋白基因,命名为Sc14-3-3(GenBank Accession Number:KJ577592)。该基因包含14-3-3基因的保守序列,有完整的开放读码框,编码256个氨基酸,且其氨基酸序列与罗炼芳等[27]报道的甘蔗Sc14-3-3蛋白相同,但编码氨基酸的碱基及该基因的3′端却有所差异(2.66%),说明是同一个14-3-3蛋白家族成员,但在自然进化或人工选择的过程中,甘蔗的不同品种之间对14-3-3基因有碱基偏好性。生物信息学分析结果显示,该编码蛋白无信号肽和跨膜螺旋区,定位于细胞质中。根据前人研究[43],拟南芥14-3-3蛋白前体无信号肽,但它们分散于细胞核、生物膜或其它细胞腔隙中,目前普遍认为14-3-3蛋白的亚细胞定位可由其靶蛋白的信号肽引导,或通过和靶蛋白的相互作用定位于它们复合体的所在区域[43-44],因此,推测该甘蔗Sc14-3-3蛋白与其靶蛋白于细胞质中进行互作。PredictProtein和Profun 2.2 Server在线软件预测显示,该Sc14-3-3蛋白具有多个潜在的磷酸化(Ser、Thr、Tyr)位点,并在N-端有4个蛋白结合区和1个多核苷酸结合区,且对翻译有一定的作用。有研究结果表明,14-3-3蛋白的每一单体都有1个靶蛋白结合位点[45-46],稳定的N-端氨基酸残基区域有助于14-3-3蛋白二聚体的形成及某些靶蛋白的结合能力,而C-端直接参与蛋白质间的相互作用[46],推测2个甘蔗Sc14-3-3蛋白单体在N-端结合形成二聚体,并在C-端形成凹穴,作为靶蛋白的作用面[27],同时,Sc14-3-3蛋白通过结合2个靶肽或1个靶肽的2个结构域,在C-端进行相互作用,增强/抑制翻译作用、调控靶蛋白的活性或与其他蛋白的结合能力等[40]。
前人研究表明,14-3-3蛋白广泛参与了植物对各种胁迫应答的信号转导过程及病原防卫应答反应。大麦在抵御白粉病菌的过程中,14-3-3蛋白的累积量增加[24];水稻OsGFl4b和OsGFl4g几乎可被所有的胁迫和激素诱导[47];小麦(Triticum aestivum)Ta14R1和Ta14R2基因均受ABA诱导而呈现上调表达,通过依赖ABA的非生物胁迫响应途径发挥作用[48];机械伤害或壳聚糖或甲基茉莉酸酮酯胁迫下,白云杉(Picea glauca)和杂交白杨(Populus tomentosa Carr)中14-3-3的转录水平增强[49-50]。本研究利用实时荧光定量PCR技术分析了甘蔗Sc14-3-3基因受SA、MeJA和ABA诱导和黑穗病菌胁迫下的表达情况,结果表明,SA、MeJA、ABA诱导后,YC05-179中,该Sc14-3-3基因均呈现上调表达趋势,且MeJA胁迫下各处理时间点的表达量都比SA和ABA高,表明该Sc14-3-3基因可能参与甘蔗对不同生物逆境[49-50]的细胞免疫响应,且对MeJA介导的信号途径应答较早;尤其值得注意的是,无论是否经黑穗病菌胁迫,ROC22中该Sc14-3-3基因都下调表达,且表达量变化不大,而YC05-179中,该基因仅在黑穗病菌胁迫后的材料中上调表达,且表达量随时间延长而增加,表明该Sc14-3-3基因在甘蔗抵御黑穗病的过程中发挥着一定的作用,但抗感材料中Sc14-3-3基因对黑穗病胁迫的应答反应不同,即在感黑穗病甘蔗材料中其调控模式应该是负调控,而在抗黑穗病材料中,黑穗病菌的诱导使其调控模式相反,可能是抗性材料中某些特異调节蛋白的改变或靶蛋白的活性变化影响了Sc14-3-3基因的表达。
综上所述,本研究从甘蔗中克隆获得了Sc14-3-3基因的全长序列,并通过生物信息学手段及RT-qPCR技术,分析了该基因的特征及其表达特性,发现该基因可能介导甘蔗对黑穗病的抗性,但由于14-3-3蛋白存在多种同工型,并且在任一时刻都同时存在多种靶蛋白,因此,要想进一步的明确该基因的功能,还需要更深入的研究。
参考文献
[1] Moore B W, Perez V J, Carlson F D. Physiological and biochemical aspects of nervous integration[M]. N J: Prentice-Hall, 1967: 343-359.
[2] Brandt J, Thordal-Christensen H, Vad K, et al. A pathogen-induced gene of barley encodes a protein showing high similarity to a protein kinase regulator[J]. The Plant Journal, 1992, 2(5): 815-820.
[3] Lu G H, DeLisle A J, de-Vetten N C, et al. Brain proteins in plants: an Arabidopsis homolog to neurotransmitter pathway activators is part of a DNA binding complex[J]. Proceedings of the National Academy of Sciences, 1992, 89(23): 11 490-11 494.
[4] Hirsch S, Aitken A, Bertsch U, et al. A plant homologue to mammalian brain 14-3-3 protein and protein kinase C inhibitor[J]. FEBS Letters, 1992, 296(2): 222-224.
[5] Ferl R J. 14-3-3 proteins and signal transduction[J]. Annual Review of Plant Biology, 1996, 47(1): 49-73.
[6] Chen F, Li Q, Sun L X, et al. The rice 14-3-3 gene family and its involvement in responses to biotic and abiotic stress[J]. DNA Research, 2006, 13(2): 53-63.
[7] YaoY, Du Y, Jiang L, et al. Molecular analysis and expression patterns of the 14-3-3 gene family from Oryza Sativa[J]. Journal of Biochemistry and Molecular Biology, 2007, 40(3): 349-357.
[8] Yang Z P, Li H L, Guo D, et al. Molecular characterization of a novel 14-3-3 protein gene(Hb14-3-3c)from Hevea brasiliensis[J]. Molecular Biology Reports, 2012, 39(4): 4 491-4 497.
[9] Mayfield J D, Paul A L, Ferl R J. The 14-3-3 proteins of Arabidopsis regulate root growth and chloroplast development as components of the photosensory system[J]. Journal of Experimental Botany, 2012, 63(8): 3 061-3 070.
[10] Chaudhri M, Scarabel M, Aitken A. Mammalian and yeast 14-3-3 isoforms form distinct patterns of dimers in vivo[J]. Biochemical and Biophysical Research Communications, 2003, 300(3): 679-685.
[11] de Vetten N C, Ferl R J. Two genes encoding GF14(14-3-3)proteins in Zea mays(structure, expression, and potential regulation by the G-box-binding complex)[J]. Plant Physiology, 1994, 106(4): 1 593-1 604.
[12] Bunney T D, van Walraven H S, de Boer A H. 14-3-3 protein is a regulator of the mitochondrial and chloroplast ATP synthase[J]. Proceedings of the National Academy of Sciences, 2001, 98(7): 4 249-4 254.
[13] May T, Soll J. 14-3-3 proteins form a guidance complex with chloroplast precursor proteins in plants[J]. The Plant Cell Online, 2000, 12(1): 53-63.
[14] Rosenquist M, Sehnke P, Ferl R J, et al. Evolution of the 14-3-3 protein family: does the large number of isoforms in multicellular organisms reflect functional specificity?[J]. Journal of Molecular Evolution, 2000, 51(5): 446-458.
[15] Bihn E A, Paul A L, Wang S W, et al. Localization of 14-3-3 proteins in the nuclei of Arabidopsis and maize[J]. The Plant Journal, 1997, 12(6): 1 439-1 445.
[16] Sehnke P C, Henry R, Cline K, et al. Interaction of a plant 14-3-3 protein with the signal peptide of a thylakoid-targeted chloroplast precursor protein and the presence of 14-3-3 isoforms in the chloroplast stroma[J]. Plant Physiology, 2000, 122(1): 235-242.
[17] Sehnke P C, Ferl R J. Plant 14-3-3s: omnipotent metabolic phosphopartners?[J]. Science Signaling, 2000, 2000(56): pe1.
[18] Gokirmak T, Paul A L, Ferl R J. Plant phosphopeptide-binding proteins as signaling mediators[J]. Current Opinion in Plant Biology, 2010, 13(5): 527-532.
[19] Wu K, Rooney M F, Ferl R J. The Arabidopsis 14-3-3 multigene family[J]. Plant Physiology, 1997, 114(4): 1 421-1 431.
[20] Shin R, Jez J M, Basra A, et al. 14-3-3 Proteins fine-tune plant nutrient metabolism[J]. FEBS Letters, 2011, 585(1): 143-147.
[21] 崔 娜, 于志海, 韓明利, 等. 植物14-3-3蛋白研究进展[J]. 西北植物学报, 2012, 32(4): 843-851.
[22] Seehaus K, Tenhaken R. Cloning of genes by mRNA differential display induced during the hypersensitive reaction of soybean after inoculation with Pseudomonas syringae pv. glycinea[J]. Plant Molecular Biology, 1998, 38(6): 1 225-1 234.
[23] Hill M K, Lyon K J, Lyon B R. Identification of disease response genes expressed in Gossypium hirsutum upon infection with the wilt pathogen Verticillium dahliae[J]. Plant Molecular Biology, 1999, 40(2): 289-296.
[24] Finnie C, Andersen C H, Borch J, et al. Do 14-3-3 proteins and plasma membrane H+-ATPases interact in the barley epidermis in response to the barley powdery mildew fungus?[J]. Plant Molecular Biology, 2002, 49(2): 137-147.
[25] 蔡英卿, 赖钟雄, 陈义挺, 等. 龙眼14-3-3基因及其启动子的克隆以及在体胚发生过程中的表达分析[J]. 热带作物学报, 2011, 32(5): 845-853.
[26] Yan J Q, Wang J, Zhang H. An ankyrin repeat-containing protein plays a role in both disease resistance and antioxidation metabolism[J]. The Plant Journal, 2002, 29(2): 193-202.
[27] 罗炼芳, 孔 冉, 苏俊波. 甘蔗14-3-3基因克隆及表达分析[J]. 南方农业学报, 2013, 44(11): 1 757-1 764.
[28] 肖 强, 郑海雷. 14-3-3蛋白与植物细胞信号转导[J]. 细胞生物学杂志, 2005, 27(4): 417-422.
[29] Diatchenko L, Lau Y F, Campbell A P, et al. Suppression subtractive hybridization: a method for generating differentially regulated or tissue-specific cDNA probes and libraries[J]. Proceedings of the National Academy of Sciences, 1996, 93(12): 6 025-6 030.
[30] 闕友雄, 许莉萍, 徐景升, 等. 甘蔗基因表达定量PCR分析中内参基因的选择[J]. 热带作物学报, 2009, 30(3): 274-278.
[31] Sehnke P C, DeLille J M, Ferl R J. Consummating signal transduction the role of 14-3-3 proteins in the completion of signal-induced transitions in protein activity[J]. The Plant Cell Online, 2002, 14(suppl 1): S339-S354.
[32] Delaney T P, Uknes S, Vernooij B, et al. A central role of salicylic acid in plant disease resistance[J]. Science, 1994, 266(5 188): 1 247-1 250.
[33] Cheong J J, Do Choi Y. Methyl jasmonate as a vital substance in plants[J]. TRENDS in Genetics, 2003, 19(7): 409-413.
[34] Audenaert K, De Meyer G B, Hofte M M. Abscisic acid determines basal susceptibility of tomato toBotrytis cinerea and suppresses salicylic acid-dependent signaling mechanisms[J]. Plant Physiology, 2002, 128(2): 491-501.
[35] Ferl R J. 14-3-3 proteins: regulation of signal-induced events[J]. Physiologia Plantarum, 2004, 120(2): 173-178.
[36] Chung H J, Sehnke P C, Ferl R J. The 14-3-3 proteins: cellular regulators of plant metabolism[J]. Trends in Plant Science, 1999, 4(9): 367-371.
[37] Würtele M, Jelich-Ottmann C, Wittinghofer A, et al. Structural view of a fungal toxin acting on a 14-3-3 regulatory complex[J]. The EMBO Journal, 2003, 22(5): 987-994.
[38] Fu H, Subramanian R R, Masters S C. 14-3-3 proteins: structure, function, and regulation[J]. Annual Review of Pharmacology and Toxicology, 2000, 40(1): 617-647.
[39] Aitken A. 14-3-3 proteins: a historic overview[J]. Seminars in Cancer Biology, 2006, 16(3): 162-172.
[40] 周 颖, 李冰樱, 李学宝. 14-3-3 蛋白对植物发育的调控作用[J]. 植物学报, 2012, 47(1): 55-64.
[41] Cotelle V, Meek S E M, Provan F, et al. 14-3-3s regulate global cleavage of their diverse binding partners in sugar-starved Arabidopsis cells[J]. The EMBO Journa, 2000, 19(12): 2 869-2 876.
[42] Ryu H, Kim K, Cho H, et al. Nucleocytoplasmic shuttling of BZR1 mediated by phosphorylation is essential in Arabidopsis brassinosteroid signaling[J]. The Plant Cell Online, 2007, 19(9): 2 749-2 762.
[43] Paul A L, Sehnke P C, Ferl R J. Isoform-specific subcellular localization among 14-3-3 proteins in Arabidopsis seems to be driven by client interactions[J]. Molecular Biology of the Cel, 2005, 16(4): 1 735-1 743.
[44] 戚传娇, 郭传龙, 孙利利, 等. 植物14-3-3基因的研究进展[J]. 安徽农业科学, 2012, 40(24): 11 942-11 945.
[45] Liu D, Bienkowska J, Petosa C, et al. Crystal structure of the zeta isoform of the 14-3-3 protein[J]. Nature, 1995, 376(6 536): 191-194.
[46] Yang X W, Lee W H, Sobott F, et al. Structural basis for protein-protein interactions in the 14-3-3 protein family[J]. Proceedings of the National Academy of Sciences, 2006, 103(46): 17 237-17 242.
[47] Rosenquist M, Alsterfjord M, Larsson C, et al. Data mining the Arabidopsis genome reveals fifteen 14-3-3 genes. Expression is demonstrated for two out of five novel genes[J]. Plant Physiology, 2001, 127(1): 142-149.
[48] 孟曉丹, 陈 新, 王亚英, 等. 两个小麦14-3-3基因的特征、亚细胞定位及其对非生物胁迫的响应[J]. 生物工程学报, 2014, 30(2): 232-246.
[49] Lapointe G, Luckevich M, Seguin A. Investigation on the induction of 14-3-3 in white spruce[J]. Plant Cell Reports, 2001, 20(1): 79-84.
[50] Lapointe G, Luckevich M D, Cloutier M, et al. 14-3-3 gene family in hybrid poplar and its involvement in tree defence against pathogens[J]. Journal of Experimental Botany, 2001, 52(359): 1 331-1 338.
责任编辑:黄 艳
