木薯SCoT—PCR反应体系优化及引物筛选
2016-05-30严华兵周慧文单建伟谢向誉
严华兵 周慧文 单建伟 谢向誉

摘要:【目的】优化木薯SCoT-PCR反应体系并进行SCoT引物筛选,为木薯辅助育种提供技术支持。【方法】以木薯品种新选048为材料,利用正交试验设计对DNA模板量、Mg2+浓度、dNTPs浓度、引物浓度、Taq DNA聚合酶量等因素进行优化,确定木薯最佳SCoT-PCR反应体系,并用其进行SCoT引物筛选。【结果】木薯最佳SCoT-PCR反应体系(20.0 μL):模板DNA 60 ng,引物浓度0.30 μmol/L,Taq DNA聚合酶 1.5 U,Mg2+ 1.50 mmol/L,dNTPs 0.25 mmol/L。利用该体系筛选出19条SCoT引物,能稳定扩增出数量多、清晰可辨且多态性丰富的条带。【结论】优化的木薯SCoT-PCR反应体系检测结果稳定,重复性好,且筛选出的19条引物均适用于木薯遗传多样性分析。
关键词: 木薯;SCoT-PCR反应体系;正交试验设计;引物筛选
中图分类号: S533 文献标志码:A 文章编号:2095-1191(2016)10-1648-05
0 引言
【研究意义】木薯(Manihot esculenta Crantz)是大戟科(Euphorbiaceae)木薯属(Manihot)植物,耐旱抗贫瘠,广泛种植于非洲、美洲和亚洲等100余个国家和地区(El-Sharkawy,2004),是世界三大薯类作物之一。广西木薯种植面积及产量均占我国总量的50%~60%,通过木薯育种提高木薯产量对广西乃至全国木薯产业发展均具有重要意义。木薯遗传背景复杂,杂合度高,为其品种选育提供了丰富的遗传资源。近年来,随着分子标记技术的发展,现已广泛应用于木薯亲本的选择、遗传图谱构建及遗传多样性分析等研究。目标起始密码子多态性分子标记(Start condon targeted polymorphism,SCoT)是Collard和Mackill(2009)以水稻为基础提出的基于单引物扩增反应(SPAR)的新目的基因分子标记,其原理是基于植物基因中ATG翻译起始位点侧翼序列的保守性,设计单引物并对基因组进行扩增,具有操作简单、引物通用、成本低廉、多态性高、能获得丰富遗传信息等优点(熊发前等,2009)。SCoT分子标记是以PCR扩增为基础,建立稳定的PCR反应体系能更好地利用SCoT分子标记。因此,开展木薯SCoT-PCR反应体系优化与引物筛选研究,对木薯分子标记辅助育种具有重要意义。【前人研究进展】目前,已有大量分子标记应用于木薯研究。Beeching等(1993)利用RFLP分子标记对木薯种质资源进行遗传多样性分析;曾霞等(2003)利用RAPD分子标记对44份木薯材料进行遗传背景研究;韦祖生等(2008)利用EST-SSR分子标记对国内原始材料和引进材料进行 遗传多样性分析,结果发现新引进材料有新的遗传类型;陈青等(2013)利用EST-SSR、SSR和SRAP分子标记构建木薯遗传连锁图谱。自2009年SCoT分子标记被开发以来,侯小改等(2009)、韩国辉等(2011)、赵瑞强等(2012)分别对牡丹、柑橘、铁皮石斛等作物的SCoT-PCR反应体系进行优化;黄秀等(2013)、李丕睿等(2013)、杨祥燕等(2013)、蒋雅琴等(2014)分别应用该技术对高牛鞭草、菊花、番木瓜、丝瓜等作物进行遗传多样性分析;此外,也有研究将该技术应用于种质鉴定、基因差异表达分析及指纹图谱和分子遗传连锁图谱构建(龙治坚等,2015)。【本研究切入点】目前,研究木薯的分子标记多为EST-SSR、SSR等,鲜见利用SCoT分子标记开展木薯PCR反应体系优化的研究报道。【拟解决的关键问题】采用正交试验设计对影响木薯SCoT-PCR扩增效果的Mg2+浓度、DNA模板量、dNTPs浓度、引物浓度、Taq DNA聚合酶量5个因素进行优化,建立最佳的木薯SCoT-PCR反应体系,并利用该体系对36条SCoT引物进行筛选,为利用SCoT分子標记进行木薯辅助育种提供技术支持。
1 材料与方法
1. 1 试验材料
20份木薯材料(表1)采集自广西农业科学院里建木薯育种基地。所用的36条SCoT引物序列(表2)参照Collard和Mackill(2009),由生工生物工程(上海)股份有限公司合成。琼脂糖、dNTPs、Taq DNA聚合酶等试剂及药品均购自宝生物工程(大连)有限公司。主要仪器设备:Applied Biosystems 2720 Thermal Cycler PCR仪[赛默飞世尔科技(中国)有限公司]、HAD-JY600C三恒电泳仪(北京亚欧德鹏科技有限公司)、DYCP- 31DN型电泳槽(北京六一仪器厂)。
1. 2 试验方法
1. 2. 1 DNA提取 采集无病虫害木薯健康植株幼嫩叶片并提取其DNA,DNA提取参考改良CTAB提取法(闫庆祥等,2010)。用紫外分光光度计检测DNA浓度,并将提取的DNA稀释成30 ng/μL,-20 ℃保存备用。
1. 2. 2 正交试验设计 选择DNA模板、Mg2+浓度、dNTPs浓度、引物浓度、Taq DNA聚合酶量等5个因素,每因素设4个水平,进行L16(45)正交试验(表3),共16个组合,每个组合设3次重复,且每个组合加入2.0 μL 10×Mg2+ Free PCR Buffer,并用ddH2O补至20.0 μL。
1. 2. 3 PCR扩增与检测 经初步筛选,选取SC21为正交试验引物,木薯品种新选048的DNA为模板。PCR扩增程序:94 ℃预变性4 min;94 ℃ 30 s,50 ℃ 1 min,72 ℃ 90 s,进行35个循环;72 ℃延伸5 min。PCR产物用0.8%琼脂糖凝胶进行电泳,设定电压为120 V,运行时间为30 min。经GoldView核酸染色剂染色后在紫外凝胶成像系统成像后拍照。
1. 2. 4 数据统计 根据电泳图谱中的电泳条带数目、清晰度及背景干净程度进行打分,参照何正文等(1998)、谢云海等(2005)的直观分析法,条带清晰、数目丰富、背景干净的记为最高分(16分),反之记为最低分(1分)。最后用Excel 2007计算平均分、极差及制作图表。
1. 2. 5 体系检验及引物筛选 以SC21为引物,20份木薯材料的DNA为模板,对优化SCoT-PCR反应体系的稳定性及扩增效果进行验证。并以新选048的DNA为模板,使用优化的SCoT-PCR反应体系从Collard和Mackill(2009)设计的36条引物中筛选适用于木薯的SCoT引物。PCR产物用0.8%琼脂糖凝胶电泳进行检测。
2 结果与分析
2. 1 PCR反应体系的正交试验结果
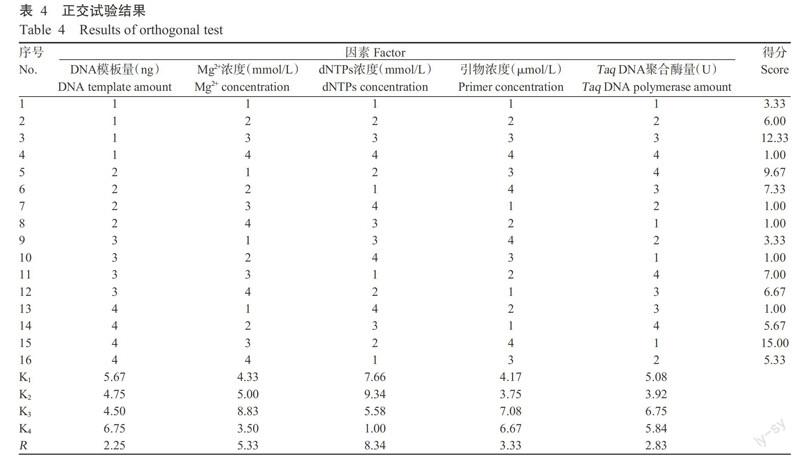
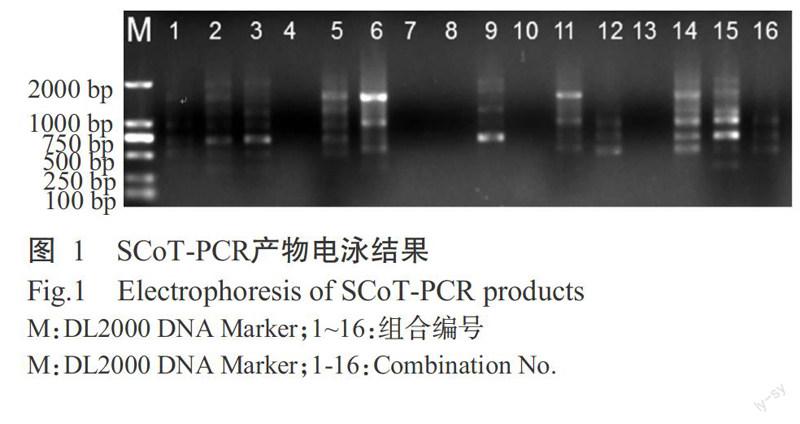
从图1可看出,16个组合的扩增效果各不相同。利用直观分析法对其进行打分(表4),由于R越大,表明该因素对SCoT-PCR反应体系影响越大;Ki越大,则表示该水平越好(桂腾琴等,2009),因此,5个因素对SCoT-PCR反应体系的影响排序为:dNTPs浓度>Mg2+浓度>引物浓度>Taq DNA聚合酶量>DNA模板量,结合Ki可初步确定最佳SCoT-PCR反应体系(20.0 μL):dNTPs 0.25 mmol/L,Mg2+1.50 mmol/L,引物浓度0.30 μmol/L,Taq DNA聚合酶1.5 U,DNA模板60 ng。
2. 2 体系验证结果
用SC21引物对20份木薯材料进行扩增,结果如图2所示,条带清晰、数目丰富,背景干净,且从不同木薯材料中扩增出多态性片段,表明优化后的SCoT-PCR反应体系扩增效果良好且稳定,可应用于木薯亲本的选择、遗传图谱构建及遗传多样性分析等研究。
2. 3 引物筛选结果
利用优化的木薯SCoT-PCR反应体系,以新选048的DNA为模板,对Collard和Mackill(2009)报道的36条引物进行筛选,以期筛选出适用于木薯的SCoT引物,结果如图3所示,有19条引物能稳定地扩增出数量多、清晰可辨且多态性丰富的条带。
3 讨论
将正交试验设计应用于SCoT-PCR反应体系优化在国内最早由何正文等(1998)提出。在此之前的PCR反应体系优化研究采用多次单因素试验,只变化一个因素而固定其他因素,仅靠经验或参照相近物种确定因素变化范围,未能考察PCR反應体系中各因素的交互作用,从而无法准确获得各因素最佳水平组合(杨水云等,2005)。以正交试验设计优化PCR反应体系省工省时,试验成本低,且兼顾各因素的交互作用,目前已有大量研究利用该方法优化了牡丹(侯小改等,2011)、柑橘(韩国辉等,2011)、苜蓿(何庆元等,2012)、铁皮石斛(赵瑞强等,2012)、菊(李丕睿等,2013)、柿(夏乐晗等,2014)等作物的SCoT-PCR反应体系。本研究采用正交试验设计,快速获得最佳的木薯SCoT-PCR反应体系,弥补了应用正交试验设计优化木薯SCoT-PCR反应体系的空白。
SCoT是一种基于PCR扩增的目的基因分子标记,而PCR是一个受多因素影响的综合反应体系(赵瑞强等,2012),因此建立最佳PCR反应体系对试验顺利开展尤为重要。影响SCoT-PCR扩增效果的主要因素因作物而异,如对于牡丹而言,Mg2+浓度是对SCoT- PCR扩增效果影响最主要的因素(侯小改等,2011);而对于铁皮石斛而言,Taq DNA聚合酶量是最主要的因素(赵瑞强等,2012)。本研究发现,dNTPs浓度对木薯SCoT-PCR扩增效果影响最大,与柑橘(韩国辉等,2011)的相同,其次是Mg2+浓度。
4 结论
木薯最佳SCoT-PCR反应体系(20.0 μL):DNA模板60 ng,引物浓度0.30 μmol/L,Taq DNA聚合酶量1.5 U,Mg2+浓度1.50 mmol/L,dNTPs浓度0.25 mmol/L,且筛选出的19条引物均适用于木薯遗传多样性分析,可为木薯的分子辅助育种提供技术支持。
参考文献:
陈青,夏志强,陈新,卢诚,王宇阳,王文泉. 2013. 木薯分子标记遗传连锁图谱的构建及相关性状QTL定位[J]. 热带作物学报, 34(5): 829-837.
Chen Q, Xia Z Q, Chen X, Lu C, Wang Y Y, Wang W Q. 2013. Construction of the moleclllar genetic linkage map and QTL analysis for related traits in cassava(Manihot esculenta Grantz)[J]. Chinese Joumal of Tropical Crops, 34(5): 829-837.
韩国辉, 向素琼, 汪卫星, 贾志刚, 洪棋斌, 梁国鲁. 2011. 柑橘SCoT分子标记技术体系的建立及其在遗传分析中的应用[J]. 园艺学报,38(7): 1243-1250.
Han G H, Xiang S Q, Wang W X, Jia Z G, Hong Q B, Liang G L. 2011. Establishment and application of SCoT molecular marker system for Citrus[J]. Acta Horticulturae Sinica, 38(7): 1243-1250.
桂腾琴,孙敏,乔爱民,王心燕. 2009. 正交设计优化果梅ISSR反应体系[J]. 果树学报,26(1): 108-112.
Gui T Q, Sun M, Qiao A M, Wang X Y. 2009. Optimization of an ISSR reaction system of Japanese apricot(Prunus mume) based on orthogonal design[J]. Journal of Fruit Science, 26 (1): 108-112.
何庆元, 王吴斌, 杨红燕, 向仕华, 周丽英, 王松华. 2012. 利用SCoT标记分析不同秋眠型苜蓿的遗传多样性[J]. 草业学报, 21(2): 133-140.
He Q Y, Wang W B, Yang H Y, Xiang S H, Zhou L Y, Wang S H. 2012. Optimization of SCoT reaction system and genetic diversity of different fall dormancy alfalfa[J]. Acta Prataculturae Sinica, 21(2): 133-140.
何正文, 刘云生, 陈立华, 曹美鸿,夏家辉. 1998. 正交设计直观分析法优化PCR条件[J]. 湖南医科大学学报, 23(4): 403-404.
He Z W, Liu Y S, Chen L H, Cao M H, Xia J H. 1998. Orthogonal design-direct analysis for PCR optimization[J]. Bulletin of Hunan Medical University, 23(4): 403-404.
侯小改, 王娟, 賈甜, 张钰乾, 侯娟, 李嘉珏. 2011. 牡丹SCoT分子标记正交优化及引物筛选[J]. 华北农学报, 26(5): 92-96.
Hou X G, Wang J, Jia T, Zhang Y Q, Hou J, Li J J. 2011. Orthogonal optimization of SCoT-PCR system and primer screening of tree peony[J]. Acta Agriculturae Boreali-Sinica, 26(5): 92-96.
黄秀, 张新全, 张瑜, 蒋晓梅, 曾捷, 刘欢, 聂刚, 张博涛, 黄琳凯. 2013. 高牛鞭草(Hemarthria altissima)及其近缘种种质资源SCoT多样性分析[J]. 热带作物学报,34(11): 2192-2199.
Huang X, Zhang X Q, Zhang Y, Jiang X M, Zeng J, Liu H, Nie G, Zhang B T, Huang L K. 2013. Genetic diversity of Hemarthria altissima and its relative species by SCoT markers[J]. Chinese Journal of Tropical Crops, 34(11): 2192-2199.
蒋雅琴, 黎炎, 李文嘉, 吴永官, 王易奎, 康德贤. 2014. SCoT分子标记技术在丝瓜上的应用[J]. 南方农业学报, 45(12): 2117-2122.
Jiang Y Q, Li Y, Li W J, Wu Y G , Wang Y K, Kang D X. 2014. Application of SCoT markers on genetic diversity analysis of Luffa Mill.[J]. Journal of Southern Agricultue, 45(12): 2117-2122.
李丕睿, 蒋甲福, 陈素梅, 管志勇, 廖园, 房伟民, 陈发棣. 2014. 菊属植物 SCoT 分子标记技术在遗传多样性分析中的应用[J]. 园艺学报, 40(10): 2015-2025.
Li P R, Jiang J F, Chen S M, Guan Z Y , Liao Y, Fang W M, Chen F D. 2014. Establishment and optimization of SCoT molecular marker system in Chrysanthemum and its application of analysis on genetic diversity[J]. Acta Horticulturae Sinica, 40(10): 2015-2025.
龙治坚, 范理璋, 徐刚, 胡尚连, 韩国辉. 2015. SCoT分子标记在植物研究中的应用进展[J]. 植物遗传资源学报, 16(2): 336-343.
Long Z J, Fan L Z, Xu G, Hu S L, Han G H. 2015. Application advance of SCoT molecular markers in plants[J]. Journal of Plant Genetic Resources, 16(2): 336-343.
夏乐晗, 杨婷婷, 杨勇, 夏宏义, 张永芳, 王仁梓. 2014. 柿SCoT-PCR体系优化及品种遗传多样性分析[J]. 西北植物学报, 34(3): 473-480.
Xia L H, Yang T T, Yang Y, Xia H Y, Zhang Y F, Wang R Z. 2014. System optimization of SCoT-PCR and analysis on genetic diversity of persimmion(Diospyros kaki Thunb.)[J]. Acta Botanica Boreali-Occidentalia Sinica, 34(3): 473-480.
韦祖生, 夏志强, 李开绵, 王文泉. 2008. 木薯种质库遗传多样性的EST-SSR标记[J]. 热带作物学报, 29(3): 304-309.
Wei Z S, Xia Z Q, Li K M, Wang W Q. 2008. Analysis of genetic diversity of cassava genepool by EST-SSR Markers[J]. Chinese Journal of Tropical Crops, 29(3): 304-309.
谢云海, 夏德安, 姜静, 林萍. 2005. 利用正交设计优化水曲柳ISSR-PCR反应体系[J]. 分子植物育种, 3(3): 445-450.
Xie Y H, Xia D A, Jiang J, Lin P. 2005. Optimization for ISSR-PCR system of Fraxinus mandshurica Rupr. using orthogonal design[J]. Molecular Plant Breeding, 3(3): 445-450.
熊发前, 唐荣华, 陈忠良, 潘玲华, 庄伟建. 2009. 目标起始密码子多态性(SCoT): 一种基于翻译起始位点目的基因标记新技术[J]. 分子植物育种, 7(3): 635-638.
Xiong F Q, Tang R H, Chen Z L, Pan L H, Zhuang W J. 2009. SCoT: a novel gene targeted marker technique based on the translation start codon[J]. Molecular Plant Bree-
ding, 7(3): 635-638.
闫庆祥, 黄东益, 李开绵, 叶剑秋. 2010. 利用改良CTAB法提取木薯基因组DNA[J]. 中国农学通报, 26(4): 30-32.
Yan Q X, Huang D Y, Li K M, Ye J Q. 2010. Genomic DNA extraction in cassava by modified CTAB method[J]. Chinese Agricultural Science Bulletin, 26(4): 30-32.
杨水云, 李续娥, 吴明宇, 孙飞龙, 李剑君. 2005. 正交试验法在PCR反应条件优化中的应用[J]. 生物数学学报, 20(2): 202-206.
Yang S Y, Li X E, Wu M Y, Sun F L, Li J J. 2005. Application of orthogonal design to optimize PCR conditions[J]. Journal of Biomathematies, 20(2): 202-206.
杨祥燕, 蔡元保, 黄秋伟, 陈涛, 覃剑峰, 彭靖茹. 2013. 番木瓜主栽品种SCoT指纹图谱构建及遗传变异分析[J]. 西北植物学报, 33(9): 1756-1761.
Yang X Y, Cai Y B, Huang Q W, Chen T, Qin J F, Peng J R. 2013. SCoT fingerprints and genetic variations of the papaya(Carica papaya L.) major cultivars[J]. Acta Botanica Boreali-Occidentalia Sinica, 33(9): 1756-1761.
曾霞, 庄南生, 李开绵. 2003. 应用RAPD技术对44份木薯材料遗传背景的研究[J]. 热带作物学报, 24(2): 59-64.
Zeng X, Zhuang N S, Li K M. 2003. Cassava genetic background revealed by RAPD[J]. Chinese Journal of Tropical Crops, 24(2): 59-64.
赵瑞强, 高燕会, 章晓玲, 斯金平. 2012. 铁皮石斛SCoT-PCR反应体系构建及优化[J]. 核农学报, 26(4):648-655.
Zhao R Q, Gao Y H, Zhang X L, Si J P. 2012. Establishment and optimization of SCoT-PCR reaction system for Dendrobium officinale[J]. Journal of Nuclear Agricultural Sciences, 26(4): 648-655.
Beeching J R, Marmey P, Gavdda M C, Noirot M, Haysom H R, Hughes M A, Charrier A. 1993. An assessment of genetic diversity within a collection of cassava(Manihot esculenta Grantz) germplasm using molecular markers[J]. Annals of Botany, 72(6): 515-520.
Collard B C Y, Mackill D J. 2009. Start codon targeted(SCoT) polymorphism: a simple, novel DNA marker technique for generating gene-targeted markers in plants[J]. Plant Molecu-
lar Biology Reporter, 27(1): 86-93.
El-Sharkawy M A. 2004. Cassava biology and physiology[J]. Plant Molecular Biology, 56(4): 481-501.
(責任编辑 陈 燕)
