中国假百合属亲缘关系及假百合遗传多样性的ISSR分析
2016-05-06周颂东何兴金
杨 梅,周颂东,何兴金,彭 昶
(四川大学 生命科学学院 生物资源与生态环境教育部重点实验室,成都 610064)
中国假百合属亲缘关系及假百合遗传多样性的ISSR分析
杨梅,周颂东*,何兴金,彭昶
(四川大学 生命科学学院 生物资源与生态环境教育部重点实验室,成都 610064)
摘要:利用7条ISSR引物对中国假百合属植物24个居群255个个体的遗传多样性进行了初步探讨。结果表明:(1)UPGMA聚类结果显示,假百合(Notholirion bulbuliferum)、钟花假百合(N.campanulatum)与大叶假百合(N.macrophyllum)分别聚为三支,在分子水平上出现了明显的分化,揭示三者是独立物种。(2)N.bulbuliferum×N.campanulatum与钟花假百合聚为一支,这可能与其母系遗传和极强的无性繁殖体系有关,但两者间基因流(Nm=0.216 0)很低,假百合与N.bulbuliferum×N.campanulatum的基因流(Nm=0.144 9)也极低,说明N.bulbuliferum×N.campanulatum正在分化。(3)假百合种下各居群间按照地理结构可明显分为4支,经Mantel检测表明存在显著的谱系地理结构(r=0.410,P=0.01)。(4)AMOVA分析显示,假百合居群间遗传变异为77.12%(P<0.01),而居群内为22.88%(P<0.01),同样表明居群间遗传分化大于居群内。研究结果揭示了横断山区假百合属植物的亲缘关系,从分子水平上为其鉴定提供了依据;并从遗传结构上为药用植物假百合的可持续利用和开发奠定了理论基础。
关键词:遗传多样性;ISSR;假百合;钟花假百合;大叶假百合
假百合属是百合科百合族较为原始的类群[1-2],属下共有5种,其中大叶假百合(Notholirionmacrophyllum)、钟花假百合(N.campanulatum)和假百合(N.bulbuliferum)等3种分布于中国西部横断山区及东喜马拉雅[3]。因其鳞茎由基生叶增厚套迭而成,外面具黑褐色的膜质鳞茎皮,与百合属的特征相似而被作为百合属的一个亚属;而后又有学者将其归为贝母属的一个亚属[4]。假百合属植物具卵形的小鳞茎,成熟后外壳稍硬,内有数片白色肉质的鳞片;柱头明显3裂,这些特征明显区别于百合族的其它属,Boissier将其提升为独立的属并得到大多数学者的支持,同时发表了大叶假百合[5-7]。Conton与Stearn又陆续发现了钟花假百合和假百合[6-7]。而有研究显示,假百合与钟花假百合之间存在杂交,即假百合×钟花假百合(N.bulbuliferum×N.campanulatum)。据文献记载以及野外观察发现这四者在未开花时很难辨认(图1),假百合为常用中药材,对其鉴定的准确与否直接关系到药材质量,所以从分子水平上对其属下种间关系探讨迫在眉睫。假百合属植物生长在2 700~4 000 m高海拔地带,且大多生长在人迹罕至的深山中,极少受到关注,更未从分子水平上对这四者进行过亲缘关系及遗传多样性的研究。
ISSR被称作简单重复区间多态性扩增,或微卫星PCR,方法是用17~22个重复碱基序列作为引物扩增出重复序列间的片段[8],因其操作步骤简单易行[9-10],应用范围十分广泛。在百合族中运用ISSR技术探讨了青岛百合[11]的遗传多样性,提出了麝香百合的分子鉴定依据[12];赵丽华等[13]采用ISSR技术对中国百合属亲缘关系进行了研究;沈呈娟等[14]对近缘类群宝兴百合与匍茎百合进行了亲缘关系与遗传多样性的研究。本实验通过ISSR分子标记对中国假百合属植物的亲缘关系进行了研究,为药用植物假百合的分子鉴定提供了参考,并对假百合进行了居群水平上的遗传多样性研究,为假百合可持续利用和保护提供了理论依据。
1材料和方法
1.1材料
实验包括24个居群,共计255个个体,其中假百合、钟花假百合、假百合×钟花假百合、大叶假百合的居群数分别为18、2、3、1个。按居群采样时,每个个体至少间隔50 m以上,将采集的新鲜叶片置入硅胶干燥备用。并将野外采集的个体引种栽培于实验室,凭证标本存放于四川大学标本馆(SZ)。采样基本覆盖中国假百合属的分布范围(表1,图2)。
1.2ISSR标记分析
对实验材料新鲜嫩叶采用植物基因组DNA试剂盒(天根生化科技有限公司,北京)提取总DNA。DNA样品用1%琼脂糖凝胶电泳检测,利用紫外分光光度计确定其浓度,将样品稀释至25 ng/μL,最后将样品置于-20 ℃冰箱中保存备用。随机从每个居群中抽取2个样本作为引物筛选的模板,根据哥伦比亚大学(UBC)公布的引物序列,从中筛选出稳定性好,重复性强且条带清晰的7条引物作为本次实验的引物(Invitrogne公司合成),引物编号及序列见表2。
从每个居群选取3个个体进行预实验,最终确定PCR扩增反应体系总体积为25 μL:包含25 ng模板DNA,1.0 UTaqDNA聚合酶,1×PCR缓冲液,1 μmol/L ISSR引物,0.5 mmol/L dNTPs,1.5 mmol/L MgCL2。PCR扩增程序为94 ℃预变性4 min;94 ℃变性45 s,51.5 ℃~55.5 ℃(视引物Tm

居群编号同表1

物种Species居群编号Populationcode采集地Location海拔Altitude/m经度Longitude(E)纬度Latitude(N)样本数Number假百合N.bulbuliferumQL陕西秦岭Qinling,Shaanxi2800107°42'33°57'10SP四川松潘Songpan,Sichuan3342103°50'32°43'10LX四川理县Lixian,Sichuan3541102°39'31°50'10HS四川黑水Heishui,Sichuan3229102°37'32°03'10JC四川金川Jinchuan,Sichuan3600102°05'31°10'10KD四川康定Kangding,Sichuan3701101°47'30°06'10YJ四川雅江Yajiang,Sichuan3638100°53'29°58'10XC四川乡城Xiangcheng,Sichuan365099°59'28°57'10ML四川木里Muli,Sichuan3414101°09'28°09'10XiC四川西昌Xichang,Sichuan3300102°22'27°35'10MN四川冕宁Mianning,Sichuan3300102°15'28°53'10MG四川美姑Meigu,Sichuan3200102°54'28°36'10YX四川越西Yuexi,Sichuan3600101°95'27°52'10LQ云南禄劝Luquan,Yunnan3987102°50'26°05'10DY云南大姚Dayao,Yunnan3357101°19'25°43'10DQ云南德钦Deqin,Yunnan334099°23'28°50'11MK西藏芒康Mangkang,Xizang381798°40'29°13'12LZ西藏林芝Linzhi,Xizang343694°43'29°41'10假百合×钟花假百合N.bulbuliferum×N.campanulatumDL云南大理Dali,Yunnan3933100°09'25°01'12ZD云南中甸Zhongdian,Yunnan334099°48'27°33'13LJ云南丽江Lijiang,Yunnan3627101°10'26°10'12钟花假百合N.campanulatumTC云南腾冲Tengchong,Yunnan350098°17'25°32'10GS云南贡山Gongshan,Yunnan334198°17'28°23'10大叶假百合N.macrophyllumJL西藏吉隆Jilong,Xizang345085°30'28°30'15
值而定)退火45 s,72 ℃延伸1 min,共35个循环;72 ℃延伸8 min。以DL2000作为分子量标准检测扩增产物,用含有Gold view的1.5%琼脂糖凝胶于120 V恒压电泳45 min。最后在凝胶成像系统仪上进行拍照并保存(Bio-Rad Gel Doc XR+imaging system),用于后续分析。
1.3数据处理与分析
ISSR作为显性分子标记视每一个多态位点为一个等位基因。以DL2000进行参照对比,在同一相对迁移位置出现清晰条带的计为1,没有或模糊的条带记为0,将条带数据信息构建成0和1的原始数据矩阵。使用POPGENE 1.32软件分析遗传变异的各项参数:如多态位点百分率(PPB)、Nei’s基因多样度指数(H)、Nei’s遗传距离(D)和遗传一致度(Hi)、平均每个位点上的观察等位基因数(Na)、平均每个位点的有效等位基因(Ne)、Shan-non多样性信息指数(I)、居群间遗传分化系数(Gst)、居群内基因多样度(Hs)、总的基因多样度(Ht)、基因流(Nm)等[15-16]。然后利用NTSYS-pc 2.10构建树状聚类图以及计算遗传相似性系数(Gs)[17],对假百合属下种间关系进行分析。遗传变异在居群内与居群间的分布情况通过AMOVA 1.55软件计算得出[18]。最后使用GenAlEX 6.5对遗传距离与地理距离之间相关性进行Mantel检验[19]。

表2 ISSR引物序列和最佳退火温度
注:Y=C/T;R=A/G。
Note:Y=C/T;R=A/G.
2结果与分析
2.1ISSR扩增产物的初步检测
图3为引物UBC815对假百合属植物进行扩增后,用1.5%琼脂糖凝胶电泳检测的结果。其它引物都达到很好的扩增效果,表明选定的反应体系和PCR扩增程序效果较好,扩增产物可用于下一步的数据分析。
2.2假百合属ISSR结果分析
从20条ISSR引物中筛选出了能扩增出清晰、稳定性和重复性较好的7条引物,对假百合属全部供试材料进行PCR分析,共计扩增出91个清晰可重复的多态性有效位点,其中多态性标记位点91个(PPB=100%)。7条引物扩增后的DNA条带数目从10到16,平均条带数目为13,变异长度从200 bp到2 000 bp。POPGENE1.32分析显示(表3):在这7条引物中平均每个位点的有效等位基因(Ne)从 1.044 0到1.248 2,平均值为1.146 3。平均每个位点上的观察等位基因数(Na)从1.155 8到1.441 6,平均值为1.259 0。Nei’s基因多样度指数(H)从0.051 8到0.262 0,平均值为0.146 8。以及Shannon多样性信息指数(I)从0.082 3到0.317 7,平均值为0.164 0。
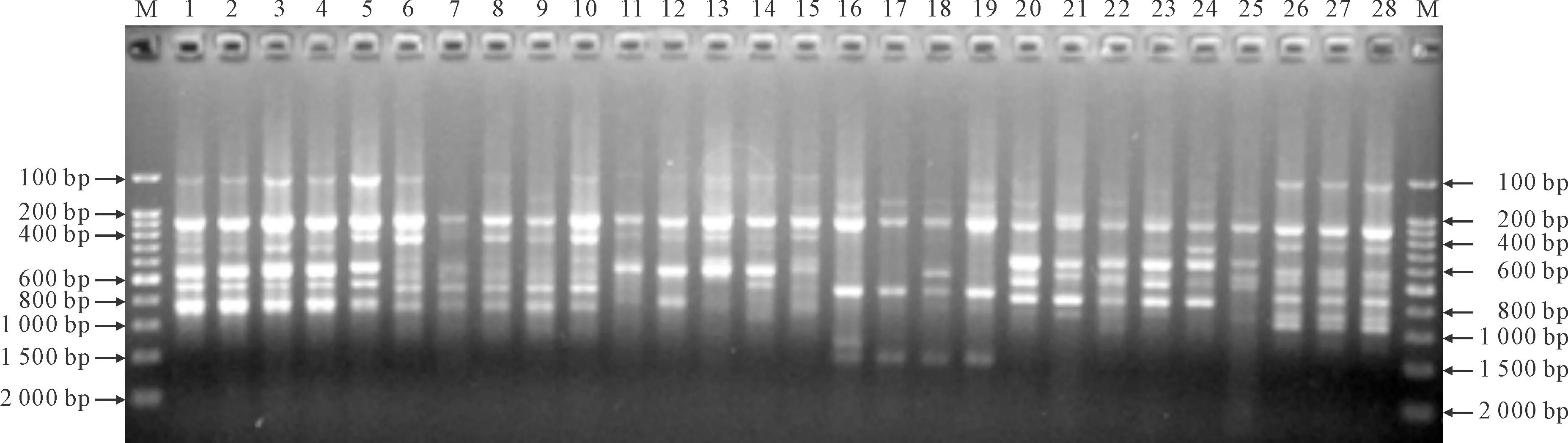
1~3.假百合,康定居群;4~9.大叶假百合;10~13.钟花假百合;14~23.假百合×钟花假百合;24~28.假百合,秦岭居群

物种Species居群编号PopulationcodeNaNeHIPPB/%假百合N.bulbuliferumQL1.4026±0.49361.223±0.35410.1295±0.18680.2964±0.267740.26SP1.3117±0.46621.1185±0.24320.1069±0.13800.1943±0.207931.17LX1.2078±0.40841.1003±0.24070.2620±0.13700.1662±0.203720.78HS1.1948±0.39861.1001±0.24130.1615±0.13780.1646±0.204719.48JC1.1948±0.39861.1003±0.24270.2617±0.13730.1646±0.204519.48KD1.4416±0.49981.2347±0.33320.1422±0.18250.3177±0.266344.16YJ1.1948±0.39861.1283±0.29730.1728±0.15890.1677±0.229219.48XC1.2727±0.44831.1492±0.29940.1084±0.16340.1843±0.238227.27ML1.2597±0.44141.1704±0.31480.1995±0.17540.1773±0.255725.97XiC1.3636±0.48421.2347±0.35150.1365±0.19290.2024±0.279736.36MN1.1558±0.36511.1021±0.27790.2564±0.14650.0830±0.208815.58MG1.1558±0.36521.0889±0.24400.0518±0.13550.0830±0.196915.58YX1.1948±0.39861.1359±0.30680.1756±0.16580.1601±0.236919.48LQ1.1948±0.39871.1017±0.24770.1821±0.13830.1655±0.205019.48DY1.1688±0.37711.1019±0.25880.0595±0.14330.1591±0.208916.88DQ1.3247±0.47131.1812±0.31660.1074±0.17550.2012±0.255432.47MK1.2597±0.44141.1223±0.27250.1737±0.14890.1773±0.217825.97LZ1.1558±0.36511.0440±0.13920.1308±0.08720.0823±0.137515.58平均值Average1.24751.13550.15660.174824.75物种水平Atspecieslevel1.6576±0.12661.4378±0.22560.4582±0.05280.3432±0.125593.9假百合×钟花假百合N.bulbuliferum×N.campanulatumDL1.3506±0.48031.2482±0.38460.1378±0.20230.2013±0.288235.06ZD1.2597±0.44141.1712±0.33470.1957±0.17980.1404±0.256825.97LJ1.2878±0.40841.1365±0.31270.0756±0.16500.1111±0.235220.78钟花假百合N.campanulatumTC1.2857±0.45481.1828±0.32900.1054±0.18040.1562±0.260728.57GS1.2987±0.46071.1979±0.35040.1113±0.18790.1637±0.268829.87大叶假百合N.macrophyllumJL1.2808±0.41751.1362±0.29990.0783±0.0.16100.1171±0.132922.08
2.3假百合属聚类分析
通过NTSYSpc-2.10软件计算出假百合属各类群间遗传相似性系数(Gs),结果显示Gs值范围从0.535 2到0.813 3,平均值为0.702 2。Gs最低值存在于大叶假百合与假百合之间为0.535 2,这表明其亲缘关系最远;最高值为钟花假百合与N.bulbuliferum×N.campanulatum为0.813 3,说明其亲缘关系最近。通过聚类分析也表明,大叶假百合与假百合相距最远,假百合与钟花假百合距离最近(图4)。
2.4假百合遗传多样性
利用筛选的7个ISSR引物(表2)对假百合进行PCR扩增,共计扩增出83个清晰可重复的有效位点,其中多态位点有78个。结果显示(表3):在物种水平上,假百合的多态位百分率(PPB)为93.9%,Ne为(1.437 8±0.225 6),H为(0.458 2±0.052 8),I为(0.343 2±0.125 5)。在居群水平上,各个居群的PPB范围从15.58%~44.16%,平均值为24.75%。假设种群处于Hardy-Weinberg平衡条件下,18个居群的平均有效等位基因为1.247 5,平均Nei’s基因多样性指数为0.135 5,平均Shannon多样性信息指数为0.174 8,各项参数均低于物种水平。
Shannon多样性指数表明各居群的遗传变异由低到高为林芝<冕宁、美姑<大姚<越西<禄劝<雅江<黑水、金川<理县<芒康、米林<乡城<松潘<德钦<西昌<秦岭<康定,与PPB值分析的结果一致。各居群间遗传差异较大,其中康定居群(KD)的遗传多样性水平最高(PPB=44.16%,Ne=1.234 7,I=0.317 7),林芝居群间的遗传多样性水平最低(PPB=15.58%,Ne=1.044 0,I=0.082 3)。

居群编号同表1
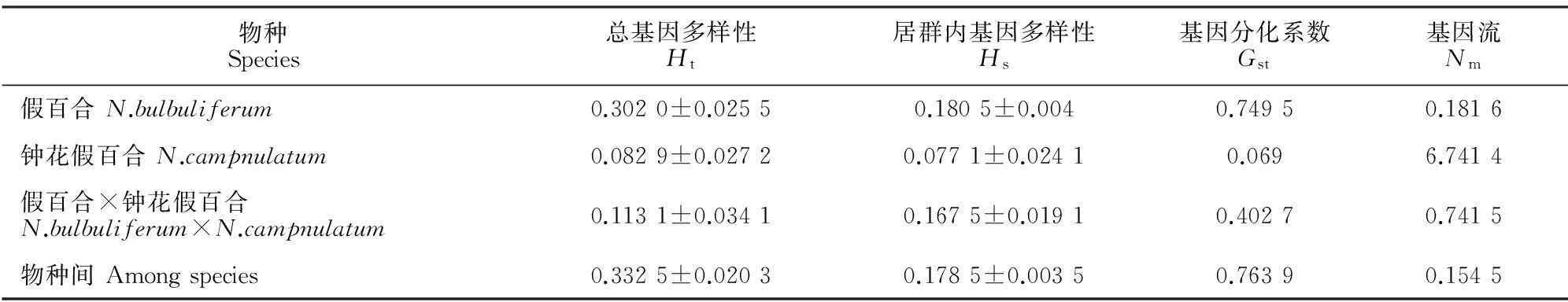
物种Species总基因多样性Ht居群内基因多样性Hs基因分化系数Gst基因流Nm假百合N.bulbuliferum0.3020±0.02550.1805±0.0040.74950.1816钟花假百合N.campnulatum0.0829±0.02720.0771±0.02410.0696.7414假百合×钟花假百合N.bulbuliferum×N.campnulatum0.1131±0.03410.1675±0.01910.40270.7415物种间Amongspecies0.3325±0.02030.1785±0.00350.76390.1545
2.5假百合遗传分化与基因流
通过POPGENE 1.32软件计算得出假百合18个居群Ht为0.302 0,Hs为0.180 5,Gst为0.749 5(表4),即74.95%的变异存在于居群之间,25.05%的变异存在于居群内,结果显示居群间的遗传分化大于居群内的分化。居群间的基因流(Nm)为0.181 6,表明居群之间的基因流水平较低。
利用AMOVA 1.55分析表明在总的遗传变异中,有77.12%(P<0.001)的遗传变异发生在居群之间,22.88%(P<0.001)的遗传变异存在于居群内,与POPGENE 1.32分析结果一致,都表明遗传变异主要存在于居群间。
2.6假百合聚类分析
POPGENE 1.32分析得出假百合18个居群两两间的Nei’s遗传一致度(Hi),范围为0.602 6~0.983 1,遗传距离(D)为0.017 1~0.464 2(表5)。乡城与雅江居群间遗传一致度最高(Hi=0.983 1),遗传距离最近(D=0.017 1),在聚类分析中也聚得最近(图4);根据Nei’s遗传相似性系数,使用NTSYSpc 2.1软件中的SHANN对假百合18个居群进行UPGMA聚类分析,18个居群按照遗传距离远近以及遗传一致度高低聚为2大支4个亚支(图4)。其中雅江和乡城遗传一致度最高,遗传距离最近首先聚在一起,然后与康定和木里聚为Clade Ⅰ;秦岭、松潘、理县、黑水和金川遗传一致度与地理距离也较近优先聚为Clade Ⅱ;西昌、冕宁、越西与美姑形成Clade Ⅲ;禄劝、大姚等与其它居群则聚为Clade Ⅳ。2个大类在0.70阈值处聚在一起。Mantel关于地理距离和分子遗传距离的检测揭示了假百合居群间的地理距离和遗传距离存在显著的相关性(r=0.410,P=0.01)。
3讨论
3.1中国假百合属遗传多样性与种间亲缘关系的探讨
生物的遗传多样性是珍贵的自然资源,也是人类赖以生存的基础。物种的遗传多样性越高说明其遗传背景越复杂,同时也具有更强的进化潜力和对恶劣环境的耐受力,且更容易扩大生境以及适应新的环境[20]。在本研究中,利用7对ISSR引物对中国假百合属4个类群进行了扩增。结果显示多态性百分率(PPB)为100%。通常来说当PPB>50%时,被试物种的遗传多样性就被视为较高,这表明中国假百合属植物具有很高的遗传多样性。根据花的形态以及颜色划分,中国假百合属下有3个种:假百合、钟花假百合与大叶假百合。而后在云南西北部发现了N.bulbuliferum×N.campanulatum。因其形态特征在未开花时差距甚微,且假百合与钟花假百合之间存在杂交,从表型上很难区分。然而假百合为名贵中药材,所以对其正确鉴定十分必要,在本研究中遗传相似性系数均值(Gs)为0.702 2,表明假百合属种间具有较高的遗传相似性;钟花假百合作为杂交个体的母本与N.bulbuliferum×N.campanulatum具有较高的遗传相似性(Gs=0.813 3)且在UPGAMA聚类分析中聚为一支,亲缘关系最近,这可能与钟花假百合作为杂交个体的母本且在种子植物中为母系遗传有关[21-22];大叶假百合与假百合在聚类分析时也相距最远,表明其亲缘关系最远。
3.2假百合遗传多样性及居群间遗传分化
对于生物遗传多样性的研究是我们探讨物种起源以及进化的基础,并且可以为人类提供合理的保护、开发和利用的理论依据[23-24]。根据ISSR分析结果显示,假百合物种水平上多态位百分率(PPB)为93.9%,Nei’s遗传多样性指数(H)为0.458 2,Shannon’s信息多样性指数(I)为0.343 2。这一较高的遗传多样性可能是由于假百合的分布从秦岭延伸到东喜马拉雅以及生长在平均海拔高度为3 000 m的横断山区,而这一地区具有复杂的地质地貌,高山峡谷纵横,生活环境多变。通常,物种具有广泛的分布就会展现出较高的遗传多样性,这种现象在竹黄菌[25-26]的研究中也有发现。一方面,长期的地理隔离导致各居群间适应不同的环境,从而产生较大的遗传变异;另一方面,也可能是假百合的祖先类群具有多样性复杂的基因库,而后由于青藏高原隆起[27]、横断山造山运动[28-29]以及第四纪冰期气候的反复波动导致生境片段化后,而出现如今不同分布地间的遗传差异。
假百合种下居群间的遗传分化系数(Gst)为0.749 5,远远高于Nybom统计的多种植物ISSR遗传分化系数均值(Gst=0.34)[30],同样也高于宝兴百合(Gst=0.642 5)、匍茎百合(Gst=0.563 7)的ISSR遗传分化系数[14],这说明假百合居群间存在较高的遗传分化。而且从NTSYSpc 2.10聚类结果以及各居群的采样分布可以看出,Clade Ⅰ与Clade Ⅱ被折多山山脉隔开;Clade Ⅲ与Clade Ⅳ被金沙江流域阻隔;而这两大支之间存在锦屏山山脉。横断山区复杂的地貌环境可能孕育了假百合的异质居群结构。而Mantel关于地理距离与遗传距离的检测存在极显著的相关性也恰好说明这一点。虽然假百合有性生殖依靠虫媒传份,这会使得居群间遗传分化降低,但是假百合生长在横断山区,该地区由高黎贡山、大雪山、锦屏山等7条几乎平行的山脉和金沙江、澜沧江与怒江等几大流域组成,高山峡谷纵横,大多数山脉海拔达到五千多米,形成了天然的地理隔离屏障。这种依靠虫媒传粉方式的植物被自然条件阻隔,从而限制了居群间的基因交流,这也是造成假百合居群间遗传分化值高的又一原因。再者,假百合居群间的基因流(Nm)为0.181 6,当Nm<1时,遗传漂变就可导致居群间明显的遗传分化[31],说明遗传漂变也可能是假百合种下居群间遗传分化较高的原因。
分布范围、种子传播机制、繁育系统等因素都可影响群体遗传结构,但在异交类型植物中群体间的分化值一般较小[32]。假百合居群间遗传分化系数远高于远交植物,这可能是因为假百合具有复杂的兼性生殖繁育系统[33]。在自然生存环境中,假百合具有大量的小鳞茎。野外取样过程中观察发现小鳞茎繁殖能力极强,在脱离母株后,大多数小鳞茎便形成无性生殖植株,同时也存在种子实生苗。假百合在漫长的进化历史中和自然选择的压力下,已经形成了适应环境要求的有性和无性两种繁育制度。通过大量观察发现,假百合聚居现象特别明显,大多成群分布,遇到有水沟的地方则沿水流方向两岸分布。据此推测依靠小鳞茎的无性繁殖体系是假百合主要的繁殖方式。较强的无性繁殖能力导致了假百合居群内遗传多样性的降低。
3.3假百合可持续利用与保护策略
对于物种来说,遗传多样性水平高低与对环境的适应能力以及变异和进化潜力的强弱是紧密相关的[34]。丰富的遗传多样性是物种具有更高的适应和存活能力的基础,从而才能提高物种的进化、繁殖以及优化遗传结构的能力[35]。基于目前的研究显示,假百合具有很高的遗传分化。因此,对集巨大药用价值与观赏价值于一体的假百合野生资源以及遗传多样性的保护是非常有意义的。假百合野生居群被药农大量采挖,加之近年来道路建设使得假百合生境破坏以及原生态旅游业的大规模开发,假百合大而艳丽的花朵吸引了游客的采摘。所以,对现存假百合居群应该采取一些有效的保护措施,比如:对假百合的生存环境适度保护,禁止人们过度采挖等;因秦岭、松潘、康定、西昌、德钦等地具有较高的遗传多样性,这些具有丰富的基因多样性资源的地区是将来进行育种的优良种质库的来源,在制定保护措施和对其利用时应多加注意。
参考文献:
[1]GAO Y D,HARRISA J,ZHOU S D,etal.Evolutionary events inLilium(includingNomocharis,Liliaceae) are temporally correlated with orogenies of the Q-T plateau and the Hengduan Mountains [J].MolecularPhylogeneticsandEvolution,2013,68(3):443-460.
[2]GAO Y D,ZHOU S D,HE X J.Chromosome diversity and evolution in tribe Lilieae (Liliaceae) with emphasis on Chinese species [J].JournalofPlantResearch,2012,125:55-69.
[3]梁松筠.百合科(狭义)植物的分布区对中国植物区系研究的意义[J].植物分类学报,1995,33(1):27-51.
LIANG S Y.Chorology of Liliaceae (S.STR) and it’s bearing on the Chinese acta phytotaxonom [J].JournalofSystematicsandEvolution,1995,33(1):27-51.
[4]BAKER J G.Revision of the genera and species ofTulipeae[J].JournaloftheLinneanSociety,1871,14:211-310.
[5]BOISSIER E.Genevaeet Basileae:H.Georg Bibliopolam[J].FloraOrientalis,1884,5:172-203.
[6]COTTON A D,STEARN W T.The genusNotholirion[J].LilyYear-Book,1934,3(19):f.6.
[7]STEARN W T,COTTON A D.A note onParadisea,DiurantheraandNotholirion[J].KewBulletin,1950,5(3):421.
[8]ZIETKIEWICE E,RAFALAKE A,LABUDA D.Genome fingerprinting by simple sequence repeat(SSR) anchored polymerase chain reaction amplification[J].Genomics,1994,20(2):176-183.
[9]DOYLE J J.Gene trees and species trees:molecular systematics as an one-character taxonomy[J].SystematicBotany,1992,71:144-163.
[10]TSUMURA Y,OHBA K,STRAUSS S H.Diversity and inheritance of inter-simple sequence repeat polymorphisms in Douglas-fir(Pseudotsugamenziesii) and sugi (Cryptomeriajapoonica) [J].TheoreticalandAppliedGenetics,1996,9:40-45.
[11]GUO W H,JEONG J,KIM Z,etal.Genetic diversity ofLiliumtsingtauensein China and Korea revealed by ISSR markers and morphological characters[J].BiochemicalSystematicsandEcology,2011,39:352-360.
[12]XI M L,SUN L N,QIU S,etal.Invitromutagenesis and identification of mutants via ISSR in lily (Liliumlongiflorum)[J].PlantCellReports,2012,31:1 043-1 048.
[13]ZHAO L H,LIU H M,CAI G Z ,etal.Assessment of the genetic diversity and genetic relationships ofLiliumin China using ISSR markers[J].BiochemicalSystematicsandEcology,2014,55:184-189.
[14]沈呈娟,周颂东,何兴金.宝兴百合与匍茎百合遗传多样性的ISSR分析[J].西北植物学报,2014,34(7):1 331-1 338.
SHEN C J,ZHOU S D,HE X J.Genetic diversity ofLiliumduhartreiandLiliumlankongenserevealed by ISSR markers [J].ActaBotanicaBoreali-OccidentaliaSinica,2014,34(7):1 331-1 338.
[15]NEI M.Estimation of average heterozygosity and genetic distance from a small number of individuals [J].Genetics,1978,89:583-590.
[16]YEH F C,YANG R C,BOTLE T B J,etal.POPGENE:The user friendly shareware for population genetic analysis.(Ver.1.32)[M].Canada:Molecular Biology and Biotechnology Centre,University of Alberta,1997.(program available from:http://www.ualberta.ca/~fyeh/).
[17]ROHLF F J.NTSYS-pc:Numerical Taxonomy and Multivariate Analysis System (Ver.2.1) Exeter Software [CP/DK].New York:Setauket,1998.
[18]EXCOFFIER L.AMOVA:Analysis of Molecular Variance (Ver.1.55) [CP/DK].Switzerland:Genetics and Biometry Laboratory,University of Geneva,1995.
[19]PEAKALL R,SMOUSE P E.GenAlEx 6.5:genetic analysis in Excel.Population genetic software for teaching and research——an update[J].Bioinformatics,2012,28:2 537-2 539.
[20]MARTINS M,TENREIRO R,OLIVEIRA M.Genetic relatedness of Portuguese almond cultivars assessed by RAPD and ISSR markers [J].PlantCellReports,2003,22:71-78.
[21]BAUMEL A,AINOUCHE M L,BAYER R J,etal.Molecular phylogeny of hybridizing species from the genusSpartina(Poaceae)[J].MolecularPhylogeneticsandEvolution,2002,22:303-314.
[22]ZHA H G,MILNE R I,SUN H.Asymmetric hybridization inRhododendronagastum:a hybrid taxon comprising mainly F1s in Yunnan,China [J].AnnalsofBotany,2010,105:89-100.
[23]HELENUR K.High levels of genetic polymorphism in the insular endemic herbJepsoniamalvifolia[J].JournalofHeredity,2001,92:427-432.
[24]LI E X,YI S,QIU Y X,etal.Phylogeography of two east asian species inCroomia(Stemonaceae) inferred from chloroplast DNA and ISSR fingerprinting variation[J].MolecularPhylogeneticsandEvolution,2008,49:702-714.
[25]FAN Y,ZHONG C G,CHENG J,etal.Genetic differentianton ofShiraiabambusicolapopulatins from east China revealed by RAPD analysis[J].GenomicsandAppliedBiology,2009,28(5):889-895.
[26]QI S S,FAN Y,GONG Z N,etal.Genetic diversity ofShiraiabambusicolafrom East China assessed using ISSR markers [J].BiochemicalSystematicsandEcology,2015,59:239-245.
[27]HARRISON T M,COPELAND P,KIDD W S F,etal.Raising Tibet [J].Science,1992,255:1 663-1 670.
[28]陈富斌.再论横断运动[J].火山地质与矿产,1996,17:14-21.
CHEN F B.Second discussion on the Hengduan movement [J].VolcanologyandMineralResources,1996,17:14-21.
[29]施雅风.晚新生代以来青藏高原的隆起与环境演化[M].广州:广州科学技术出版社,1998:6-20.
[30]NYBOM H,BARTISH I V.Effects of life history traits and sampling strategies on genetic diversity estimates obtained with RAPD markers in plants[J].PerspectivesinPlantEcologyEvolutionandSystematics,2000,3(2):93-114.
[31]SLATKIN M.Gene flow in natural population [J].AnnualReviewofEcology,Evolution,andSystematics,1985,16:393-430.
[32]HAMRICK J L,GODT M J W.Plant Population Genetics,Breeding,and Genetic Resources[M].Sinauer:Sunderland,Mass,1990:43-63.
[33]BUSSEL J D.The distribution of random amplified polymorphic(RAPD)diversity amongst population ofIsotomapetraea(Lobeliaceae)[J].MolecularEcology,1999,8:775-789.
[34]HOLSINGER K E.Genetics and Conservation of Rare Plant [M].New York:Oxford University Press,1991:20-55.
[35]FRANKHAM R,BALLOU J D,BRISCOE D A.Introduction to Conversation Genetics[M].Cambridge:Cambridge University Press,2002:10-36.
(编辑:宋亚珍)
Genetic Relationships ofNotholirionand Genetic Diversity ofN.bulbuliferumin China Revealed by ISSR Markers
YANG Mei,ZHOU Songdong*,HE Xingjin,PENG Chang
(Key Laboratory of Bio-Resources and Eco-Environment of Ministry of Education,College of Life Sciences,Sichuan University,Chengdu 610064,China)
Abstract:To understand the sibship of Notholirion and the genetic differentiation of N.bulbuliferum,the genetic diversity of 255 individuals from 24 populations was analyzed by using seven inter-simple sequence repeat (ISSR) markers.(1)The results of the UPGMA analysis exhibited significant genetic differentiation between N.bulbuliferum,N.campanulatum,and N.macrophyllum,which indicated that they are separate species.(2)N.bulbuliferum×N.campanulatum clustered together with N.campanulatum,which may be a result from the matrilineal inheritance and strong asexual reproduction capacity;its low gene flow (Nm=0.216 0) implied that N.bulbuliferum×N.campanulatum may be undergoing differentiation.(3)The Mantel test showed a substantial significant correlation between genetic and geographic distances in N.bulbuliferum (r=0.410,P=0.01),and clustered four groups.(4)The analysis of molecular variance (AMOVA) revealed that the variation of N.bulbuliferum among the populations was 77.12% (P<0.01),and the variation within the population was 22.88% (P<0.01).These findings clearly evidenced the genetic relationship of Notholirion in the Hengduan Mountains.Importantly,the high diversity of N.bulbuliferum found in this study indicated that reasonable measures should be taken for the conservation and restoration for medicinal and ornamental plant species.
Key words:genetic diversity;inter-simple sequence repeats (ISSR);Notholirion bulbuliferum;N.campanulatum;N.macrophyllum
中图分类号:Q346.+5;Q789
文献标志码:A
作者简介:杨梅(1989-),女,在读硕士研究生,主要从事植物系统进化研究。E-mail:meiyang100418@163.com*通信作者:周颂东,副教授,硕士生导师,主要从事植物分类与系统进化相关方面的研究。E-mail:songdongzhou@ aliyun.com
基金项目:国家自然科学基金(31270241,31470009,31570198);国家基础条件平台;国家标本平台教学标本子平台(http://mnh.scu.edu.cn);科技部基础性研究专项(2013FY112100)
收稿日期:2015-12-02;修改稿收到日期:2016-02-01
文章编号:1000-4025(2016)03-0449-09
doi:10.7606/j.issn.1000-4025.2016.03.0449
