多重PCR靶向富集结合高通量测序筛查结直肠癌中MMR基因的突变
2016-04-21陈慧杰刘方奇
黄 凯 陈慧杰 刘方奇 徐 烨 李 轩 南 蓬
(1复旦大学生命科学学院生物多样性与生态工程教育部重点实验室 上海 200433;2上海同达科信生物技术发展有限公司 上海 200080;
3上海生物信息技术研究中心 上海 201203; 4复旦大学附属肿瘤医院大肠外科 上海 200032;
5中国科学院上海生命科学研究院植物生理生态研究所 上海 200032)
多重PCR靶向富集结合高通量测序筛查结直肠癌中MMR基因的突变
黄凯1,2陈慧杰3刘方奇4徐烨4李轩5南蓬1△
(1复旦大学生命科学学院生物多样性与生态工程教育部重点实验室上海200433;2上海同达科信生物技术发展有限公司上海200080;
3上海生物信息技术研究中心上海201203;4复旦大学附属肿瘤医院大肠外科上海200032;
5中国科学院上海生命科学研究院植物生理生态研究所上海200032)
【摘要】目的探讨多重PCR靶向富集结合高通量测序技术在结直肠癌 (colorectal cancer,CRC)中检测错配修复 (mismatch repair,MMR)基因种系突变的应用。方法收集17例CRC患者和14例正常人的血液并提取基因组DNA;设计和优化寡聚核苷酸探针,使其能对5个基因MLHl、MSH2、PMS1、PMS2和MSH6的73个外显子序列进行有效的PCR扩增和富集;应用多重PCR技术靶向富集样本MMR基因的外显子序列;扩增产物进行文库构建和高通量测序,检测MLHl、MSH2、PMS1、PMS2和MSH6基因的突变情况。结果31个样本共得到2.7Gb的数据,平均reads数为287 048,测序数据中平均82.18%可与参考序列进行比对,外显子序列平均覆盖度为99.9%,平均测序深度为2 282。在MMR基因的外显子区域共发现13种非同义单核苷酸变异 (single nucleotide variation,SNV)、2种同义 SNV,其中MSH6的c.G3205C:p.G1069R为未见报道SNV。检测结果经Sanger法测序验证,结果一致。结论多重PCR靶向富集结合高通量测序技术是一套通量高、速度快、费用低、准确性高的MMR基因突变筛查方法。
【关键词】错配修复基因;Lynch综合征;靶向富集;高通量测序
*This work was supported by the National Key Basic R&D Program of China (973 Plan,2012CB316501),the National Key Technologies R&D Program of China (863 Plan,2012AA02A602) and the National Natural Science Foundation of China (31271409,31401128).
结直肠癌 (colorectal cancer,CRC)是全世界第三大常见恶性肿瘤[1]。研究发现35%的CRC存在家族易感性[2],其中最常见的是Lynch综合征,即遗传性非息肉病性结直肠癌 (hereditary nonpolyposis colorectal cancer,HNPCC),占所有病例的2%~5%[3]。错配修复 (mismatch repair,MMR)基因发生胚系突变是Lynch综合征的遗传学发病机制[4-5]。研究发现至少5种MMR基因 (MLH1、MSH2、MSH6、PMS1、PMS2)的胚系突变会导致Lynch综合征,其中MLH1的胚系突变占50%,MSH2占40%,MSH6占7%,剩下的基因占3%[6]。在家系中,MMR基因种系突变携带者为发生Lynch综合征相关肿瘤的高危人群,检测MMR基因种系突变能很好地预测Lynch综合征相关肿瘤的发生危险[7]。
直接基因测序法是检测MMR基因胚系突变最灵敏和最特异的方法,但Sanger法测序不仅费时而且价格昂贵。除了直接测序外的筛检方法还有:MMR蛋白表达免疫组化分析 (immunohistoch-meistry,IHC)和微卫星不稳定性 (microsatellite instability,MSI)检测。IHC检测可以确定受累基因,从而指导直接测序,不足之处是某些突变只导致MMR蛋白功能受损,而并没有严重影响到MMR的表达,此时IHC检测可能为阴性[8-10]。另外,有些MSH6胚系突变只导致MMR受损但不会引起MSI[11]。
与传统的Sanger法测序相比,第二代高通量测序技术具有通量高、速度快、费用低等优点,结合特定核酸序列靶向富集技术,可以实现对疾病最相关的基因进行深度测序。目前已有多种疾病的相关基因运用该技术进行了深度测序,并取得了一定的研究成果[12-14]。本研究对17例CRC病例和14例正常人样本进行多重PCR靶向富集MMR基因外显子序列,结合高通量测序技术,检测5个基因 (MLH1、MSH2、MSH6、PMS1、PMS2)的胚系突变,探讨多重PCR靶向富集结合高通量测序在MMR基因胚系突变检测中的应用,以期为Lynch综合征的临床诊断和研究提供新的参考。
资 料 和 方 法
研究资料CRC患者17例,年龄63~81岁,其中男8例,女9例;健康志愿者14例,年龄30~50岁,其中男6例,女8例。CRC样本由复旦大学附属肿瘤医院提供,正常人来自健康志愿者,所有患者及志愿者均知情同意,并签署了经上海市人类遗传资源委员会批准的知情同意书。
靶向富集探针的设计使用在线引物设计工具探针 3设计PCR靶向富集MMR基因所需探针,对5个基因的73个外显子总共11 419个碱基设计了87对探针,PCR扩增产物长度为257~528 bp,该组合探针能覆盖5个MMR基因 (表1)的所有外显子区域。引物合成由上海生工生物工程有限公司合成。
MMR相关基因的靶向富集及高通量测序将设计和优化的87对探针分成11组对样本基因组DNA 进行多重PCR扩增,每组多重PCR的DNA模板量为50 ng,其中第1~8和11组使用多重PCR 5× Master Mix试剂盒(美国NEB公司)进行多重PCR扩增,第9和10组使用GB缓冲液扩增(日本TaKaRa公司LA Tag DNA聚合酶)试剂盒进行多重PCR扩增,扩增条件为:95 ℃预变性1 min;95 ℃变性20 s、63 ℃退火60 s、68 ℃延伸30 s,30个循环;68 ℃延伸5 min。扩增产物经AMpure磁珠(美国Beckman公司)纯化后等量混合,取200 ng使用TflexTM快速DNA-Seq试剂盒(美国NEX公司)构建测序文库,然后用Miseq测序仪进行高通量测序。
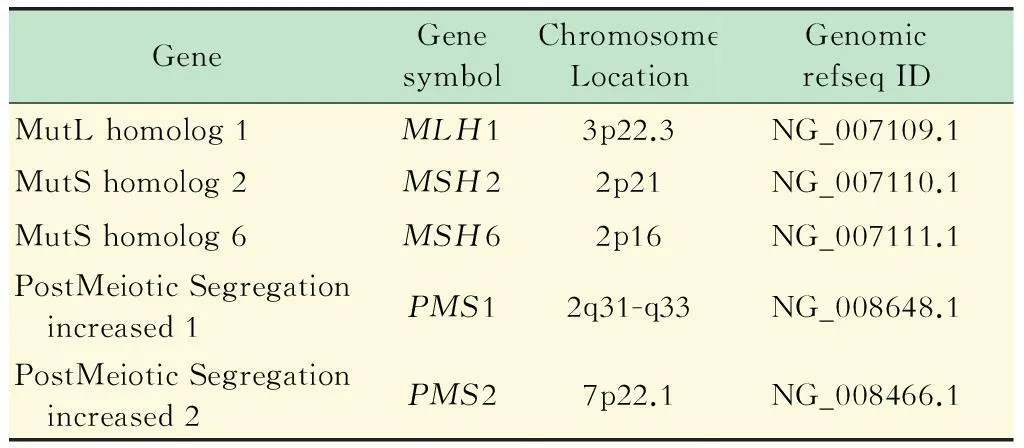
表1 靶向富集探针设计参考序列信息表
生物信息学分析及Sanger测序法验证测序数据分析使用Bowtie2软件[15]与人类基因组参考序列 (hg19)进行比对,用SAM工具软件[16]对结果进行突变筛查分析 (筛选条件:比对质量>20且测序深度>100),用ANNOVAR软件[17]对筛选的突变进行功能注释。分析所得结果用Sanger法测序验证。测序样本的制备和Sanger测序由上海生工生物工程有限公司完成,测序所用试剂和仪器为BigDye Terminator v1.1 Cycle Sequencing kit 和ABI 3730 测序仪。
结果
靶向富集及测序结果通过多重PCR靶向富集MMR基因和高通量测序,31个样本共得到2.7 gigabases (Gb)数据,平均每个样本86 Mb,平均reads数为287 048。其中30个样本的5个筛查基因 (MLH1、MSH2、MSH6、PMS1、PMS2)外显子测序覆盖度为100%,1个为96.8%,平均覆盖度为99.9%,外显子区域平均测序深度为2 284。经过生物信息分析,31个测序样本共发现13种非同义单核苷酸变异 (single nucleotide variation,SNV)、2种同义SNV,在非同义SNV中,位于MLH1基因的有3个:c.A655G:p.I219V、c.T1151A:p.V384D和c.C2101A:p.Q701K;位于MSH2基因的有3个:c.C1168T:p.L390F、c.A1690G:p.T564A和c.G2425A:p.E809K;位于MSH6基因的有3个:c.G116A:p.G39E、c.G3205C:p.G1069R和c.A3488T:p.E1163V;位于PMS2基因的有4个:c.G59A:p.R20Q、c.A1621G:p.K541E、c.C1408T:p.P470S和c.C1454A:p.T485K。2个同义SNV是MSH6基因c.T3306A和PMS2基因c.C780G;基因PMS1未检测到SNV。这些SNV与数据库dbSNP和InSight[18](http://www.insight-group.org/mutatioons)进行检索,发现14种已经报导,其中MSH6的c.G3205C:p.G1069R未见报导 (表2)。
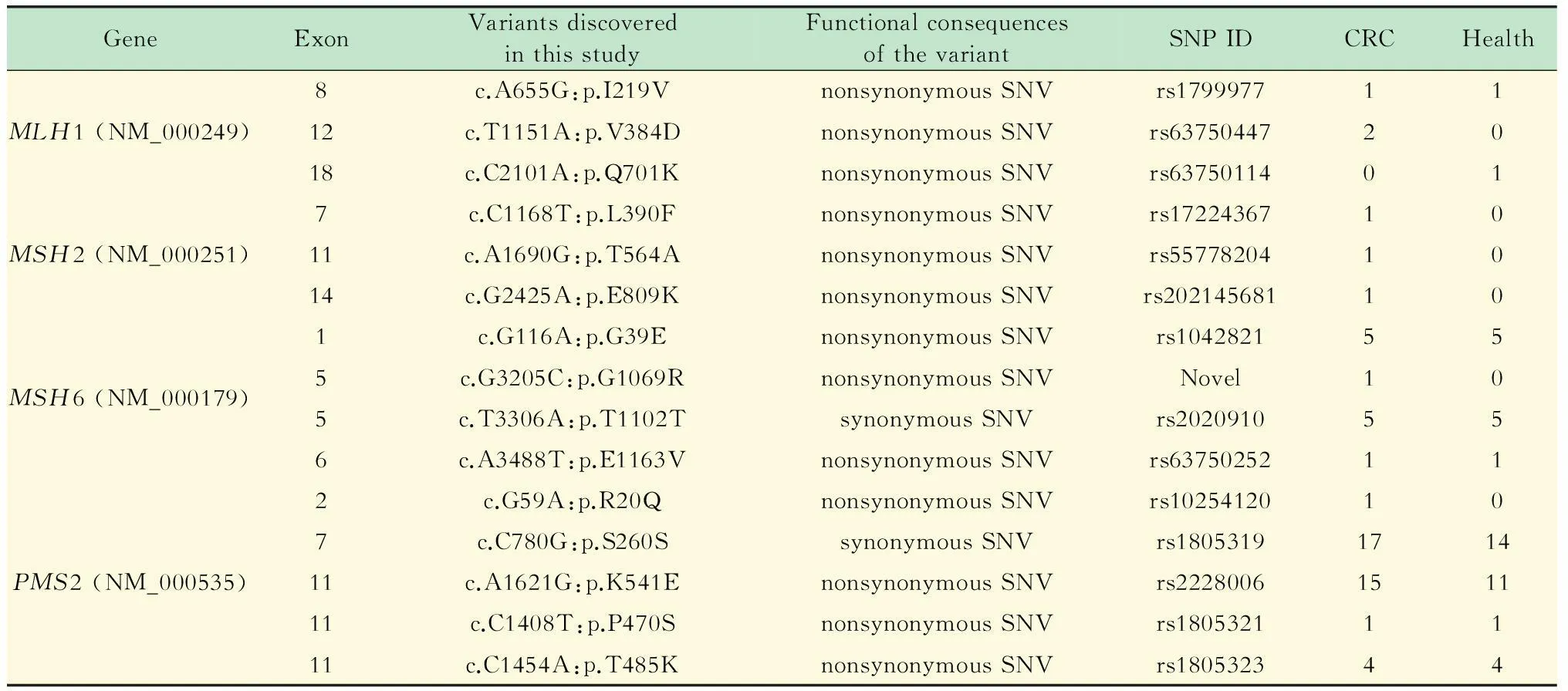
表2 MMR基因突变筛查结果
Sanger测序验证为了验证多重PCR靶向富集高通量测序的检测结果,我们对每种单核苷酸变异类型选取了一个样本进行了单管的PCR扩增,扩增产物纯化后进行Sanger测序。单管PCR扩增及Sanger测序引物与多重PCR靶向富集高通量测序所用的引物相同。结果显示:Sanger测序验证结果与多重PCR靶向富集高通量测序结果一致 (图1)。
讨论
目前,新一代测序以其低成本、高通量的优势成为生命科学研究和临床疾病基因检测的重要工具,结合特定核算序列靶向富集技术,可以更加高效经济的对疾病最相关的基因进行深度测序和研究。Keller等[19]应用靶向富集高通量测序研究多形性成胶质细胞瘤 (glioblastoma multiforme,GBM)SNP,结合全基因组关联研究 (the genome-wide association study,GWAS)和已知的疾病相关联SNP,发现一些SNP与吸烟、体质指数、乳腺癌和高分级胶质瘤相关联。Ying等[20]运用目标区域捕获高通量测序技术对6个苯丙酮尿症相关基因 (PAH、PTS、GCH1、QDPR、PCBD1和GFRP)的所有外显子进行了突变检测,共发现了PAH基因中23个已知变异,以及6个PAH和PTS的新型突变。
CRC是我国最常见的恶性肿瘤之一,并有逐年上升的趋势,部分CRC具有家族遗传易感性。由于我国的计划生育政策和城市化人口迁徙,家系正变得越来越小,家系成员也越来越趋于分散,这对遗传性CRC的研究和诊断带来一定的挑战。因此,对普通CRC患者或健康人员进行遗传学检查,以确认或排除肿瘤遗传易感性是具有一定应用价值的。MMR基因的胚系突变被认为是最常见的遗传性CRC-Lynch综合征发生的分子遗传学基础,检测MMR基因种系突变能很好地预测Lynch综合征相关肿瘤的发生危险。目前检测MMR基因突变的方法依然是单个基因逐一检测且以一代测序为主[21-23],但此方法费时、通量低且价格昂贵,不适合临床大样本量的检测,而高通量测序应用于MMR基因检测的报道极少。因此,我们设计了一套探针,通过多重PCR靶向富集MMR基因,结合高通量测序检测MMR基因的胚系突变。
为了能够有效的多重PCR靶向富集5个MMR基因MLH1、MSH2、MSH6、PMS1、PMS2的外显子序列,我们设计了87对探针,覆盖了5个基因73个外显子共11 419个碱基序列,PCR产物的长度为257~528 bp。设计的探针不仅包含了外显子区域,也包含了外显子与内含子的结合位置,同时,这些探针具有相近的TM值,这样多重PCR时可以使用相同的退火条件。经过实验优化,我们将87对探针分成11组进行多重PCR,每组包含2~11对探针,其中第9、10组扩增区域GC含量较高,使用TaKaRa LA Tag with GC Buffer试剂盒能有效扩增,其余的多重PCR使用NEB MixMultiplex PCR 5× Master Mix试剂盒进行扩增。多重PCR反应条件经过多次实验优化后可使每对探针都能够有效扩增,其扩增条件为:95 ℃预变性1 min;95 ℃变性20 s、63 ℃退火60 s、68 ℃延伸30 s,30个循环;68 ℃延伸5 min。多重PCR产物经过纯化后可进行测序文库制备,在测序文库构建时加入标签序列,可以实现多样本的检测。本研究使用了31种标签序列,对17例CRC病例和14CRC正常人的样本进行了标记。通过引入标签序列,使得多样本基因检测更加高效快捷。以高通量测序平台Miseq和一代Sanger测序平台ABI 3730为例,我们将自己设计的多重PCR靶向富集结合高通量测序的方法和传统的单管PCR-Sanger测序法检测5个MMR基因外显子所用时间和费用进行了比较。单管PCR-Sanger测序需对每个基因的外显子进行单管PCR扩增,扩增产物纯化后进行Sanger法测序,每检测一个样本的MMR基因胚系突变需要分别进行87个单管PCR及87个Sanger测序反应;多重PCR技术可同时扩增多个MMR基因外显子序列,扩增产物纯化后进行文库构建并测序,每检测1个样本只需11管多重PCR和1次文库构建。如表3所示:由于受合成引物费用的影响,当检测少量样本时,例如检测1个样本,所产生的费用主要是引物合成费用;同时,二代测序仪运行通量高,每次运行可同时检测上百个样本,当检测单个样本时需要和其他样本混合测序,时间上比一代测序花费更多;但当样本数量达到100个或更多时,检测时间和费用主要受限于PCR扩增和测序技术,靶向富集结合高通量测序检测MMR基因突变的时间和费用要远远小于单管PCR-Sanger法测序。因此,多重PCR靶向富集结合高通量测序技术是一套通量高、速度快、费用低的MMR基因突变筛查方法,适合临床大样本量的MMR基因突变筛查。
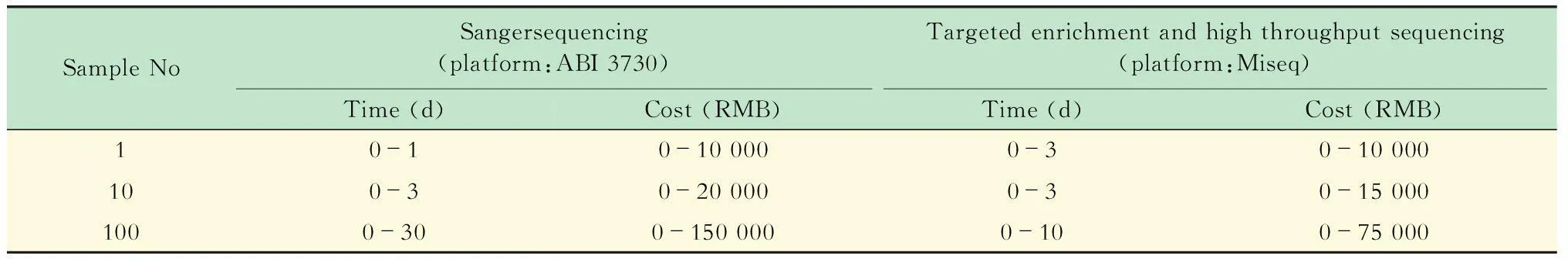
表3 MMR基因外显子测序费用比较
利用多重PCR靶向富集高通量测序,我们在17例DRC病例和14例正常人的样本中共发现外显子区域13种非同义单核苷酸变异、2种同义单核苷酸变异。与dbSNP数据库进行检索,发现MSH6的c.G3205C:p.G1069R为未报导的单核苷酸变异,其余14种为已报导单核苷酸变异。将这些已知单核苷酸变异位点进行数据库ClinVar (http://www.ncbi.nlm.nih.gov/clinvar/)检索发现:位于MLH1基因第18外显子c.C2101A:p.Q701K为致病或可能致病的突变;位于MSH2基因第14外显子c.G2425A:p.E809K为致病性不确定的突变;其余10种非同义单核苷酸变异为可容忍的变异,可能与CRC的发生没有关联。致病或可能致病的突变c.C2101A:p.Q701K由范怡梅等[22]于2005年首次发现,并在随后的功能分析表明此变异造成MLp和PMS2互动效率降低,可能提高突变携带者患胃肠肿瘤的风险[24]。在另一项研究中,c.C2101A:p.Q701K在2例肿瘤样本中检测出,而在100例非肿瘤对照样本中并没有发现该突变,同时对这两例携带该突变的肿瘤样本进行MSI检测,5个常规位点检测出4个阳性结果[25]。本研究在一例健康人 (年龄<50岁)中检测出该突变,提示该志愿者有可能存在肿瘤遗传易感性,应询问是否有患病家族史,并做进一步的检测 (IHC或者MSI)和随访;致病性不确定的突变c.G2425A:p.E809K在1例CRC患者中检测出,同时检索1 000 Genomes Projects (2012 release)数据库发现该等位基因的频率只有0.000 5,经Mutation Taster预测该单核苷酸变异为有害变异[26],可能会影响MMR的功能,提示该变异可能与CRC的发生有关。在本研究中新发现一种单核苷酸变异,即位于MSH6的第5外显子c.G3205C:p.G1069R,此变异在1例CRC患者中检测出,经Mutation Taster预测该单核苷酸变异为有害变异,推测可能与CRC的发生有关。然而,进一步的功能分析研究是必要的,以确认MSH2基因c.G2425A:p.E809K和MSH6基因c.G3205C:p.G1069R的临床意义。每种类型的非同义SNV我们都挑选了一个样本进行Sanger测序,结果与高通量测序一致,表明靶向富集高通量测序技术对于发现MMR基因的胚系突变具有高度的准确性。
多重PCR靶向富集结合高通量测序技术用于MMR基因的外显子突变检测准确且经济、高效,其与临床表型对照研究,可为Lynch综合征遗传风险的评估和治疗方案的制定提供新的参考。
参考文献
[1]PETO J.Cancer epidemiology in the last century and the next decade[J].Nature,2001,411 (6835):390-395.
[2]LICHTENSTEIN P,HOLM NV,VERKASALO PK,etal.Environmental and heritable factors in the causation of cancer-analyses of cohorts of twins from Sweden,Denmark,and Finland[J].NEnglJMed,2000,343 (2):78-85.
[3]SAMOWITZ WS,CURTIN K,LIN HH,etal.The colon cancer burden of genetically defined hereditary nonpolyposis colon cancer[J].Gastroenterology,2001,121 (4):830-838.
[4]JASS JR.Role of the pathologist in the diagnosis of hereditary non-polyposis colorectal cancer[J].DisMarkers,2004,20 (4-5):215-224.
[5]BOLAND CR.Decoding hereditary colorectal cancer[J].NEnglJMed,2006,354 (26):2815-2817.
[7]李晓鸥,盛剑秋,付蕾,等.错配修复基因突变检测对遗传性非息肉病性结直肠癌患病风险的预测[J].中华消化杂志,2009,29 (11):721-725.
[8]ROBINSON KL,LIU T,VANDROVCOVA J,etal.Lynch syndrome (hereditary nonpolyposis colorectal cancer)diagnostics[J].JNatlCancerI,2007,99 (4):291-299.
[9]LYNCH HT,LYNCH JF,LYNCH PM.Toward a consensus in molecular diagnosis of hereditary nonpolyposis colorectal cancer (Lynch syndrome)[J].JNatlCancerI,2007,99 (4):261-263.
[10]RAECAARA TE,KORHONEN MK,LOHI H,etal.Functional significance and clinical phenotype of nontruncating mismatch repair variants of MLp[J].Gastroenterology,2005,129 (2):537-549.
[11]SALOVAARA R,LOUKOLA A,KRISTO P,etal.Population-based molecular detection of hereditary nonpolyposis colorectal cancer[J].JClinOncol,2000,18 (11):2193-2200.
[12]GERRARD G,VALGANON M,FOONG HE,etal.Target enrichment and high-throughput sequencing of 80 ribosomal protein genes to identify mutations associated with Diamond-Blackfan anaemia[J].BrJHaematol,2013,162 (4):530-536.
[13]SCOTT CA,PLAGNOL V,NITOIU D,etal.Targeted sequence capture and high-throughput sequencing in the molecular diagnosis of ichthyosis and other skin diseases[J].JInvestDermatol,2013,133 (2):573-576.
[14]LIN HH,SINNER MF,BRODY JA,etal.Targeted sequencing in candidate genes for atrial fibrillation:the cohorts for heart and aging research in genomic epidemiology (CHARGE) targeted sequencing study[J].HeartRhythm,2014,11 (3):452-457.
[15]LANGMEAD B,SALZBERG S.Fast gapped-read alignment with Bowtie 2[J].NatMethods,2012,9 (4):357-359.
[16]LI H,HANDSAKER B,WYSOKER A,etal.The Sequence Alignment/Map format and SAM tools[J].Bioinformatics,2009,25 (16):2078-2079.
[17]WANG K,LI MY,HAKONARSON H.ANNOVAR:Functional annotation of genetic variants from next-generation sequencing data[J].NucleicAcidsRes,2010,38 (16):e164.
[18]KOHONEN-CORISH MR,MACRAE F,GENUARDI M,etal.Deciphering the colon cancer genes-report of the InSiGHT-Human Variome Project Workshop,UNESCO,Paris 2010[J].HumanMutat,2011,32 (4):491-494.
[19]KELLER A,HARZ C,MATZAS M,etal.Identification of novel SNPs in glioblastoma using targeted resequencing[J].PLoSOne,2011,6 (6):e18158.
[20]GU Y,LU KM,YANG GH,etal.Mutation spectrum of six genes in Chinese phenylketonuria patients obtained through next-generation sequencing[J].PLoSOne,2014,9 (4):e94100.
[21]WEI WQ,LIU FQ,LIU L,etal.Distinct mutations in MLp and MSh1 genes in hereditary non-polyposis colorectal cancer (HNPCC) families from china[J].BMBRep,2011,44 (5):317-322.
[22]FAN YM,LIU XR,ZHANG H,etal.Variations in exon7 of the MSh1 gene and susceptibility to gastrointestinal cancer in a Chinese population[J].CancerGenetCytogenet,2006,170 (2):121-128.
[23]KIM YM,CHOE CG,CHO SK,etal.Three novel germline mutations in MLp and MSh1 in families with Lynch syndrome living on Jeju island,Korea[J].BMBRep,2010,43 (10):693-697.
[24]FAN YM,WANG W,ZHU M,etal.Analysis of hMLp missense mutations in East Asian patients with suspected hereditary nonpolyposis colorectal cancer.[J].ClinCancerRes,2007,13 (24):7515-7521.
[25]YAP HL,CHIENG WS,LIM RC,etal.Recurring MLp deleterious mutations in unrelated Chinese Lynch syndrome families in Singapore[J].FamCancer,2009,8 (2):85-94.
[26]SCHWARZ JM,RODELSPERGER C,SCHUELKE M,etal.Mutation Taster evaluates disease-causing potential of sequence alterations[J].NatMethods,2010,7 (8):575-576.
Distinct mutations ofMMRgene in colorectal cancer by targeted enrichment and high-throughput next generation sequencing
HUANG Kai1,2,CHEN Hui-jie3,LIU Fang-qi4,XU Ye4,LI Xuan5,NAN Peng1△
(1MinistryofEducationKeyLaboratoryforBiodiversityScienceandEcologicalEngineering,SchoolofLifeSciences,FudanUniversity,Shanghai200433,China;2Tongji-SCBITBiotechnologyCo.,Ltd.,Shanghai200080,China;3ShanghaiCenterforBioinformationTechnology,Shanghai201203,China;4DepartmentofColorectalSurgery,ShanghaiCancerCenter,FudanUniversity,Shanghai200032,China;5InstituteofPlantPhysiologyandEcology,ShanghaiInstitutesforBiologicalSciences,ChineseAcademyofSciences,Shanghai200032,China)
【Abstract】ObjectiveTo detect the germline mutations of mismatch repair (MMR) genes in colorectal cancer (CRC) using targeted enrichment and high-throughput next generation sequencing,and to explore its applications in research and clinical diagnosis of Lynch syndrome.MethodsGenomic DNA was extracted from 17 patients diagnosed with colorectal cancer and 14 healthy adults, Primers,which could amplify all the 73 exons and flanking regions of 5 MMR genes (MLH1,
MSH2,MSH6,PMS1,PMS2) by multiplex PCR were designed and optimized,PCR products were then sequenced by Illumina Miseq sequencer.ResultsWe obtained approximately 2.7 giga-base sequence data,and the average reads number of individual samples was 287048.On average,82.18% of the reads could be mapped to the reference human genome (HG19).Average coverage and sequencing depth of targeted regions were 99.9% and 2282-fold respectively.After bioinformatic analysis,we found 14 previously annotated single-nucleotide variants (SNVs) in 5 mismatch repair (MMR) genes and 1 novel mutations inMSH6 genes (c.G3205C:p.G1069R).These results were confirmed by sanger sequencing.ConclusionsTargeted enrichment combined with high-throughput next generation sequencing can be used to detect mutations in MMR genes with high sensitivity and lower cost than conventional methods.
【Key words】mismatch repair gene;lynch syndrome;targeted enrichment;high-throughput sequencing
(收稿日期:2015-07-24;编辑:段佳)
【中图分类号】R735.3,Q781
【文献标识码】A
doi:10.3969/j.issn.1672-8467.2016.02.014
国家重点基础研究发展计划(973计划)项目 (2012CB316501);国家高技术研究发展计划(863计划)项目 (2012AA02A602);国家自然科学基金 (31271409,31401128)
△Corresponding authorE-mail:nanpeng@fudon.edu.cn
