奉化江底泥碳、氮、硫转化功能基因的定量研究*
2016-03-13叶丹华
顾 卿 张 旭 叶丹华 郑 毓 孙 静 魏 铮 周 莹
(1.浙江省环境监测中心,浙江 杭州 310015;2.上海大学环境与化学工程学院,上海 200444)
水体中的微生物与区域环境有着密切的联系,其在物质循环与能量流动中起着非常重要的作用。在水生态系统中,微生物既是各种有机物质的分解者和转化者,又是物质和能量的贮存者,同时有些微生物还是食物链中的重要生产者[1]。微生物能直接利用动植物生命活动过程中产生的和死后分解形成的各类碎屑、生物大分子、氨基酸等,在分解过程中释放营养盐为水域植物进行光合作用提供养分。微生物种类繁多,分布广泛,根据微生物的生理特性可将微生物分为不同的功能类群,如产甲烷菌、脂肪降解菌、纤维素降解菌、淀粉降解菌、磷细菌、氨化细菌、硝化细菌、反硝化细菌等。各功能类群的微生物一方面表现在需要特定的生长条件,另一方面表现在物质转化中具有特定的功能,它们在生态环境中发挥着重要的作用。
微生物对环境条件的变化十分敏感,人类活动(例如污染)引起的水体化学循环的改变会导致生境化学待征变化,因而从种类、数量和质量上影响生境中的生物群落,微生物功能类群的种类、数量、分布及变动直接反映着所处生境的污染状态[2]。本研究以奉化江为研究对象,采集奉化江4大支流及干流下游断面底泥中的微生物,以碳、氮、硫关键转化功能基因作为分子标记,对不同的功能类群微生物的丰度进行定量研究。分析河流底泥的碳、氮、硫转化的功能基因丰度与主要环境影响因子的相关性,了解河流底泥物质生物转化过程的主要影响因素,从而为河流水质调控提供参考。
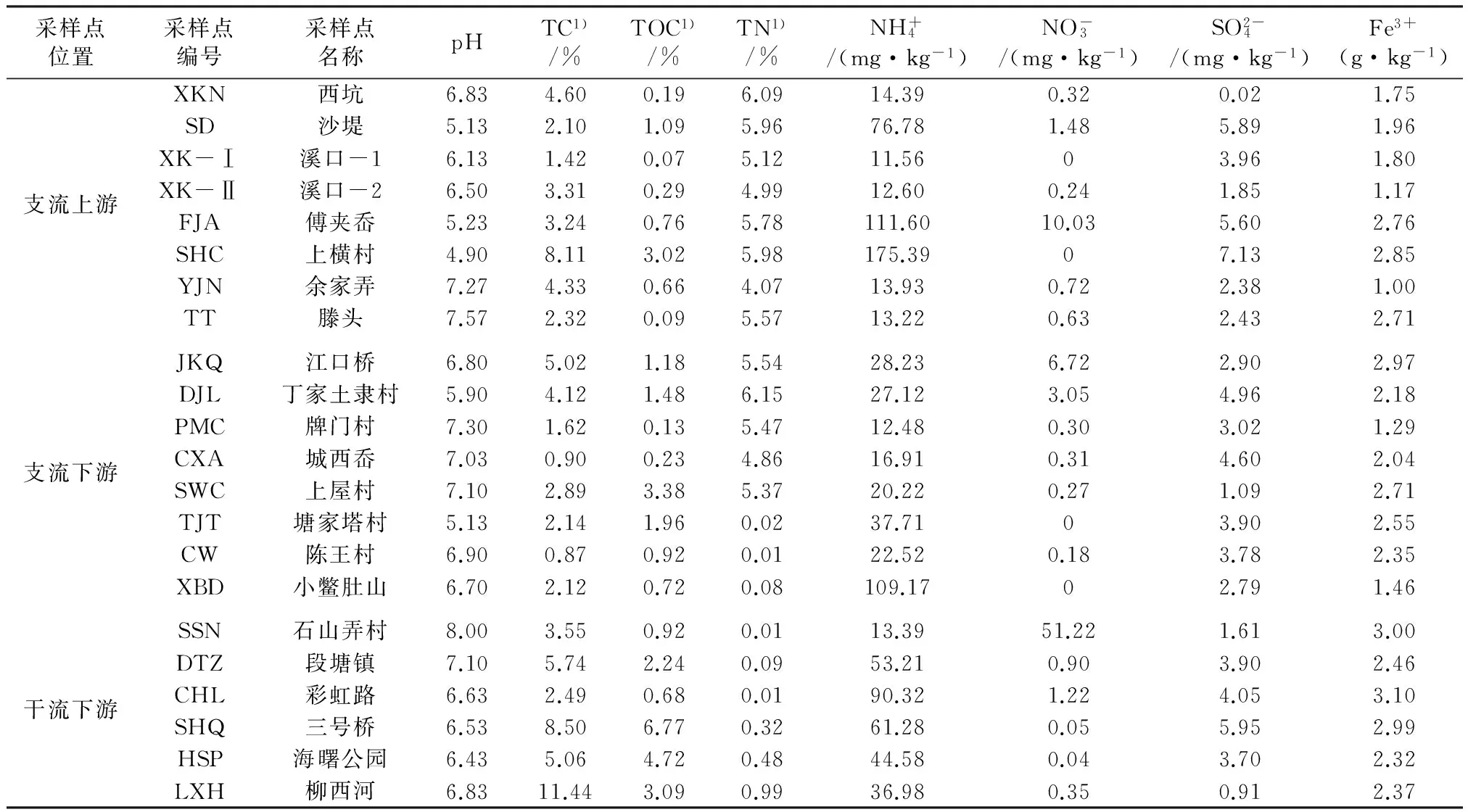
表1 不同采样点底泥理化性质
注:1)以质量分数计。
1 材料与方法
1.1 底泥采集与理化性质分析
底泥样品采自宁波市奉化江及其支流,共设22个采样点,每个采样点用抓斗式采样器采集表层沉积物,每个采样点取3个重复样,贮存于无菌自封袋中,置于干冰中迅速运回实验室,于-80 ℃冰箱保存,并尽快提取DNA。

1.2 DNA提取
底泥DNA采用土壤DNA提取试剂盒(美国MoBio UltraClean公司)提取,提取的DNA用Nanodrop 2000c紫外分光光度计(美国Thermo Fisher Scientific公司)检测浓度与纯度,并在-20 ℃下保存。
1.3 聚合酶链式反应(PCR)扩增
1.3.1 标准质粒制备
以样品DNA为模板,进行各个功能基因的普通PCR扩增,扩增条件参考表2。扩增产物经琼脂糖凝胶电泳检测,切下目的条带,由DNA凝胶回收试剂盒(美国Axygen公司)回收纯化,用pGEM-T克隆载体试剂盒(美国Promega公司)进行纯化产物的酶连,并转化至感受态细胞JM109,在氨苄青霉素平板上进行蓝白斑筛选,选取阳性克隆(白斑)扩繁菌液进行测序分析,进一步鉴定克隆结果。菌液质粒采用 QIAprep试剂盒(德国Qiagen公司)提取,测定其浓度并计算拷贝数,以十倍梯度进行梯度稀释,以不同浓度标准品作为模板进行荧光定量PCR扩增,绘制标准曲线,各功能基因的标准曲线参数见表2。
1.3.2 荧光定量PCR
使用iQ5型荧光PCR仪(美国Bio Rad 公司)对不同功能基进行定量扩增。定量PCR体系:染料法荧光定量试剂(SYBR premix Ex Taq) 10.0 μL,20 μmol/L上下游引物各0.5 μL,DNA模板1.0 μL,双蒸水补充至20 μL。
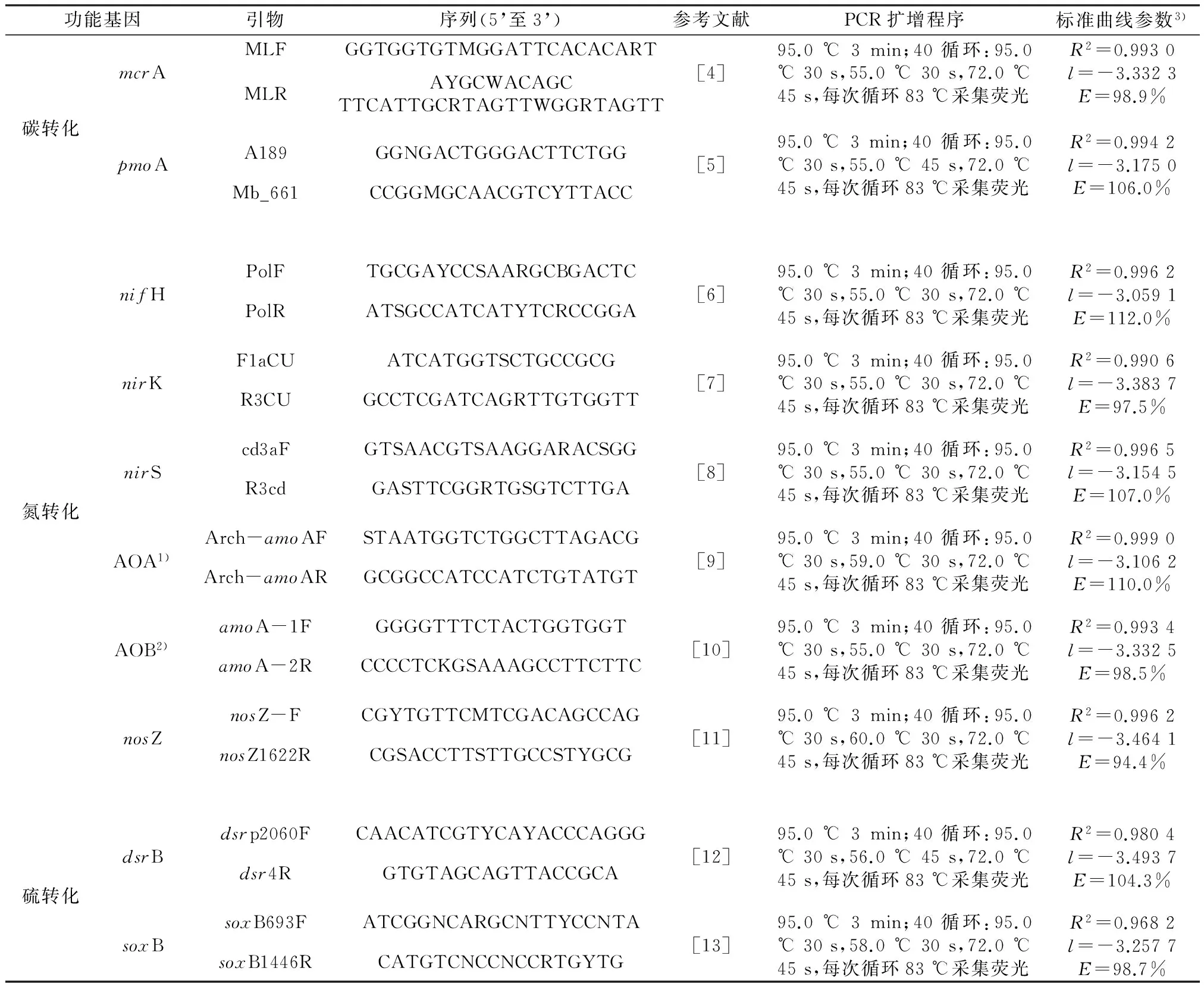
表2 各功能基因引物序列及定量PCR扩增程序和标准曲线参数
注:1)氨氧化古菌硝化基因amoA;2)氨氧化细菌硝化基因amoA;3)R2为标准曲线相关系数,l为标准曲线的斜率,E为扩增效率。
1.4 统计分析
采用 OriginPro 8和Excel 2010 软件进行数据处理和制图,SPSS 22.0软件进行单因素方差分析(AVNOA)和相关性分析。
2 结果与讨论
2.1 碳转化功能基因定量分析
以单位质量DNA提取基因的拷贝数表征基因丰度,对奉化江底泥样品中各功能基因进行定量分析,2种碳转化功能基因(产甲烷基因mcrA与甲烷氧化基因pmoA)的定量分析结果见图1。由图1可见,干流下游mcrA的丰度显著高于上游,可能受到下游溶解氧和养分的影响,奉化江干流下游养分较多,微生物消耗大量溶解氧,造成底泥的厌氧环境,有利于碳的转化,促进了产甲烷菌的生长。而pmoA的丰度在支流下游(采样点DJL、SWC、CXA)、支流上游中的汇合处(采样点SHC)、干流下游(采样点HSP、LXH)较高,这可能与溶解氧和甲烷的存容相关。分析原因,由于干流下游采样点TC和有机碳含量较高,促进了产甲烷菌和甲烷氧化菌的生长,从而促进了底泥中碳的转化。
碳循环是自然界基本的物质循环之一,碳转化功能类群微生物对所处生境的变化十分敏感,群落结构会迅速发生变化来适应各种有机或无机污染物的变化[14]。淡水沉积物每年对甲烷排放的贡献率占40%~50%[15]。表层沉积物中存在着活跃的甲烷氧化和甲烷生成循环,而微生物对有机物矿化作用主要发生在深层淡水沉积物的厌氧环境中[16]。厌氧环境中,古菌中的产甲烷古菌对甲烷产生起着重要作用;而好氧环境下,产甲烷细菌的作用相对更多一些,并且不同水深处的功能细菌也有较大差别[17]。DEUTZMANN等[18]在研究贫营养湖康士坦茨湖底泥时发现,底泥中存在着显著的甲烷氧化活动,甲烷氧化活动和NC10属的甲烷氧化菌有关, 底泥中pmoA基因丰度较高。KOJIMA等[19]在研究中等营养的日本琵琶湖时得出相似结论,NC10属的Methylomirabilisoxyfera菌是优势菌种,湖底表层沉积物为其提供一个稳定环境,是该菌种生长的良好栖息地。

图1 底泥样品中碳转化功能基因(mcrA、pmoA)的丰度Fig.1 The abundance of functional genes involved in carbon transformation (mcrA and pmoA) in the sediments
2.2 氮转化功能基因定量分析
底泥样品中固氮基因nifH和硝化基因AOA、AOB的丰度见图2。由图2可见,奉化江底泥样品中nifH的丰度为4.39×103~1.41×104拷贝数/ng,支流上游(采样点XKN、SD、XK-Ⅱ、SHC、TT)中nifH的丰度要显著高于奉化江干流下游(采样点CHL、SHQ、HSP)。奉化江底泥中AOA和AOB的丰度差异不显著,其中采样点SD、FJA、DJL、SWC、DTZ的AOB丰度显著高于AOA,说明以上点位属于富营养区域,其他采样点的AOA丰度均大于AOB。
淡水生态系统的反硝化作用对全球氮损失的贡献率约为19%[20]。淡水中的微生物参与到氮循环的各个机制有氮的固定、氨化、氨氧化、硝化和反硝化作用[21]。由于淡水生态系统的氮循环中同时存在硝化作用和反硝化作用,二者互相影响,对于保持水圈、生物圈和大气圈氮的动态平衡均起着相当重要的作用。SANTORO等[22]对地下河口沉积物中AOB和AOA基因丰度的研究中发现,AOA在低氧、微盐环境中是AOB的10倍,但在好氧环境中AOB却是AOA的约30倍。
底泥样品中反硝化基因(nirK,nirS,nosZ)的丰度见图3。由图3可见,奉化江干流下游采样点的反硝化基因(nirS,nosZ)丰度总体高于支流,尤其是HSP、LXH两个采样点的nirS基因丰度分别高达9.56×103、7.69×103拷贝数/ng,Nir属于亚硝酸盐还原酶,能催化亚硝酸盐转化为氧化氮,这是反硝化过程中最重要的限速步骤,也是反硝化作用有别于其他硝酸盐代谢的标志性反应,而Nir是催化此反应的限速酶。HSP、LXH两个采样点有机质含量较高,可能促进了反硝化作用导致nirS基因丰度较高。一氧化二氮还原酶(Nos)用于催化一氧化二氮至氮气的转化过程,普遍认为其活性受到溶解氧的限制。由于该酶催化反硝化的最后一步,终产物为氮气,因此nosZ基因常常被作为检测可进行完全反硝化作用的微生物。在底泥样品中发现下游的nosZ基因总体高于上游清洁点,说明下游的反硝化作用要强于上游,这与河流底泥中的溶解氧、养分有关。然而,支流上游SHC采样点nosZ基因丰度达1.83×103拷贝数/ng,这是因为SHC采样点的有机碳含量较高,微生物降解有机质消耗水中的溶解氧,造成底泥的厌氧环境促进了反硝化菌的生长和反硝化过程。nirK基因与nirS基因趋势相反,在上游清洁点的含量较高,如FJA、TT等采样点,这可能与沉积物理化性质不同有关。GASPAR等[23]在研究切萨皮克湾沉积物中与氮循环相关的功能基因丰度特征时发现,盐度、无机氮和溶解有机碳对amoA、nifH、nirK、nirS的分布均有较大影响。
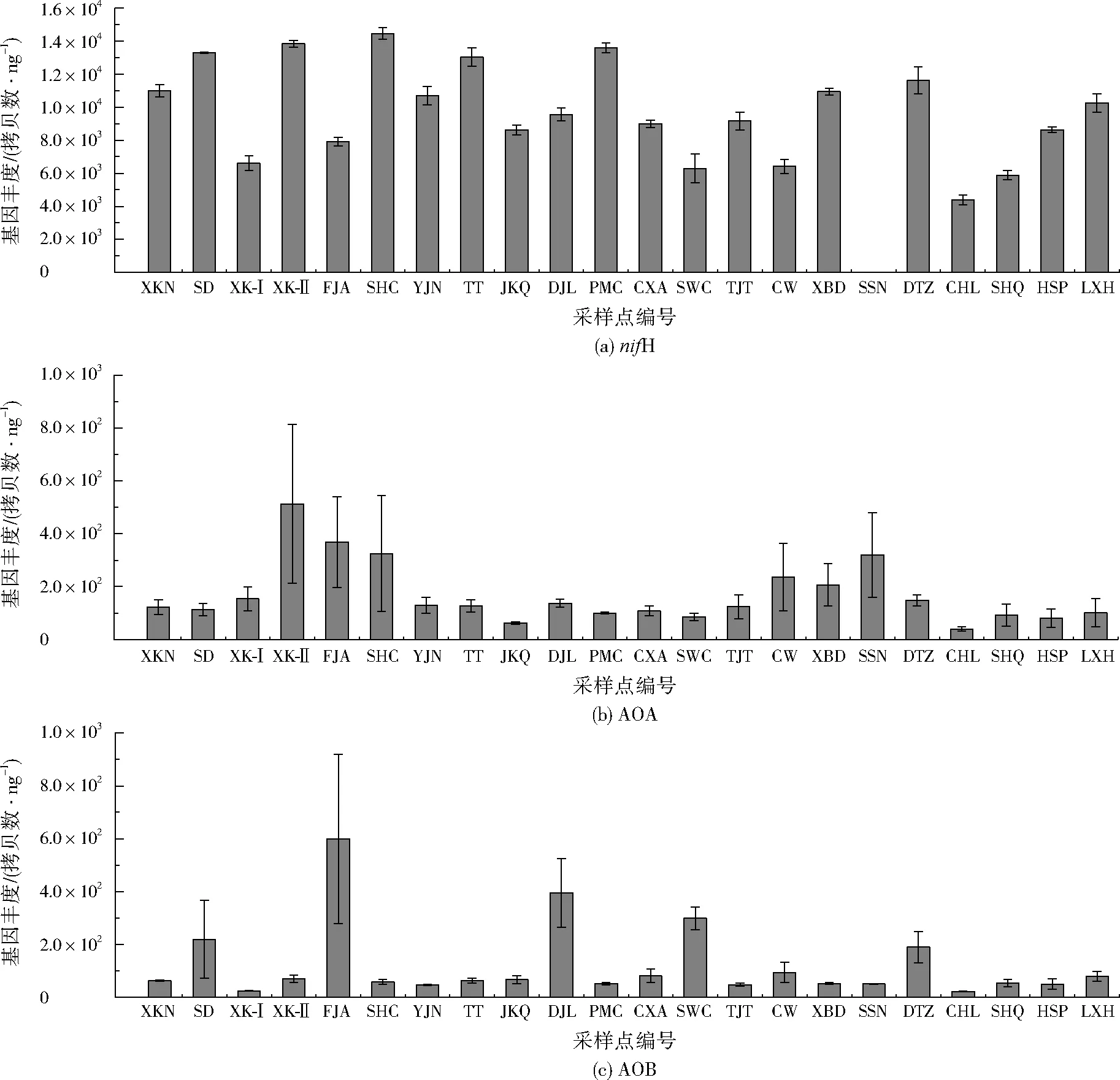
注:由于样品采集的原因,部分数据缺失。图3同。图2 底泥样品中固氮基因(nifH)和硝化基因(AOA、AOB)的丰度Fig.2 The abundance of functional genes involved in nitrogen fixation (nifH) and nitrification (AOA and AOB) in the sediments
2.3 硫转化功能基因定量分析
奉化江河流底泥中存在着较丰富的硫化物,因此底泥中存在一定数量的硫转化功能菌,包括硫氧化菌(soxB基因)和硫酸盐还原菌(dsrB基因)。奉化江底泥样品中硫转化功能基因(dsrB,soxB)的丰度见图4。由图4可见,CXA采样点的dsrB丰度最大,达到1.16×103拷贝数/ng,DJL采样点的soxB丰度最大,达到2.45×103拷贝数/ng,两个采样点均位于居民区,底泥中硫酸盐含量显著高于其他采样点,因此促进了硫的地球化学转化。
硫元素在淡水中的复杂转化是河流湖泊黑臭的重要原因之一,硫元素转化的同时也会对碳、氮、磷等元素的循环产生一定影响。淡水沉积物中硫的氧化还原与硫酸盐还原菌和硫氧化菌关系密切,但目前对此方面的研究相对较少[24]。王明义等[25]用稀释培养测数法(MPN)测定阿哈湖沉积物中硫酸盐还原菌,并对比湖底沉积物中和海洋沉积物中硫酸盐还原菌含量,发现淡水湖泊沉积物中硫酸盐还原菌有102~103个/g,低于海洋沉积物几个数量级。同时,硫酸盐还原菌也呈现季节和空间分布差异,秋季含量高于春季;一定深度范围内,随着深度增加,硫酸盐还原菌含量增加。SASS等[26]和LI等[27]也得到相似结论。
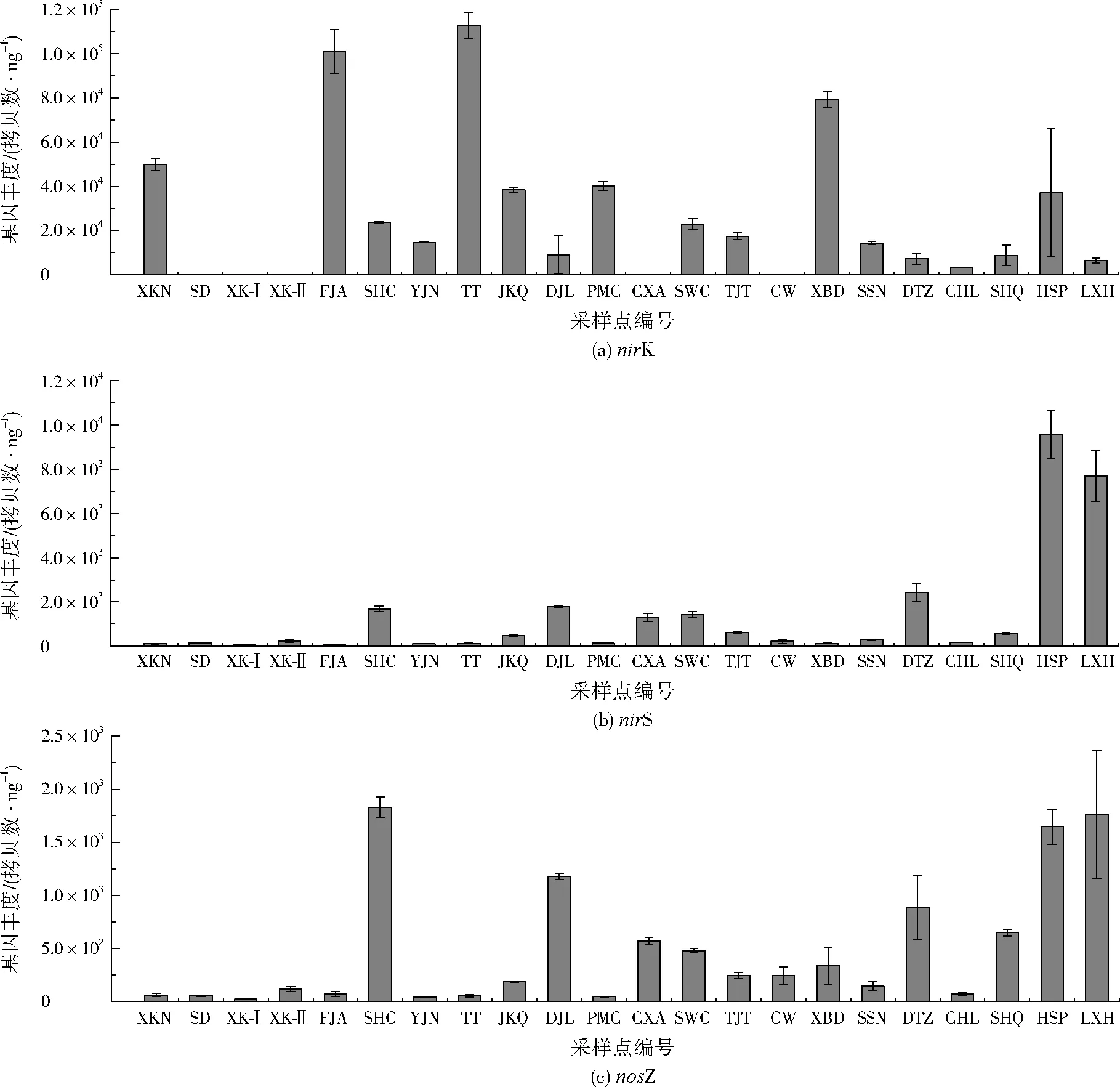
图3 底泥样品反硝化基因(nirK、nirS、nosZ)的丰度Fig.3 The abundance of functional genes involved in denitrification (nirK,nirS and nosZ) in the sediments
2.4 微生物基因丰度与环境因子的相关性分析
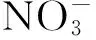
3 结 论
(1) 干流下游采样点TC和TOC含量较高,促进了产甲烷菌和甲烷氧化菌的生长,从而促进了底泥中碳的转化,奉化江干流下游的碳转化基因mcrA丰度显著高于上游清洁点,受溶解氧和甲烷存容影响,pmoA的丰度在支流下游、支流上游汇合处、干流下游较高。
(2) 采样点SD、FJA、DJL、SWC、DTZ的AOB丰度显著高于AOA,说明以上点位属于富营养区域,其他采样点的AOA丰度均大于AOB。
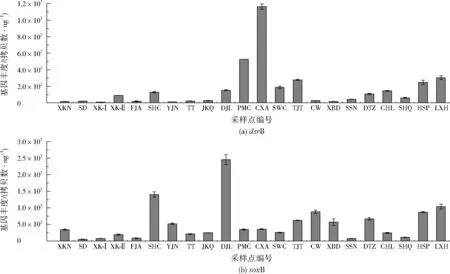
图4 底泥样品硫转化功能基因(soxB、dsrB)的丰度Fig.4 The abundance of functional genes involved in sulfur transformation (soxB and dsrB) in the sediments
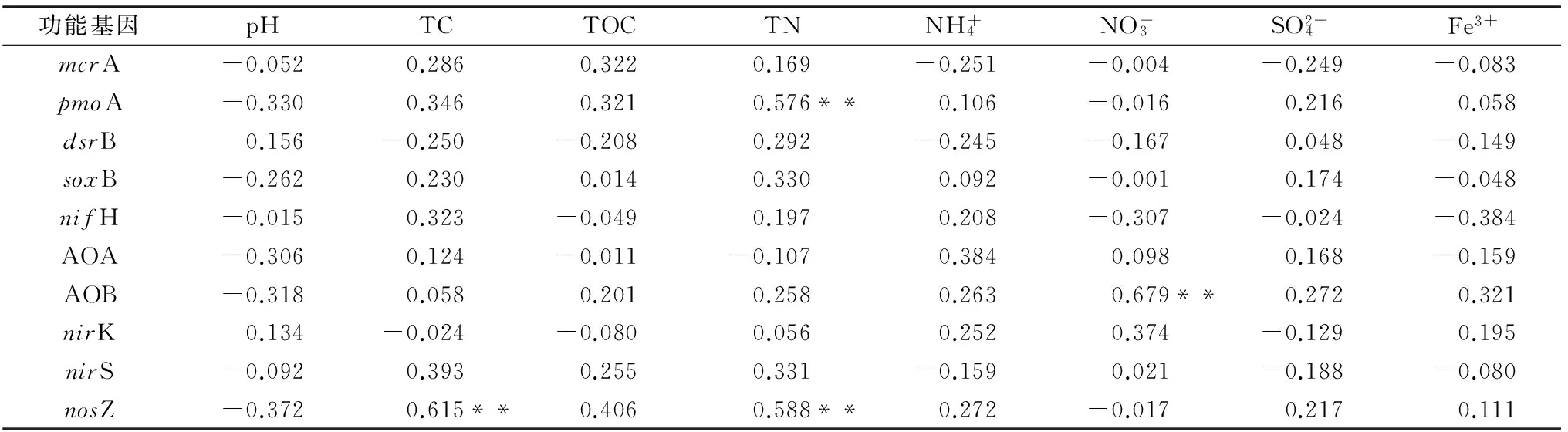
功能基因pHTCTOCTNNH+4NO-3SO2-4Fe3+mcrA-0.0520.2860.3220.169-0.251-0.004-0.249-0.083pmoA-0.3300.3460.3210.576**0.106-0.0160.2160.058dsrB0.156-0.250-0.2080.292-0.245-0.1670.048-0.149soxB-0.2620.2300.0140.3300.092-0.0010.174-0.048nifH-0.0150.323-0.0490.1970.208-0.307-0.024-0.384AOA-0.3060.124-0.011-0.1070.3840.0980.168-0.159AOB-0.3180.0580.2010.2580.2630.679**0.2720.321nirK0.134-0.024-0.0800.0560.2520.374-0.1290.195nirS-0.0920.3930.2550.331-0.1590.021-0.188-0.080nosZ-0.3720.615**0.4060.588**0.272-0.0170.2170.111
注:1)**表示差异达到1%极显著水平。
(3) 奉化江底泥样品中,干流下游的nosZ基因显著高于上游清洁点,说明下游的反硝化作用要强于上游。
(4) 居民区附近采样点河流底泥中硫转化功能基因丰度相对较大,其中硫还原基因dsrB丰度最高达1.16×103拷贝数/ng,硫氧化基因soxB丰度最高达2.45×103拷贝数/ng。
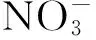
[1] 白洁,李岿然,李正炎,等.渤海春季浮游细菌分布与生态环境因子的关系[J].青岛海洋大学学报(自然科学版),2003,33(6):841-846.
[2] SMITH J E,PERDEK J M.Assessment and management of watershed microbial contaminants[J].Critical Reviews in Environmental Science and Technology,2004,34(2):109-139.
[3] 鲍士旦.土壤农化分析[M].3版.北京:中国农业出版社,2000.
[4] LUTON P E,WAYNE J M,SHARP R J,et al.ThemcrA gene as an alternative to 16S rRNA in the phylogenetic analysis of methanogen populations in landfill[J].Microbiology,2002,148(11):3521-3530.
[5] LIN Juling,JOYE S B,SCHOLTEN J M,et al.Analysis of methane monooxygenase genes in mono lake suggests that increased methane oxidation activity may correlate with a change in methanotroph community structure[J].Applied & Environmental Microbiology,2005,71(10):6458-6462.
[6] POLY F,JOCTEUR M L,BALLY R.Improvement in RFLP procedure to study the community of nitrogen fixers in soil through the diversity ofnifH gene[J].Research in Microbiology,2001,152(1):95-103.
[7] HENRY S,BAUDOIN E,LOPEZ GUTIERREZ J C,et al.Quantification of denitrifying bacteria in soils bynirK gene targeted real-time PCR[J].Journal of Microbiological Methods,2004,59(3):327-335.
[8] THROBACK I N,ENWALL K,JARVIS A,et al.Reassessing PCR primers targetingnirS,nirK andnosZ genes for community surveys of denitrifying bacteria with DGGE[J].FEMS Microbiology Ecology,2004,49(3):401-417.
[9] FRANCIS C A,O’MULLAN G D,WARD B B.Diversity of ammonia monooxygenase(amoA)genes across environmental gradients in Chesapeake Bay sediments[J].Geobiology,2003,1(2):129-140.
[10] ROTTHAUWE J H,WITZEL K P,LIESACK W.The ammonia monooxygenase structural geneamoA as a functional marker:molecular fine-scale analysis of natural ammonia-oxidizing populations[J].Applied & Environmental Microbiology,1997,63(12):4704-4712.
[11] HENRY S,BRU D,STRES B,et al.Quantitative detection of thenosZ gene,encoding nitrous oxide reductase,and comparison of the abundances of 16S rRNA,narG,nirK,andnosZ genes in soils[J].Applied & Environmental Microbiology,2006,72(8):5181-5189.
[12] GEETS J,BORRERNANS B,DIELS L,et al.DsrB gene-based DGGE for community and diversity surveys of sulfate-reducing bacteria[J].Journal of Microbiological Methods,2006,66(2):194-205.
[13] YANG Jian,JIANG Hongchen,DONG Hailiang,et al.Abundance and diversity of sulfur-oxidizing bacteria along a salinity gradient in four Qinghai-Tibetan lakes,China[J].Geomicrobiology Journal,2013,30(9):851-860.
[14] WINTER C,HEIN T,KAVKA G,et al.Longitudinal changes in the bacterial community composition of the Danube River:a whole-river approach[J].Applied & Environmental Microbiology,2007,73(2):421-431.
[15] CHAUDHARY P P,BRABLCOVL,BURINKOVI,et al.Molecular diversity and tools for deciphering the methanogen community structure and diversity in freshwater sediments[J].Applied Microbiology and Biotechnology,2013,97(17):7553-7562.
[16] URBAN N R,BREZONIK P L,BAKER L A,et al.Sulfate reduction and diffusion in sediments of Little Rock Lake,Wisconsin[J].Limnology & Oceanography,1994,39(4):797-815.
[17] CONRAD R.The global methane cycle:recent advances in understanding the microbial processes involved[J].Environmental Microbiology Reports,2009,1(5):285-292.
[18] DEUTZMANN J S,SCHINK B.Anaerobic oxidation of methane in sediments of lake constance,an oligotrophic freshwater lake[J].Applied & Environmental Microbiology,2011,77(13):4429-4436.
[19] KOJIMA H,TSUTSUMI M,ISHIKAWA K,et al.Distribution of putative denitrifying methane oxidizing bacteria in sediment of a freshwater lake,Lake Biwa[J].Systematic and Applied Microbiology,2012,35(4):233-238.
[20] SEITZINGER S,HARRISON J A,BOHLKE J K,et al.Denitrification across landscapes and waterscapes:a synthesis[J].Ecological Applications,2006,16(6):2064-2090.
[21] 刘正辉.东江氨氮污染河段的微生物群落特征[D].广州:华南理工大学,2011.
[22] SANTORO A E,FRANCIS C A,DE SIEYES N R,et al.Shifts in the relative abundance of ammonia-oxidizing bacteria and archaea across physicochemical gradients in a subterranean estuary[J].Environmental Microbiology,2008,10(4):1068-1079.
[23] GASPAR T O,GRINER E M,FRANCIS C A,et al.Oligonucleotide microarray for the study of functional gene diversity in the nitrogen cycle in the environment[J].Applied & Environmental Microbiology,2003,69(2):1159-1171.
[24] HOLMER M,STORKHOLM P.Sulphate reduction and sulphur cycling in lake sediments:a review[J].Freshwater Biology,2001,46(4):431-451.
[25] 王明义,张伟,梁小兵,等.阿哈湖和洱海沉积物硫酸盐还原菌研究[J].水资源保护,2007,23(3):9-10.
[26] SASS H,CYPIONKA H,BABENZIEN H D.Vertical distribution of sulfate-reducing bacteria at the oxic-anoxic interface in sediments of the oligotrophic Lake Stechlin[J].FEMS Microbiology Ecology,1997,22(3):245-255.
[27] LI Jianhua,PURDY K J,TAKII S,et al.Seasonal changes in ribosomal RNA of sulfate-reducing bacteria and sulfate reducing activity in a freshwater lake sediment[J].FEMS Microbiology Ecology,1999,28(1):31-39.
