慢性乙型肝炎和肝硬化“异病同证”的全基因组甲基化分析*
2016-03-06呼雪庆陈启龙陆奕宇苏式兵
呼雪庆,陈 健,陈启龙,陆奕宇,苏式兵
(上海中医药大学中医复杂系统研究中心 上海 201203)
慢性乙型肝炎和肝硬化“异病同证”的全基因组甲基化分析*
呼雪庆,陈 健,陈启龙,陆奕宇,苏式兵**
(上海中医药大学中医复杂系统研究中心 上海 201203)
目的:本文从全基因组DNA甲基化的角度探讨慢性乙型肝炎(慢乙肝)和慢乙肝后肝硬化(肝硬化)湿热内蕴证、肝郁脾虚证和肝肾阴虚证异病同证的生物学基础。方法:采取慢乙肝及肝硬化湿热内蕴证、肝郁脾虚证和肝肾阴虚证患者和健康志愿者外周血样本,提取DNA进行HumanMethylation450K芯片检测及分析。结果:慢乙肝和肝硬化异病同证湿热内蕴证特异性差异甲基化位点有9个,覆盖9个基因;肝郁脾虚证有30个,覆盖20个基因;肝肾阴虚证有22个,覆盖14个基因。其中,与正常组相比,湿热内蕴证中KCTD2和NAV1,肝郁脾虚证中LGR6和SH2D4B及肝肾阴虚证中CYP2E1、PCSK6、DEXI、HIST1H3B和SULT1C2的差异位点甲基化程度变化较大(|Delta Beta|>0.15)。结论:慢乙肝与肝硬化基因甲基化可能与其湿热内蕴证、肝郁脾虚证和肝肾阴虚证的形成有关。
DNA甲基化 慢性乙型肝炎 肝硬化 证候 异病同证
中医证候是中医学理论体系指导下,一定阶段的临床症状与体征所反应的病理实质的概括。中医证候具有相兼性、动态性和复杂性,且缺乏微观层次的认识和辨证方法,为证候的客观化,即证候形成和演变机制及证候诊断标准带来了挑战。不同疾病,其发病及转归机制可能存在相同或相似点,即异病同证。不同疾病的患者可能有相似的物质群变化,可采取异病同治。异病同证的深入研究为现代个体化诊疗和精准医学发展提供依据,为疾病发病机制探索和治疗提供参考[1]。
全球约有2.4亿慢性乙型肝炎(慢乙肝)患者,其发病机制主要涉及机体免疫和炎症反应[2]。慢乙肝的形成因素包括HBV基因多型性、机体免疫相关基因多态性以及宿主免疫细胞的功能失调。慢乙肝后肝硬化(肝硬化)由慢乙肝发展形成,长期持续的炎症反应激活肝星状细胞(Hepatic Stellate Cell,HSCs),肝内结缔组织异常增生,肝内弥漫性细胞外基质(Extracellular Matrix,ECM)过度沉淀,最终导致肝硬化。宿主感染HBV后,通过上调甲基化转移酶(DNA Methyltransferase,DNMT)对病毒基因组进行甲基化修饰,抑制其复制,另一方面,DNMT的升高也使宿主某些基因高甲基化,而利于病毒复制,甚至激活HSC和促进肝硬化的发展[3]。HBV感染的急、慢性阶段及肝硬化中均存在差异甲基化基因[4-6]。本文从全基因组DNA甲基化的角度探讨慢乙肝及肝硬化异病同证(湿热内蕴证、肝郁脾虚证和肝肾阴虚证)的生物学基础,为证候的客观化及慢乙肝和肝硬化的个体化诊疗提供依据。
1 材料与方法
1.1 研究对象
实验组慢乙肝及肝硬化患者各15例,正常组健康志愿者6例均来自上海中医药大学附属曙光医院,包括慢乙肝和肝硬化的湿热内蕴证各5例、脾虚湿盛证各5例和肝肾阴虚证各5例。患者就诊时由主治医生辨证,并通过证候问卷表采集患者证候相关临床信息,交由3名正高职称的中医肝病专家确定证候类型,两次辨证一致的患者纳入本研究。采集患者和健康者空腹外周静脉血,置入抗凝管内后,及时分离血浆和血细胞,分别速冻于-80℃冰箱,血细胞用于提取DNA。
1.2 诊断标准、纳入标准和排除标准
慢乙肝的诊断标准参照2005年12月中华医学会肝病学分会、感染病学分会制订的《慢性乙型肝炎防治指南》[7],其证候诊断标准参照《病毒性肝炎中医辨证标准(试行)》[8]。肝炎后肝硬化诊断标准参照《病毒性肝炎防治方案》[9],其证候诊断标准参照《肝硬化临床诊断,中西辨证和疗效评定标准(试行方案)》[10]。纳入标准:①符合慢乙肝及肝硬化疾病诊断标准;②中医辨证为湿热内蕴证、脾虚湿盛证和肝肾阴虚证;③年龄18-65岁;④签署知情同意书。排除标准:①年龄<18岁或>65岁;②合并其它嗜肝病毒感染、脂肪肝、自身免疫性疾病等肝脏疾病;③慢性重型肝炎、肝硬化失代偿;④伴有心、肾、肺、内分泌血液、代谢及胃肠道严重原发病;⑤精神病患者、孕妇或哺乳期妇女。
1.3 DNA提取和全基因组甲基化检测
采用血液基因组DNA提取试剂盒TIANamp Blood DNA Kit(北京天根生化科技有限公司,货号:DP318)提取DNA。采用HumanMethylation450K芯片技术检测全基因组甲基化,委托上海欧易生物医学科技有限公司实施。该芯片检测485、577个甲基化位点,覆盖96%的已知CpG岛和99%的NCBI参考序列基因。
1.4 全基因组甲基化分析
将原始数据导入Genomestudio(美国Illunina公 司,版 本:genomestudio software 2011.1)进 行BMIQ标准化。CpG位点的甲基化程度由Beta值衡量,Illunina甲基化芯片针对每一个CpG位点设计两种探针,用双色荧光信号signalB和signalA来检测甲基化和非甲基化的等位基因。Beta值即为signal B强度与signal A,Signal B强度之和比值。Beta值的取值范围为0到1,0表示该位点无甲基化,1表示该位点完全甲基化。组间甲基化差异统计采用One-Way ANOVA计算方法。差异甲基化位点的判断标准为Diffscore值小于-13或大于13,且Beta差值大于0.1或小于-0.1。将差异甲基化位点所在的基因导入DAVID基因功能分析平台** https://david.ncifcrf.gov,进行Gene ontology(GO)功能和KEGG通路分析。
2 结果与分析
2.1 慢乙肝和肝硬化患者证候与健康者临床资料
慢乙肝和肝硬化湿热内蕴证、肝郁脾虚证、肝肾阴虚证及正常组7组的年龄、性别在统计学上无差异。在ALT水平上,慢乙肝,肝硬化和正常组三组间有统计学差异(P=0.035)。各证候在慢乙肝与肝硬化间无统计学差异。在AST水平上,慢乙肝,肝硬化和正常组三组间无统计学差异(P=0.079)。各证候在慢乙肝与肝硬化间无统计学差异。在ALb水平上,组别之间都无统计学差异(表1)。
表1 慢乙肝和肝硬化患者证候与正常组的临床资料(±s)
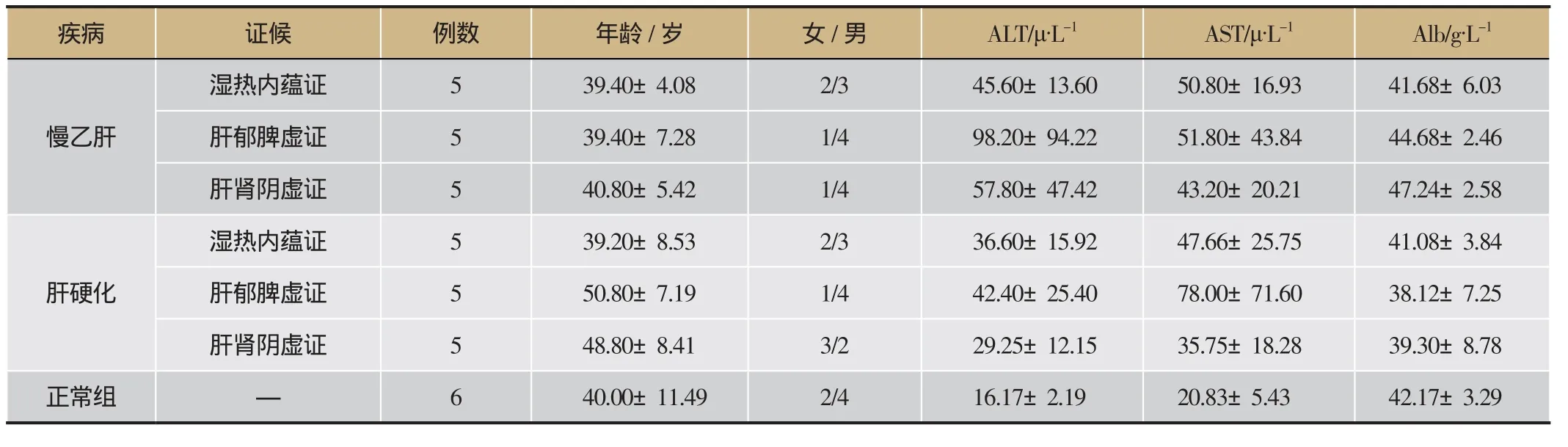
表1 慢乙肝和肝硬化患者证候与正常组的临床资料(±s)
正常组 — 6 40.00±11.49 2/4 16.17±2.19 20.83±5.43 42.17±3.29
2.2 慢乙肝和肝硬化异病同证证候特异性差异甲基化基因
慢乙肝和肝硬化湿热内蕴证特异性差异甲基化位点有9个,覆盖9个基因;肝郁脾虚证有30个,覆盖20个基因;肝肾阴虚证有22个,覆盖14个基因(图1)。慢乙肝和肝硬化与正常组相比,在同一证型中,差异甲基化位点的甲基化变化方向一致,即同为高甲基化或低甲基化。同一基因可能会出现多个差异甲基化位点,分布于基因的不同区域及CpG岛相关区域,且同一基因的不同甲基化位点的甲基化变化方向一致。如肝郁脾虚证中,慢乙肝和肝硬化在GREM2基因上存在4个差异甲基化位点,与正常组相比,均发生了去甲基化,其中有3个位点位于基因body区,1个位点位于3’UTR。2个位点位于CpG岛上,另外2个位点位于CpG岛的shore区域。又如,肝肾阴虚证中,慢乙肝和肝硬化在CYP2E1基因上存在8个差异甲基化位点,与正常组相比,均发生了高甲基化,8个差异甲基化位点都分布在基因Body区,其中,4个位点位于CpG岛上,另外4个位点位于CpG岛的shore区域。慢乙肝和肝硬化与正常组相比,|DeltaBeta|>0.15的差异甲基化基因信息如表2。
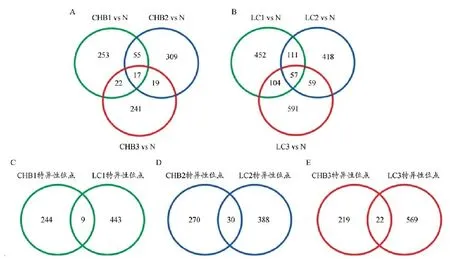
图1 慢乙肝和肝硬化湿热内蕴证、肝郁脾虚证和肝肾阴虚证差异甲基化位点VENN分析
慢乙肝和肝硬化湿热内蕴证、肝郁脾虚证和肝肾阴虚证差异高甲基化位点比例均低于低甲基化位点(湿热内蕴证:30%/70%;肝郁脾虚证:22%/78%;肝肾阴虚证:45%/55%)。湿热内蕴证和肝郁脾虚证的差异甲基化位点主要位于1号染色体,分别占11.11%和30.00%,而在肝肾阴虚证中,差异甲基化位点主要位于10号染色,占31.82%。各证型差异甲基化位点主要存在于基因Body区(分别占44.44%,43.33%和77.27%),TSS200(分 别 占22.22%,23.33%和4.55%)和TSS1500(分 别 占22.22%,3.33%和9.09%),3个证型基因区域构成比差异无统计学意义(图2)。慢乙肝和肝硬化与正常组相比,以|Delta Beta|>0.15为阀值,湿热内蕴证中,KCTD2在两疾病中甲基化程度均较高,NAV1甲基化程度均为最低。肝郁脾虚证中,LGR6基因4个位点及SH2D4B基因1个位点在两疾病中的甲基化程度均较高。肝肾阴虚证中,CYP2E1的7个位点和PCSK6的1个位点在两疾病中的甲基化程度均较高。DEXI、HIST1H3B和SULT1C2在两疾病中的甲基化程度均较低(表2)。
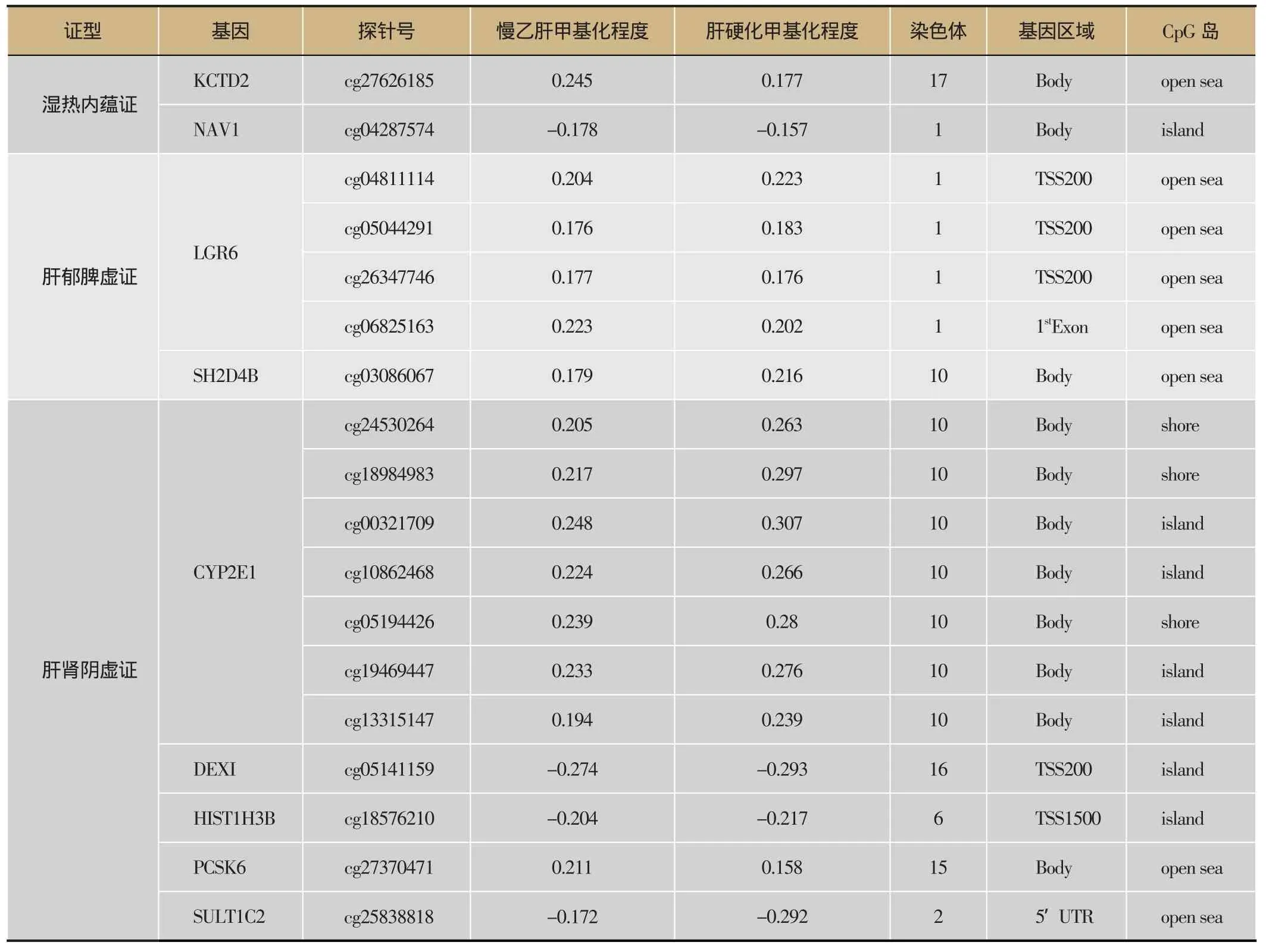
表2 慢乙肝和肝硬化异病同证主要差异甲基化基因的分布和甲基化程度

图2 慢乙肝和肝硬化异病同证全基因组差异甲基化位点区域分布
2.3 慢乙肝和肝硬化证候特异性差异甲基化基因GO功能和KEGG通路分析
慢乙肝和肝硬化不同证候的差异甲基化基因参与不同的GO功能及KEEG通路。如图3所示,慢乙肝和肝硬化的湿热内蕴证差异甲基化基因主要参与一些代谢过程,比如喹啉代谢,二羧酸代谢和类维生素A代谢等以及脂肪酸的氧化作用和降解过程等;肝郁脾虚证的差异甲基化基因主要参与免疫调节相关生物学过程,如免疫反应,刺激反应和信号调控,以及移植物抗宿病、甲状腺癌和哮喘等与免疫相关的疾病;肝肾阴虚证的差异甲基化基因参与免疫反应及一些代谢过程,如对刺激的细胞反应,信号调控,以及亚油酸代谢和类固醇激素的合成等过程。
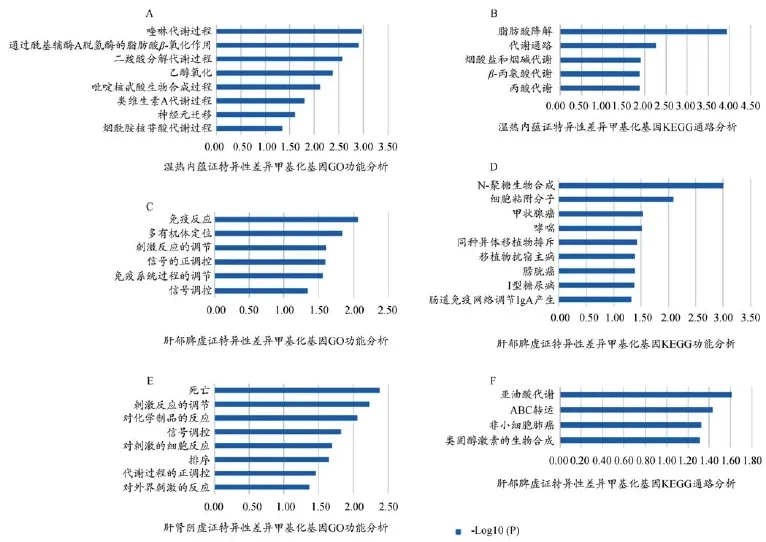
图3 慢乙肝和肝硬化证候特异性差异甲基化基因GO功能和KEGG通路分析
3 讨论
表观遗传学和中医学存在理论上的共性,如表观遗传修饰水平受环境、情绪和饮食的影响与中医整体观、七情致病理论存在共性。表观遗传DNA序列不变,表达水平可变与中医“同病异证”和“异病同证”存在相似之处[11,12]。DNA甲基化是表观遗传学最重要的修饰方式之一。导致DNA甲基化的不同途经可形成不同的证候特征[13,14]。
近年来,从DNA甲基化角度探索中医证候实质的研究有了初步进展,如急性骨髓性白血病患者中,与气阴两虚证相比,血瘀痰凝证和热毒蕴结证ID4基因甲基化程度较高[15]。我们之前的研究发现HLA-DRB1和ADAMTS9,NUDT1和YES1,APOA5和PRKCZ等基因的差异甲基化可能分别与乙肝后肝硬化湿热内蕴证,脾虚湿盛证和肝肾阴虚证证候的形成有关[16]。目前从DNA甲基化分子机制研究证候的生物学基础仍在初始阶段,且大多局限于单个或几个基因与证候间的关系,缺乏从全基因组甲基化层面阐释异病同证实质的研究。本研究从全基因组DNA甲基化的角度探讨慢乙肝及肝硬化异病同证的生物学基础。
本课题组研究发现,慢乙肝和肝硬化的同一证型存在共同的差异甲基化基因,湿热内蕴证特异性差异甲基化位点有9个,覆盖9个基因;肝郁脾虚证有30个,覆盖20个基因;肝肾阴虚证有22个,覆盖14个基因。同一基因上可能存在几个差异甲基化位点,且不同疾病在同一差异位点上的甲基化方向一致,如肝郁脾虚证中的GREM2上有4个差异甲基化位点,且在慢乙肝和肝硬化中都发生了去甲基化。LGR6上4个差异甲基化位点在两疾病中都呈高甲基化。肝肾阴虚证中CYP2E1上8个差异甲基化位点均呈高甲基化。差异甲基化位点分布于基因不同区域(主要集中于基因Body区域)及不同染色体上。其中,湿热内蕴证和肝郁脾虚证的差异甲基化位点主要分布于1号染色体,肝肾阴虚证主要分布于10号染色体。进一步分析发现慢乙肝和肝硬化的湿热内蕴证特异性差异甲基化基因主要参与某些代谢过程;肝脾脾虚证主要参与免疫相关过程;而肝肾阴虚的差异甲基化基因同时参与代谢过程及免疫反应。研究提示不同疾病相同证候可能存在共同的差异甲基化基因和差异甲基化位点,并具有类似的生物学过程。
湿热内蕴证证候特异性甲基化基因中,KCTD2在慢乙肝和肝硬化中甲基化程度均较高,KCTD2参与cAMP依赖性蛋白激酶(PKA)及神经性疼痛信号通路。研究表明,KCTD2的基因多态性与阿尔茨海默病的发生有关[17]。NAV1在两个疾病中均发生了去甲基化,NAV1参与神经系统发育和再生及神经元迁移[18]。目前尚未查到KCTD2和NAV1与中医证候或其甲基化研究的相关报道。肝郁脾虚证证候特异性甲基化基因中,慢乙肝和肝硬化在LGR6的4个位点和SH2D4B的1个位点上呈现高甲基化。LGR6抑制Wnt信号通路的激活,LGR6是指甲干细胞,表皮干细胞的标记[19,20],也有报道称LGR6是一个潜在的抑癌基因[21]。SH2D4B与淋巴细胞性血管炎有关,也是T细胞特异性的适应蛋白之一,参与细胞内信号传导[22]。肝肾阴虚证证候特异性甲基化基因中,慢乙肝和肝硬化在CYP2E1的7个位点及PCSK6呈高甲基化,DEXI、HIST1H3B和SULT1C2均发生了去甲基化。CYP2E1参与胆固醇、类固醇等脂质的合成,其基因多态性可能与的鼻咽癌、肺癌等疾病的发生有关[23,24]。研究发现,胎儿肝中,CYP2E1基因的不表达可能与其5’UTR的甲基化有关[25],人体肺、肾及足月胎盘中,CYP2E1基因第一个外显子及第一个内含子的甲基化影响其表达水平[26],人胚胎干细胞源性肝细胞中,CYP2E1的低表达与其第一个内含子的CpG位点的高甲基化有关[27]。另有研究指出,CYP2E1基因启动子区的甲基化程度与甲苯暴露及吸烟有关[28]。PCSK6的高表达与风湿性关节炎、鳞状上皮细胞癌和骨关节炎疼痛等有关[29-31]。DEXI基因功能不明确,研究称CLEC16A基因的多态性通过影响邻近基因DEXI的表达,介导1型糖尿病及多发性硬化的发生[32,33]。结直肠癌及胃癌中,DEXI呈高甲基化状态,且可能参与对化疗药喜树碱的耐药过程[34]。HIST1H3B编码组蛋白,弥漫性的内源性脑干胶质瘤中常出现HIST1H3B基因突变[35]。SULT1C2编码的蛋白属磺基转移酶,磺基转移酶催化激素、神经传递素,外源性物质等的硫酸盐共轭。胆固醇生物合成中间体会影响大鼠原代肝细胞中SULT1C2的表达[36]。
上述基因甲基化与参与证候形成的具体机制仍需进一步探索。目前关于甲基化的研究,主要集中于其参与调控基因的转录水平,从而影响疾病的发生发展。一般认为,基因启动子区CpG位点的甲基化通常抑制转录,而去甲基化通常促进转录。近年来,关于基因其它区域的CpG位点的甲基化所与转录关系的研究增多,研究称基因Body区域的甲基化能促进转录[37],也有研究表明基因Body区域甲基化程度极高或极低能够抑制转录,而中等水平的甲基化程度则促进转录[38-41]。基因第一个外显子上的甲基化与转录抑制有关[42]。因此,基因甲基化也可能通过调控其转录水平,参与证候的形成。
综上,湿热内蕴证中KCTD2和NAV1,肝郁脾虚证中LGR6和SH2D4B及肝肾阴虚证中CYP2E1、PCSK6、DEXI、HIST1H3B和SULT1C2的甲基化程度的改变可能与其相应证候的形成有关,其机制之一可能是通过影响基因转录,从而影响其生物学过程,参与证候形成,具体机制还需进一步的研究。该研究因样本量较少,存在一定的局限性,基因的甲基化程度需后续大样本的验证。
参考文献
1 陈健,陈启龙,苏式兵.中医药精准医疗的思考与探索.世界科学技术-中医药现代化,2016,18(4):557-562.
2 Ott J J, Stevens G A, Groeger J, et al. Global epidemiology of hepatitis B virus infection: new estimates of age-specific HBsAg seroprevalence and endemicity. Vaccine, 2012, 30(12): 2212-2219.
3 凌青霞,陈明泉,施光峰. DNA甲基化修饰在乙肝病毒持续感染相关肝病中的研究现状.肝脏,2014,19(5):362-364.
4 张旻.慢性乙型肝炎患者PD-1启动子区域甲基化状态的研究.长沙:中南大学博士学位论文,2012.
5 李凤彩.慢加急性重型乙型病毒性肝炎患者血清IFN-γ水平及其启动子甲基化状态研究.济南:山东大学硕士学位论文,2010.
6 Zhao Y, Xue F, Sun J, et al. Genome-wide methylation profiling of the different stages of hepatitis B virus-related hepatocellular carcinoma development in plasma cell-free DNA reveals potential biomarkers for early detection and high-risk monitoring of hepatocellular carcinoma. Clin Epigenetics, 2014, 6(1): 30.
7 中华医学会肝病学分会、感染病学分会.慢性乙型肝炎防治指南.中华肝脏病杂志,2005,13(12):881-891.
8 中国中医药学会内科肝病志业委员会.病毒性肝炎中医辨证标准(试行).中医杂志,1992(5):39-40.
9 中华医学会传染病与寄生虫病学分会、肝病学分会.病毒性肝炎防治方案.中华肝脏病杂志,2000,8(6):324-329.
10 张育轩,危北海.肝硬化临床诊断、中医辨证和疗效评定标准(试行方案).新消化病杂志,1994,2(2):126.
11 成细华,饶春梅,喻嵘,等.表观遗传学在中医药研究中的应用.中国中医药信息杂志,2016,23(1):134-136.
12 苏式兵,许锦文.生命科学前沿技术与中医药研究.上海浦江教育出版社,2013:145-177.
13 牟新.中医证候实质与表观遗传学相关性初探.浙江中西医结合杂志,2013,23(6):443-446.
14 玄丽娜.不同中医证型骨髓增生异常综合征RIZ1、ID4基因甲基化异常表达及临床意义.山东中医药大学硕士学位论文,2012.
15 刘菲,徐瑞荣.急性髓系白血病中医证型与ID4基因启动子区甲基化相关性研究.中国中西医结合杂志,2012,32(4):471-473.
16 呼雪庆,苏式兵.基于全基因组甲基化的乙肝后肝硬化典型证候生物学基础观察.中华中医药杂志,2016,31(5):1868-1873.
17 Boada M, Antunez C, Ramirez-Lorca R, et al. ATP5H/KCTD2 locus is associated with Alzheimer's disease risk. Mol Psychiatry, 2014, 19(6): 682-687.
18 Martinez-Lopez M J, Alcantara S, Mascaro C, et al. Mouse neuron navigator 1, a novel microtubule-associated protein involved in neuronal migration. Mol Cell Neurosci, 2005, 28(4): 599-612.
19 Lehoczky J A, Tabin C J. Lgr6 marks nail stem cells and is required for digit tip regeneration. Proc Natl Acad Sci U S A, 2015, 112(43): 13249-13254.
20 Snippert H J, Haegebarth A, Kasper M, et al. Lgr6 marks stem cells in the hair follicle that generate all cell lineages of the skin. Science, 2010, 327(5971): 1385-1389.
21 Gong X, Carmon K S, Lin Q, et al. LGR6 is a high affinity receptor of R-spondins and potentially functions as a tumor suppressor. PLoS One, 2012, 7(5): e37137.
22 Lapinski P E, Oliver J A, Bodie J N, et al. The T-cell-specific adapter protein family: TSAd, ALX, and SH2D4A/SH2D4BJ. Immunol Rev, 2009, 232(1):240-254.
23 Ben C A, Abaza H, Douik H, et al. Genetic polymorphism of cytochrome P450 2E1 and the risk of nasopharyngeal carcinoma. Bull Cancer, 2015, 102(12): 967-972.
24 Shen Z T, Wu X H, Li B, et al. CYP2E1 Rsa Iota/Pst Iota polymorphism and lung cancer susceptibility: A meta-analysis involving 10,947 subjects. J Cell Mol Med, 2015, 19(9): 2136-2142.
25 Jones S M, Boobis A R, Moore G E, et al. Expression of CYP2E1 during human fetal development: methylation of the CYP2E1 gene in human fetal and adult liver samples. Biochem Pharmacol, 1992, 43(8):1876-1879.
26 Vieira I, Pasanen M, Raunio H, et al. Expression of CYP2E1 in human lung and kidney during development and in full-term placenta: A differential methylation of the gene is involved in the regulation process. Pharmacol Toxicol, 1998, 83(5):183-187.
27 Park H J, Choi Y J, Kim J W, et al. Differences in the epigenetic regulation of cytochrome P450 genes between human embryonic stem cell-derived hepatocytes and primary hepatocytes. PLoS One, 2015, 10(7): e132992.
28 Jimenez-Garza O, Baccarelli A A, Byun H M, et al. CYP2E1 epigenetic regulation in chronic, low-level toluene exposure: Relationship with oxidative stress and smoking habit. Toxicol Appl Pharmacol, 2015, 286(3): 207-215.
29 Wang F, Wang L, Jiang H, et al. Inhibition of PCSK6 may play a protective role in the development of rheumatoid arthritis. J Rheumatol, 2015, 42(2):161-169.
30 Bassi D E, Cenna J, Zhang J, et al. Enhanced aggressiveness of benzopyrene-induced squamous carcinomas in transgenic mice overexpressing the proprotein convertase PACE4 (PCSK6). Mol Carcinog, 2015, 54(10): 1122-1131.
31 Malfait A M, Seymour A B, Gao F, et al. A role for PACE4 in osteoarthritis pain: Evidence from human genetic association and null mutant phenotype. Ann Rheum Dis, 2012, 71(6):1042-1048.
32 Tomlinson M T, Pitsillides A, Pickin R, et al. Fine mapping and functional studies of risk variants for type 1 diabetes at chromosome 16p13.13. Diabetes, 2014, 63(12): 4360-4368.
33 Leikfoss I S, Mero I L, Dahle M K, et al. Multiple sclerosis-associated single-nucleotide polymorphisms in CLEC16A correlate with reduced SOCS1 and DEXI expression in the thymus. Genes Immun, 2013, 14(1): 62-66.
34 Miyaki Y, Suzuki K, Koizumi K, et al. Identification of a potentepigenetic biomarker for resistance to camptothecin and poor outcome to irinotecan-based chemotherapy in colon cancer. Int J Oncol, 2012, 40(1):217-226.
35 Reyes-Botero G, Giry M, Mokhtari K, et al. Molecular analysis of diffuse intrinsic brainstem gliomas in adults. J Neurooncol, 2014, 116(2):405-411.
36 Rondini E A, Pant A, Kocarek T A. Transcriptional regulation of cytosolic sulfotransferase 1C2 by intermediates of the cholesterol biosynthetic pathway in primary cultured rat hepatocytes. J Pharmacol Exp Ther, 2015, 355(3):429-441.
37 Yang X, Han H, De Carvalho D D, et al. Gene body methylation can alter gene expression and is a therapeutic target in cancer. Cancer Cell, 2014, 26(4):577-590.
38 Zhang X, Yazaki J, Sundaresan A, et al. Genome-wide high-resolution mapping and functional analysis of DNA methylation in arabidopsis. Cell, 2006, 126(6):1189-1201.
39 Zilberman D, Gehring M, Tran R K, et al. Genome-wide analysis of Arabidopsis thaliana DNA methylation uncovers an interdependence between methylation and transcription. Nat Genet, 2007, 39(1):61-69.
40 Li X, Zhu J, Hu F, et al. Single-base resolution maps of cultivated and wild rice methylomes and regulatory roles of DNA methylation in plant gene expression. BMC Genomics, 2012, 13: 300.
41 Zemach A, McDaniel I E, Silva P, et al. Genome-wide evolutionary analysis of eukaryotic DNA methylation. Science, 2010, 328(5980): 916-919.
42 Brenet F, Moh M, Funk P, et al. DNA methylation of the first exon is tightly linked to transcriptional silencing. PLoS One, 2011, 6(1): e14524.
Genome-Wide Methylation Analysis in the Same Syndrome Between the Chronic Hepatitis B and Hepatitis B-Related Cirrhosis
Hu Xueqing, Chen Jian, Lu Yiyu, Su Shibing
(Research Center for Traditional Chinese Medicine Complexity System, Shanghai University of Traditional Chinese Medicine, Shanghai 201203, China)
This study aimed to explore the biological basis of the same syndrome of Traditional Chinese Medicine (TCM) in different diseases, involving dampness and heat excess (DH) syndrome, liver stagnation and spleen deficiency (LSSD) syndrome and liver- and kidney-yin deficiency (LKYD) syndrome in chronic hepatitis B (CHB) and hepatitis B-related cirrhosis (LC). The CHB and LC patients with DHS, LSSDS and LKYDS (10 cases apiece) and healthy volunteers (6 cases) were enrolled. Blood samples were prepared and DNAs were extracted for HumanMethylaiton450K detection and analyses. As a result, 9 loci (covering 9 genes) were specifically and differently methylated in DH syndrome in both the two diseases, while 30 loci (covering 20 genes) in LSSD syndrome and 22 loci (covering 14 genes) in LKYD syndrome. Among the genes, KCTD2 and NAV1 in DH syndrome, LGR6 and SH2D4B in LSSD syndrome, and CYP2E1, PCSK6, DEXI, HIST1H3B and SULT1C2 in LKYD syndrome were significantly differently methylated compared with the healthy group (|Delta Beta| > 0.15). In conclusion, methylation level alternation may involve in the formation of DH, LSSD and LKYD syndromes in CHB and LC.
DNA methylation, chronic hepatitis B, cirrhosis, TCM syndrome, the same TCM syndrome in different diseases?
10.11842/wst.2016.09.004
R2-031
A
(责任编辑:朱黎婷,责任译审:朱黎婷)
2016-07-12
修回日期:2016-08-25
* 国家自然科学基金委重点项目(81330084):基于系统生物学的原发性肝癌和大肠癌“异病同证”和“异病同治”的研究,负责人:苏式兵。
** 通讯作者:苏式兵,本刊编委,教授,博士生导师,主要研究方向:中医药系统生物学。
