鱼类环境DNA研究中通用引物的筛选验证
2016-02-26赵良杰凡迎春张新磊刘其根
刘 军,赵良杰,凡迎春,张新磊,刘其根
(上海海洋大学农业部淡水水产种质资源重点实验室,上海 201306)
鱼类环境DNA研究中通用引物的筛选验证
刘军,赵良杰,凡迎春,张新磊,刘其根
(上海海洋大学农业部淡水水产种质资源重点实验室,上海201306)
摘要:为了筛选一个通用性和适用性良好的能够运用于环境DNA(eDNA)研究的鱼类引物,从相关文献中选取了鱼类线粒体基因组部分片段的5对引物,分别对线粒体D-loop区、16S rRNA基因、COI基因以及Cytb基因部分片段进行扩增。对千岛湖48种鱼类基因组DNA进行扩增后比较发现,引物16s和COI均可以取得良好的扩增效果,通用性优于其他几对引物。引物16s的扩增产物经凝胶电泳检测均出现明亮的目的条带,引物COI则有3种鱼的条带经凝胶电泳检测亮度较暗。利用上述引物对环境样品eDNA扩增时发现,只有16s和COI的引物具有良好的扩增效果,能够得到明显单一的亮带。对该两种引物的PCR产物克隆后测序比对发现,16s的PCR产物均为千岛湖常见鱼类物种的基因片段,COI的PCR产物则为细菌COI基因的部分片段。综上,我们认为引物16s在通用性和适用性上都更为适合作为鱼类群落结构eDNA研究的通用引物。
关键词:引物筛选;线粒体标记;环境DNA;千岛湖
环境DNA(eDNA)是指从有机体脱落释放进入到自然环境中(如空气、水、土壤等)的DNA,包括细胞中的DNA和细胞破碎后游离出细胞外的DNA分子。eDNA技术就是直接从水、土壤、沉积物等环境样本中提取DNA片段后通过DNA测序和qPCR等分子生物学检测技术来定性或定量目标生物,从而确定目标生物在该环境中的分布及功能特征的研究方法[1-2]。目前eDNA在生态学方面的应用主要包括对物种多样性的研究[3]、动物食性分析[4]、入侵及稀有物种调查以及生物量和分布的估测[5-7]。eDNA在水生动物方面的应用在国外已见一些文献报道[8-9],特别是在鱼类稀有种和入侵种的监测以及生物量估测方面eDNA技术显示出了较传统方法更简单、快捷、灵敏和低成本的诸多优点。
但是,对于鱼类群落结构的eDNA研究还需要寻求到一个通用性和适用性都比较理想的通用引物。通过通用引物对于环境样品的有效扩增,利用克隆测序、二代测序以及凝胶电泳等方案,分析鱼类群落多样性的分子生物学信息。在以往关于鱼类通用引物的研究中,研究者往往只是考虑提供扩增单一鱼类DNA模板的通用性引物,因为模板DNA中没有其他生物来源的DNA组分,因此只需要考虑引物的通用性[10-11];而环境样品的组分是复杂的,这就要求引物的通用性是适中的:即需要对于不同的鱼类物种具有通用性,又需要对于鱼类物种的扩增具有一定的特异性,不能扩增或者过多的扩增出其他物种的目的条带。另外运用eDNA对鱼类进行研究,几乎都是在欧美或者日本开展的[6,8,12],由于鱼类区系的差异,我国是一个以鲤科为主要类群的国家[13],水域生态系统中主要鱼类组成为鲤科鱼类,因此国外研究中提供的鱼类通用引物的通用性和适用性也有待论证。

1材料与方法
1.1样品采集和DNA提取
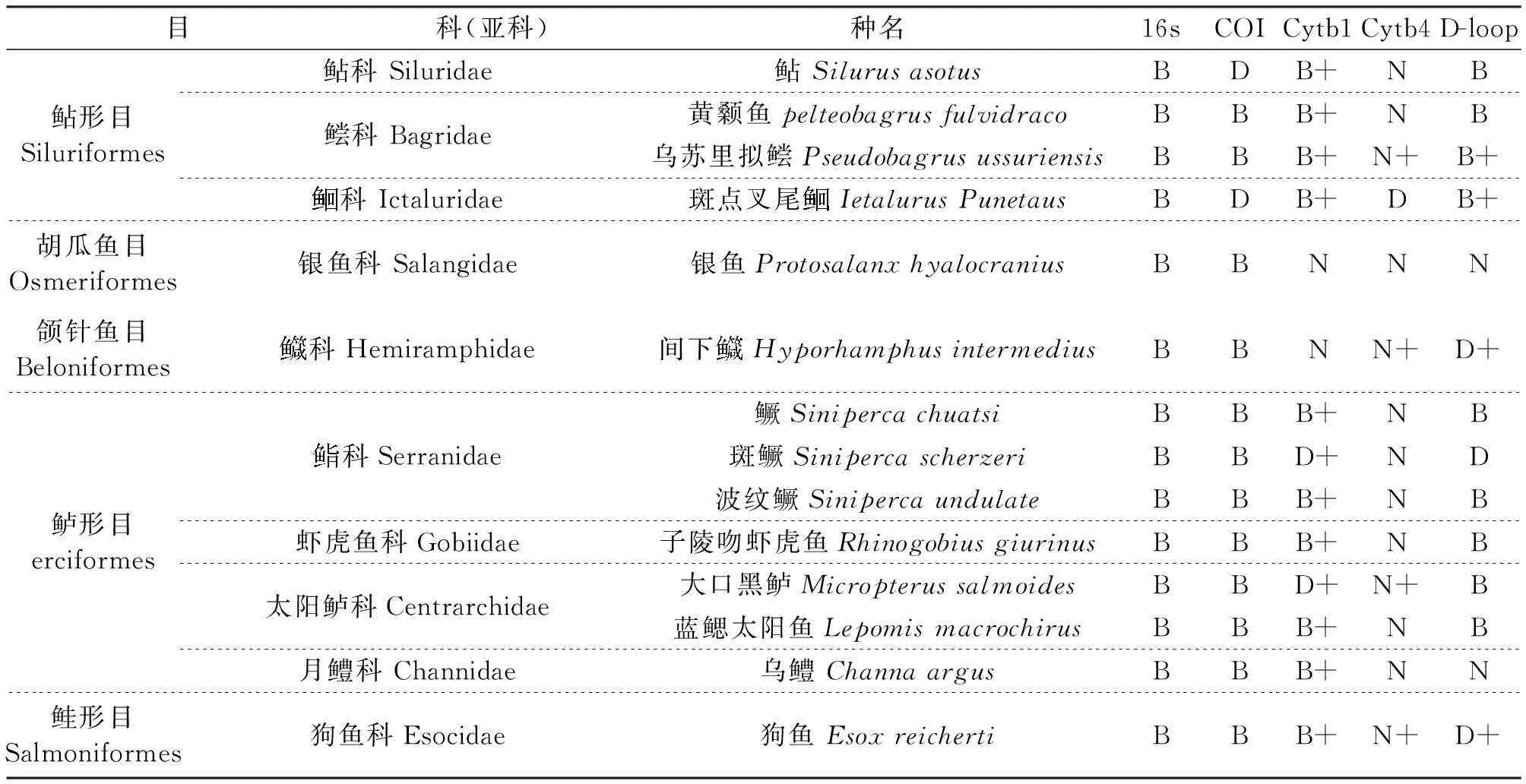
2014年7月在千岛湖通过刺网捕捞、向渔民收购,收集鱼类48种(见附录),保存在无水乙醇中,使用改进的酚氯仿法[15]从鱼类肌肉组织中提取基因组DNA。同时在千岛湖上游库区随机选取10个点采集水样,每份样品1 L,放入带冰的保温箱中带回千岛湖发展有限公司水科所实验室,24 h内对水样进行抽滤处理,使用滤膜孔径为1.2 μm,使用QIAGEN公司生产的试剂盒QIAamp DNA Micro Kit提取滤膜中的环境DNA。提取好的DNA溶于TE缓冲液中,-20 ℃保存备用。
1.2实验引物的选择
鱼类线粒体DNA与核DNA相比具有分子小、结构简单、易于提取且是母系遗传的特点,因此选择线粒体DNA片段作为分析鱼类种群结构、物种分类鉴定的分子标记[16]。参照文献[17-21]找到5对线粒体序列的鱼类通用引物,由上海生工生物工程有限公司合成,见表1。

表1 PCR所用的5对通用引物
1.3PCR扩增及电泳
使用选取的5对通用引物分别对鱼类DNA和环境DNA进行扩增,反应条件和体系见表2。 扩增后对PCR产物进行凝胶电泳检测,各取5 μL的PCR产物于1%的琼脂糖凝胶中,电压100 V,电泳60 min,用溴化乙锭染色8 min,最后在凝胶成像系统上拍照,并统计每对引物的扩增效果。

表2 5对通用引物进行PCR扩增的反应体系和条件
1.4克隆测序
挑选出对鱼类样品和环境样品都能够良好扩增的引物,再随机挑选环境样品的扩增产物进行切胶克隆测序,然后通过GENBANK进行Blast比对,以验证环境样品中扩增出来的产物是否为鱼类序列。克隆方法参见《Molecular Cloning:A Laboratory Manual-IV》[22]。
2结果
2.1鱼类样品的扩增结果
按照1.3中的反应体系和条件分别用5对引物对提取的48种鱼类DNA进行PCR扩增,扩增产物的凝胶电泳图像如图1-图5。

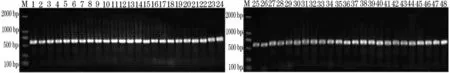
图1 使用16s扩增鱼类样品的结果


图2 使用COI扩增鱼类样品的结果
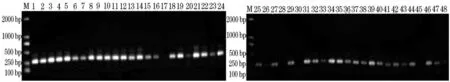
图3 使用Cytb1扩增鱼类样品的结果
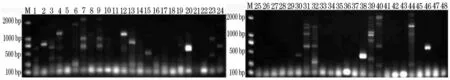
图4 使用Cytb4扩增鱼类样品的结果
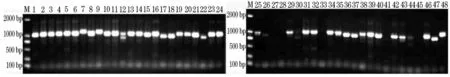
图5 使用D-loop扩增鱼类样品的结果

2.2环境样品的扩增结果
参照鱼类样品扩增的体系和条件,用该5对引物分别对10个环境样品进行扩增,扩增结果如图6。
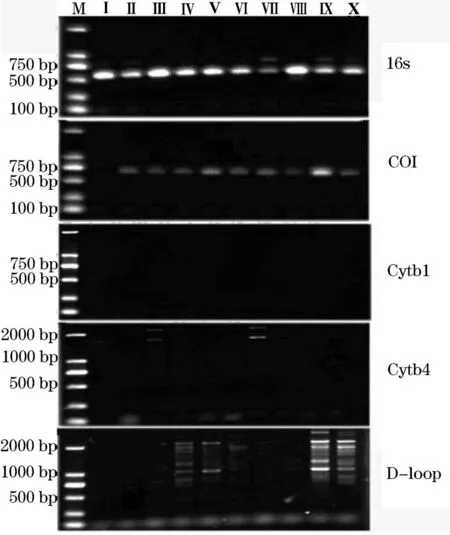
图6 使用5对引物(16s、COI、Cytb1、Cytb4、D-loop)
由图6可知,引物16s扩增出了所有环境样品中的16S rRNA基因片段,且目的条带明亮清晰,但是在Ⅰ、Ⅱ、Ⅶ、Ⅸ、Ⅹ泳道上相同的位置出现了一条暗淡的非特异性条带。引物COI的扩增条带干净清晰,无非特异性条带,无拖带现象,但是对I号环境样品没有扩增。而对鱼类样品有较好扩增效果的引物Cytb1,无法扩增出10个环境样品的中的目的基因。引物Cytb4的扩增结果显示部分泳道在1 500 ~2 000 bp间出现亮带,而Cytb4的目的条带为500 bp,说明该引物对目的基因片段没有扩增。至于引物D-loop的扩增结果显示只有4个环境样品得到了明显扩增,而且杂带较多,无法判断目的条带,对环境样品的扩增效果无法满足后续的实验要求。
2.3克隆测序结果
结合鱼类样品和环境样品的扩增结果,可知通用引物16s和COI对鱼类样品和环境样品都有较好的扩增效果,目的片段清晰、明亮。为了进一步了解它们扩增的基因片段是否为鱼类片段,将16s和COI的扩增产物送上海生工生物工程有限公司进行克隆测序。测得序列在GENBANK中进行Blast对比,对比结果显示(见表3)引物16s的扩增产物克隆测序得到15个阳性序列,经比对检测出6条黄尾鲴(Xenocyprisdavidi)序列、5条蓝鳃太阳鱼(Lepomismacrochirus)序列以及细鳞鲴(Plagiognathopsmicrolepis)、蒙古鲌(Chanodichthysmongolicus)、鲢(Hypophthalmichthysmolitrix)、鳙(Hypophthalmichthysnobilis)序列各1条,全部为千岛湖常见鱼类。而COI扩增产物克隆测序得到的5条阳性序列,经对比发现均为细菌的细胞色素氧化酶第一亚基。

表3 使用引物16s和COI扩增环境样品产物的比对结果
3讨论
环境DNA是直接从外界提取的总DNA,而从水样中提取的环境DNA来源也很丰富,包括水中的微生物、水生动物的粪便、尿液和粘液,水中动植物死亡后的组织细胞,以及陆生动植物遗留在水中和由于人类活动进入到水体中的DNA源[23]。因此本试验提取的环境DNA是一种成分多样的混合DNA,同时由于承载DNA源介质的特殊性也注定混合DNA里面存在的目的基因很微量。针对水样中环境DNA“成分多”“浓度低”的特点,找到一对特异性好、适用性高的通用引物是关键。特异性好是指从环境DNA中只能够扩增出鱼类的基因片段,而适用性高主要是针对鱼类DNA在环境DNA中所占比例低的情况。

鱼类mtDNA的D-loop区属于遗传高变区,进化速度快,多态性丰富,很适合鱼类种内种群和个体间的遗传分化研究。Jerde等[8]使用2对D-loop区特异性引物分别扩增出了水样DNA中鲢和鳙的基因片段,以此估测鲢和鳙在密西西比河中的入侵范围。这与本初衷有一定的相似性,都是希望能够扩增出水环境DNA中的鱼类基因片段用作进一步分析,不同的是本研究是期望找到一对能够扩增水环境DNA中多数鱼类基因的引物。本实验中应用的D-loop引物能够扩增绝大部分亚洲鲤科鱼类[21],同时从本实验结果也可以看到该引物对淡水鱼类有较高的通用性,但是其对环境DNA的扩增效率很低,且非特异性条带出现较多,这可能是由于环境中其他物种来源的DNA被该引物非特异性扩增的结果,因此不能满足我们实验的扩增要求。
另外,引物的特异性也反映出鱼类间的进化关系,一般的,在线粒体这样的序列中,亲缘关系越近的类群,由于DNA序列上面的差异小,其被同一对通用引物所扩增出来越容易。而在我们研究中,胡瓜鱼目中的银鱼,鲤形目中的鳡同时不能被Cytb1和D-loop这两对引物扩增出来,胡瓜鱼目本就独属一目,所以扩增情况可以反映出引物对于不同类群之间的通用性;而鳡作为东亚鲤科鱼类的代表[13],传统的研究认为:东亚鲤科鱼类是一个单系群[26],而这两对引物的扩增上面却暗示该种在系统发生中存在着与鲤科其他类群的较大差异。鳡由于强烈的特化,已经进化成为凶猛的食肉性鱼类,其形态与鲤科其他类群之间有着较大的差异,上述的结果,一方面提示我们鳡的进化生物学研究是一个值得关注的领域,另一方面也提示我们,在进行通用引物的筛选或设计时,要特别注意引物对于鳡的通用性。
引物COI对鱼类DNA有很高的扩增效率,Ivanova等[18]使用该引物扩增了94种鱼类DNA,有效扩增达到了96.8%,同时对环境DNA也有较高的扩增效率。但就本实验而言从克隆测序结果可知,COI从环境DNA 中扩增出来的基因片段属于细菌微生物细胞色素氧化酶第一亚基基因,没有发现鱼类基因。由此说明COI引物没有足够的“特异性”,在微生物基因组存在的情况下,不能用来扩增环境DNA中的鱼类片段。引物16s扩增产物克隆测序结果显示了不同种类的鱼类基因,鉴定出来的物种均是千岛湖的常见鱼类,且结果中不仅有鲤科鱼类,还存在近年来在调查中普遍较多存在的外来物种—蓝鳃太阳鱼[14],因此,该引物符合实验的要求。
本实验采用千岛湖eDNA样品的目的是为了验证各引物对环境样品的扩增效果,由于不是用于鱼类群落组成的研究,因此并不需要考虑取样点鱼类群落的分布特点、种群密度、以及资源量的实际情况,但在使用所筛选的引物进行后续的鱼类群落研究时,我们还需要考虑上述问题对采样点进行设计。另外,由于各对引物对于鱼类线粒体部分序列的通用性验证已经通过48种鱼类DNA的PCR扩增实验得到检验,为了进一步验证上述的引物对eDNA样品进行PCR扩增的适用性,我们通过克隆测序对eDNA的PCR产物进行分析,目的是为了了解各组引物对于eDNA样品中鱼类DNA的扩增效果(即确定不同引物是否在组分复杂的eDNA样品中可以有效地扩增出鱼类的线粒体基因片段,而不是为了获取详细的鱼类群落组成信息),因此并未大量进行克隆测序,也使得获得的测序结果只有48种千岛湖的主要鱼类物种。在后续的鱼类群落研究时,我们可以采用测序深度更大的二代测序技术来替代克隆测序以此获取更为丰富的物种组成信息。
引物16s既能扩增全部实验鱼类的DNA,也能在水样DNA中扩增出鱼类基因,并且能够通过从水样中扩增出来的鱼类基因达到鉴别鱼类种类的目的,可以满足水样中eDNA应用研究的要求,这对探讨水环境中鱼类种类分布,种群结构及生物多样性具有一定现实意义。但是,目前水环境DNA的应用仍处于初级阶段,由于水环境DNA成分复杂、受影响因子多,因此各研究在提取时采取的方法也不尽相同[23],不同方法对环境DNA提取造成的影响仍不清楚。另外,水环境DNA中目的基因浓度对扩增效果的影响也制约了eDNA技术在水生动物方面的应用,因此引物对目的基因浓度的扩增下限有待进一步研究。
参考文献:
[1]Levy B D J,Campbell R G,Gulden R H,et al.Cycling of extracellular DNA in the soil environment[J].Soil Biol Biochem,2007,39(12):2977-2991.
[2]Pietramellara G,Ascher J,Borgogni F,et al.Extracellular DNA in soil and sediment:fate and ecological relevance[J].Biol Fertility Soils,2009,45(3):219-235.
[3]Blaalid R,Carlsen T,Kumar S,et al.Changes in the root-associated fungal communities along a primary succession gradient analysed by 454 pyrosequencing[J].Mole Ecol,2012,21(8):1897-1908.
[4]Bohmann K,Monadjem A,Lehmkuhl Noer C,et al.Molecular diet analysis of two African free-tailed bats(Molossidae)using high throughput sequencing[J].PLoS One,2011,6(6):e21441.
[5]Olson Z H,Briggler J T,Williams R N.An eDNA approach to detect eastern hellbenders (Cryptobranchus a.alleganiensis) using samples of water[J].Wildlife Res,2012,39(7):629-636.
[6]Teruhiko T,Toshifumi M,Hiroki Y,et al.Estimation of fish biomass using environmental DNA[J].PLoS One,2012,7(4):e35868.
[7]Pilliod D S,Goldberg C S,Arkle R S,et al.Estimating occupancy and abundance of stream amphibians using environmental DNA from filtered water samples[J].Can J Fisher Aquatic Sci,2013,70(8):1123-1130.
[8]Jerde C L,Mahon A R,Chadderton W L,et al.“Sight-unseen”detection of rare aquatic species using environmental DNA[J].Conserv Letters,2011,4(2):150-157.
[9]Teruhiko T,Toshifumi M,Hideyuki D.Using Environmental DNA to Estimate the Distribution of an Invasive Fish Species in Ponds[J].PloS One,2013,8(2):e56584.
[10]Xiao W H,Zhang Y P,Liu H Z.Molecular Systematics of Xenocyprinae (Teleostei:Cyprinidae):Taxonomy,Biogeography,and Coevolution of a Special Group Restricted in East Asia[J].Mole Phyl Evol,2001,18(2):163-173.
[11]陈文炳,赵晨,邵碧英,等.PCR方法检测河豚鱼的引物筛选及反应体系优化[J].食品科学,2010,31(20):376-381.
[12]Goldberg C S,Sepulveda A,Ray A,et al.Environmental DNA as a new method for early detection of New Zealand mudsnails (Potamopyrgusantipodarum)[J].Fresh Sci,2013,32(3):792-800.
[13]陈宜瑜.中国动物志,硬骨鱼纲,鲤形目(中)[M].北京:科学出版社,1998.
[14]刘其根,汪建敏,何光喜.千岛湖鱼类资源[M].上海:上海科学出版社,2011:48-64.
[15]Sambrook J,Fritach E F,Maniatis T.Molecular cloning:A Laboratory Manual[M].2nd edn.New York:Cold Spring Harbor Laboratory,1989.
[16]郭新红,刘少军,刘巧,等.鱼类线粒体DNA研究新进展[J].遗传学报,2004,31(9):983-1000.
[17]Palumbi S R.Nucleic acids II:the polymerase chain reaction[J].Mole System,1996,2(1):205-247.
[18]Ivanova N V,Zemlak T S,Hanner R H,et al.Universal primer cocktails for fish DNA barcoding[J].Mole Ecol Notes,2007,7(4):544-548.
[19]Toshifumi M,Hiroki Y,Teruhiko T,et al.Surveillance of fish species composition using environmental DNA[J].Limnol,2012,13(2):193-197.
[20]Kyle C J,Wilson C C.Mitochondrial DNA identification of game and harvested freshwater fish species[J].Forensic Sci Internat,2007,166(1):68-76.
[21]Liu H,Tzeng C S,Teng H Y.Sequence variations in the mitochondrial DNA control region and their implications for the phylogeny of the Cypriniformes[J].Can J Zool,2002,80(3):569-581.
[22]Green M R.Molecular Cloning:A Laboratory Manual-IV[M].London:Cold Spring Harbor Laboratory Press 4 Lab ed,2012.
[23]Rees H C,Maddison B C,Middleditch D J,et al.The detection of aquatic animal species using environmental DNA-a review of eDNA as a survey tool in ecology[J].J Appl Ecol,2014,51(5):1450-1459.
[24]Thomsen P F,Kielgast J,Iversen L L,et al.Detection of a diverse marine fish fauna using environmental DNA from Seawater Samples[J].PloS One,2012,7(8):e41732.
[25]Thomsen P F,Kielgast J,Iversen L L,et al.Monitoring endangered freshwater biodiversity using environmental DNA[J].Mole Ecol,2012,21(11):2565-2573.
[26]He S,Liu H,Chen Y,et al.Molecular phylogenetic relationships of Eastern Asian Cyprinidae (Pisces:Cypriniformes) inferred from cytochrome b sequences[J].Sci China Series C:Life Sciences,2004,47(2):130-138.
(责任编辑:张潇峮)
续附录1
Universal primer screening and verification for
fish environment DNA research
LIU Jun,ZHAO Liang-jie,FAN Ying-chun,ZHANG Xin-lei,LIU Qi-gen
(KeyLaboratoryofFreshwaterFisheryGermplasmResources,MinistryofAgriculture,ShanghaiOceanUniversity,
Shanghai201306,China)
Abstract:In order to screen the primer of well universality and applicability for eDNA research,five pairs of universal primer of fish mitochondrial genome fragment were chosen from the previous references to amplify the partial sequences of D-loop region,16S rRNA gene,COI gene and Cytb gene.These primers were used to amplify genome of 48 species of fish in Qiandao Lake to test the universality,it was found that the 16s and COI were better than the other primers.Gel electrophoresis showed that all 16s DNA bands of the 48 species of fish was light,but 3 COI gene bands of the fish species was dark.The eDNA from Qiandao Lake were amplified by these five pairs of primer,and the electrophoretogram showed that the PCR product of 16s DNA and COI gene presented a clear bright band.According to the cloned sequencing and BLAST results,the PCR product of 16s DNA was coincident to the fragments of fishes from Qiandao Lake,but the PCR product of COI gene was in accord with the COI gene of bacteria.In summary,the primer of 16S rRNA was the most suitable universal primer to study on eDNA of fish community structure in the Qiandao Lake.
Key words:primer screening;mitochondrial marker;environment DNA;Qiandao Lake
中图分类号:S917.4
文献标识码:A
文章编号:1000-6907-(2016)01-0009-09
作者简介:第一刘军(1990-),男,硕士研究生,专业方向为分子生态学。E-mail:liujuncool2000@163.com通讯作者:刘其根。E-mail:qgliu@shou.edu.cn
收稿日期:2015-03-04;
修订日期:2015-06-23
资助项目:公益性行业(农业)专项“湖泊水库养殖容量及生态增养殖技术研究与示范”(201303056);教育部博士点基金项目(20123104110002);上海市高校知识服务平台项目(ZF1206)
