牛支原体蛋白-蛋白相互作用网络的构建
2016-01-13
牛支原体蛋白-蛋白相互作用网络的构建

刘威1,袁芳艳1,周丹娜1,刘泽文1,杨克礼1,段正赢1,郭锐1,肖少波2,田永祥1*
(1. 湖北省农业科学院畜牧兽医研究所/动物胚胎与分子育种湖北省重点实验室,湖北武汉 430064;2. 华中农业大学农业微生物学国家重点实验室,湖北武汉 430064)
摘要:病原菌的相互作用组学研究是揭示潜在信号转导通路和新型药物靶标的有力工具。通过同源蛋白映射的方法构建了牛支原体的蛋白-蛋白相互作用网络,所构建的互作网络包含138个蛋白和693个相互作用关系。通过蛋白的互作频率分析,确定了在牛支原体中互作频率最高的20个蛋白,这些蛋白主要与转录、翻译、分子伴侣等功能相关。进一步分析发现,伴侣蛋白DnaK和ClpB发生互作的频率较高,能与核糖体蛋白、代谢相关蛋白等多种功能蛋白发生相互作用。结果提示,若DnaK和ClpB的功能受到抑制,将会对牛支原体的正常生命活动造成较大的影响。
关键词:牛支原体;蛋白相互作用;互作网络
牛支原体(Mycoplasmabovis)是感染牛的一种重要的致病性病原体,可导致牛肺炎、耳炎、乳腺炎、关节炎、角膜炎、流产或不孕等多种疾病[1-2]。该病在世界范围内广泛存在,给养牛业造成了巨大的经济损失[3]。据初步统计,欧洲每年约25%~33%的犊牛肺炎是由牛支原体引起,造成的直接经济损失高达1.44亿~1.92亿欧元,其中英国每年约190万头牛患支原体肺炎;美国每年因牛支原体感染造成的经济损失约1.40亿美元[4]。2008年,在中国湖北省首次发现了疑似牛肺疫的呼吸道疾病,发病2 476头(发病率高达50%~100%),死亡610头,随后贵州省和宁夏回族自治区也报道了类似病例[5]。通过检测核酸,发现此呼吸道疾病并非牛肺疫复发,而是一种我国从未出现过的新发支原体传染病[6]。目前,已有内蒙古、广西、天津、重庆、四川、河南等10多个省市自治区报道了类似疫情,牛支原体已经成为威胁我国养牛业的重要病原[7]。
牛支原体感染后的临床症状与牛肺疫类似,感染初期症状不明显,待出现明显症状时,病情已十分严重。感染初期,病牛体温升高至39℃~40℃,食欲不振,精神沉郁,呼吸急促,干咳,有黏液性或化脓性鼻分泌物[8]。随着病情加剧,体温继续升高至40℃~41℃,采食量急剧下降,清晨和采食后咳嗽明显,使用抗生素治疗后病情略有好转,但病情反复,随后病畜开始死亡,大部分病牛剖检后肺部可观察到明显的粘连[9]。目前世界上仅有两家美国公司的牛支原体疫苗获得美国农业部授权,但是并没有充足的数据证实疫苗保护的有效性[10]。一方面,牛支原体的抗体水平与保护率并不一致,即使抗体在机体中处于较高水平,但是长途运输和饲养环境变化,依然可以诱发牛支原体肺炎的暴发。另一方面,牛支原体菌株表面存在一个由13个基因组成的Vsp家族,Vsp蛋白容易发生漂移,继而引起抗原表位的变化,这也是疫苗保护效果并不理想的又一个重要原因[11]。在药物治疗方面,支原体由于缺乏细胞壁,对常用的作用于细胞壁的抗生素不敏感。一些牛支原体敏感的药物,如四环素类、喹诺酮类药物,因为在养殖生产中的大量使用甚至滥用,催生出了许多耐药菌株,显著降低了抗生素的治疗效果[12]。利用现代生物信息学和分子生物学技术寻找牛支原体新的药物靶标,将为牛支原体病的防控奠定坚实的基础,为新型药物的研发提供理论依据。
1材料与方法
1.1材料
1.1.1基因组序列与数据库牛支原体Hubei-1的全基因组序列来源于NCBI数据库,氨基酸序列通过Artemis导出。直系同源分析时涉及到的数据来源于Database of Interaction Protein(DIP)数据库(数据更新至20150101)。
1.2方法
1.2.1同源蛋白映射DIP数据库中蛋白与蛋白间的互作关系已经通过酵母双杂交、免疫共沉淀等试验验证确定。同源蛋白映射(HPM)是通过整合试验中已确定的蛋白-蛋白相互作用信息,来预测未知蛋白间的互作网络[13]。图1展示了同源蛋白映射的原理。如DIP数据库中C与D能发生互作,而蛋白A与C同源,B与D同源,那么A和B极可能发生相互作用。基于此通过HPM构筑蛋白-蛋白相互作用网络。
1.2.2相互作用网络的构建DIP数据库中的氨基酸序列从DIP网站上下载获得(20150101)。Blast软件包和牛支原体的基因组序列从NCBI FTP服务器下载。利用Formatdb将牛支原体的氨基酸序列建库,DIP数据库中的氨基酸序列与牛支原体氨基酸序列的同源性通过Blastp确定(E-value 1e-30)(表1)。Blastp得到的初步比对结果通过本实验室设计的script程序映射成互作网络,以获得最终的牛支原体蛋白-蛋白相互作用网络。蛋白-蛋白相互作用网络通过可视化软件Cytoscape[14]展示。
2结果
2.1牛支原体蛋白-蛋白相互作用网络
牛支原体蛋白-蛋白相互作用网络通过同源蛋白映射的方法构建完成(图1)。预测的蛋白互作网络中包含138个蛋白和693个相互作用关系,蛋白用圆形的节点表示,相互作用关系用直线表示。

图1 同源蛋白映射方法的原理

程序/数据库名称Software/database主要用途Mainuses网络资源NetworkresourceArtemis基因组可视化Visualizationofgenomehttp://www.sanger.ac.uk/resources/software/artemis/Blastp同源比对Sequencehomologyanalysishttp://www.ncbi.nlm.nih.gov/Cytoscape可视化分析Visualizationanalysiswww.cytoscape.org/COG蛋白质功能分类Classificationofproteinfunctionhttp://www.ncbi.nlm.nih.gov/COGDIP蛋白相互作用数据库DatabaseofInteractingProteinhttp://dip.doe-mbi.ucla.edu/dip/Main.cgiPerlPerl语言Perllanguagehttp://www.perl.org/
2.2蛋白相互作用发生频率的分析
在蛋白-蛋白相互作用网络中,互作频率表示某单一蛋白与其他蛋白发生相互作用的数量。互作频率较高的蛋白往往参与细胞的多种生命活动,在细胞的生存中起着十分关键的作用。表2列出了互作频率最高的20个蛋白,在这20个蛋白中,大多数蛋白与转录、翻译、分子伴侣等功能相关。MMB0149和 MMB0664分别编码伴侣蛋白DnaK和ClpB,这两个蛋白的互作频率较高,且都能与核酸代谢蛋白(如MMB0047等)、运输相关蛋白(如SecA等)、DNA复制与修复相关蛋白(如MMB0242,MMB0606等)、氨基酸合成相关蛋白(如MMB0192等)发生相互作用。若DnaK和ClpB的功能受到抑制,将严重影响牛支原体的生存和繁殖,结果提示DnaK和ClpB可以作为新型药物的潜在靶标。
2.3蛋白相互作用网络中不同功能蛋白的分布
蛋白质直系同源簇(COGs)数据库是对细菌、藻类和真核生物的21个完整基因组的编码蛋白,根据系统进化关系分类构建而成。COGs库常用于未知蛋白的功能预测和蛋白功能的分类研究中。我们对牛支原体蛋白相互作用网络中互作频率高于10次的蛋白进行了功能分布分析,首先将互作频率高于10次的蛋白根据蛋白质直系同源簇(COGs)数据库进行功能分类,随后对不同COGs功能类别的蛋白个数进行统计分析。结果如图3所示:互作频率较高的功能蛋白主要与核糖体结构、复制、翻译、分子伴侣和防御等功能相关。核糖体是细胞内一种核糖核蛋白颗粒,由RNA和蛋白质构成,其唯一功能是按照mRNA的指令将氨基酸合成蛋白质多肽链,是细胞内蛋白质合成的分子机器。MMB0182编码DNA拓扑异构酶,拓扑异构酶是生物体内的基础酶类,参与到所有涉及DNA拓扑结构变化的生命活动中,如DNA的复制,转录和重组。细菌细胞中,三分之一的蛋白质是在合成后被转运到细胞质外才发挥功能的。其中大多数蛋白是通过Sec途径(即分泌途径secretion pathway)进行跨膜运动的。MMB0288编码水解ATP的动力蛋白SecA,SecA和膜蛋白三聚体SecYEG共同构成了Sec转运酶的核心。MMB0192编码磷酸核糖焦磷酸,是一种重要的代谢中间物,参与嘌呤核苷酸与嘧啶核苷酸的从头合成和补救合成、某些核苷酸类辅酶如辅酶Ⅰ和辅酶Ⅱ、以及某些氨基酸如组氨酸和色氨酸的合成。上述蛋白在维持细胞正常的生命活动中都起着十分关键的作用,若这些蛋白的功能受到抑制将严重影响牛支原体的生存与繁殖。

图2 牛支原体蛋白-蛋白相互作用网络
3讨论
病原菌互作蛋白组学分析是解析毒力相关信号通路和挖掘潜在药物靶标的重要工具[13]。本研究通过HPM的方法构建了牛支原体的蛋白-蛋白相互作用网络,所构建的互作网络包括138个蛋白和693个相互作用关系。HPM的方法主要通过计算蛋白序列的同源性,因此会造成少数假阳性的结果。为了减少假阳性的发生,我们在构建互作网络时,通过结合序列覆盖率和E-value[15]值综合评价单一蛋白和互作关系的准确性,以确定最终的蛋白互作网络。
在蛋白-蛋白相互作用网络中,蛋白发生互作的频率越高,提示这一蛋白在病原菌的生命活动中越重要。在完成了牛支原体蛋白互作网络的构建后,通过COGs对互作频率最高的20个蛋白进行了功能分类。在这20个蛋白中,大多数蛋白与转录、翻译、分子伴侣等功能相关。分子伴侣和核糖体蛋白往往在细胞的生命活动中起着关键的作用,如伴侣蛋白能帮助蛋白正确折叠和降解,这对细胞内病原菌抵抗宿主细胞的环境压力十分重要。在牛支原体蛋白互作网络中,DnaK和ClpB发生互作的频率较高,能与核糖体蛋白、代谢相关蛋白等多种蛋白发生相互作用。一旦DnaK和ClpB的功能受到抑制,将会对牛支原体的正常生命活动造成不小的影响。综上所述,通过HPM的方法构建了牛支原体蛋白-蛋白相互作用网络,蛋白互作网络的构建将为新型药物靶标的寻找、新的信号分子的筛选提供依据。
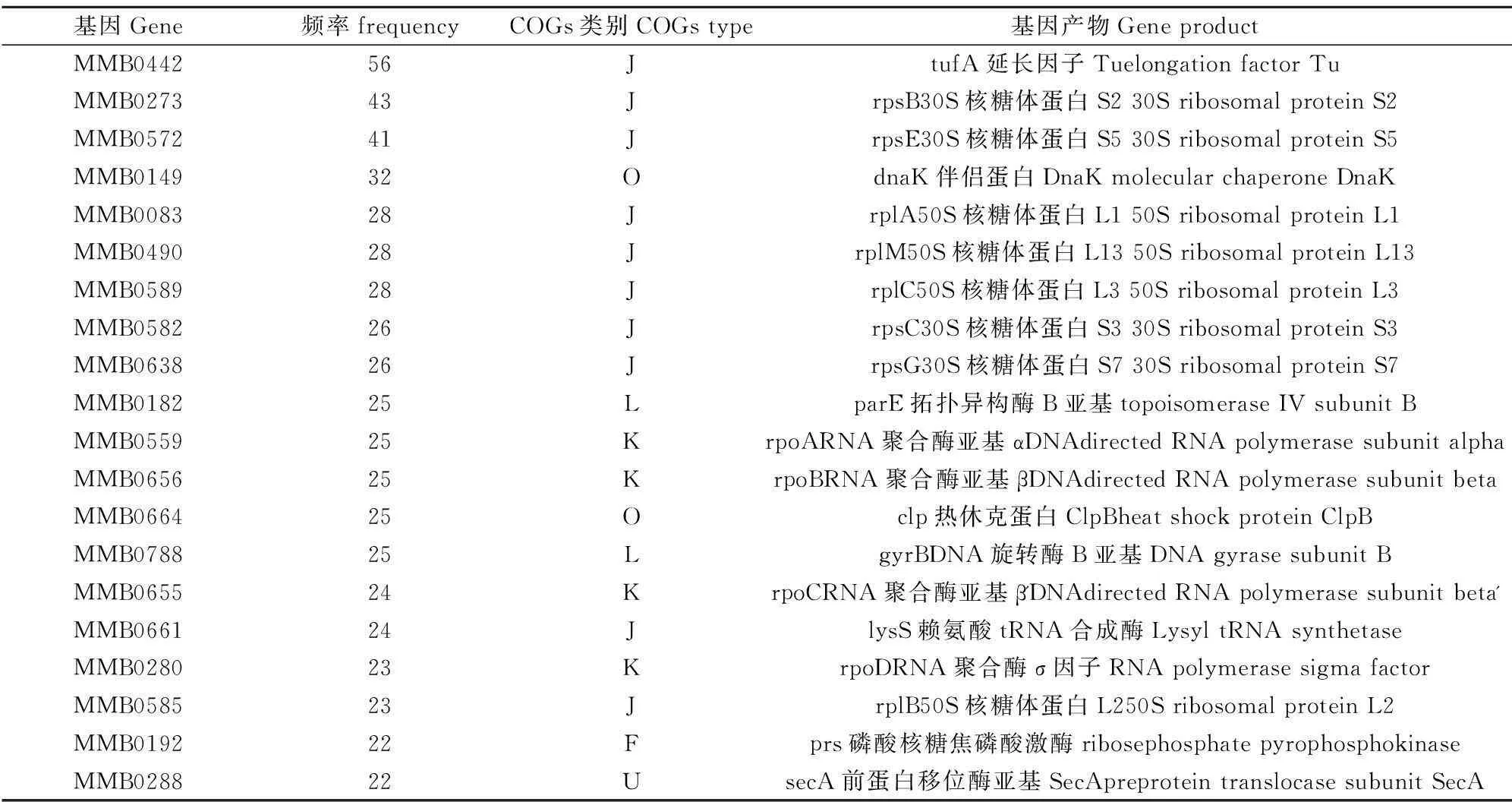
表2 相互作用网络中互作频率较高的20个蛋白

C.能量产生和转换;E.氨基酸运输和代谢;F.核酸运输和代谢;G.碳水化合物运输和代谢;H.辅酶代谢;J.翻译,核糖体结构和产生;K. 转录;L. DNA复制,重组和修复;O.转译后的修饰,伴侣蛋白;R.常规功能;U.细胞内运输,分泌和囊膜运输;V.防卫机制
C.Energy production and conversion;E.Amino acid transport and metabolism;F.Nucleotide transport and metabolism;G. Carbohydrate transport and metabolism;H.Coenzyme metabolism;J.Translation, ribosomal structure and biogenesis;K.Transcription;L.DNA replication, recombination and repair;O.Posttranslational modification, protein turnover, chaperones;R.General function prediction only;U.Intracellular trafficking, secretion and vesicular transport;V.Defense mechanisms
图3 牛支原体蛋白相互作用网络中不同功能蛋白的分布
Fig.3The COGs distribution in the protein-protein interaction network ofMycoplasmabovis
参考文献:
[1]Morton J,Malmo J,House J,et al.Mycoplasmabovisin Australian dairy herds[J]. Aust Vet J,2014, 92(9):322-323.
[2]张瑞,崔朋,巴晓亮,等.牛支原体临床分离株牛体毒力和免疫原性比较[J].动物医学进展,2013,34(6):126-132.
[3]Aebi M,van den Borne BH,Raemy A,et al.Mycoplasmabovisinfections in Swiss dairy cattle: a clinical investigation[J]. Acta Vet Scand,2015,57:10. doi: 10.1186/s13028-015-0099-x.
[4]胡长敏,石磊,龚瑞,等. 牛支原体病研究进展[J]. 动物医学进展,2009,30(8):73-77.
[5]石磊,龚瑞,尹争艳,等. 肉牛传染性牛支原体肺炎流行的初步诊断[J]. 华中农业大学学报,2008,27(4):572-572.
[6]辛九庆,李媛,郭丹,等. 国内首次从患肺炎的犊牛肺脏中分离到牛支原体[J]. 中国预防兽医学报,2008,30(9):661-664.
[7]伍晓红,储岳峰,张轩,等.牛支原体的分离鉴定及16S rRNA基因序列分析[J].动物医学进展,2012,33(12):35-37.
[8]冉智光,谢建华,骆璐,等. 我国部分地区牛支原体肺炎和关节炎的病原体诊断[J]. 中国预防兽医学报,2010,32(1):40-43.
[9]Radaelli E,Luini M,Loria G R,et al. Bacteriological, serological, pathological and immunohistochemical studies ofMycoplasmabovisrespiratory infection in veal calves and adult cattle at slaughter[J]. Res Vet Sci,2008,85(2): 282-290.
[10]Mulongo M,Prysliak T,Perez C J,et al. Vaccination of feedlot cattle with extracts and membrane fractions from twoMycoplasmabovisisolates results in strong humoral immune responses but does not protect against an experimental challenge[J]. Vaccine,2013,31: 1406-1412.
[11]Li Y,Zheng H,Liu Y,et al. The complete genome sequence ofMycoplasmabovisstrain Hubei-1[J]. PLoS One,2011,6(6): e20999.doi: 10.1371/Journal.pone.0020999.
[12]Bürki S,Frey J,Pilo P,et al. Virulence, persistence and dissemination ofMycoplasmabovis[J]. Vet Microbiol,2015,pii: S0378-1135(15)00079-6. doi: 10.1016/j.vetmic.
[13]Cui T,Zhang L,Wang X,et al.Uncovering new signaling proteins and potential drug targets through the interactome analysis of Mycobacterium tuberculosis[J]. BMC Genomics,2009,10:118.doi:10.1186/1471-2164-10-118.
[14]Shannon P,Markiel A,Ozier O,et al.Cytoscape:a software environment for integrated models of biomolecular interaction networks[J]. Genome Res,2003,13:2498-2504.
[15]Zhaxybayeva O,Gogarten J P.Bootstrap, Bayesian probability and maximum likelihood mapping: exploring new tools for comparative genome analyses[J]. BMC Genomics,2002,3:4.

动物医学进展,2015,36(11):20-24ProgressinVeterinaryMedicine
Construction of the Protein-protein Interaction Network ofMycoplasmabovis
LIU Wei1, YUAN Fang-yan1, ZHOU Dan-na1, LIU Ze-wen1, YANG Ke-li1,
DUAN Zheng-ying1,GUO Rui1, XIAO Shao-bo2, TIAN Yong-xiang1
(1.HubeiProvincialKeyLaboratoryofAnimalEmbryoandMolecularBreedingofAnimalHusbandry
andVeterinaryResearchInstituteofHubeiAcademyofAgriculturalSciences,Wuhan,Hubei,430064,China;
2.StateKeyLaboratoryofagriculturalmicrobiology,HuazhongAgriculturalUniversity,Wuhan,Hubei,430064,China)
Abstract:Analysis of the pathogen interactome is a powerfull approach for dissecting potential signal trasducion and new drug targets. We constructed a comprehensive protein-protein interaction network for Mycoplasma bovis consisting of 138 proteins and 693 interaction pairs. Our analysis unraveled 20 proteins with highest interactions in the network, most of them are related to the function of transcription, translation and molecular chaperone. The MMB0149/ MMB0664 gene and its gene product DnaK/ClpB were analyzed in detail because they showed high interactions with ribosomal and metabolism related proteins. Our analyses indicated that the chaperone DnaK and ClpB may be critical for the survival of Mycoplasma bovis. Collectively, this study therefore provides valuable clues in exploring new signaling protein and new drug targets.
Key words:Mycoplasma bovis; protein interaction; interaction network
文章编号:1007-5038(2014)11-0020-05
中图分类号:S852.62;S852.653
文献标识码:A
作者简介:刘威(1985-),男,湖北武汉人,助理研究员,博士,主要从事动物传染病研究。 *
通讯作者
基金项目:湖北省农业科技创新中心资助项目(2015-620-004-001)
收稿日期:2015-05-12