微生物合成鼠李糖脂生物表面活性剂的研究进展
2016-01-08杜瑾,郝建安,张晓青等
微生物合成鼠李糖脂生物表面活性剂的研究进展
杜瑾,郝建安,张晓青,王静,张雨山
(国家海洋局天津海水淡化与综合利用研究所,天津300192)
摘要:微生物合成的鼠李糖脂是一类重要的生物表面活性剂,具有易被生物降解、表面活性良好等优点,应用前景广阔。目前用于发酵合成鼠李糖脂的菌株主要为假单胞菌属。综述了产鼠李糖脂菌株的发现与筛选、鼠李糖脂的生物合成途径与代谢调控关系、鼠李糖脂发酵过程的优化与控制、高产鼠李糖脂工程菌株的构建等方面的研究进展,并对其研究趋势进行了展望。
关键词:鼠李糖脂;生物表面活性剂;假单胞菌;微生物发酵;代谢工程
基金项目:国家自然科学基金青年科学基金资助项目(21406042),海洋公益性行业科研专项项目(201105026,201305022-5)
收稿日期:2014-12-08
作者简介:杜瑾(1985-),女,天津人,博士,工程师,研究方向:海水利用技术及合成生物学,E-mail:dujin111@126.com;通讯作者:张雨山,教授级高级工程师,E-mail:yushanzhang@hotmail.com。
doi:10.3969/j.issn.1672-5425.2015.04.002
中图分类号:Q 81文献标识码:A
生物表面活性剂是天然的两亲化合物,与化学合成的表面活性剂相比,具有无毒、易被生物降解、高效的起泡性、良好的破乳性、对特定界面的选择性、化学结构的多样性等优点,在化工、医药、农业和环境修复等领域具有广阔的应用前景[1]。其中,鼠李糖脂是研究和应用最广泛的生物表面活性剂[2]。如在石油开采中,添加微生物合成的鼠李糖脂来降低水-原油-岩石间的界面张力,是提高原油采收率的有效方法[3]。1989年发生在美国阿拉斯加的Exxon Valdez号油轮溢油事件,通过使用微生物表面活性剂鼠李糖脂,成功地在6周内分解石油中几乎全部的正烷烃[4]。微生物合成的鼠李糖脂还具有抗细菌、抗真菌、抗病毒等生物活性[5]。因此,鼠李糖脂生物表面活性剂的生产受到了广泛关注,如2004年美国Jeneil生物表面活性剂公司研制的生物基鼠李糖脂获得了美国联邦环保署颁发的“总统绿色化学挑战奖”(Presidential Green Chemistry Challenge)[6]。因此,开发高效的微生物合成鼠李糖脂的方法具有重要的社会和环境效益。
作者从产鼠李糖脂菌株的发现与筛选、鼠李糖脂的生物合成途径与代谢调控关系、鼠李糖脂发酵过程的优化与控制、高产鼠李糖脂工程菌株的构建等4个方面综述了国内外的相关研究进展,并对其研究趋势进行了展望。
1产鼠李糖脂菌株的发现与筛选
鼠李糖脂最早于1949年由Jarvis和Johnson[7]在铜绿假单胞菌(Pseudomonasaeruginosa)中发现。P.aeruginosa也是目前发现的合成鼠李糖脂的最主要种属,在不同条件下能合成多种结构的鼠李糖脂,主要有双糖双脂结构R1(Rha-Rha-C10-C10)、单糖双脂结构R2(Rha-C10-C10)、双糖单脂结构R3(Rha-Rha-C10)和单糖单脂结构R4(Rha-C10)4种结构类型。由于脂肪链的长度及饱和程度不同,且各亚种菌株合成不同结构鼠李糖脂的比例不同,造成不同菌株合成的表面活性剂的性能各异。P.aeruginosa是条件致病菌,它合成表面活性剂的行为与其致病性相关,致使该菌的发酵生产受到限制。近年来,Pseudomonas属其它具有鼠李糖脂合成能力的菌种也陆续被分离,如P.putida[8]、P.chlororaphis[9]、P.fluorescens[10]、P.nitroreducens[11]等。
Burkholderia属是合成鼠李糖脂的另一重要菌种来源,能合成带有C14-C14脂结构的鼠李糖脂。其中,B.plantarii[12]和B.pseudomallei[13]产鼠李糖脂的过程与其对人和动物的致病性有关。而B.thailandensis[14]为非致病菌,与P.aeruginosa相比,它合成的鼠李糖脂脂链较长、双鼠李糖脂比例较高,具有良好的表面活性,是理想的生产菌之一。
很多合成鼠李糖脂的微生物是从受烃类污染的环境中获得的。如Nayak等[15]筛选到的产鼠李糖脂菌株Pseudoxanthomonassp.PNK-04能够激活其降解邻氯苯甲酸、间氯苯甲酸和1-甲基萘等污染物,可用于生物修复。Rooney等[16]从生物柴油污染的土壤中筛选到Acinetobactercalcoaceticus、Enterobacterhormaechei、Pantoeastewartii和E.asburiae等多种细菌,能够以甘油为唯一碳源生成含C10-C10脂结构的单糖或双糖鼠李糖脂,其中A.calcoaceticus和E.hormaechei合成鼠李糖脂的水平与Pseudomonas属相近。
多种革兰氏阳性菌也被发现具有合成鼠李糖脂的能力,如Tetragenococcuskoreensis[17]、Renibacteriumsalmoninarum[18]等。Christova等[19]首次报道了一株具有鼠李糖脂合成能力的Bacillussubtilis,并能够降解烃类物质。Cheng等[20]从胜利油田地下废水中分离的B.subtilisTU2能够同时生成脂肽和鼠李糖脂混合生物表面活性剂。此外还有真菌合成鼠李糖脂的报道,如Kiran等[21]从海绵中分离获得的共生真菌Aspergillussp.MSF1。
2鼠李糖脂的生物合成途径与代谢调控关系
2.1 生物合成途径及关键酶
目前,针对P.aeruginosa的鼠李糖脂生物合成途径研究比较深入。Burger等于1963年利用放射性标记前体物质追踪酶提取物,首次提出了鼠李糖脂的从头生物合成途径,并认为dTDP-L-鼠李糖和β-羟基脂肪酸是其主要前体。在该途径的基础上,逐步阐明其遗传背景、分子机制和代谢调控关系,为构建代谢工程菌株提供了可能。针对P.aeruginosa的鼠李糖脂生物合成途径如图1所示。
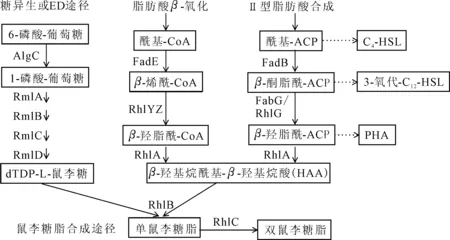
图1 鼠李糖脂生物合成途径 Fig.1 Biosynthetic pathway of rhamnolipid
由图1可知,前体dTDP-L-鼠李糖为鼠李糖脂提供糖基部分。它可经糖异生途径或Entner-Doudoroff(ED)途径起始合成,由6-磷酸-葡萄糖在磷酸葡萄糖变位酶(AlgC)的作用下转化为1-磷酸-葡萄糖[22],然后经rmlBDAC基因簇编码的1-磷酸葡萄糖胸腺嘧啶转移酶(RmlA)、dTDP-D-葡萄糖-4,6-脱水酶(RmlB)、dTDP-4-酮-6-脱氧-D-葡萄糖-3,5-异构酶(RmlC)和dTDP-4-酮-6-脱氧-D-甘露糖还原酶(RmlD)4步反应生成dTDP-L-鼠李糖[23]。
前体β-羟基脂肪酸提供鼠李糖脂的脂链部分。它的从头合成经过Ⅱ型脂肪酸合成酶系催化,在酮脂酰还原酶(FabG)作用下将β-酮脂酰-ACP还原为β-羟脂酰-ACP。2分子β-羟脂酰-ACP在肽转移酶(RhlA)作用下生成β-羟基烷酰基-β-羟基烷酸(HAA)[24]。该途径中酰基-ACP、β-酮脂酰-ACP等中间产物同时为某些群体感应(QS)信号分子的合成提供前体,在酰化高丝氨酸内酯合成酶RhlI和LasI的作用下,合成C4-HSL和3-氧代-C12-HSL等信号分子,对鼠李糖脂合成途径进行表达调控[25-26]。近期有研究表明脂肪酸的β-氧化也可为鼠李糖脂提供前体HAA,该途径中β-烯酰-CoA在水合酶/异构酶RhlYZ催化下生成β-羟脂酰-CoA,再通过RhlA作用生成HAA[27]。
RhlB与RhlA构成一种复合酶鼠李糖基转移酶1,可使前体dTDP-L-鼠李糖和HAA合成为单鼠李糖脂,再在鼠李糖基转移酶2(RhlC)作用下进一步合成双鼠李糖脂[28-29]。RhlB和RhlC均包含跨膜的疏水性区域,可锚定于细胞内膜,因此鼠李糖脂可能是在内膜的细胞质一侧合成后再转运至胞外的。
RhlAB和RhlC催化了鼠李糖脂生物合成的3个关键反应,其中RhlAB在基因组中以操纵子rhlABRI的形式编码,在启动子Prhl操纵下受QS系统调控表达。在产鼠李糖脂菌株B.thailandensis和B.pseudomaller的研究中也发现了rhlA、rhlB和rhlC基因[14],但与P.aeruginosa不同的是,3个基因以rhlABC基因簇的形式编码在一起,并在基因组中有2个相同的拷贝。
2.2 鼠李糖脂生物合成途径的调控机制
受QS信号分子在基因转录水平的调控,微生物合成鼠李糖脂的过程与细胞密度紧密相关。如P.aeruginosa中相关的QS信号分子主要有N-酰基丝氨酸内酯(AHL)和2-庚基-3-羟基-4-喹诺酮(PQS)两类,相应的QS系统有AHL-依赖的RhlR/I系统、LasR/I系统和PqsR/A/H系统[30]。其中RhlR/I系统的编码基因以操纵子rhlABRI的形式存在,直接调控rhlAB的转录表达。当细胞生长至一定密度时,RhlI合成的信号分子C4-HSL的浓度达到阈值,与转录调控子RhlR结合形成RhlR/C4-HSL,解除RhlR对转录的阻遏,激活rhlAB上游启动子Prhl开始转录[31]。鼠李糖基转移酶Ⅱ的编码基因rhlC与rhlAB位于基因组中不同位置,但也同样受RhlR/I系统调控[28]。LasR/I系统通过调控RhlR/I系统的转录来间接控制鼠李糖脂的合成途径[32]。
P.aeruginosa中QS系统在直接调控鼠李糖脂合成途径的同时,也受到其它多种转录调控因子的调控。其中对QS系统起负调控作用的转录因子,如RsaL可与lasI基因的启动子区域结合,抑制lasR和lasI基因的转录表达,间接影响鼠李糖脂的合成[33];AlgR在生物被膜形成过程中可与rhlI基因和rhlAB基因启动子部分结合,抑制鼠李糖脂合成基因的转录;PtxR通过下调PQS系统调控的基因表达来抑制鼠李糖脂的合成途径[34]。对QS系统起正调控作用的转录因子Vfr能够直接激活lasR基因和rhlR基因的转录过程,并可构成正反馈调控环[35];LysR型转录调控因子MvfR能够激活PQS系统编码基因pqsABCDE操纵子的转录[36];再如全局调控因子VqsR[37]和VqsM[38]能够促进QS系统的转录以间接调控鼠李糖脂的合成途径。感受环境压力的sigma因子(σ),如氮代谢的调控因子RpoN(σN)和菌株生长进入稳定期的调控因子RpoS(σS)均可在特定环境条件下调控QS系统的表达[39]。
另一些调控因子则在转录后对QS系统进行调控,进而影响P.aeruginosa中鼠李糖脂的合成。如QscR作为LasR和RhlR的同源蛋白,可以与信号分子AHL结合形成QscR/AHL复合物,阻碍AHL与阻遏蛋白RhlR或LasR的结合,抑制鼠李糖脂合成基因rhlAB的转录[40]。转录后调控蛋白RsmA通过与rhlI或lasI基因的转录产物mRNA相结合,阻碍mRNA翻译过程的起始,抑制QS信号分子合成[41],但同时RsmA还可能促进rhlAB基因的翻译,增强鼠李糖脂的合成[42];转录后调控蛋白DksA的调控机制与RsmA相似[43]。
上述调控元件之间的相互作用可用图2表示。
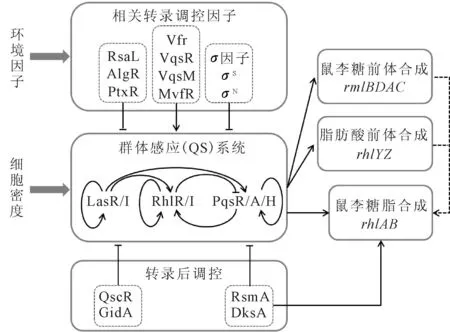
图2 鼠李糖脂生物合成途径的基因调控 Fig.2 Genetic regulation of rhamnolipid biosynthetic pathway
图2是鼠李糖脂生物合成途径的调控网络,生物合成鼠李糖脂的产量和产率受细胞密度等多种因素的复合控制。同时,鼠李糖脂前体(如鼠李糖、脂肪酸)合成途径中涉及的rmlBDAC、rhlYZ等基因也受不同QS系统调控转录[27,44]。因此,不同生长状态下,生物表面活性剂合成的完整途径中,各反应步骤受调控表达的水平不同,造成产物中单、双鼠李糖脂及代谢中间产物的比例不同,产物的表面活性、乳化性等亦有区别,从而影响发酵产品性能的稳定性。
3鼠李糖脂发酵过程的优化与调控
3.1 培养基成分的优化及低值原料的利用
微生物合成鼠李糖脂的成分和活性均受到培养基组成的影响,特别是其中的碳源底物。这是由于大部分具备表面活性剂合成能力的微生物是与其摄取生长环境中疏水性底物相适应的(如降解烷烃等),其细胞代谢与环境条件相适应[45]。研究表明,P.aeruginosa可利用多种碳源合成鼠李糖脂,如甘露醇或苯蒽烯等作为唯一碳源[46]。碳源底物的亲、疏水性可改变产物鼠李糖脂与副产物PHA和HAA的组成比例,从而改变发酵产物的表面活性和乳化性[47]。但疏水性底物往往抑制细胞生长,降低了鼠李糖脂的产量。通过添加适量葡萄糖碳源,控制油类与葡萄糖在培养基中的比例,可提高鼠李糖脂的表面活性及产量[48],但会增加原料成本。
培养基中氮源对鼠李糖脂的合成也有显著影响。如硝酸盐、谷氨酸盐等氮源能促进鼠李糖脂的合成,而铵盐、谷氨酰胺、精氨酸等氮源则抑制其合成[49]。Lee等[50]发现,以鱼油为碳源时,P.aeruginosaBYK-2合成鼠李糖脂的最佳氮源为尿素。不仅碳源和氮源的种类影响鼠李糖脂的生物合成,C/N值也有重要的影响。Guerra-Santos等[51]报道,P.aeruginosaDSM2569在以葡萄糖为碳源、硝酸盐为氮源的培养基中,C/N值在16~18之间时,鼠李糖脂产量最高,而C/N值低于11时不能合成鼠李糖脂。
此外,限制某些金属离子的浓度也有利于提高鼠李糖脂产量。Abalos等[52]报道,当培养基中FeSO4·7H2O的浓度由13.7 g·L-1降至7.4 g·L-1时,鼠李糖脂的产量明显提高。
可见,权衡原料底物的特异性、产物的表面活性及组成成分、产量的稳定性,是利用微生物发酵产鼠李糖脂的研究重点。越来越多的研究者致力于寻求廉价原料作为碳、氮源发酵合成鼠李糖脂。如Haba等[53]利用葵油和橄榄油的烹饪废油作原料时,P.aeruginosa发酵产鼠李糖脂达6.75~9.25 g·L-1;但以油类精炼废弃物等作原料时,发酵产物成分增多,甚至可能由多达六七种具有表面活性的同系物组成[54-55],反而增加了产物分离纯化的成本。
3.2 不同发酵工艺的选择
目前报道的鼠李糖脂发酵工艺主要包括分批发酵、补料分批发酵、连续发酵、休止细胞培养、固态发酵等。补料分批发酵工艺可以保持较低的底物水平并控制细胞的比生长速率,产量较高。但由于微生物合成鼠李糖脂的代谢途径和基因调控复杂,补料分批发酵生产鼠李糖脂的效果并不理想,产量在6~95 g·L-1之间,与分批发酵(5~112 g·L-1)没有显著差别[56]。如Giani等[57]将P.aeruginosaDSM7107或7108菌株在250 g·L-1豆油和15 g·L-1NaNO3培养基中进行分批发酵,鼠李糖脂产量达112 g·L-1,是目前文献报道中最高的。在补料分批发酵过程中,控制pH值可以提高发酵效率。如Chen等[58]研究发现,过多补加葡萄糖会增加酸性代谢物的积累,而葡萄糖不足则会降低鼠李糖脂产量,故以6%葡萄糖进行补料,可将P.aeruginosaS2菌株发酵体系的pH值维持在6.8,产量达6.06 g·L-1。Zhu等[59]对P.aeruginosaO-2-2菌株5 L发酵体系的pH值进行分段控制,并补加大豆油,鼠李糖脂发酵产量达70.56 g·L-1。
由于鼠李糖脂具有表面活性,其液态发酵过程中产生的大量泡沫降低了生产效率。Sarachat等[60]在发酵的同时耦合了分离方法,通过简单处理后对鼠李糖脂进行分离浓缩,在收集溢出泡沫的同时对罐内补料,实现发酵过程的连续性;但其操作和控制均较复杂,产物纯度较低,染菌几率高,使该发酵工艺的应用受到限制。固态发酵法以固体底物为发酵原料,避免了液态发酵中的泡沫问题,但目前仅限于摇瓶实验。如Camilios-Neto等[61]以质量比50∶50的蔗渣和玉米糠为固体底物、以6%(体积分数)甘油和大豆油为浸渍液,对P.aeruginosaUFPEDA 614菌株进行固态发酵生产鼠李糖脂。
4高产鼠李糖脂工程菌株的构建
鼠李糖脂的微生物合成途径由多步酶催化反应构成,并受到群体感应、底物种类等多种因素在转录、代谢等水平的复杂调控,对发酵过程的稳定高效造成影响。运用代谢工程手段和合成生物技术构建高产鼠李糖脂的工程菌株,有望实现人工调控微生物产鼠李糖脂代谢途径,降低生产成本,提高合成效率。
目前,构建鼠李糖脂工程菌的研究工作主要集中于关键基因rhlAB在非致病菌株的异源表达。Ochsner等[62]最早异源表达了rhlAB基因,在P.fluorescens和P.putida中合成鼠李糖脂。Wang等[63]将rhlAB基因簇导入不产表面活性剂的菌株E.coliBL21和P.aeruginosa-rhlA中,可赋予其产鼠李糖脂的活性并用于提高原油采收率。
某些宿主细胞合成鼠李糖脂前体的能力存在缺陷,限制了异源表达rhlAB时合成鼠李糖脂的水平。Cabrera-Valladares等[64]在E.coli中表达P.aeruginosa的rhlAB的同时,表达dTDP-L-鼠李糖合成基因rmlBDAC,保证了前体供应,使重组E.coli以葡萄糖为唯一碳源,合成单鼠李糖脂产量达120 mg·L-1。
Cao等[65]克隆了P.aeruginosaBSFD5基因组中rhlABRI整个基因簇,并整合入非致病菌P.putidaKT2440基因组中,实现rhlABRI基因簇稳定表达,鼠李糖脂产量达1.68 g·L-1,并对芘污染土壤具有良好的修复作用。Wittgens等[66]以非致病菌P.putidaKT2440作为底盘细胞异源表达rhlAB,同时基于对底盘细胞代谢通量平衡分析,敲除副产物PHA合成途径中的关键基因phaC,实现非生长依赖、以葡萄糖为底物的鼠李糖脂合成,产量达0.15 g·g-1。研究者还试图寻找更多适合表达鼠李糖脂合成基因的宿主细胞,如Tavares等[67]在Burkholderia菌株中异源表达P.aeruginosaPAO1的rhlAB基因,可提高产量并使产物结构由以双鼠李糖脂为主转变为以单鼠李糖脂为主。
5展望
围绕发酵法生产鼠李糖脂表面活性剂这一研究课题,国内外研究人员已在菌株筛选、代谢合成机制和调控机理、发酵条件和工艺优化、高产工程菌株构建等方面取得了可喜进展,但是还存在着原料成本高、产量低、产物成分不稳定、分离纯化成本高等问题,制约了鼠李糖脂生物表面活性剂的大规模产业化生产。因此,综合运用系统生物学和合成生物技术将成为未来鼠李糖脂生物合成研究的重点,主要集中于以下几个方面:(1)通过分析和比较产鼠李糖脂菌株的转录组、蛋白组、代谢物组、通量组的规律和变化,从代谢网络全局深入解析鼠李糖脂合成途径及其调控关系;(2)基于组学分析,解析鼠李糖脂合成关键反应步骤,设计、合成和优化可调控的功能模块,并选择相适配的底盘细胞,构建高效合成鼠李糖脂人工细胞;(3)通过基因改造和进化筛选,提高菌株对廉价底物的利用效率,降低原料成本,提高微生物合成鼠李糖脂的经济性。
参考文献:
[1]BANAT I M,FRANZETTI A,GANDOLFI I,et al.Microbial biosurfactants production,applications and future potential[J].Appl Microbiol Biotechnol,2010,87(2):427-444.
[2]MULLER M M,KUGLER J H,HENKEL M,et al.Rhamnolipids—Next generation surfactants?[J].J Biotechnol,2012,162(4):366-380.
[3]KRYACHKO Y,NATHOO S,LAI P,et al.Prospects for using native and recombinant rhamnolipid producers for microbially enhanced oil recovery[J].Int Biodeterior Biodegrad,2013,81:133-140.
[4]HARVEY S,ELASHVILI I,VALDES J,et al.Enhanced removal of Exxon Valdez spilled oil from Alaskan gravel by a microbial surfactant[J].Nat Biotechnol,1990,8(3):228-230.
[5]CORTES-SANCHEZ A D J,HERNANDEZ-SANCHEZ H,JA-RAMILLO-FLORES M E.Biological activity of glycolipids produced by microorganisms:New trends and possible therapeutic alternatives[J].Microbiol Res,2013,168(1):22-32.
[6]BORNSCHEUER U,HUISMAN G,KAZLAUSKAS R,et al.Engineering the third wave of biocatalysis[J].Nature,2012,485(7397):185-194.
[7]JARVIS F G,JOHNSON M J.A glyco-lipide produced byPseudomonasaeruginosa[J].J Am Chem Soc,1949,71(12):4124-4126.
[8]TULEVA B,IVANOV G,CHRISTOVA N.Biosurfactant production by a newPseudomonasputidastrain[J].Z Naturforsch C,2002,57(3-4):356-360.
[9]GUNTHER N W,NUNEZ A,FETT W,et al.Production of rhamnolipids byPseudomonaschlororaphis,a nonpathogenic bacterium[J].Appl Environ Microbiol,2005,71(5):2288-2293.
[10]VASILEVA-TONKOVA E,GALABOVA D,STOIMENOVA E,et al.Production and properties of biosurfactants from a newly isolatedPseudomonasfluorescensHW-6 growing on hexadecane[J].Z Naturforsch C,2005,61(7-8):553-559.
[11]ONWOSI C O,ODIBO F J C.Effects of carbon and nitrogen sources on rhamnolipid biosurfactant production byPseudomonasnitroreducensisolated from soil[J].World J Microb Biot,2012,28(3):937-942.
[12]HOERMANN B,MULLER M M,SYLDATK C,et al.Rhamnolipid production byBurkholderiaplantariiDSM 9509T[J].Eur J Lipid Sci Tech,2010,112(6):674-680.
[13]HAUSSLER S,NIMTZ M,DOMKE T,et al.Purification and characterization of a cytotoxic exolipid ofBurkholderiapseudomallei[J].Infect Immun,1998,66(4):1588-1593.
[14]DUBEAU D,DEZIEL E,WOODS D E,et al.Burkholderiathailandensisharbors two identicalrhlgene clusters responsible for the biosynthesis of rhamnolipids[J].BMC Microbiol,2009,9(1):263.
[15]NAYAK A S,VIJAYKUMAR M,KAREGOUDAR T.Characterization of biosurfactant produced byPseudoxanthomonassp.PNK-04 and its application in bioremediation[J].Int Biodeterior Biodegrad,2009,63(1):73-79.
[16]ROONEY A P,PRICE N P,RAY K J,et al.Isolation and characterization of rhamnolipid-producing bacterial strains from a biodiesel facility[J].FEMS Microbiol Lett,2009,295(1):82-87.
[17]LEE M,KIM M K,VANCANNEYT M,et al.Tetragenococcuskoreensissp.nov.,a novel rhamnolipid-producing bacterium[J].Int J Syst Evol Microbiol,2005,55(4):1409-1413.
[18]CHRISTOVA N,TULEVA B,LALCHEV Z,et al.Rhamnolipid biosurfactants produced byRenibacteriumsalmoninarum27BN during growth onn-hexadecane[J].Z Naturforsch C,2004,59(1-2):70-74.
[19]CHRISTOVA N,TULEVA B,NIKOLOVA-DAMYANOVA B.Enhanced hydrocarbon biodegradation by a newly isolatedBacillussubtilisstrain[J].Z Naturforsch C,2004,59(3-4):205-208.
[20]CHENG F Y,YANG H,CHEN Y.Characterization of a blend-biosurfactant of glycolipid and lipopeptide produced byBacillussubtilisTU2 isolated from underground oil-extraction wastewater[J].J Microbiol Biotechn,2013,23(3):390-396.
[21]KIRAN S G,THAJUDDIN N,HEMA T,et al.Optimization and characterization of rhamnolipid biosurfactant from sponge associated marine fungiAspergillussp.MSF1[J].Desalin Water Treat,2010,24(1-3):257-265.
[22]OLVERA C,GOLDBERG J B,SANCHEZ R,et al.ThePseudomonasaeruginosaalgCgene product participates in rhamnolipid biosynthesis[J].FEMS Microbiol Lett,1999,179(1):85-90.
[23]RAHIM R,BURROWS L L,MONTEIRO M A,et al.Involvement of the rml locus in core oligosaccharide and O polysaccharide assembly inPseudomonasaeruginosa[J].Microbiology,2000,146(11):2803-2814.
[24]ZHU K,ROCK C O.RhlAConvertsβ-hydroxyacyl-acyl carrier protein intermediates in fatty acid synthesis to theβ-hydroxydecanoyl-β-hydroxydecanoate component of rhamnolipids inPseudomonasaeruginosa[J].J Bacteriol,2008,190(9):3147-3154.
[25]GOULD T A,SCHWEIZER H P,CHURCHILL M E.Structure of thePseudomonasaeruginosaacyl-homoserinelactone synthase LasI[J].Mol Microbiol,2004,53(4):1135-1146.
[26]SCHAEFER A L,VAL D L,HANZELKA B L,et al.Generation of cell-to-cell signals in quorum sensing:Acyl homoserine lactone synthase activity of a purifiedVibriofischeriLuxI protein[J].PNAS,1996,93(18):9505-9509.
[27]ABDEL-MAWGOUD A M,LEPINE F,DEZIEL E.A stereospecific pathway divertsβ-oxidation intermediates to the biosynthesis of rhamnolipid biosurfactants[J].Chem Biol,2014,21(1):156-164.
[28]RAHIM R,OCHSNER U A,OLVERA C,et al.Cloning and functional characterization of thePseudomonasaeruginosarhlCgene that encodes rhamnosyltransferase 2,an enzyme responsible for di-rhamnolipid biosynthesis[J].Mol Microbiol,2001,40(3):708-718.
[29]OCHSNER U A,FIECHTER A,REISER J.Isolation,characterization,and expression inEscherichiacoliof thePseudomonasaeruginosarhlABgenes encoding a rhamnosyltransferase involved in rhamnolipid biosurfactant synthesis[J].J Biol Chem,1994,269(31):19787-19795.
[30]STOVER C,PHAM X,ERWIN A,et al.Complete genome sequence ofPseudomonasaeruginosaPAO1,an opportunistic pathogen[J].Nature,2000,406(6799):959-964.
[31]WILLIAMS P,CAMARA M.Quorum sensing and environmental adaptation inPseudomonasaeruginosa:A tale of regulatory networks and multifunctional signal molecules[J].Curr Opin Microbiol,2009,12(2):182-191.
[32]PESCI E C,PEARSON J P,SEED P C,et al.Regulation of las and rhl quorum sensing inPseudomonasaeruginosa[J].J Bacteriol,1997,179(10):3127-3132.
[33]RAMPIONI G,SCHUSTER M,GREENBERG E P,et al.Contribution of the RsaL global regulator toPseudomonasaeruginosavirulence and biofilm formation[J].FEMS Microbiol Lett,2009,301(2):210-217.
[34]CARTY N L,LAYLAND N,COLMER-HAMOOD J A,et al.PtxR Modulates the expression of QS-controlled virulence factors in thePseudomonasaeruginosastrain PAO1[J].Mol Microbiol,2006,61(3):782-794.
[35]CRODA-GARCIA G,GROSSO-BECERRA V,GONZALEZ-VALDEZ A,et al.Transcriptional regulation ofPseudomonasaeruginosarhlR:Role of the CRP orthologue Vfr (virulence factor regulator) and quorum-sensing regulators LasR and RhlR[J].Microbiology,2011,157(9):2545-2555.
[36]XIAO G,HE J,RAHME L G.Mutation analysis of thePseudomonasaeruginosamvfRandpqsABCDEgene promoters demonstrates complex quorum-sensing circuitry[J].Microbiology,2006,152(6):1679-1686.
[37]LIANG H,DENG X,JI Q,et al.ThePseudomonasaeruginosaglobal regulator VqsR directly inhibits QscR to control quorum-sensing and virulence gene expression[J].J Bacteriol,2012,194(12):3098-3108.
[38]DONG Y H,ZHANG X F,XU J L,et al.VqsM,a novel AraC-type global regulator of quorum-sensing signalling and virulence inPseudomonasaeruginosa[J].Mol Microbiol,2005,58(2):552-564.
[39]POTVIN E,SANSCHAGRIN F,LEVESQUE R C.Sigma factors inPseudomonasaeruginosa[J].FEMS Microbiol Rev,2008,32(1):38-55.
[40]LEQUETTE Y,LEE J H,LEDGHAM F,et al.A distinct QscR regulon in thePseudomonasaeruginosaquorum-sensing circuit[J].J Bacteriol,2006,188(9):3365-3370.
[41]PESSI G,WILLIAMS F,HINDLE Z,et al.The global posttranscriptional regulator RsmA modulates production of virulence determinants andN-acylhomoserine lactones inPseudomonasaeruginosa[J].J Bacteriol,2001,183(22):6676-6683.
[42]HEURLIER K,WILLIAMS F,HEEB S,et al.Positive control of swarming,rhamnolipid synthesis,and lipase production by the posttranscriptional RsmA/RsmZ system inPseudomonasaeruginosaPAO1[J].J Bacteriol,2004,186(10):2936-2945.
[43]JUDE F,KOEHLER T,BRANNY P,et al.Posttranscriptional control of quorum-sensing-dependent virulence genes by DksA inPseudomonasaeruginosa[J].J Bacteriol,2003,185(12):3558-3566.
[44]AGUIRRE-RAMIREZ M,MEDINA G,GONZALEZ-VALDEZ A,et al.ThePseudomonasaeruginosarmlBDACoperon,encoding dTDP-l-rhamnose biosynthetic enzymes,is regulated by the quorum-sensing transcriptional regulator RhlR and the alternative sigma factorσS[J].Microbiology,2012,158(Pt 4):908-916.
[45]REIS R S,da ROCHA S L G,CHAPEAUROUGE D A,et al.Effects of carbon and nitrogen sources on the proteome ofPseudomonasaeruginosaPA1 during rhamnolipid production[J].Process Biochem,2010,45(9):1504-1510.
[46]DEZIEL E,PAQUETTE G,VILLEMUR R,et al.Biosurfactant production by a soil pseudomonas strain growing on polycyclic aromatic hydrocarbons[J].Appl Environ Microbiol,1996,62(6):1908-1912.
[47]CHOI M H,XU J,GUTIERREZ M,et al.Metabolic relationship between polyhydroxyalkanoic acid and rhamnolipid synthesis inPseudomonasaeruginosa:Comparative13CNMR analysis of the products in wild-type and mutants[J].J Biotechnol,2011,151(1):30-42.
[48]PANSIRIPAT S,PORNSUNTHORNTAWEE O,RUJIRAVANIT R,et al.Biosurfactant production byPseudomonasaeruginosaSP4 using sequencing batch reactors:Effect of oil-to-glucose ratio[J].Biochem Eng J,2010,49(2):185-191.
[49]ARINO S,MARCHAL R,VANDECASTEELE J P.Identification and production of a rhamnolipidic biosurfactant by aPseudomonasspecies[J].Appl Microbiol Biotechnol,1996,45(1-2):162-168.
[50]LEE K M,HWANG S H,HA S D,et al.Rhamnolipid production in batch and fed-batch fermentation usingPseudomonasaeruginosaBYK-2 KCTC 18012P[J].Biotechnol Bioproc E,2004,9(4):267-273.
[51]GUERRA-SANTOS L H,KAPPELI O,FIECHTER A.Dependence ofPseudomonasaeruginosacontinous culture biosurfactant production on nutritional and environmental factors[J].Appl Microbiol Biotechnol,1986,24(6):443-448.
[52]ABALOS A,MAXIMO F,MANRESA M A,et al.Utilization of response surface methodology to optimize the culture media for the production of rhamnolipids byPseudomonasaeruginosaAT10[J].J Chem Technol Biotechnol,2002,77(7):777-784.
[53]HABA E,ESPUNY M,BUSQUETS M,et al.Screening and production of rhamnolipids byPseudomonasaeruginosa47T2 NCIB 40044 from waste frying oils[J].J Appl Microbiol,2000,88(3):379-387.
[54]GEORGE S,JAYACHANDRAN K.Production and characterization of rhamnolipid biosurfactant from waste frying coconut oil using a novelPseudomonasaeruginosaD[J].J Appl Microbiol,2013,114(2):373-383.
[55]ABALOS A,PINAZO A,INFANTE M,et al.Physicochemical and antimicrobial properties of new rhamnolipids produced byPseudomonasaeruginosaAT10 from soybean oil refinery wastes[J].Langmuir,2001,17(5):1367-1371.
[56]MULLER M M,HAUSMANN R.Regulatory and metabolic network of rhamnolipid biosynthesis:Traditional and advanced engineering towards biotechnological production[J].Appl Microbiol Biotechnol,2011,91(2):251-264.
[57] GIANI C,MEIWES J,ROTHERT R,et al.Pseudomonasaeruginosaand its use in a process for the biotechnological preparation of L-rhamnose:US,5501966[P].1996-03-26.
[58] CHEN S Y,WEI Y H,CHANG J S.Repeated pH-stat fed-batch fermentation for rhamnolipid production with indigenousPseudomonasaeruginosaS2[J].Appl Microbiol Biotechnol,2007,76(1):67-74.
[59]ZHU L,YANG X,XUE C,et al.Enhanced rhamnolipids production byPseudomonasaeruginosabased on a pH stage-controlled fed-batch fermentation process[J].Bioresour Technol,2012,117:208-213.
[60]SARACHAT T,PORNSUNTHORNTAWEE O,CHAVADEJ S,et al.Purification and concentration of a rhamnolipid biosurfactant produced byPseudomonasaeruginosaSP4 using foam fractionation[J].Bioresour Technol,2010,101(1):324-330.
[61]CAMILIOS-NETO D,BUGAY C,de SANTANA-FILHO A P,et al.Production of rhamnolipids in solid-state cultivation using a mixture of sugarcane bagasse and corn bran supplemented with glycerol and soybean oil[J].Appl Microbiol Biotechnol,2011,89(5):1395-1403.
[62]OCHSNER U A,REISER J,FIECHTER A,et al.Production ofPseudomonasaeruginosarhamnolipid biosurfactants in heterologous hosts[J].Appl Environ Microb,1995,61(9):3503-3506.
[63]WANG Q,FANG X,BAI B,et al.Engineering bacteria for production of rhamnolipid as an agent for enhanced oil recovery[J].Biotechnol Bioeng,2007,98(4):842-853.
[64]CABRERA-VALLADARES N,RICHARDSON A P,OLVERA C,et al.Monorhamnolipids and 3-(3-hydroxyalkanoyloxy) alkanoic acids(HAAs) production usingEscherichiacolias a heterologous host[J].Appl Microbiol Biotechnol,2006,73(1):187-194.
[65]CAO L,WANG Q,ZHANG J,et al.Construction of a stable genetically engineered rhamnolipid-producing microorganism for remediation of pyrene-contaminated soil[J].World J Microb Biot,2012,28(9):2783-2790.
[66]WITTGENS A,TISO T,ARNDT T T,et al.Growth independent rhamnolipid production from glucose using the non-pathogenicPseudomonasputidaKT2440[J].Micro Cell Fact,2011,10(1):80.
[67]TAVARES L F,SILVA P M,JUNQUEIRA M,et al.Characterization of rhamnolipids produced by wild-type and engineeredBurkholderiakururiensis[J].Appl Microbiol Biot,2013,97(5):1909-1921.
Research Progress in Biosynthesis of Rhamnolipid Biosurfactant
DU Jin,HAO Jian-an,ZHANG Xiao-qing,WANG Jing,ZHANG Yu-shan
(TheInstituteofSeawaterDesalinationandMultipurposeUtilization,
StateOceanicAdministration,Tianjin300192,China)
Abstract:Rhamnolipid is an important biosurfactant with the advantages of biodegradability and high surface activities,and it has broad application potentials.Rhamnolipid is produced mainly by Pseudomonassp..The recent progress in screening and isolation of rhamnolipid-producing strains,relationship between synthetic pathway and metabolic regulation,optimization and control of fermentation process,construction of metabolic engineering strains with high-yield rhamnolipid-producing were reviewed.And the research trends were prospected.
Keywords:rhamnolipid;biosurfactant;Pseudomonassp.;microbial fermentation;metabolic engineering
