犬源伪狂犬病毒的分离与鉴定
2015-12-30靳红亮李志君赵敬慧王述超张守峰扈荣良
靳红亮,刘 晔,李 楠,李志君,3,赵敬慧,王述超,张守峰,扈荣良
(1.吉林农业大学动物科学技术学院,长春130118;2.军事医学科学院军事兽医研究所,长春130122;3.吉林大学公共卫生学院,长春 130021)
·研究论文·
犬源伪狂犬病毒的分离与鉴定
靳红亮1,2,刘 晔2,李 楠2,李志君2,3,赵敬慧2,王述超2,张守峰2,扈荣良2
(1.吉林农业大学动物科学技术学院,长春130118;2.军事医学科学院军事兽医研究所,长春130122;3.吉林大学公共卫生学院,长春 130021)
2012与2013年冬季,本实验室从长春市某宠物诊所连续收到疑似伪狂犬病毒感染的2份犬脑组织病料,接种Vero细胞,分离到2株病毒,经电镜观察、动物回归试验及PCR扩增鉴定为伪狂犬病毒,命名为JLCC-2012和JLCC-2013株。PCR扩增病毒gC基因序列,经遗传进化分析显示,2株PRV gC基因同源性为100%,且与国内犬源分离株BJ/RD、BJ/YT同源性分别为99.93%、100%,与猪源病毒分离株同源性为94.33%~100%,与国外猪源病毒分离株同源性为93.58%~94.81%。结果表明本研究分离株为与最近报道的犬源毒株系相近的流行毒株。
伪狂犬病毒;分离;鉴定;感染;基因分析;犬
伪狂犬病毒(Pseudorabies virus,PRV)属于I型疱疹病毒,为疱疹病毒科甲亚科家族中的一员,感染猪后可引起一种以脑脊髓炎为主要临床症状的急性高度传染性传染病[1]。除了猪以外,PRV可感染多种其他哺乳动物,包括反刍动物、食肉动物和啮齿类动物等[2,3,5]。犬通过摄食未经煮熟的猪骨头和内脏也可感染。犬感染的潜伏期较短,一般1~6 d,多在发病出现临床症状后几天内死亡,死亡率100%[4]。目前猪群通过接种减毒活疫苗或灭活苗,患病率已大大降低[6-8]。但由于多种原因,尤其是因漏免导致的潜伏感染,常有带毒猪产品进入市场,犬在食用这些带毒猪产品后,常有伪狂犬病发生。
PRV基因组为线性DNA,长约150 kb,由长独特区(UL)、短独特区(US)以及US两侧的末端重复序列(TR)和内部重复序列(IR)构成,可编码70~100种蛋白[1]。其中gC蛋白与PRV的毒力有关,也是病毒复制的非必需区[9]。gC蛋白通过和细胞表面的肝素样受体相互作用,在PRV攻击靶细胞中起着非常重要的作用[10,11]。另外,gC蛋白也是PRV主要保护性抗原之一,可以刺激机体产生保护性抗体[10,12]。
本研究从具有神经症状的病犬脑组织中分离和鉴定了2株伪狂犬病毒,对其gC基因进行了扩增、测序及比对分析,旨在阐明其分子流行病学特点,为该病的预防提供依据。
1 材料和方法
1.1 病料和细胞本研究所用病料分别于2012和2013年冬季采自长春市某宠物诊所送检的病犬。按照送检人的叙述,病犬表现为瘙痒症状,用爪极力抓挠头部,后期采食能力丧失,最终死亡,据此初步诊断为伪狂犬病。采集脑组织后置-20℃保存备用。Vero细胞由军事兽医研究所流行病学实验室保存,细胞在含有5%胎牛血清(FBS)的DMEM培养基(购自Gibco公司)中培养。
1.2 引物根据参考文献[13]已发表的PRV gC基因序列(GenBank登录号:KC981239)设计其全长扩增引物,扩增产物大小为1600 bp,引物由苏州金唯智生物科技有限公司合成。其序列如下:
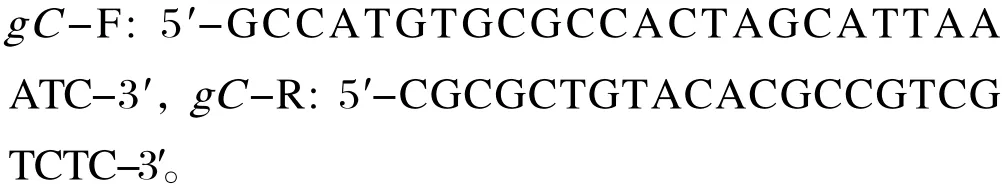
1.3 病毒分离无菌条件取病料剪碎后,加入适量的生理盐水和细胞培养基,用组织研磨棒研碎,制成悬液,反复冻融3次,10 800×g 离心 15 min,取上清加入双抗,4℃过夜;用 0.22 μm一次性滤器过滤除菌,取500 μL病毒悬液接种至80%单层Vero细胞,37℃感作1 h;加入含2%胎牛血清的DMEM培养基,继续培养;每天观察细胞病变(cytopathic effect,CPE),出现CPE后,用2%磷钨酸负染,透射电镜下观察。
1.4 病毒TCID50测定使用Vero细胞在96孔细胞培养板内测定,并按Käber法计算分离毒TCID50[14]。
1.5 本动物回归试验3月龄健康犬9只均分为3组。第1组接种JLCC-2012株病毒细胞培养物(107.5/0.1mL TCID50),3 mL/只,肌肉注射和滴鼻途径各1.5 mL;第2组接种JLCC-2013株病毒细胞培养物(107.75/0.1 mL TCID50),3 mL/只,肌肉注射和滴鼻途径各1.5 mL;第3组注射相同剂量的无菌DMEM培养基作对照。相同条件分笼饲养,每天观察其发病死亡情况。
1.6 病毒基因组DNA提取及gC基因扩增及测序取90%以上细胞病变的培养物,反复冻融3次。取200 μL置于DEPC水处理过的1.5 mL灭菌离心管中,使用病毒DNA小量试剂盒(Axygen)提取病毒DNA。取病毒基因组DNA进行gC基因扩增时,PCR反应体系为50μL,反应管依次加入灭菌ddH2O 28 μL、5×GC buffer 10 μL、DMSO 1.5 μL、10 mmol/ mL dNTP 1 μL、上下游引物各2.5 μL、模板DNA 4 μL,最后加入Phusion DNA polymerase 0.5μL。gC基因扩增条件:98℃预变性30 s;98℃变性10 s,58℃退火1 min,72℃延伸1 min 30 s,35个循环;最后72℃延伸10 min。扩增产物用1%TAE琼脂糖凝胶电泳,回收扩增产物送吉林省库美生物科技有限公司测序。
1.7 PRV gC基因序列分析采用DNAStar软件对测序结果进行序列比对,将其核苷酸序列及其推导的氨基酸序列与GenBank中其他PRV毒株的gC基因进行同源性分析比较,并基于gC基因序列用MEGA5.2分析软件以Neighbor-joining法绘制遗传进化树进行遗传进化分析。
2 结果
2.1 病毒分离、电镜观察和PCR检测病料滤液接种Vero细胞24 h后开始出现细胞圆缩、聚集,48 h后细胞开始慢慢脱落。取冻融细胞培养物上清用2%磷钨酸进行负染,透射电镜下观察,可见典型呈圆形、带囊膜的成熟病毒粒子。结果初步表明所分离的2株病毒均为伪狂犬病毒,将其命名为JLCC-2012和JLCC-2013株(图1)。
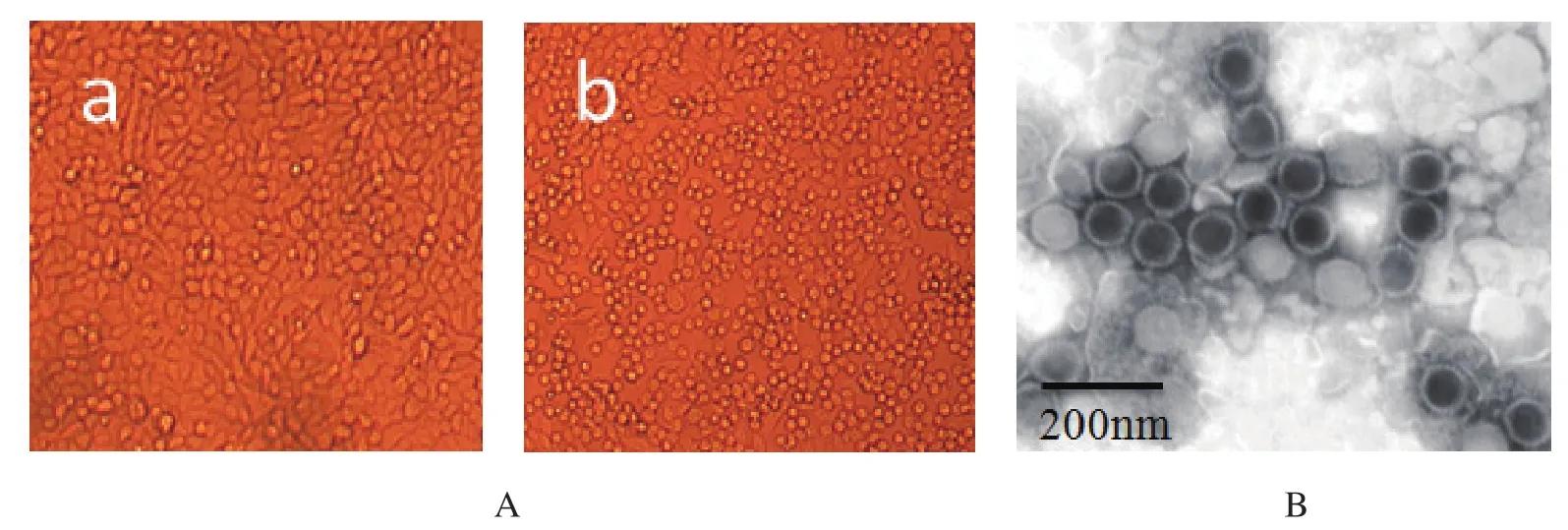
图1 犬病料的诊断结果Fig.1 Diagnosis results of brain samples from the diseased dogs
2.2 病毒TCID50测定取分离株连续培养至第5代的病毒,在Vero细胞上测定TCID50。按Käber法计算,JLCC-2012与JLCC-2013株的TCID50分别为107.5/0.1 mL、107.75/0.1 mL。
2.3 本动物回归试验3月龄健康犬接种JLCC-2012与JLCC-2013株病毒细胞培养物24 h后体温升高,最高可达42℃。48 h后开始出现明显临床症状,表现为精神不振,接种部位出现典型瘙痒症状,频频回头撕咬接种部位及周围组织,用爪极力抓挠头部,后期四肢麻痹,卧地不起,最终衰竭而死。结果表明JLCC-2012与JLCC-2013株对3月龄犬具有致病性,并且表现出典型的伪狂犬病神经症状。
2.4 PRV gC基因的扩增利用PCR从病毒基因组DNA中扩增gC基因,经1%琼脂糖凝胶电泳后,JLCC-2012与JL-CC2013均扩增出约为1.6 kb大小的特异性条带(图2),与预期相符,并经序列分析验证为伪狂犬病毒的gC基因序列,由此,进一步证实2株分离株均为伪狂犬病毒。
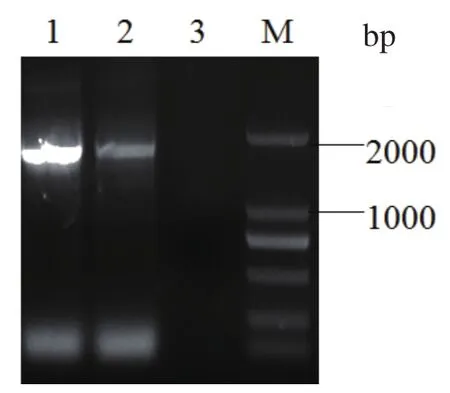
图2 JLCC-2012株与JLCC-2013株 gC基因PCR产物Fig.2 PCR product of gC gene of JLCC-2012 and JLCC-2013
2.5 PRV gC基因序列分析JLCC-2012和JLCC-2013 株gC基因均包含一个大小为1464 bp完整的开放性阅读框(open reading frame,ORF),编码487个氨基酸。用Clustalw软件基于gC基因核苷酸序列绘制遗传进化树,结果显示,JLCC-2012和JLCC-2013株同源性为100%,与国内犬源分离株BJ/RD、BJ/YT同源性分别为99.93%、100%;与猪源伪狂犬病毒分离株同源性为94.33%~100%;与国外猪源性狂犬病毒分离株的核苷酸序列及推导的氨基酸序列变异率较高,同源性为93.58~94.81%(图3)。
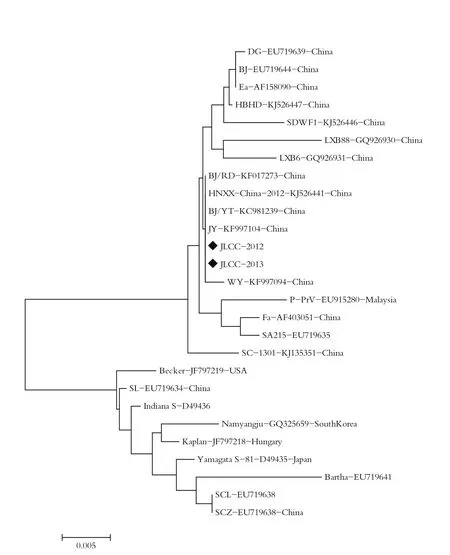
图3 伪狂犬病毒 gC基因遗传进化树Fig.3 Phylogenetic analysis based on gC gene of Pseudorabies virus
3 讨论
近年来,尽管伪狂犬病减毒活疫苗和灭活疫苗的应用在预防和控制猪群感染发病取得了很好的效果,一些地区仍不断有该病暴发的报道。大量研究证实,疫苗株和强毒力株在猪群中发展为潜伏感染,在一定条件下通过某些途径传播感染其他哺乳动物(犬、猫、牛、羊等)和野生动物(狼、狐狸、臭鼬等)[15-20]。犬主要是通过摄食未经煮熟猪的骨头和内脏而感染,一旦发病后几天内死亡。长春市某宠物诊所每年都收到一些具有神经症状的犬病例,其中一些诊断为犬瘟热,个别为狂犬病,另有一些怀疑为伪狂犬病,但由于诊断条件有限,很少获得确定性证据。本实验室从宠物诊所收到的具有神经症状的病犬脑组织病料,经Vero细胞接种、电镜观察、动物回归试验、PCR特异性检测确诊病犬死于PRV感染。
本研究从疑似伪狂犬病的2份犬脑组织病料中分离鉴定了2株伪狂犬病毒,我们曾对PRV的TK、gE、gC和gD共4个基因进行了扩增。由于有关 I 型疱疹病毒gE和TK基因遗传多样性数据匮乏,我们选择了 I 型疱疹病毒基因组中研究最多的gC蛋白基因,gC基因已广泛用于PRV毒株的遗传演化和分子流行病学分析[21,22]。本研究所采病料来自吉林省长春市,该地区未曾有PRV gC基因的分析报道。本研究基于gC基因核苷酸序列绘制遗传进化树,分析结果显示,JLCC-2012与JLCC-2013株同源性为100%,与国内犬源分离株BJ/RD、BJ/YT间同源性分别为99.93%、100%,与猪源伪狂犬病毒分离株同源性为94.33%~100%,与国外猪源性狂犬病毒分离株的核苷酸序列及推导的氨基酸序列变异率较高,同源性为93.58~94.81%,可以初步推测犬伪狂犬病各毒株间变异较小,对该病的预防提供了一定理论基础。
TK基因缺失株PRV弱毒对犬的安全性未见报道,潜伏感染PRV对犬的致病性也不清楚。本研究的2株分离株TK基因ORF完整,因此可排除疫苗株引起感染的可能性。2株分离株gE和gC基因均变异较小,由于PRV毒力与很多因素有关,这些变异是否与其毒力有关仍有待于进一步研究。
目前尚无犬用的伪狂犬病疫苗。由于PRV在猪体内具有潜伏感染特性,我们通过本研究分析认为,防止犬伪狂犬病发生的措施主要是避免犬生吃猪产品尤其是病猪肉或骨头,从长远来看,尤其是对那些大型养犬场,如不能避免摄食生肉或骨头,建议试用安全有效的伪狂犬病活疫苗或灭活疫苗。
[1] Mettenleiter T C.Molecular biology of pseudorabies (Aujeszky's disease) virus[J].Comp Immunol Microbiol Infect Dis, 1991, 14(2)∶ 151-163.
[2] Pomeranz L E, Reynolds A E, Hengartner C J.Molecular biology of pseudorabies virus∶ impact on neurovirology and veterinary medicine[J].Microbiol Mol Biol Rev, 2005, 69(3)∶ 462-500.
[3] Mettenleiter T C, Keil G M, Fuchs W.Molecular biology of animal herpesviruses[M]//Mettenleiter T C, Sobrino F.Animal viruses∶ molecular biology, Caister Academic Press, 2008∶ 375-455.
[4] Egberink H F.Aujeszky's disease in dogs and cats[J].Tijdschr Diergeneesk, 1990, 115(8)∶ 349-353.
[5] Fonseca Jr A A, Camargos M F, de Oliveira A M, et al.Molecular epidemiology of Brazilian pseudorabies viral isolates[J].Vet Microbiol, 2010, 141(3)∶ 238-245.
[6] Van Rooij E M A, Moonen-Leusen H W, De Visser Y E, et al.A DNA vaccine coding for gB and gD of pseudorabies virus (suid herpes type 1) primes the immune system in the presence of maternal immunity more efficiently than conventional vaccines[J].Vaccine, 2006, 24(9)∶ 1264-1273.
[7] Mettenleiter T C, Klupp B G, Weiland F, et al.Characterization of a quadruple glycoprotein-deleted pseudorabies virus mutant for use as a biologically safe live virus vaccine[J].J Gen Virol, 1994, 75(Pt7)∶ 1723-1733.
[8] Vannier P.Experimental infection of fattening pigs with pseudorabies (Aujeszky's disease) virus∶ efficacy of attenuated live-and inactivated-virus vaccines in pigs with or without passive immunity[J].Am J Vet Res, 1985, 46(7)∶ 1498-1502.
[9] Rue C A, Ryan P.Pseudorabies virus glycoprotein C attachment-proficient revertants isolated hrough a simple,targeted mutagenesis scheme[J].J Virol Methods, 2008, 151(1)∶ 101-106.
[10] Hampl H, Ben-Porat T, Ehrlicher L, et al.Characterization of the envelope proteins of pseudorabies virus[J].J Virol, 1984, 52(2)∶ 583-590.
[11] Pensaert M, Gielkens A L J, Lomniczi B, et al.Round table on control of Aujeszky's disease and vaccine development based on molecular biology[J].Vet Microbiol, 1992, 33(1)∶ 53-67.
[12] Wathen M W, Wathen L M.Characterization and mapping of a nonessential pseudorabies virus glycoprotein[J].J Virol, 1986, 58(1)∶ 173-178.
[13] Pérez L J, Arce H D.Development of a polymerase chain reaction assay for the detection of pseudorabies virus in clinical samples[J].Braz J Microbiol, 2009, 40(3)∶ 433-438.
[14] 殷震, 刘景华.动物病毒学[M].北京∶ 科学出版社, 1997∶329-331.
[15] Lipowski A.Evaluation of efficacy and safety of Aujeszky's disease vaccines[J].Pol J Vet Sci, 2005, 9(1)∶75-79.
[16] Wittmann G.Spread and control of Aujeszky's disease (AD)[J].Comp Immunol Microb, 1991, 14(2)∶ 165-173.
[17] Puentes E, Eiras A, Cancio E, et al.Comparison of the protective efficacy of Aujeszky's disease (pseudorabies) virus glycoproteins obtained from different sources[J].Vet Microbiol, 1993, 35(1)∶ 45-59.
[18] Kong H, Zhang K, Liu Y, et al.Attenuated live vaccine (Bartha-K16) caused pseudorabies (Aujeszky’s disease) in sheep[J].Vet Res Commun, 2013, 37(4)∶ 329-332.
[19] Verpoest S, Cay A B, Bertrand O, et al.Isolation and characterization of pseudorabies virus from a wolf (Canis lupus) from Belgium[J].Eur J Wildlife Res, 2014, 60(1)∶149-153.
[20] Mettenleiter T C.Aujeszky's disease (pseudorabies) virus∶the virus and molecular pathogenesis-state of the art, June 1999[J].Vet Res, 2000, 31(1)∶ 99-115.
[21] Ishikawa K, Tsutsui M, Taguchi K, et al.Sequence variation of the gC gene among pseudorabies virus strains[J].Vet Microbiol, 1996, 49(3)∶ 267-272.
[22] Goldberg T L, Weigel R M, Hahn E C, et al.Comparative utility of restriction fragment length polymorphism analysis and gene sequencing to the molecular epidemiological investigation of a viral outbreak[J].Epidemiol Infect, 2001, 126(3)∶ 415-424.
ISOLATION AND IDENTIFICATION OF PSEUDORABIES VIRUS FROM DOGS
JIN Hong-liang1,2, LIU Ye2, LI Nan2, LI Zhi-jun2,3, ZHAO Jing-hui2, WANG Shu-chao2, ZHANG Shou-feng2, HU Rong-liang2
(1.College of Animal Science and Technology, Jilin Agricultural University, Changchun 130118, China; 2.Military Veterinary Research Institute, Science, Academy of Military Medical Sciences, Changchun 130122, China; 3.School of Public Health, Jilin University, Changchun 130021, China)
In the winter of 2012 and 2013, some canine cases were initially diagnosed as Pseudorabies virus(PRV) infection according to their clinical signs at a pet animal hospital in Changchun.The brain tissues of the diseased dogs were submitted to our laboratory for confi rmation.The examination under electron microscope, animal experiment and PCR on the brain tissues confi rmed positive results.Two virus strains were obtained and designated as JLCC-2012 and JLCC-2013.Their gC gene fragments were amplifi ed using respective primers.Sequence analysis of gC gene showed 100% homology between JLCC-2012 and JLCC-2013.Further phylogenetic analysis of nucleotide sequence of gC gene revealed that these two strains had identity at 99.93% and 100% with recently isolated canine BJ/RD and BJ/YT strains, 94.33%-100% with swine PRV strains kept in our laboratory and 93.58%-94.81% with foreign swine PRV strains published in the literature.The results confi rmed that those canine cases were infected with PRV.
Pseudorabies virus; isolation; identifi cation; infection; phylogenetic analysis; dogs
S852.659.1
:A
:1674-6422(2015)01-0009-06
2014-08-27
863计划(2011AA10A212)
靳红亮,男,硕士研究生,预防兽医学专业
扈荣良,E-mail:ronglianghu@hotmail.com
