生活史性状和取样策略对植物微卫星遗传多样性参数估算的影响
2015-12-22杜夏瑾黎啸峰
杜夏瑾,黎啸峰,吴 敏,王 丽
(中南大学生命科学学院,中国长沙 410013)
遗传变异是物种长期存在和进化的物质基础.因此,遗传多样性的度量和描述是种群和进化遗传学研究、生物多样性保护和管理中的中心问题.Hamrick,Godt,Nybom,Bartish,Nybom 和Duminil 等[1-4]的研究表明,植物遗传多样性水平及其遗传结构与物种本身的繁育系统、种子传播方式和分布范围等生活史性状关系密切.合理的取样策略在遗传多样性研究、生物多样性保护和利用中具有重要的意义.取样理论近几十年来被广泛研究[5-8],随着更多的遗传标记的出现,该理论需要不断更新,因为不同的遗传标记和方法所得出的结果有差异,如何在物种保护中合理取样和有效地分析遗传数据也有待完善.
微卫星(microsatellites)亦称简单重复序列(simples sequence repeats,SSRs),由于其共显性、重复性好及高变异性等优点,已在植物群体遗传学研究、品种鉴定、连锁图谱构建、系统发育和分子标记辅助育种等方面得到了广泛的应用.近二十年来,大量群体遗传学研究借助于微卫星分子标记技术展开,但其种群遗传变异的分析主要是估测一系列有代表性的参数,如观测杂合度(Observed heterozygosity,HO),期望杂合度(Expected heterozygosity,HE),NA(Observed number of alleles,观测等位基因数),NE(Effective number of alleles,有效等位基因数),FIS(Breeding coefficient,近交系数)和FST(Genetic differentiation index,遗传分化指数)等.
为了探讨植物生活史性状和取样策略对微卫星遗传参数估算的影响,本文对139 篇1999—2012年所报道的150 种野生种子植物基于微卫星分子标记的遗传多样性参数进行了统计分析,以期为种子植物遗传多样性的相关研究提供有价值的参考.
1 材料与方法
通过Web of science 和Google scholar 检索关于种子植物遗传多样性的文献,整理近十年发表在国外知名期刊上有关SSR 标记的野生种群遗传多样性的研究,得到各项取样策略参数和遗传多样性参数,主要有取样种群数、取样个体数、最大取样距离、SSR 标记数、观测杂合度(HO)、期望杂合度(HE)、观测等位基因数(NA)和遗传分化系数(FST与RST).由于检测方法(银染和测序)对多态性高低有直接的影响,因此检测方法也作为一种因素用于分析.
数据整理原则:(1)限于种子植物,即裸子植物、双子叶植物和单子叶植物,不统计蕨类、藻类及真菌等;(2)限于野生植物种群,不统计栽培品种;(3)限于研究的野生种群数大于或等于2 的文献.另外,如果一篇文献中对两种或两种以上的植物进行了研究,分析不同的植物时作为独立的文献进行统计.
本文将植物生活史特性分为5 个方面:分类地位、繁育系统、生活型、分布范围和种子传播.分类地位包括裸子植物、双子叶植物和单子叶植物.繁育系统包括自交、混交和异交.生活型包括一年生植物、短寿多年生植物(多年生草本植物)和长寿多年生植物(多年生乔灌木和藤本植物).分布范围包括特有种、窄域种、地方种和广布种.种子传播包括重力传播、附着传播、吞食传播和风/水传播.
利用SPSS 19.0(IBM Company,2010)计算各项参数的平均值和标准差,对各取样策略参数之间、遗传多样性参数之间以及取样策略与遗传多样性参数之间进行了Pearson 相关分析,为了进一步探讨取样策略参数和遗传参数之间的关系以及不同生活史性状内(主要是繁育系统和分类地位)二者之间的关系,作者以4 个取样策略参数作为自变量,4 个遗传多样性参数作为因变量进行了回归分析.为了进行不同生活史类型植物间遗传参数的比较,还将不同生活史变量与4 种遗传参数分别进行了单因素方差分析(One-way ANOVA).
2 结果
作者收集了330 篇关于种子植物遗传多样性的文献,统计得到341 种植物的遗传多样性参数值,剔除数据不全的文献和物种,最终用于分析的物种有150 种.其中裸子植物24 种,双子叶植物95 种,单子叶植物31种;遗传多样性参数个数不少于3 个的有110 种.
2.1 参数统计
150 种植物的平均取样种群数为11(变化范围2~42,SD=7.77),每个种群的平均取样个体数为31(变化范围4~144,SD=23.58),取样种群间的平均最大取样距离为716.06 km(变化范围0.01~3 333.33,SD=830.27),SSR 标记数为9.37(变化范围2~25,SD=4.86)(表1).Pearson 相关性分析表明,平均取样种群数与每个种群的平均取样个体数之间有极显著的负相关性(r=-0.250,df=149,P <0.01),与平均最大取样距离之间具有极显著的正相关性(r=0.296,df=149,P <0.001),与标记数之间有一定的负相关关系,但相关性不显著(r=-0.153,df=149,P=0.062);每个种群的平均取样个体数与平均最大取样距离之间具有显著的负相关性(r=-0.174,df=149,P <0.05),与标记数之间也有一定的负相关关系,相关性不显著(r=-0.156,df=149,P=0.057);平均最大取样距离与标记数无相关性(r=0.043,df=149,P=0.598).
统计所有的150 种植物,得到105 个FST,平均值为0.220,标准差为0.200;37 个RST,平均值为0.227,标准差为0.205(表1).统计28 种同时采用了FST和RST这两种种群分化指数的植物,成对样本的t 检验(Paired-Samples T Test)结果表明二者之间没有显著性差异(平均值分别为FST=0.238,RST=0.256,P >0.05,t=-2.096,df=27).统计得到114 个HO,平均值为0.450,标准差为0.212;得到119 个HE,平均值为0.545,标准差为0.197;得到93 个NA,平均值为6.099,标准差为3.916.统计106 种同时采用了HO和HE这两种指数的植物,经成对样本t 检验,二者之间差异极显著(平均值分别为HO=0.456,HE=0.546,P <0.001,t=-7.238,df=105).而且,在统计的所有植物中,有91 种植物遗传多样性分析得到的HE>HO,仅有15 种植物遗传多样性分析得到的HE<HO.Pearson 相关性分析表明,种群分化指数FST与多样性指数HO(r=-0.748,df=77,P <0.001)和HE(r=-0.744,df=80,P <0.001)均呈极显著负相关关系,种群分化指数RST与多样性指数HO(r=-0.735,df=30,P <0.001)和HE(r=-0.703,df=26,P <0.001)也均呈极显著负相关关系.
2.2 取样策略对遗传参数的影响
把4 个取样策略参数作为自变量,4 个遗传多样性参数作为因变量进行回归分析(表2),结果表明,取样种群数和取样个体数对各遗传参数没有影响,种群间多样性指数FST随着SSR 标记数的增加而增加,多样性指数HO和HE随着SSR 标记数增加而减少,多样性指数HO也会随着取样距离的加大而增加,Pearson 相关分析表明取样距离与种群间多样性指数FST和RST均呈正相关(图1).

图1 种群间多样性指数与取样距离的相关关系示意图Fig.1 The relationship between on the one hand logarithmic values for maximum distance between collected populations,and on the other hand between-population diversity estimates,FST(filled symbols)and RST(open symbols)
2.3 检测方法对遗传参数的影响
统计的150 种植物中有58 种植物采用电泳银染技术进行结果检测,有89 种采用测序技术,有3种使用琼脂糖EB 检测方法,由于琼脂糖检测技术用得非常少,在此仅针对前两种方法展开分析.使用测序检测方法得出的HO,HE和NA(平均值分别为0.486,0.570,6.68;标准差分别为0.211,0.199,3.985)均高于电泳银染技术得出的HO,HE和NA(平均值分别为0.431,0.529,5.43;标准差分别为0.185,0.182,3.734),而使用测序检测方法得出的FST和RST(平均值分别为0.201,0.200;标准差分别为0.200,0.163)却低于电泳银染技术得出的FST和RST(平均值分别为0.233,0.221;标准差分别为0.175,0.193).
2.4 不同生活史类型植物间的遗传参数比较
上文(2.2)提到遗传参数HO,HE和FST会随着SSR 标记数的变化而变化,为了进一步探讨各遗传参数在不同繁育系统类型和不同分类地位植物中是否会受SSR 标记数的影响,作者分别进行了种群间多样性指数HO,HE和FST与SSR 标记数的回归分析,结果表明,在不同繁育系统类型植物中除在异交类群中HO会随着SSR 标记数的增加有一定增加外,其他类群各参数都不会受SSR 标记数的影响,双子叶植物的遗传参数受SSR 标记数影响较大,裸子植物和单子叶植物受SSR 标记数影响较小(表2).

表2 取样策略参数与遗传参数回归分析结果Tab.2 Regression analysis with four sampling strategy parameters and genetic parameters
本文所统计的150 种植物,按不同的生活史类型划分得到的遗传参数数目差异较大,例如,附着传播的植物得到的遗传参数数目为0,长寿多年生的植物得到的遗传参数数目远远多于一年生或者短寿多年生的植物(表3).对不同生活史变量与4 种遗传参数分别进行单因素方差分析(One-way ANOVA),结果表明,对于不同的繁育系统和生活型的植物,多样性指数HO和HE差异极为显著(P <0.001),种群间多样性指数FST差异也极为显著(P <0.001),RST在不同生活型的植物间差异也极为显著(P <0.001),但在不同繁育系统的植物间差异显著(P <0.01).组间两两比较(Post Hoc Multiple Comparisons)结果表明,自交植物的HO和HE(平均值分别为HO=0.120,HE=0.303)均明显低于混交(平均值分别为HO=0.410,HE=0.575)或异交(平均值分别为HO=0.503,HE=0.563)植物,但自交植物的遗传分化系数FST和RST(平均值分别为FST=0.520,RST=0.555)均明显高于混交(平均值分别为FST=0.233,RST=0.155)或异交(平均值分别为FST=0.181,RST=0.183)植物.一年生植物(平均值分别为HO=0.012,HE=0.203)的多样性指数HO和HE均明显低于短寿多年生(平均值分别为HO=0.369,HE=0.513)或长寿多年生(平均值分别为HO=0.490,HE=0.580)植物,遗传分化系数FST的平均值为0.590,明显高于多年生植物,由于统计得到的RST数目较少,甚至缺少一年生植物的RST值,因此不予比较.
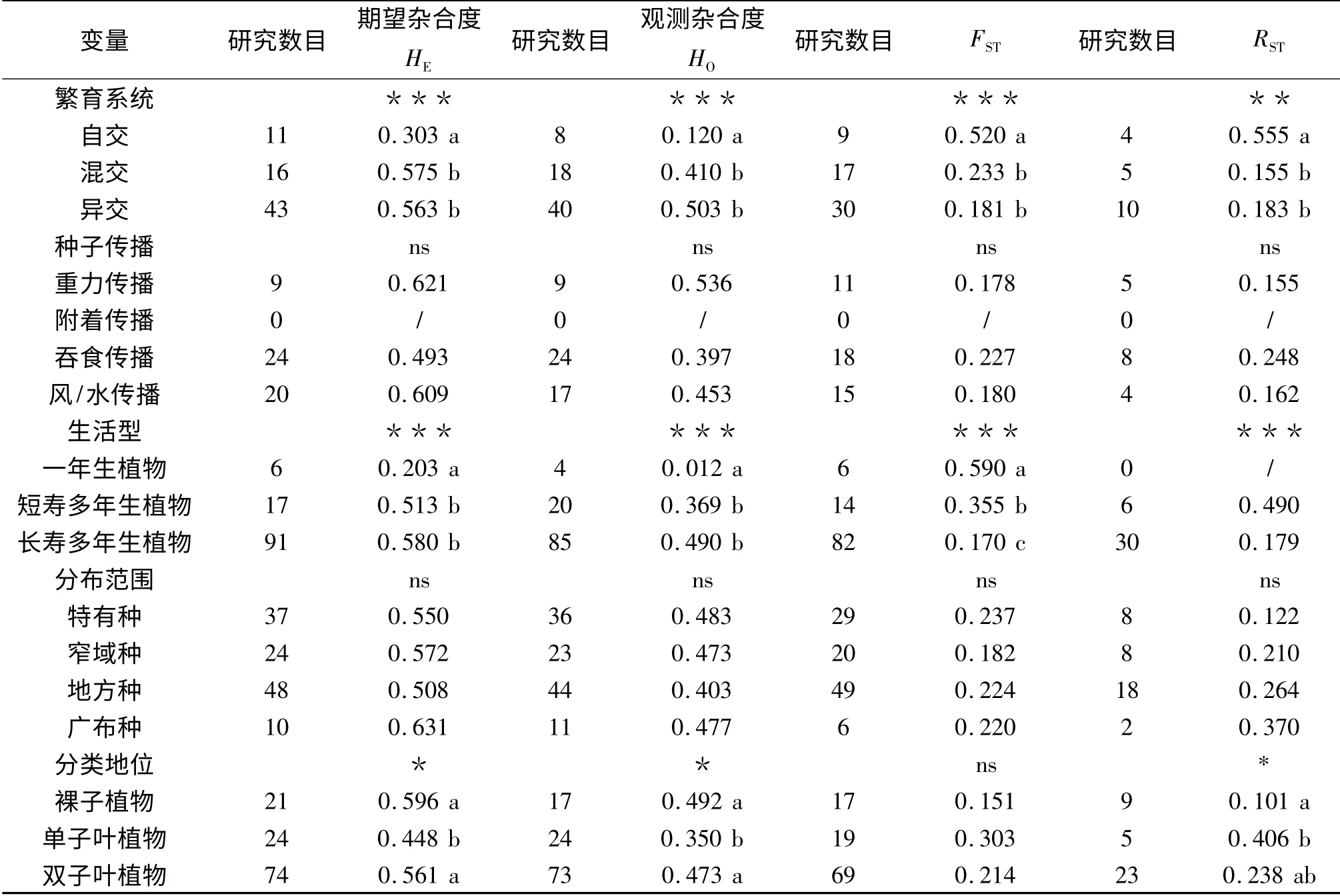
表3 不同生活史组变量与遗传参数的方差分析Tab.3 Analysis of variance conducted with different life history traits as group variable,and with mean genetic parameters HE,NA,FST and RST as dependent variables
单因素方差分析(One-way ANOVA)上述4 个遗传参数结果表明,不同种子传播类型或分布范围的植物均没有显著性差异(P >0.05).但可看出靠重力传播的物种HO和HE高于其他3 种类型的植物;地方种的HO和HE略低于其他3 种类型的植物,广布种的HO和HE最高,说明广布种具有高水平的种群间多样性.
对于不同分类地位的植物,多样性指数HO和HE差异较显著(P <0.05),遗传分化系数RST差异较显著(P <0.05),但遗传分化系数FST之间没有显著性差异(P >0.05).裸子植物(平均值分别为HO=0.492,HE=0.596)和双子叶植物(平均值分别为HO=0.473,HE=0.561)的多样性指数HO和HE均明显地高于单子叶植物(平均值分别为HO=0.350,HE=0.448),从本研究的统计结果看,裸子植物在种群水平上具有比被子植物更高的遗传多样性,但裸子植物和双子叶植物的遗传分化系数RST则低于单子叶植物.
3 讨论
3.1 取样策略
SSR 标记在遗传多样性和遗传分化研究中是一个非常重要的工具,而在SSR 相关研究中取样策略的制定对SSR 研究结果解释的可靠性至关重要[2-3,5].本次研究统计得到的平均取样种群数为11,种群的平均取样个体数为31.本次研究发现统计的150 种植物中,平均取样种群数小于4 的植物仅有16 种,每个种群平均取样个体数小于10 的植物仅有5 种,平均取样个体数大于50 的植物有17 种.取样种群数和取样个体数对本研究选取的4 个遗传多样性参数均没有影响,而SSR 标记数与遗传分化系数FST呈显著正相关,与多样性指数HO和HE呈显著负相关,这可能是由于单个标记的多态性较低时,研究者们都倾向于探讨更多的标记从而增加结果的可靠性.
Nybom 和Bartish[2]的研究表明,取样种群数、取样个体数和RAPD 标记数对各遗传参数(ΦST,GST,Hpop)没有明显影响,但是ΦST、GST值会随着种群间的取样距离的增加而增加,Hpop随着取样距离的增加而减小,进一步的分析发现只在异交植物类群中ΦST、GST值会随着种群间取样距离的增加而增加,自交植物类群中ΦST,GST与取样距离之间无显著相关关系.作者的统计结果表明,遗传参数HO会随着取样距离的增加减少,HE,FST和RST与取样距离之间相关关系虽然不显著,但FST和RST值也是随取样距离的增加而增加的.两种标记得出的结论基本一致.
3.2 遗传参数的估测
本次统计的106 种同时采用了HO和HE这两种多样性指数的植物中,有91 种植物HE>HO,仅有15 种植物HE<HO.这可能是SSR 标记中短链等位基因优势或者无效等位基因的出现造成的[9-10].在研究群体遗传结构时,无限等位基因模型(Infinite Allele Model,IAM)[11-12]是广泛采用的模型,基于这一模型的种群间遗传分化指数一般以FST来表示,它通过等位基因频率计算得到,但它需要种群处于Hardy-Weinberg 平衡时才有意义[13-14].RST则是基于逐步突变模型(Stepwise Mutation Model,SMM)[15]得到的遗传分化指数,因为大多数微卫星座位的突变都是由一小部分重复单元的增加或减少引起的,所以在SSR 遗传多样性分析中SMM模型更贴切[16].一般认为,遗传较近或者地理距离较近的种群遗传分析比较适合选用FST指数;而考虑到距离隔离的影响,距离较远一些的种群遗传分析更适合RST指数[17-19].此外,选择FST或者RST指数,还需要考虑一些取样策略因素,例如取样种群大小、取样个体数目和SSR 标记数目等[20].一些研究表明,对于SSR 标记而言,基于IAM 模型得到的遗传分化值FST经常低于基于SMM 模型得到的遗传分化值RST[16,19].本文中,对28 种同时采用了FST和RST这两种种群遗传分化指数的植物进行统计分析,结果表明二者之间没有显著性差异,但FST平均值略低于RST平均值.
3.3 不同生活史类型植物间的遗传多样性指数比较
本次研究发现,繁育系统、生活型和分类地位与种群遗传参数具有较大关联,而种子传播类型和分布范围对各遗传参数影响不显著.
Hamrick,Godt[1]和Ellstrand,Elam[21]认为植物的繁育系统可作为解释遗传多样性水平的主要因素,在自交种群和其他类型种群之间差异显著.本研究发现,自交植物在种群水平上的遗传多样性水平明显低于其他类型植物,而遗传分化程度则相对较高,这与已有的研究结论一致[1-3,22].
作者还发现,不同生活型的植物各遗传多样性指数差异极为显著.一年生植物的多样性指数HO和HE值均明显低于短寿多年生或长寿多年生植物,且长寿多年生植物HO和HE的平均值最大;长寿多年生植物的遗传分化系数FST和RST均较低.而在Hamrick,Godt[1],Nybom,Bartish[2]及Nybom[3]的统计中,长寿多年生植物的H 参数也都比较高,而遗传分化值比较低.
从本研究的统计结果看,分类地位与种群多样性具有一定关联.总体上,裸子植物的遗传多样性水平高于被子植物,但裸子植物和双子叶植物的HO和HE值差异不明显,均明显地高于单子叶植物.裸子植物种群间遗传分化值(FST和RST)均相对较低,这与Nybom,Bartish[2]和Hamrick,Godt[1]的统计结果一致.因此,裸子植物和双子叶植物较高的种群内遗传多样性可能与其长寿多年生和异交繁殖等生活史特征有关[2,23].
Hamrick,Godt[1]和Karron[24]的研究结果都表明,特有或濒危种的遗传变异水平比广布种低,特别是在种群水平上.但是,也有研究认为,地理分布范围并不是决定物种的遗传多样性水平和遗传结构的重要因素.近年来有不少研究表明,一些特有和濒危物种却保持着较高水平的遗传变异,如太白红杉Larix potaninii var.chinensis,尾叶桉Eucalyptus urophylla 和短叶雪松Cedrus brevifolia 等[25-27].本文中,分布范围对多样性指数影响不显著,这与Nybom,Bartish[2]基于RAPD 标记的统计结果基本一致,而与Hamrick 和Godt[1]基于等位酶与Nybom[3]基于STMS 标记的统计结果不同,Nybom[3]的结果表明不同分布范围HO和HE差异较显著(P <0.05),而FST的差异不显著,鉴于张德全[22]的研究,HO和HE不适合在不同标记之间进行比较,但遗传分化参数在不同标记中的研究结果基本一致,即无论是RAPD 还是SSR,不同分布范围对FST的值影响不是很大.即使如此,本文与已有的研究都表明,广布种一般都表现出较高的遗传多样性.因此,作者认为,广布种在通常情况下具有较特有或濒危种更高的遗传多样性,但这不是绝对的;一些人为因素导致的濒危种,如黄山梅Kirengeshoma palmata,由于其遗传基础较丰富,其野生种群数量和个体数量可能很少,仍然可能维持着较高的遗传多样性[28].
本研究还发现,不同种子传播类型的植物统计得到的遗传参数之间均没有显著性差异,这与Nybom[3]的统计结果不一致,却与Nybom 和Bartish[2]的统计结果一致.但Nybom 和Bartish[2]在附着传播和风/水传播的统计植物均只有3 种,其统计的随机误差可能较大.
综上所述,鉴于自交植物、一年生植物和单子叶植物的遗传多样性指数水平较低,考虑到SSR 标记数与各遗传参数有显著的负相关关系,因此建议在进行自交植物、一年生植物和单子叶植物的相关研究时可选用较少的SSR 标记,而在异交植物、多年生植物、裸子植物和双子叶植物的相关研究中可选用较多的SSR 标记.
4 结论
本文探讨了基于SSR 标记的取样策略和植物生活史特性对遗传多样性参数的影响.统计结果表明,SSR标记数目与各种群遗传参数显著相关,研究者增加标记的使用数目,可以有效提高种群遗传分析中的遗传分化值;而取样距离对种群间遗传多样性指数的正相关影响,已经得到许多相关研究的证明.对不同生活史特性植物遗传多样性的统计分析表明,繁育系统、生活型和分类地位与种群遗传多样性指数具有较大关联,自交植物的遗传多样性水平明显低于其他类型植物;一年生植物的遗传多样性水平明显低于短寿多年生或长寿多年生植物,且长寿多年生植物的遗传多样性水平最高.传播类型和分布范围对各遗传指数影响不显著.考虑到SSR 标记数与遗传参数(HO,HE)有显著的负相关关系,因此建议在进行异交植物、多年生植物、裸子植物和双子叶植物的相关研究中可选用较多的SSR 标记.此外,检测方法对遗传参数也有一定的影响,使用测序技术得出的遗传参数HO,HE和NA均高于电泳银染技术.
[1]HAMRICK J L,GODT M J W.Plant population genetics,breeding,and genetic resources[M].Massachusetts:Sinauer Associates Inc,1990.
[2]NYBOM H,BARTISH I V.Effect of life history traits and sampling strategies on genetic diversity estimates obtained with RAPD markers in plants[J].Perspect Plant Ecol,Evol Syst,2000,3(2):93-114.
[3]NYBOM H.Comparison of different nuclear DNA markers for estimating intraspecific genetic diversity in plants[J].Mol Ecol,2004,13(5):1143-1155.
[4]DUMINIL J,FINESCHI S,HAMPE A,et al.Can population genetic structure be predicted from life-history traits?[J].Am Nat,2007,169(5):662-672.
[5]LEBERG P L.Estimating allelic richness:effects of sample size and bottleneck[J].Mol Ecol,2002,11(11):2445-2449.
[6]MARIETTE S,LE C V,AUSTERLITZ F,et al.Sampling with the genome for measuring within-population diversity:trade-offs between markers[J].Mol Ecol,2002,11(7):1145-1156.
[7]KHANLOU K M,VANDEPITTE K,ASL L K,et al.Towards an optimal sampling strategy for assessing genetic variation within and among white clover(Trifolium repens L.)cultivars using AFLP[J].Genet Mol Biol,2011,34(2):252-258.
[8]STUDNICKI M,MADRY W,SCHMIDT J.Comparing the efficiency of sampling strategies to establish a representative in the phenotypic-based genetic diversity core collection of orchardgrass(Dactylis glomerata L.)[J].Czech J Genet Plant,2013,49(1):36-47.
[9]WATTIER R,ENGEL CR,SAUMITOU-LAPRADE P,et al.Short allele dominance as a source of heterozygote deficiency at microsatellite loci:experimental evidence at the dinucleotide locus Gv1CT in Gracilaria gracilis(Rhodophyta)[J].Mol Ecol,1998,7(11):1569-1573.
[10]CRAFT KJ,ASHLEY M V,KOENIG W D.Limited hybridization between Quercus lobata and Quercus douglasii(Fagaceae)in a mixed stand in central coastal California[J].Am J Bot,2002,89(11):1792-1798.
[11]WRIGHT S.The genetical structure of populations[J].Ann Hum Genet,1951,15(4):323-334.
[12]WRIGHT S.The interpretation of population structure by F-statistics with special regard to systems of mating[J].Evolution,1965,19(3):395-420.
[13]LYNCH M,MILLIGAN B G.Analysis of population genetic structure with RAPD markers[J].Mol Ecol,1994,3(2):91-99.
[14]PALACIOS C,GONZÁLEZ-CANDELAS F.Analysis of population genetic structure and variability using RAPD markers in the endemic and endangered Limonium dufourii(Plumbaginaceae)[J].Mol Ecol,1997,6(12):1107-1121.
[15]SLAKTIN M.A measure of population subdivision based on microsatellite allele frequencies[J].Genetics,1995,139(1):457-462.
[16]COLLEVATTI R G,GRATTAPAGLIA D,HAY J D.Population genetic structure of the endangered tropical tree species Caryocar brasiliense,based on variability at microsatellite loci[J].Mol Ecol,2001,10(2):349-356.
[17]OLSEN K M,SCHAAL B A.Microsatellite variation in cassava(Manihot Esculenta,Euphorbiaceae)and its wild relatives:further evidence for a southern Amazonian origin of domestication[J].Am J Bot,2001,88(1):131-142.
[18]RAYBOULD AF,MOGG RJ,ALDAM C,et al.The genetic structure of sea beet(Beta vulgaris ssp.maritima)populations.ⅢDetection of isolation by distance at microsatellite loci[J].Heredity,1998,80(1):127-132.
[19]REUSCH T B H,STAM W T,OLSEN J L.A microsatellite-based estimation of clonal diversity and population subdivision in Zostera marina,a marine flowering plant[J].Mol Ecol,2000,9(2):127-140.
[20]GAGGIOTTI O E,LANGE O,RASSMANN K,et al.A comparison of two indirect methods for estimating average levels of gene flow using microsatellite data[J].Mol Ecol,1999,8(9):1513-1520.
[21]ELLSTRAND N C,ELAM D R.Population genetic consequences of small population size:implications for plant conservation[J].Ann Rev Ecol Syst,1993,24:217-242.
[22]张德全,杨永平.几种常用分子标记遗传多样性参数的统计分析[J].云南植物研究,2008,30(2):159-167.
[24]KARRON J D.Genetics and Conservation of Rare Plants[M].New York:Oxford University Press Inc,1991.
[25]ELIADES N H,GAILING O,LEINEMANN L,et al.High genetic diversity and significant population structure in Cedrus brevifolia Henry,a narrow endemic Mediterranean tree from Cyprus[J].Plant Syst Evol,2011,294(3-4):185-198.
[26]OLEAS N H,MEEROW A W,FRANCISCO-ORTEGA J.Population dynamics of the endangered plant,Phaedranassa tunguraguae,from the tropical Andean hotspot[J].J Hered,2012,103(4):557-569.
[27]PAYN K G,DVORAK W S,JANSE B J H,et al.Microsatellite diversity and genetic structure of the commercially important tropical tree species Eucalyptus urophylla,endemic to seven islands in eastern Indonesia[J].Tree Genet Genomes,2008,4(3):519-530.
[28]ZHANG X P,LI X H,QIU Y X.Genetic diversity of the endangered species Kirengeshoma palmata(Saxifragaceae)in China[J].Biochem Syst Ecol,2006,34(1):38-47.
