新疆传统馕发酵面团中酵母菌的多样性分析
2015-12-20米尔班古丽阿卜杜如苏力玛依古丽库尔班努斯热提古丽安外尔艾合买提江艾海提吐尔逊阿依麦麦提依明艾科拜尔木哈塔尔木合塔尔阿布都克里木祖丽皮亚玉努斯艾尔肯热合曼
米尔班古丽·阿卜杜如苏力,玛依古丽·库尔班,努斯热提古丽·安外尔,艾合买提江·艾海提,吐尔逊阿依·麦麦提依明,艾科拜尔·木哈塔尔,木合塔尔·阿布都克里木,祖丽皮亚·玉努斯,艾尔肯·热合曼
(新疆大学生命科学与技术学院,新疆 乌鲁木齐 830046)
新疆传统馕发酵面团中酵母菌的多样性分析
米尔班古丽·阿卜杜如苏力,玛依古丽·库尔班,努斯热提古丽·安外尔,艾合买提江·艾海提,吐尔逊阿依·麦麦提依明,艾科拜尔·木哈塔尔,木合塔尔·阿布都克里木,祖丽皮亚·玉努斯*,艾尔肯·热合曼*
(新疆大学生命科学与技术学院,新疆 乌鲁木齐 830046)
采用平板分离与纯化、26S rDNA D1/D2区域序列测定和系统发育分析方法,对新疆乌鲁木齐、阿勒泰、伊犁、喀什、阿克苏及和田6 个地区民间传统馕面团发酵酵母菌多样性进行了研究。从中分离得到227 株酵母菌,它们隶属于10 个属14 个种:Saccharomyces cerevisiae、Saccharomyces servazzii、Pichia fermentans、Pichia membranifacies、Pichia kudriavzevii、Pichia anomala、Candida humilis、Hanseniaspora uvarum、Torulaspora delbrueckii、Wickerhamomyces anomalus、Cladosporium ramotenellum、Cryptococcus albidus、Kodamaea ohmeri、Issatchenkia orientalis;其中Saccharomyces cerevisiae为优势种,占总分离株的55.51%(126 株),在系统发育树上形成2 个大分枝4 个小分枝,A1-1分枝由115 株菌株构成,是馕面团发酵的优势品系菌群。研究结果阐明了新疆民间传统馕面团中酵母菌的多样性,为开发利用传统馕面团的酵母菌资源提供了理论依据。
馕面团;酵母菌;26S rDNA D1/D2区域
馕是以面粉为主要原料,经发酵后放少许盐,在特制的馕坑中烘烤而成的,大小、形状、厚薄不一的圆形饼。馕作为一种干粮,在中国新疆、中亚、南亚、西亚、中东及东南欧地区广泛地被烤制和食用,品种多达50多种,具有香、脆、酥等特点,已被人类食用了数千年[1-2]。
馕面团发酵是采用民间传统面团发酵方法,在新疆广大城乡居民家庭中至今仍被广泛应用,所谓馕面团是上次打馕留下来的发酵面团,一直在邻居之间相互借用,受到空间距离与地理条件的限制,一般不容易产生远距离的借用传播现象。馕面团发酵的酵母菌在馕的烤制工艺中是一个非常重要的关键因素,发酵菌种的不同会直接影响到馕的风味、成色与质量。本研究中采集样品的6 个地区都是相互之间有荒漠隔离区的绿洲,相互直线距离均远于300 km,一个地区选取的2~3 个馕房的相互直线距离也不低于30 km,所以每个样品的酵母菌的多样性不会受到其他不同样品的影响。
国外对传统发酵剂酸面团的研究报道较多,Succi[3]、Luangsakul[4]、Vrancken[5]、Meroth[6]等对不同地区不同来源的酸面团和酵面中的微生物进行过研究,Huys等[7]对40 篇原始的有关酸面团的文章做了综述,同时也将重复出现的菌株进行了报道,总结出S. cerevisiae、Candida humilis、Pchia kudriavzevii、Kazachstania exigua、Torulaspora delbrueckii、W. anomalus 6 个物种是酸面团中最常见的酵母类群。国内目前关于传统发酵剂中酵母菌的研究较少。马智刚等[8]研究了馒头面团长时间发酵过程中的酵母菌、醋酸菌、乳酸菌、大肠杆菌等微生物的变化。李自红[9]从民间采集的馒头老酵头中分离酵母菌25 株,筛选出制作馒头的优良酵母菌被鉴定为酿酒酵母。吴斯日古冷[10]从28 份酸面团样品中共分离出酵母菌85 株,其中S. cerevisiae占50.59%。潘向辉[11]从内蒙古采集的酸面团中分离筛选出5 株酵母菌并对其进行发酵特性研究。到目前为止,尚未见馕发酵面团酵母菌多样性有关的报道。因此,本实验以采自新疆乌鲁木齐、阿勒泰、伊犁、阿克苏、喀什及和田的馕面团为对象,以26S rDNA D1/D2区域的碱基序列分析为依据,对分离酵母菌株进行分子鉴定,了解馕面团中存在的酵母菌群落结构,对其分布概况和多样性进行分析。
1 材料与方法
1.1 材料与试剂
样品采自新疆维吾尔自治区乌鲁木齐、阿勒泰、伊犁、阿克苏、喀什与和田6 个地区,每地区选择2~3 个相互隔离的馕房采集适量的馕面团,保存于灭菌的采样管内,4 ℃条件下运回实验室,保存备用。
乙二胺四乙酸二钠(ethylenediamine tetraacetic disodium salt,EDTA-2Na)、十六烷基三甲基溴化铵(cetyl trimethyl ammonium bromide,CTAB)、Tris、十二烷基硫酸钠(sodium dodecyl sulphate,SDS)、溴化乙锭(ethidium bromide,EB)、琼脂糖、Marker DL 15 000 bp 上海Sangon生物公司;DNA聚合酶、dNTP、10×聚合酶链式反应(polymerase chain reaction,PCR)Buffer、溴酚蓝(DNA Loading Buffer)天根生化科技有限公司;引物NL1、NL4 北京鼎国昌盛生物技术有限公司。
1.2 仪器与设备
PCR 仪 美国Bio-Rad公司;Alphaimage2200TM凝胶成像仪 美国Alphaimage公司;DYY-8C水平电泳槽北京六一仪器厂。
1.3 方法
1.3.1 菌株分离与纯化
菌株分离采用PDA(马铃薯20%、葡萄糖2%、琼脂2%)和YPD(葡萄糖2%、蛋白胨2%、酵母浸粉1%、琼脂2%)培养基。采用梯度稀释涂布平板法进行分离,称取馕面团样品10 g,溶解于90 mL含有玻璃珠的灭菌生理盐水中,振荡30 min,分别取10-1~10-55 个梯度浓度,每皿100 μL,涂布分离。28 ℃培养48~120 h,挑取不同形态单菌落,编号,经多次划线纯化,获得纯培养物[12]。
1.3.2 菌株基因组DNA的提取
菌株DNA的提取采用CTAB法[13-15]。
1.3.3 26S rDNA D1/D2区域PCR扩增和序列测定
用引物NL1(5’-GCATATCAATAAGCGGAGGAAAAG-3’)和NL4(5’-GGTCCGTGTTTCAAGACGG-3’)扩增所分离菌株的26S rDNA D1/D2区域[16]。PCR扩增程序为:94 ℃ 4 min;94 ℃ 1 min,55 ℃ 30 s,72 ℃ 1 min,共35个循环;72 ℃ 10 min,最后于4 ℃保存[17-18]。1%琼脂糖凝胶电泳检测扩增的目标产物[19]。所得产物送生工生物工程(上海)股份有限公司和北京鼎国昌盛生物技术有限公司进行测序。
1.3.4 序列分析及系统发育树的构建
测序结果用Seqman软件序列图谱人工校对。校正后所测菌株的26S rDNA D1/D2区域序列在GenBank数据库中进行同源序列搜索(BLAST search),比较供试菌株与已知酵母菌之间的亲缘关系及其系统地位并将序列提交到GenBank核酸序列数据库得到登录号[20-21]。根据同源序列搜索结果,下载相关模式菌种的D1/D2区域序列,与供试菌的序列一起用Clustal X软件进行序列校准排齐,用Mega 5.0软件的邻接法(Neighbor-Joining),进行1 000 次Bootstrap检验后构建系统发育树[22-23]。
2 结果与分析
2.1 酵母菌鉴定结果
本研究从新疆6 个地区的馕面团样品中共分离到酵母菌227 株,对其进行了形态学观察及26S rDNA D1/D2区域序列分析。目前国际上公认的酵母菌分类标准一般认为,如果测试菌株的26S rDNA D1/D2区域序列与已知种的相似性为99%以上,并有形态特征的支持时,则对其作出种的鉴定;与最近缘的已知种的相似性为98%或以下时,则可初步确定为新种;相似性在98%~99%之间时,则需要结合ITS序列分析和生理生化特征比较对其作出分类学处理[24-26]。通过BLAST search发现,分离菌株26S rDNA D1/D2区域序列在500~630 bp,符合酵母菌26S rDNA D1/D2区域理论预期值(约为600 bp)。分离到的227 株菌(表1)序列比对与相应的模式菌株同源性均为99%~100%,未发现潜在新种。这227 株酵母菌隶属于10 个属,分别为酿酒酵母属(Saccharomyces)、假丝酵母属(Candida)、毕赤酵母属(Pichia)、有孢汉逊酵母属(Hanseniaspora)、有孢圆酵母属(Torulaspora)、隐球酵母属(Cryptococcus)、伊萨酵母属(Issatchenkia)、Wickerhamomyces属、Kodamaea属和Cladosporium属,共有14 个种,其中Saccharomyces属有2 个种,Pichia属有4 个种,其余各属仅鉴定出一个种。每个种选出一个代表菌株与相应已知种的26S rDNA D1/D2进行比较,构建系统发育树(图1)。在系统发育树上,分离菌株与相同的已知菌种被归在一个主分枝内,表明它们具有较近的亲缘关系,而不同的种却位于不同的亚分枝上,显示它们在D1/D2区域序列上具有明显区别。上述结果表明,新疆馕面团含有相对丰富的酵母种类。

表1 馕面团采集地点及采集样品、分离菌株、属和种数量Table 1 Sampling location of ‘NNaanngg’ dough and the numbers of samples, yeast strains, genera and species

图1 基于26S rDNA D1/D2区域序列和Neighbour-Joining分析绘制的系统发育树Fig.1 Phylogenetic tree drawn from Neighbor-Joining analysis based on 26S rDNA D1/D2 domain sequence alignment
2.2 酵母菌种群分布及优势类群
在各地区分离到的酵母菌类型及数量见表2。由表2可知,上述地区馕面团酵母菌群分布不太均匀,各样品中酵母菌的差异性很大。如S. cerevisiae在所有的采样点均可发现;C. humilis、P. fermentans、P. membranifacies、P. kudriavzevii、T. delbrueckii、W. anomalus、C. ramotenellum等物种同时出现在2 个地区的样品中;但是,S. servazzii、P. anomala、H. uvarum、C. albidus、K. ohmeri、I. orientalis等物种只在一个地区的样品中出现,这可能与所使用的面粉、水的来源及区域环境有关。

表2 馕面团酵母种类及其地理分布Table 2 Distribution of yeast species in ‘Nang’ dough 株
在鉴定出的14 个种中,S. cerevisiae为优势种,广泛分布于6 个采样地点的馕面团中,占总分离菌株的55.51%。C. humilis、P. fermentans这2 个物种所占比例比其他物种要高,分别占总分离菌株的11.45%和10.10%,但它们只在部分样品中出现,所以不能认为是新疆馕面团中的优势种,而只能是特定环境发酵条件中参入的菌群。
2.3 Saccharomyces cerevisiae进化分析
本研究发现S. cerevisiae为新疆馕面团的优势种,在227 株菌中有126 株S. cerevisiae,占总分离菌株的55.51%,每个采样地点的馕面团中均高频率地出现。对馕面团中的S. cerevisiae菌株26S rDNA D1/D2区域序列进行进化分析,对它们的进化发育及多样性分布情况进行检测。如图2所示,126 株菌株在系统发育树上形成了A 和B两个大分枝。相对于A枝,B枝是一个较小分枝,仅有9 株菌株构成。A分枝117 株菌株又分成A1(116 株)和A2(1 株)两个亚分枝,其中A1枝又分成A1-1 (115 株)和A1-2(1 株)小分枝。从整体上看,A1-1分枝占据了126 株S. cerevisiae中的115 株,该分枝是新疆馕面团发酵酵母菌种适应性最强、分布面最广、分化程度最高的酵母菌群。虽然这126 株菌株都属于S. cerevisiae一个物种,具有共同的祖先,但它们在系统发育树上形成2 个大分枝,4 个小分枝,结果显示,不同的环境条件导致它们选择了不同的进化与分化途径,基因组结构上已产生了相应的碱基差异。
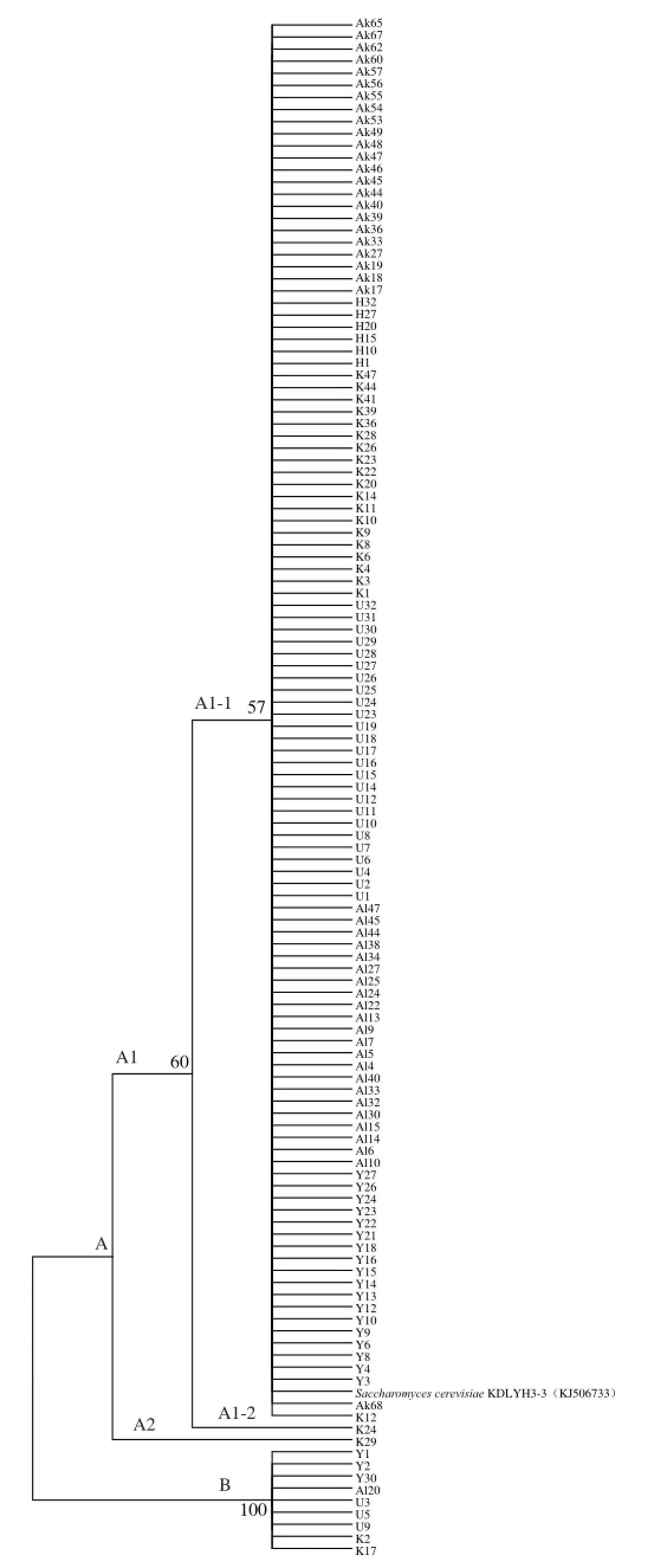
图2 基于26S rDNA D1/D2序列构建的S. cerevisiae菌种的系统发育树Fig.2 Phylogenetic tree based on the 26S rDNA D1/D2 domain of S. cerevisiae
3 讨 论
新疆地区具有独特的气候条件和特殊的地理环境,各地区的自然环境条件也存在着较大的差异,这对微生物资源的多样性提供了较大的生存和选择空间。研究结果显示,在乌鲁木齐、阿勒泰、伊犁、喀什、阿克苏及和田6 个地区中,和田的馕面团发酵酵母菌多样性最高,阿克苏的馕面团总分离菌株最多,多样性次于和田,这2 个地区的酵母菌群很相似;乌鲁木齐、阿勒泰地区馕面团的分离菌株分别属于4 个属4 个种;喀什地区馕面团的总分离菌株较多,但是它们只属于2 个属4 个种;伊犁地区馕面团发酵酵母菌多样性最低,分离株只属于S. cerevisiae一个物种,可能是总分离菌株少而导致其他酵母菌种漏检。上述结果揭示,新疆的馕面团发酵酵母菌资源丰富,每个地区酵母菌资源的分布有差异,这种差异与新疆各地区之间地理隔离与环境条件的差异相关。
研究还发现馕面团中存在P. anomala、C. ramotenellum、C. albidus、K. ohmeri等物种,数量较少,只在个别的样品中出现,这些物种可能来自于采集地的水、面粉及空气等周围环境。烤制馕饼的过程中,发酵好的面团成形后放置于160 ℃以上的馕坑里烘烤0.5 h左右,因此成品中不可能有活的菌体存在,并且代谢产物中也没有任何有害或有毒物质,一般不可能对馕制品食用者的健康造成危害。
在分离的227 株酵母菌中,126 株被鉴定为S. cerevisiae,占总分离菌株的55.51%,6 个采样点中均可发现,是新疆地区馕面团样品中的优势种。这一结果与吴斯日古冷等[10]的报道相似。在系统发育树上,126 株S. cerevisiae中有115 株聚集在A1-1分枝上,是新疆馕面团酵母中的优势品系菌群,在馕面团发酵过程中起着关键性的作用。这一结果揭示同一种内突变品系的类型众多,可为馕面团的发酵提供不同的代谢产物与不同的味感。这些馕面团发酵酵母菌株可能是本地区保存的特殊酵母品系,它们与酵母粉制品中的酵母是不同的酵母品种或品系。这也是新疆馕烤制工艺中依然采用传统的民间馕面团发酵工艺,现代酵母粉产品未被新疆原居民广泛采用和得到口感认可的原因。对土著酵母菌株的对比分析及其发酵代谢产物的深入探索研究极有可能为馕面团的现代化工业发酵提供优良酵母菌株。
[1] 热莎拉提·玉苏普. 新疆维吾尔族的特色食品-馕的基本解读[J]. 康定民族师范高等专科学校学报, 2007, 16(2): 17-20.
[2] 王苹, 房玉霞. 也谈维吾尔族的馕[J]. 喀什师范学院学报, 2013, 34(1): 37-40.
[3] SUCCI M, REALE A, ANDRIGHETTO A, et al. Presence of yeasts in southern Italian sourdoughs from Triticum aestivum fl our[J]. FEMS Microbiology, 2003, 255(1): 143-148.
[4] LUANGSAKUL N, KEERATIPIBUL S, JINDAMORAKOT S, et al. Lactic acid bacteria and yeasts isolated from the starter doughs for Chinese steamed buns in Thailand[J]. LWT-Food Science and Technology, 2009, 42(8): 1404-1412.
[5] VRANCKEN G, de VUYST L, van der MEULEN R, et al. Yeast species composition differs between artisan bakery and spontaneous laboratory sourdoughs[J]. FEMS Yeast Research, 2010, 10(4): 471-481.
[6] MEROTH C B, HAMMES W P, HERTEL C. Identification and population dynamics of yeasts in sourdough fermentation processes by PCR-denaturing gradient gel electrophoresis[J]. Applied and Environmental Microbiology, 2003, 69(12): 7453-7461.
[7] HUYS G, DANIAL H M, VUYST L. Handbook on sourdough biotechnology: taxonomy and biodiversity of sourdough yeasts and lactic acid bacteria[M]. New York: Springer Science, 2013: 105-154.
[8] 马智刚, 刘长虹, 冯忠军. 馒头面团发酵过程中的微生物变化[J]. 食品工业, 2003, 24(5): 11-12.
[9] 李自红. 传统发酵剂微生物的筛选、鉴定及对馒头品质的影响[D].郑州: 河南工业大学, 2011: 28-29.
[10] 吴斯日古冷. 内蒙古西部地区酸面团中酵母菌和乳酸菌的分离鉴定及其生物多样性研究[D]. 呼和浩特: 内蒙古农业大学, 2011: 20-23.
[11] 潘向辉. 酸面团酵母菌筛选及发酵特性研究[D]. 保定: 河北农业大学, 2011: 18-21.
[12] 沈萍, 陈向东. 微生物学实验[M]. 4版. 北京: 高等教育出版社, 2007: 14-35.
[13] NISIOTOU A A, SPIROPOULOS A S, NYCHAS G J. Yeast community structures and dynamics in healthy and botrytisaffected grape must fermentations[J]. Applied and Environmental Microbiology, 2007, 73(21): 6705-6713.
[14] 张颖慧, 魏东盛, 邢来君, 等. 一种改进的丝状真菌DNA提取方法[J].微生物学通报, 2008, 35(3): 466-469.
[15] 昂莎莎, 荚荣, 卢伟. 白腐真菌总DNA提取方法的研究[J]. 生物学杂志, 2009, 26(4): 82-85.
[16] KURTZMAN C P, ROBNETT C J. Identifi cation and phylogeny of ascomycetous yeasts from analysis of nuclear large subunit (26S) ribosomal DNA partial sequences[J]. Antonie van Leeuwenhoek, 1998, 73(4): 331-371.
[17] WANG Chunxiao, LIU Yanlin. Dynamic study of yeast species and Saccharomyces cerevisiae strains during the spontaneous fermentations of Muscat blanc in Jingyang, China[J]. Food Microbiology, 2013, 33(2): 172-177.
[18] 祝春梅, 姚新奎, 孟军, 等. 新疆自然发酵酸马奶中酵母菌的分离鉴定[J]. 食品与发酵工业, 2013, 39(4): 42-47.
[19] 魏群. 分子生物学实验指导[M]. 2版. 北京: 高等教育出版社, 2007: 37-39.
[20] PARAMITHIOTIS S, TSIASIOTOU S, DROSINOS E H. Comparative study of spontaneously fermented sourdoughs originating from two regions of Greece: Peloponnesus and Thessaly[J]. European Food Research and Technology, 2010, 231(6): 883-890.
[21] 卿蔓君, 白梅, 张勇, 等. 西藏曲拉和云南乳饼中酵母菌的鉴定及其生物多样性[J]. 微生物学报, 2010, 50(9): 1141-1146.
[22] 周新丽, 李治滢, 杨丽源. 云南程海湖酵母菌多样性及应用[J]. 微生物学报, 2011, 51(4): 547-553.
[23] TAMURA K, PETERSON D, PETERSON N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28(10): 2731-2739.
[24] 周巧, 李治滢, 杨丽源, 等. 云南5个地区戟叶酸模花中酵母菌和类酵母的多样性[J]. 微生物学通报, 2013, 40(4): 567-575.
[25] FELL J W, NOEKHOUT T, FONSECA A, et al. Biodiversity and systematics of basidiomycetous yeasts as determined by large-subunit rDNA D1/D2 domain sequence analysis[J]. International Journal of Systematic and Evolutionary Microbiology, 2000, 50(3): 1351-1371.
[26] 孙剑秋, 刘雯雯, 臧威, 等. 基于26S rDNA D1/D2序列分析酱香型白酒酒醅中酵母菌的群落结构[J]. 微生物学报, 2012, 52(10): 1290-1296.
Yeast Diversity in Traditional Xinjiang ‘Nang’ Dough
Mihribangul ABDURUSUL, Marygul KURBAN, Nusratgul ANWAR, Ahmatjan AHAT, Tursunay MAMTIMIN, Akbar MUHTAR, Muhtar ABDUKERIM, Zulfi ya YUNUS*, Erkin RAHMAN*
(College of Life Science and Technology, Xinjiang University, Ürümqi 830046, China)
Traditional ‘Nang’ (traditional Xinjiang food) doughs were sampled from Ürümqi, Altay, Ili, Kashgar, Aksu and Hotan in Xinjiang Uygur Autonomous Region and investigated for yeast diversity by plate separation and purification, 26S rDNA D1/D2 sequence analysis and phylogenetic analysis. Totally 227 yeast strains were isolated and identified as belonging to 14 species in 10 genera, i.e., Saccharomyces cerevisiae, Saccharomyces servazzii, Pichia fermentans, Pichia membranifacies, Pichia kudriavzevii, Pichia anomala, Candida humilis, Hanseniaspora uvarum, Torulaspora delbrueckii, Wickerhamomyces anomalus, Cladosporium ramotenellum, Cryptococcus albidus, Kodamaea ohmeri and Issatchenkia orientalis. S. cerevisiae was the most widely distributed and predominant species (126 isolates), representing 55.51% of all isolates. In the phylogenetic tree, S. cerevisiae formed two big and four small branches. Among them, A1-1 branch was constituted by 115 strains and were the most predominant strains of ‘Nang’ dough during fermentation. The diversity of yeasts in traditional ‘Nang’ dough can provide useful information for further utilization of yeasts in traditional fermented ‘Nang’ dough.
‘Nang’ dough; yeast; 26S rDNA D1/D2 region
TS201.3
A
1002-6630(2015)19-0199-05
10.7506/spkx1002-6630-201519036
2014-12-23
乌鲁木齐市科技创新种子资金项目(21061710);国家自然科学基金新疆联合基金项目(U1203101);
国家自然科学基金地区科学基金项目(31060002)
米尔班古丽·阿卜杜如苏力(1988-),女,硕士研究生,研究方向为资源微生物。E-mail:mihribangul@sina.com
*通信作者:祖丽皮亚·玉努斯(1970-),女,副教授,博士,研究方向为资源微生物。E-mail:zulfi ya_yunus@126.com
艾尔肯·热合曼(1953-),男,教授,博士,研究方向为资源微生物。E-mail:erkin1106@163.com
