鸡堆型艾美尔球虫3-1E抗原表位的生物信息学预测
2015-11-30李广兴丛培君黄艺华任玉东黄小丹
李广兴,丛培君,潘 龙,马 玲,冯 俪,黄艺华,任玉东,黄小丹
(1.东北农业大学动物医学学院,哈尔滨 150030;2.东北农业大学电气与信息学院,哈尔滨 150030)
鸡堆型艾美尔球虫3-1E抗原表位的生物信息学预测
李广兴1,丛培君1,潘 龙1,马 玲1,冯 俪1,黄艺华1,任玉东2,黄小丹1
(1.东北农业大学动物医学学院,哈尔滨 150030;2.东北农业大学电气与信息学院,哈尔滨 150030)
应用生物信息学分析和预测鸡堆型艾美尔球虫3-1E蛋白二级结构和抗原表位,为确定和筛选优势表位,研制安全、高效表位疫苗奠定基础。以克隆获得的鸡堆型艾美尔球虫3-1E蛋白氨基酸一级结构为基础,采用DNAStar软件和生物信息学在线分析程序ProtParam、SOSUI、IEDB、SYFPEITHI等预测3-1E蛋白理化性质和二级结构,并分析其序列跨膜区、可溶性、亲水性、可及性、柔韧性参数以及抗原指数,预测B细胞表位和T细胞表位的可能区域。3-1E蛋白由170个氨基酸组成,分子式C818H1257N213O272S3,分子质量18.5234 ku,理论等电点为4.25,半衰期为10 h;无跨膜区均为膜外区,是一种可溶性蛋白。其二级结构中α螺旋占31.76%,β转角为12.35%。B细胞表位预测区域:44-49、65-67、86-87、111-116;分值较高的T细胞表位区域:52-60、94-102、97-105、154-162、157-165。运用生物信息学方法确定3-1E存在多个抗原表位,对进一步研究3-1E的抗原性和研发优势表位疫苗具有重要意义。
鸡堆型艾美尔球虫;3-1E蛋白;抗原表位;生物信息学
网络出版时间2015-4-30 14:45:00 [URL]http://www.cnki.net/kcms/detail/23.1391.S.20150430.1445.015.html
李广兴,丛培君,潘龙,等.鸡堆型艾美尔球虫3-1E抗原表位的生物信息学预测[J].东北农业大学学报,2015,46(5):38-43.
Li Guangxing,Cong Peijun,Pan Long,et al.Bioinformatics prediction on 3-1E antigen epitopes ofEimeria acervulina[J]. Journal of Northeast Agricultural University,2015,46(5):38-43.(in Chinese with English abstract)
鸡球虫病(Coccidiosis)是由艾美尔球虫属多种球虫引起的严重危害鸡生长发育的寄生性原虫病,其中堆型艾美尔球虫(Eimeria acervulina)可引起感染鸡十二指肠严重损伤,导致增重和饲料转化率下降[1-2]。同时临床用于预防和治疗球虫病所用药物增加饲养成本,目前防治球虫病手段主要依赖于药物,但存在药物耐药性、新药研制费用高昂及药物残留等不足,亟待研发一种安全、稳定、高效且易于使用的疫苗防治鸡球虫病。
鸡球虫相关免疫保护性抗原鉴定较为困难。其中鸡堆型艾美尔球虫3-1E基因在子孢子和裂殖子阶段均有表达,基因序列高度保守,所编码蛋白具有良好免疫原性,已证明其免疫后能诱导机体产生细胞免疫和体液免疫应答[1],抵抗同种球虫攻击,明显减少攻虫鸡排放卵囊的数量,具有很好免疫保护效果[2],适合作为鸡球虫基因工程疫苗候选抗原。
随着生物信息网络技术发展和数据库扩展,多参数多方法综合预测已成为当前表位预测主流,可显著提高预测的准确性[3]。李晋涛等运用多种方法对附睾蛋白酶抑制蛋白(Epidydimal protease inhibitor,Eppin)二级结构和表面特性,如亲水性、理化性质、可及性、免疫原性和可塑性等方面进行分析,发现存在多个潜在抗原表位[4]。
经体外实验证明,所预测抗原表位区域基本能与免疫抗血清发生抗原特异性反应。李章球等应用SYFPEITHI等7种软件对人、鼠源血小板糖蛋白GPⅡb/b/Ⅲa抗体蛋白进行HLA-A*0201、HLA-A*1101和HLA-A*2401限制性表位预测[5],发现GPⅡb/Ⅲa抗体蛋白的T细胞表位。本文利用生物信息学方法分析3-1E蛋白结构与抗原表位,预测该蛋白免疫原性,为进一步研究其免疫保护性提供参考。
1 材料与方法
1.1 基因及蛋白序列
鸡堆型艾美耳球虫3-1E基因由东北农业大学动物医学学院动物病理学教研室鉴定和保存[2]。基因全长510 bp,用DNAStar软件推导出3-1E蛋白抗原氨基酸序列,编码170个氨基酸残基:MGEE ADTQAWDTSVKEWLVDTGKVYAGGIASIADGCRL FGAAIDNGEDAWSQLVKTGYQIEVLQEDGSSTQED CDEAETLRQAIVDGRAPNGVYIGGIKYKLAEVKRD FTYNDQNYDVAILGKNKGGGFLIKTPNDNVVIALY DEEKEQNKADALTTALAFAEYLYQGGF。
1.2 方法
1.2.1 堆型艾美耳球虫3-1E蛋白理化性质及可溶性分析
应用ExPASy(蛋白分析专家系统)中ProtParam (http://web.expasy.org/protparam/)和SOSUI(http://bp. nuap.nagoya-u.ac.jp/sosui/sosui_submit.html)分析3-1E蛋白理化性质和可溶性。
1.2.2 堆型艾美耳球虫3-1E蛋白跨膜区域及二级结构预测
TMHMM Sever2.0软件(http://www.cbs.dtu.dk/ services/TMHMM/)分析蛋白跨膜区;SOPMASev⁃er(file:///NPS%20@%EF%BC%9ASOPMA%E4%BA% 8C%E7%BA%A7%E7%BB%93%E6%9E%84%E9% A2%84%E6%B5%8B.htm)预测蛋白二级结构。
1.2.3 堆型艾美耳球虫3-1E蛋白B细胞表位预测
采用DNAStar软件Protean程序以及在线预测网站IEDB(http://www.iedb.org/)结合亲水性参数、可及性参数、抗原性指数、柔韧性参数对3-1E抗原B细胞表位进行综合预测。
1.2.4 堆型艾美耳球虫3-1E蛋白T细胞表位预测
通过在线网络资源使用以下两个网站:SYFP EITHI(http://www.syfpeithi.de/Scripts/MHCServer.dll/ EpitopePrediction.htm)和BIMAS(http://www-bimas.cit. nih.gov/molbio/hla_bind/),通过其各种算法对3-1E抗原的T细胞表位进行预测。
2 结果与分析
2.1 堆型艾美耳球虫3-1E蛋白理化性质及可溶性
经ProtParam分析,3-1E基因编码蛋白由170个氨基酸组成,其中含量最多的是甘氨酸(11.2%)和丙氨酸(11.2%),分子式C818H1257N213O272S3,相对分子质量为18.5234 ku,理论等电点为4.25,半衰期为10 h。经SOSUI分析3-1E蛋白平均疏水性为-0.410000,是可溶性蛋白。
2.2 堆型艾美耳球虫3-1E蛋白跨膜结构及二级结构预测
用TMHMM对3-1E跨膜序列进行分析,3-1E不存在跨膜区域,1-170区域都属于膜外区(见图1)。
对3-1E二级结构进行预测,发现3-1E基因中α螺旋占31.76%,无规则卷曲占33.53%,β转角占12.35%。各种结构在3-1E抗原中的分布情况见图2。
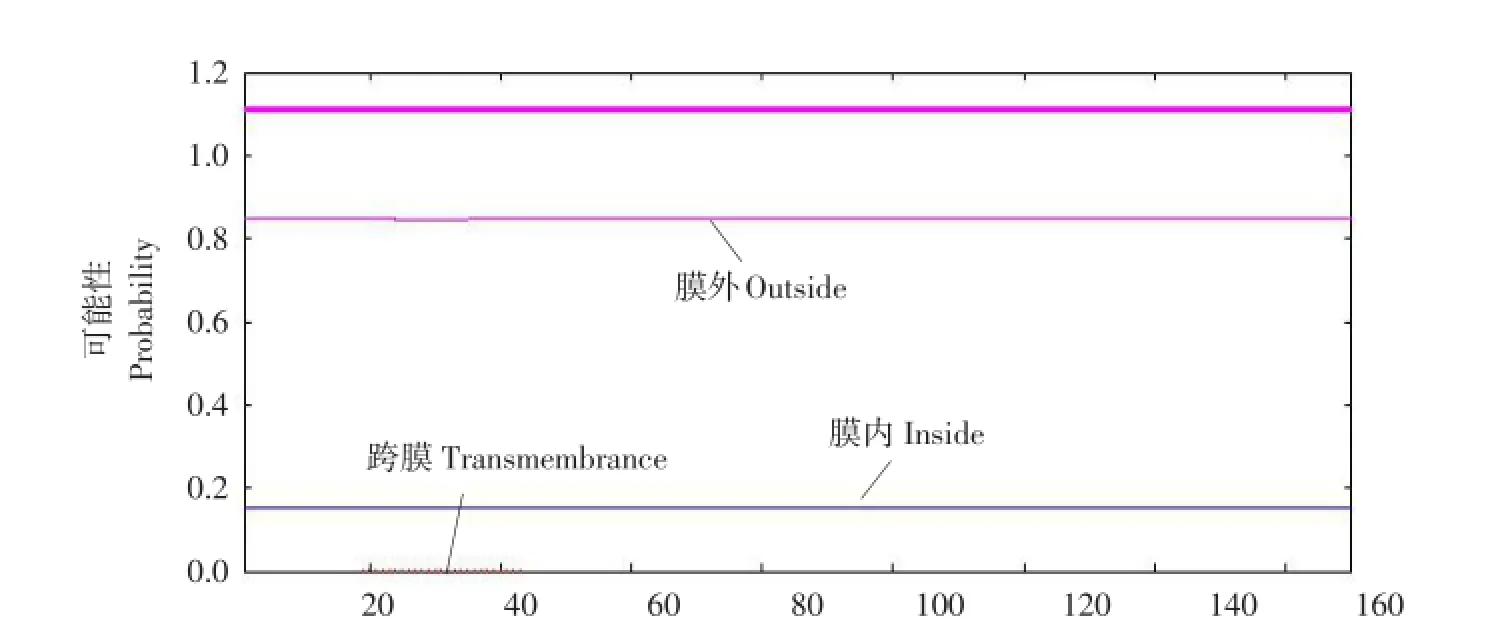
图1 TMHMM分析的E 3-1蛋白跨膜结构Fig.1 Transmembrane structure of 3-1E protein using TMHMM analysis

图2 SOPMA分析的3-1E蛋白二级结构Fig.2 Secondary structure of 3-1E protein using SOPMA analysis
2.3 堆型艾美耳球虫3-1E蛋白B细胞表位预测结果
2.3.1 DNAStar软件对3-1E蛋白B细胞表位预测
DNAStar软件对3-1E蛋白预测结合以下有代表性参数综合预测方案(见图3)。
以Hopp和Woods方案预测的3-1E蛋白亲水性区 域 为 : 1-15、 43-52、 60-81、 103-116、143-154。用Emini蛋白可及性方案分析,分值较高区 域 为 : 3-7、 64-74、 104-116、 132-136、143-153。经Karplus-Sohulz柔性区域分析,3-1E蛋白可塑性较强区域为:5-16、19-23、44-49、54-57、 64-80、 86-92、 98-100、 105-109、111-116、 122-128、 132-137、 143-152。 采 用Jameson-Wolf法对3-1E蛋白抗原性指数进行预测,抗原性指数较高的表位区域为:19-27、33-38、42-50、 61-82、 83-93、 100-117、 122-128、132-139、143-154。联合以上参数和二级结构进行综合分析,得出3-1E蛋白有7个潜在B细胞抗原表位,分别为:35-37、40-49、64-80、86-87、105-109、111-116、143-152。
2.3.2 在线软件IEDB预测3-1E蛋白B细胞表位
通过在线软件IEDB对3-1E蛋白进行表位预测(见图4)。① Chou&Fasman β转角预测,分值较高区域为:64-73、86-92、110-118、121-129、131-138。②对3-1E蛋白进行Emini表面可及性分析,分值较高区域为:65-73、105-116、143-151。③在Karplus&Schulz柔韧性区域预测中,3-1E蛋白分值高区域为:4-16、19-24、44-50、64-82、85-92、122-128、144-152。④ 通过Kolaskar&Tongaonkar抗原性指数预测,可能存在表位区域为:15-41、51-57、59-65、80-87、91-106、116-121、136-143。⑤Parker亲水性预测显示3-1E蛋白高亲水性区域为:4-12、42-50、63-80、86-90、108-118、124-126、143-156。⑥ 通过Bepipred预测抗原表位,3-1E蛋白存在多个Bepipred线性抗原表位区域为:1-12、23-25、43-50、64-79、85-93、109-116、123-126、134-136、145-154。综合以上参数分析,得出3-1E蛋白潜在抗原表位区域也为7个,分别是:4-12、23-24、44-50、65-73、86-92、110-116、121-128。
通过综合以上两个分析软件结果,将两个软件预测为表位相近区域确定为最终3-1E蛋白B细胞抗原表位区域,分别为:44-49、65-73、86-87、111-116。
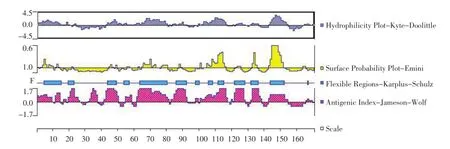
图3 DNAStar分析3-1E蛋白的各项参数Fig.3 Parameters of 3-1E protein using DNAStar analysis

图4 IEDB分析3-1E蛋白B细胞抗原表位Fig.4 B antigenic epitopes of 3-1E protein using IEDB analysis
2.4 堆型艾美耳球虫3-1E蛋白T细胞表位预测结果
2.4.1 SYFPEITHI预测3-1E蛋白T细胞表位
运用在线软件SYFPEITHI预测T细胞表位后,选取分值高的13个T细胞抗原表位,预测结果见图5。
2.4.2 BIMAS预测3-1E蛋白T细胞表位
为提高预测准确性,同时用在线预测工具BIMAS对3-1E蛋白T细胞表位进行预测,结果见图6。

图5 SYFPEITHI预测3-1E蛋白T细胞表位Fig.5 T cell epitope of 3-1E protein using SYFPEITHI

图6 BIMAS预测3-1E蛋白T细胞表位Fig.6 T cell epitope of 3-1E protein using BIMAS
综合两个在线软件预测结果,确定在两种方法中分值均较高的5个表位为3-1E蛋白的T细胞表位区域,分别为:52-60、94-102、97-105、154-162、157-165。
3 讨论
本研究利用生物信息学技术对鸡堆型艾美尔球虫3-1E蛋白理化性质及可溶性进行分析,发现其性质稳定且为可溶性蛋白,不易发生氨基酸变异和表位形成[6]。用TMHMM对3-1E跨膜序列进行分析,结果证明3-1E蛋白不存在跨膜区域,全部属于膜外区,这与目前堆型艾美尔球虫3-1E基因研究相符,3-1E蛋白是堆型艾美尔球虫子孢子、裂殖子的表面抗原[7]。膜外区产生抗原表位可能性也较大。
蛋白质二级结构对抗原表位有很大影响。α螺旋、β折叠的化学键键能比较高,能够维持蛋白高级结构,很难与抗体嵌合,且经常处于蛋白质内部,一般不作为抗原表位。而蛋白质转角及无规卷曲则较松散,易于发生扭曲、盘旋并展示在蛋白表面,结构突出,有利于与抗体嵌合,成为抗原表位可能性较大[8]。本研究用SOPMA Sever对3-1E蛋白进一步分析发现其无规则卷曲占33.53%,β转角占12.35%,由无规则卷曲和β转角组成的柔性区域占45.88%,表明在3-1E蛋白的这些区域中,抗原表位将容易产生并发挥特异性反应。
在免疫应答中,T细胞抗原受体和B细胞抗原受体所识别的抗原表位不同,分别称为T细胞表位和B细胞表位。B细胞表位是抗原分子中与抗体结合的特异性位点,通常由5~15个氨基酸组成。用抗原表位预测经典方案对3-1E蛋白进行多参数预测,通常需多种方案综合考虑,其中亲水性、表面可及性、二级结构和抗原性预测较为重要[9]:①Hopp和Woods为代表的蛋白质亲水性方案(Hy⁃drophilicity),此方案认为蛋白质抗原各氨基酸残基可分为亲水残基和疏水残基两类。在机体内,疏水性残基一般埋在蛋白内部,而亲水性残基位于表面,因此蛋白亲水部位与蛋白抗原表位有密切联系[10]。②Emini蛋白可及性方案(Accessibili⁃ty),指蛋白质抗原中氨基酸残基被溶剂分子接触的可能性,反映蛋白质抗原内和抗原外各层残基分布情况[7]。③Karplus-Sohulz柔性区域预测方案(Flexibility)认为蛋白抗原构象并非刚性,其多肽链骨架有一定程度的活动性,即蛋白质柔性区域。活动性强的氨基酸残基即可塑性大的位点,易形成抗原表位[9]。④Jameson-Wolf抗原性指数方案(Antigenic index)通过对20个已研究蛋白质69个连续位点606个氨基酸的统计分析,用各氨基酸残基在已知B细胞表位中出现的百分率与其通常在蛋白质中出现的百分率比值的对数建立抗原性刻度,并以此计算蛋白中各亚序列的抗原性[11]。⑤β转角预测方案认为β转角是蛋白质二级结构的基本类型之一,由4个氨基酸残基组成,其中第一个残基的CO基团和第四个残基的NH基团之间形成氢键,使多肽链的方向发生U形改变,使表位容易在β转角存在区域形成。本试验中将这些方案综合运用,选出不同方案结果中相同或相近区域,提高B细胞表位预测准确性。
在T细胞预测中,MHCI类抗原表位预测的准确率较高,可达90%,MHCI类表位由9个氨基酸残基构成短肽,最常用是HLAA 0201限制的抗原表位[12]。因此本研究对3-1E进行HLAA 0201 MHC I类9肽限制性表位预测。Tong等发现,根据蛋白质氨基酸的分布,运用各种软件对氨基酸构成和分布进行计算得出一个T细胞表位分值,以表格形式显示[13]。本研究将两种预测法分值排在前13位的表位进行综合分析,发现两种不同方法结果重复多,说明综合两种方法能提高预测准确性。联合各参数预测3-1E含有4个潜在的B细胞表位分别为:44-49、65-73、86-87、111-116;5个分值较高的T细胞表位是52-60、94-102、97-105、154-162、157-165。
4 结论
目前对鸡堆型艾美尔球虫3-1E蛋白结构研究较少,本试验在实验室克隆鉴定3-1E基因基础上,对3-1E蛋白进行理化性质、可溶性、跨膜结构、二级结构以及抗原表位等进行全面深入分析和预测,对进一步研究3-1E抗原性和根据优势表位研发疫苗具有重要意义。
[1] Ma D X,Ma C X,Pan L.et al.Vaccination of chickens with DNA vaccine encoding Eimeria acervulina 3-1E and chicken IL-15 of⁃fers protection against homologous challenge[J].Experimental Par⁃asitology,2011,127(1):208-214.
[2] Lillehoj S H,Ding X,Quiroz M A.et al.Resistance to intestinal coccidiosis following DNA immunization with the cloned 3-1E Ei⁃meria gene plus IL-2,IL-15,and IFN-γ[J].Avian Diseases, 2005,49(1):112-117.
[3] Sompuram S R,Bastas G,Vanil K.et al.Accurate identification of paraprotein antigen targets by epitope reconstruction[J].Blood, 2008,111(1):302-308.
[4]李晋涛,陈正琼,梁志清.Eppin抗原的二级结构分析B细胞表位预测[J].第三军医大学学报,2008,30(24):2254-2257.
[5]李章球,张梅霞,胡海燕,等.GPⅡb/Ⅲa抗体的二级结构及T、B细胞表位的预测[J].生物医学工程学杂志,2010,27(5):1146-1151.
[6] Sihic K,Tomic S,Carugo O.Systematic comparison of crystal and NMR protein structures deposited in the protein data bank[J].The Open Biochemistry Journal,2010,3(4):83-95.
[7] Sy S M,Chen J,Huen M S.The 53BP1-EXPAND1 connection In chromatin structure regulation[J],Nucleus,2010,1(6):472-474.
[8] Blytbe M.Flower D.Benchmaking B cell epitope prediction:An⁃derperformance of existing methods[J].Protein Science,2005(14): 1246-1248.
[9] Florea L,Halldorsson B,Kohlbacher O.et al.Epitope prediction algorithms for peptide-based vaccine design[J].Proceedings of Computer Society Bioinformatics Conference,2003(2):17-26.
[10] Sikic K,Tomic S,Carugo O.Systematic comparison of crystal and NMR protein structures deposited in the protein data bank[J],The Open Biochemistry Journal,2010,3(4):83-95.
[11] Casella G,Ferrante F,Saielli G.A.A DFT study of the Kar⁃plus-type dependence of vicinal(3)J(Sn-C-X-C),X=N,O,S in organotin(iv)compounds:Application to conformationally flexible systems[J].Organic& Biomolecular Chemistry,2010,8(12): 2711-2718.
[12] Yan C,Wang R,Li J.et al.HLA-A gene polymorphism defined by high-resolution sequence-based typing in 161 Northern Chi⁃nese Han people[J].Genomics,Proteomics&Bioinformatics, 2003,1(4):304-309.
[13] Tong J C,Tan T W,Ranganathan S.Methods and protocols for prediction of immunogenic epitopes[J].Briefings in Bioinformat⁃ics,2007,8(2):96-108.
Bioinformatics prediction on 3-1E antigen epitopes ofEimeria acervuli⁃na
LI Guangxing1,CONG Peijun1,PAN Long1,MA Ling1,FENG Li1,HUANG Yihua1,REN Yudong2,
HUANG Xiaodan1(1.School of Veterinary Medicine,Northeast Agricultural University,Harbin 150030, China;2.School of Electric and Information Engineering,Northeast Agricultural University,Harbin 150030,China)
To predict the immunogenicity of the protein coded byEimeria acervulina3-1E gene and characterize its structure and epitopes from a bioinformatics approach.The protein secondary structure was analyzed by TMHMM,and B and T cell epitope of 3-1E were predicted by DNAStar,IEDB and SYFPEITHI. The physico-chemical properties,solubility were analyzed and predicted using the ProtParam and SOSUI. The results showed that 3-1E protein consists of 170 amino acids and the molecular formula was C818H1257N213O272S3,the molecular mass was 18.5234 ku,its theoretical isoelectric point was 4.25,half-life time was 10 h.Many distinct antigenic epitopes of 3-1E were identified by computation,the assumed B cell epitopes of which were in the regions of 44-49,65-67,86-87 and 111-116.The high score of T cell epitopes were in the regions of 52-60,94-102,97-105,154-162 and 157-165.In general,the analysis of 3-1E antigen epitopes with contemporary bioinformatic software,it should provide significant assistances in the future experimental research of 3-1E antigenicity and development of dominant epitope vaccine.
chickenEimeria acervulina;3-1E protein;antigen epitope;bioinformatics
S852.31
A
1005-9369(2015)05-0038-06
2014-03-22
黑龙江省自然科学基金项目(ZJN0702-01);国家自然科学基金项目(31172295;31272569)
李广兴(1968-),男,教授,博士,博士生导师,研究方向为动物病理学。E-mail:ligx@neau.edu.cn
