辽藁本及其混伪品DNA条形码鉴定研究
2015-11-30秦祎婷董学会
李 雪,秦祎婷,董学会
(中国农业大学农学与生物技术学院,北京 100193)
辽藁本及其混伪品DNA条形码鉴定研究
李 雪,秦祎婷,董学会*
(中国农业大学农学与生物技术学院,北京 100193)
文章对比分析不同DNA条形码候选序列对辽藁本及其混伪品的鉴定能力。以辽藁本(Ligusticum jeholense Nakai et Kitag)、新疆藁本(Conioselinum vaginatium)及藁本混伪品白芷(Angelica dahurica)、石防风(Peucedanum terebinthaceum)、当归(Radix angelica sinensis)为鉴定材料,提取总DNA,利用4对候选序列(NrITS、ITS2、accD和trnH-psbA)分别进行PCR扩增,产物进行双向测序。得到的序列采用CodonCode Aligner V2.06进行拼接,Clustal X2.1进行多序列比对,MEGA 5.0计算K-2-P距离,构建系统发育NJ树。结果表明,NrITS和ITS2序列能够鉴别辽藁本与新疆藁本及其他混伪品。
辽藁本;混伪品;DNA条形码;分子鉴定
网络出版时间2015-4-30 14:44:00 [URL]http://www.cnki.net/kcms/detail/23.1391.S.20150430.1444.013.html
李 雪,秦祎婷,董学会.辽藁本及其混伪品DNA条形码鉴定研究[J].东北农业大学学报,2015,46(5):22-31.
Li Xue,Qin Yiting,Dong Xuehui.DNA barcode identification ofLigusticum jeholenseand its adulterants[J].Journal of Northeast Agricultural University,2015,46(5):22-31.(in Chinese with English abstract)
辽藁本(Ligusticum jeholense Nakai et Kitag)是伞形科藁本属多年生草本植物,以干燥根和根茎入药,具有抑菌、散风、祛寒、祛湿止痛功效,主治风寒头痛、寒湿腹痛、风湿肢节痹痛等症[1-2]。目前市场上常用的藁本药材主要来自辽藁本、藁本(Ligusticum sinense)和新疆藁本(Conioselinum vaginatium),还混有部分同科不同属的其他植物。这些植物形态特征极为相似,容易被混淆,充当辽藁本或藁本药材进行出售。而《中华人民共和国药典(一部)》收载的只有辽藁本和藁本,新疆藁本由于药理活性和化学成分区别于辽藁本,仅限当地习用。因此,辽藁本种质资源及中药材的正确鉴定工作十分重要。目前,关于辽藁本鉴定研究主要集中在形态[3-6]、显微观察[7]、色谱和光谱分析[8-9]等方面,鲜少有关于DNA分子鉴定方面研究报道。而分子鉴定技术作为研究药用植物分类和亲缘关系的重要手段,已经在多种药用植物分类鉴定中得到运用。其中,DNA条形码序列在多种药用植物鉴定中获得较好结果。
DNA条形码(DNA barcoding)由加拿大动物学家Hebert等2003年首次提出[10],已在动物物种分类研究中应用广泛,其中,CO1基因被认为是动物界中的标准DNA条形码。近年来,DNA条形码亦被植物分类学家广泛应用于植物物种分类和遗传进化研究中。但由于植物在进化过程中容易发生杂交、网状进化等情况,同一基因在不同类群的进化速率不同,使植物DNA条形码研究进展较为缓慢。迄今为止,仍未发现可作为植物通用条形码的DNA序列。2009年,生命条形码联盟植物工作组(CBOL)推荐以rbcL+matK组成的复合序列作为整个植物界DNA条形码序列,但其在物种水平的鉴定成功率仅为72%[11]。陈士林等研究药用植物及其近缘种的4 800个物种的6 600个样本,发现ITS2(Internal transcribed spacer 2)序列鉴定成功率高达90%以上[12-13]。高婷等运用ITS2序列对藁本及其常见混伪品骨缘当归、长鞘当归、细叶藁本、泽芹等进行分子鉴定,认为ITS2序列可用作药材藁本的混伪品鉴定[14]。现在,ITS2序列已被广泛应用于菊科、蓼科、爵床科等药用植物的分子鉴定研究中[15-17]。
本研究使用在植物鉴定上应用较多的ITS2、NrITS、trnH-psbA、accD四种DNA条形码候选序列对辽藁本及其混伪品石防风、当归和白芷进行鉴定研究,考查四种DNA条形码候选序列的鉴定能力,以期为进一步的辽藁本种属DNA分子鉴定研究提供参考依据。
1 材料与方法
1.1 植物材料
植物研究材料均为实地采集,并经中国农业大学农学与生物技术学院董学会教授鉴定。具体样品信息见表1。

表1 采集材料的样品信息Table 1 Source of samples for Ligusticum jeholense and adulterants
1.2 方法
1.2.1 样品DNA提取
取新鲜植物叶片组织100 mg,加入液氮充分研磨。利用植物基因组DNA提取试剂盒(RransGen Biotech Co,China)提取总DNA。
1.2.2 PCR扩增和测序
试验采用ITS2、NrITS、trnH-psbA、accD 4种序列引物对样品DNA进行扩增。
反应体系为25 μL,分别包含2.5 mmol·L-1dNTPs 2 μL、5×PCR buffer 5 μL、10 μmol·L-1引物各0.25 μL、2.5 U·μL-1TaqDNA聚合酶0.4 μL和模板DNA约30 ng。引物对序列和扩增程序参照文献[18-19],详见表2。
扩增完成后在凝胶成像分析仪下检测扩增结果,然后将扩增成功的PCR产物送至Life technolo⁃gies公司北京合成部测序。

表2 引物序列和扩增程序Table 2 Primers sequence and amplification procedure
1.2.3 序列拼接和数据分析
测序峰图采用序列拼接软件CodonCode Align⁃er V2.06(CodonCode Co.,USA)校对拼接,去除低质量序列区及引物区,进行多序列比对并人工校验,得到完整的各DNA条形码序列。利用DNA⁃STAR软件分析得到各DNA序列长度及各碱基组成,利用Clustal X2.1进行多序列比对,用MEGA 5.0计算K-2-P距离,对不同序列种间、种内变异大小进行比较,构建其系统进化树,评价不同序列的鉴定能力。
2 结果与分析
2.1 序列特点分析
运用MEGA 5.0等软件分析得到的各序列特点见表3,序列比对结果见图1~4。结果表明,4条考查序列长度为297~721 bp不等,其中trnH-psbA序列最短,为297~325 bp,NrITS序列最长,为712~721 bp。各序列GC含量各不相同,NrITS序列GC含量最高,达到53.65%~55.88%,trnH-psbA序列GC含量最低,为33.00%~35.20%。

表3 4条序列的序列特点Table 3 Sequencing information
2.1.1 NrITS序列特点分析
由表3可知,NrITS序列长度为712~721 bp, GC含量为53.65%~55.88%,具体序列比对结果见图1。


图1 不同产地辽藁本及其混伪品NrITS序列比对结果Fig.1NrITS blast results forLigusticum jeholenseand adulterants
2.1.2 ITS2序列特点分析
从表3中可以看出,ITS2序列长度为474~479 bp,GC含量为53.67%~55.46%,具体序列比对结果见图2。
2.1.3 accD序列特点分析
从表3中可以看出,accD序列长度为395~409 bp,GC含量为38.31%~39.75%,具体序列比对结果见图3。
2.1.4 trnH-psbA序列特点分析
从表3中可以看出,trnH-psbA序列长度为297~325 bp,GC含量为33.00%~35.20%,具体序列比对结果见图4。


图2 不同产地辽藁本及其混伪品ITS2序列比对结果Fig.2 ITS2 blast results for Ligusticum jeholense and adulterants


图3 不同产地辽藁本及其混伪品accD序列比对结果Fig.3 accD blast results for Ligusticum jeholense and adulterants

图4 不同产地辽藁本及其混伪品trnH-psbA序列比对结果Fig.4trnH-psbA blast results forLigusticum jeholenseand adulterants
2.2 种内种间遗传距离考查
利用MEGA 5.0软件对试验测得序列进行遗传距离分析,采用K-2-P模型计算遗传距离。分析结果表明,NrITS序列平均种间距离为0.046,ITS2序列平均种间距离为0.037,accD序列平均种间距离为0.004,trnH-psbA序列平均种间距离为0.030。各DNA条形码序列的遗传距离差异矩阵见表4~7。

表4 辽藁本及其混伪品NrITS序列的遗传距离差异矩阵Table 4 Differences matrix of NrITS genetic distances for Ligusticum jeholense and adulterants

表5 辽藁本及其混伪品ITS2序列的遗传距离差异矩阵Table 5 Differences matrix of ITS2 genetic distances for Ligusticum jeholense and adulterants

表6 辽藁本及其混伪品accD序列的遗传距离差异矩阵Table 6 Differences matrix of accD genetic distances for Ligusticum jeholense and adulterants

表7 辽藁本及其混伪品trnH-psbA序列的遗传距离差异矩阵Table 7 Differences matrix of trnH-psbA genetic distances for Ligusticum jeholense and adulterants
由表4~7可知,NrITS序列种间遗传距离差异明显,种内遗传距离差异较大;ITS序列种间遗传距离差异较大,种内遗传距离差异较小;trnH-ps⁃bA序列种间遗传距离差异明显,种内遗传距离差异较大;accD序列种间遗传距离和种内遗传距离差异均不明显。
2.3 构建系统发育NJ树
利用MEGA 5.0对各样品NrITS、ITS2、accD 和trnH-psbA序列进行分析,使用NJ法构建基于Kimura 2-parameter模型算法的系统聚类分析树,结果见图5~8。

图5 不同产地辽藁本及其混伪品NrITS序列的系统发育NJ树Fig.5 NJ tree constructed with NrITS sequences for Ligusticum jeholense and adulterants

图6 不同产地辽藁本及其混伪品ITS2序列的系统发育NJ树Fig.6 NJ tree constructed with ITS2 sequences for Ligusticum jeholense and adulterants

图7 不同产地辽藁本及其混伪品accD序列的系统发育NJ树Fig.7 NJ tree constructed with accD sequences for Ligusticum jeholense and adulterants
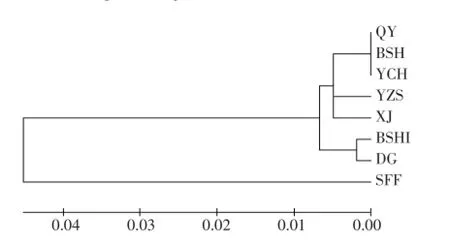
图8 不同产地辽藁本及其混伪品trnH-psbA序列的系统发育NJ树Fig.8 NJ tree constructed with trnH-psbA sequences for Ligusticum jeholense and adulterants
由图5~8可知,由NrITS、ITS2、accD和trnH-psbA序列构建的聚类进化树均明显分为两个部分。在NrITS序列的NJ树中,药植所辽藁本与白山辽藁本聚为一支,伊春辽藁本和清源辽藁本聚为一支,然后再与新疆藁本聚为一支为第一部分;石防风、白芷和当归均各自一支,然后聚为一支为第二部分。在ITS2序列的NJ树中,辽藁本和新疆藁本分支情况同NrITS序列,但石防风与当归聚为一支后再与白芷聚为另外一支。与NrITS、ITS2序列不同的是,在accD序列的NJ树中,伊春辽藁本和白山辽藁本聚为一支后,再与新疆藁本、当归、药植所辽藁本和白芷聚为一支;清源辽藁本和石防风聚为另外一支。在trnH-psbA序列的NJ树中,不同产地辽藁本与新疆藁本聚为一支后,再与白芷和当归聚为一支为第一部分;石防风单独一支为第二部分。
因此,利用NrITS和ITS2序列能够鉴别辽藁本与新疆藁本及其他混伪品种;trnH-psbA序列不能区分辽藁本与新疆藁本,但对鉴别藁本混伪品有参考价值;而accD序列不能鉴别辽藁本与新疆藁本及其他混伪品种。
3 讨论与结论
本试验使用四种候选条形码序列(NrITS、ITS2、accD和trnH-psbA)对不同产地辽藁本、新疆藁本及其混伪品石防风、当归、白芷的DNA进行扩增、测序,并对得到的序列进行拼接、比对和聚类分析,发现NrITS和ITS2序列能较好地区分辽藁本、新疆藁本及其混伪品,trnH-psbA序列对区分辽藁本及其混伪品有一定价值。这与高婷等认为ITS2序列能够反映出藁本及其易混伪品间差异的结论相吻合,增加NrITS和trnH-psbA两种序列用于辽藁本及其易混伪品的分子鉴别。结论可为辽藁本规范化种植过程中种源筛选和鉴定提供理论依据和技术支持。
但由于本试验分析样本数量较少,NrITS、ITS2以及trnH-psbA三种序列能否作为辽藁本种属间鉴定条形码序列,尚需后续大量样本研究验证。而accD序列对辽藁本及其混伪品鉴定效果不理想,后续研究意义不大。
[1] 中华人民共和国药典委员会.中华人民共和国药典(一部)[S].北京:化学工业出版社,2005.
[2] 于英,张永刚,刘国辉,等.辽藁本主要生物学特性的研究[J].吉林农业大学学报,2005,27(4):401-404,407.
[3] 高淑范,李玉梅,苗伟艳.辽藁本与新疆藁本的鉴别[J].黑龙江医药,1996,9(5):267-268.
[4] 包玮鸳,肖宁.藁本及其混淆品的鉴别[J].中国药业,1999,8 (6):53.
[5] 许彦,陈乃霞.藁本及草藁本的比较鉴别[J].黑龙江中医药, 1998(5):47-48.
[6]陈若芸,于德泉.藁本化学和药理研究[J].中医药通报,2002,1 (1):44-48.
[7]傅湘琦,侯燕鸣.药房中药的形态学研究——“风”药藁本组织的扫描电镜观察[J].新疆中医药,1988(3):36-37.
[8]张迎春,陈畅,李邵菁,等.藁本、辽藁本和新疆藁本挥发油化学成分分析及其血管活性观察[J].中国实验方剂学杂志, 2011,17(14):159-164.
[9] 戴斌.四种藁本药材挥发油的气相色谱-质谱分析比较[J].药学学报,1988,23(5):361-369.
[10] Hebert P D N,Cywinska A,Ball S L,et al.Biological identifica⁃tions through DNA barcodes[J].Proc R Soc Biol Sci Ser B,2003, 270:313.
[11] Kress W J,Erickson D L,Jones F A,et al.Plant DNA barcodes and a community phylogeny of a tropical forest dynamics plot in pnnama[J].Proc Natl Acad Sci USA,2009,106(44):18621.
[12] 陈士林,郭宝林,张贵君,等.中药鉴定学新技术新方法研究进展[J].中国中药杂志,2012,37(8):1043-1055.
[13] Chen S L,Yao H,Han J P,et al.Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species[J]. PLoS One,2010(5):8613.
[14] 高婷,姚辉,陈士林.基于ITS2序列的藁本与常见混伪品的分子鉴定[J].世界科学技术:中医药现代化,2011,13(2):418-423.
[15] 胡志刚.菊科药用植物DNA条形码及叶绿体基因组研究[D].武汉:湖北中医药大学,2012.
[16] 刘美子,宋经元,罗焜,等.DNA条形码序列对9种蒿属药用植物的鉴定[J].中草药,2012,43(7):1393-1397.
[17] 梁欣健.南板蓝根与山药的DNA条形码鉴定研究[D].广州:广州中医药大学,2012.
[18] Kress W J,Wurdack K J,Zimmer E A,et al.Use of DNA bar⁃codes to identify flowering plants[J].Proceedings of the National Academy of Sciences,2005,102:869-874.
[19] Sass C,Little D P,Stevenson D W,et al.DNA barcoding in the cy⁃cadales:Testing the potential of proposed barcoding markers for species identification of Cycads[J].PLoS ONE,2007,2(11):1154.
DNA barcode identification ofLigusticum jeholenseand its adulterants
LI Xue,QIN Yiting,DONG Xuehui(School of Agriculture and Biotechnology,China Agricultural University,Beijing 100193,China)
The article contrastively analyzed the abilities of different DNA barcodes to identify Ligusticum jeholenseand its adulterants.Total DNA was extracted fromLigusticum jeholense,Conioselinum vaginatiumand its adulterantsAngelica dahurica,Peucedanum terebinthaceumandRadix angelica sinensis. Their NrITS,ITS2,accD and trnH-psbA DNA sequences were amplified and PCR products were sequenced. Sequences assembly were performed using the CodonCode Aligner V2.06 and blasted using the Clustal X2.1.The Kimura 2-Parameter(K2P)distances were calculated using software MEGA 5.0 and then Neighbor-Joining(NJ)tree was constructed.The results indicated that the NrITS and ITS2 sequences could be effectively used as DNA barcodes for identifyingLigusticum jeholenseand its adulterants.
Ligusticum jeholense;adulterant;DNA barcode;molecular identification
S155.2+6
A
1005-9369(2015)05-0022-10
2014-12-18
国家科技支撑计划(2006BAI06A15-2)
李雪(1984-),女,博士研究生,研究方向为种子分子生物学。E-mail:rebeccayou@163.com
董学会,教授,博士生导师,研究方向为种子分子生物学。E-mail:xuehuidong@cau.edu.cn
