跑节省化标志的有氧能力相关尿游离miRNA表达谱特征
2015-11-19魏胜辉
魏胜辉,李 岩
1.Sichuan Normal University,Chengdu Sichuan 610101 2.Ludong University,Yantai Shandong 264025
杰出的运动能力是具有遗传优势的个体在系统水平对长期训练发生良性适应性重塑的结果[1]。表观遗传学基因调控机制在机体响应环境压力的适应性重塑过程中起到关键性作用,其基因调控模式具有遗传性个体差别,导致适应能力在不同个体间显著不同[2]。适应能力越强,则运动能力的可训练性越好,发展潜力越大[3]。目前对于表观遗传学基因调控模式与运动能力训练敏感性的具体关联所知甚少,因而无法将其作为指标应用于运动员选材和个性化训练。
microRNA基因表达调控作用是表观遗传学基因调控的主要途径,是环境因素诱导的基因选择性表达的主要调控方式[4]。研究表明,microRNAs分子(以下简称miRNAs)参与了运动性适应基本生理过程的调控,如肌组织肥大和心肌/骨骼肌收缩力增强[5]、血管增生[6]、线粒体合成与酶活性提高[7]等。miRNAs的表达受转录因子系统和表观遗传修饰等因素调控,其调控模式本身存在个体差异,形成个体之间对环境因素适应能力的差别。miRNA调控模式差别表现为个体之间的miRNA差异表达谱[8]。近期研究证实:尿液游离miRNA(Urine cell-free miRNA,以下简称UCF-miRNA)与组织细胞内和循环miRNA表达谱存在密切关联[9-10]。因此推测:运动性适应过程中UCF-miRNA特异表达谱可作为反映运动能力的可训练性的“分子指纹”,在选材阶段准确预估运动能力的发展潜力,或用以评估训练效果,作为制订个性化训练方案的依据。
有氧耐力水平是决定运动能力的主要因素之一[11],长期周期性训练使机体反复不断的对负荷做出响应和适应,其积累效应为机体适应性重塑和有氧运动能力的提高[12]。个体间表观遗传学调控模式差异使每个周期的适应效果产生差别,最终形成表型水平的显著个体差异。本研究以UCF-miRNA作为miRNA调控模式的生物标志,分析其与不同训练敏感水平表型之间的关联,从而获得高敏感表型的miRNA基因表达调控模式特征。跑节省化(Running Economy,以下简称RE)能够客观反映具体个体的有氧运动能力[13],本研究以RE变化率作为反映有氧运动能力训练敏感性指标,建运动性适应UCF-miRNA表达谱其与有氧运动能力训练敏感性的关联模型,作为早期预测和评估有氧运动能力发展潜力和适应特点的指标应用于运动员选材和个性化训练方案制订。
1 研究对象与方法
1.1 研究对象
表1 参加实验人员基本情况s)

表1 参加实验人员基本情况s)
参训人员 样本(n) 年龄(yr) 身高(cm) 体重(kg)入选实验人员 127 18.7±0.6 172.6±9.9 63.10±10.05完成实验人员91 18.6±0.5 173.2±7.45 64.68±8.93
1.2 有氧耐力训练方案
参加实验学员进行为期17周的系统性有氧运动能力训练。训练方案:每周日、二、四下午16:30-17:30进行5 000米长跑训练,负荷强度[14]:2-3周60%个体最大心率±3次/min;4-14周70%个体最大心率±3次/min;15-17周75%个体最大心率±3次/min(注:个人最大心率=220-年龄)。以polar表(瑞典产)监控靶心率维持速度。
1.3 有氧运动能力相关指标检测
所有指标在17周有氧耐力训练前后各检测1次,分别记为基础值(baseline)和训练值(postex)。
(1)5 000米最好成绩测试(Personal Best 5000,简称PB5000):参训学员佩戴polar表在400米标准跑道上全力完成5 000米跑,记录完成时间。
(2)最大摄氧量(VO2max)、通气无氧阈(VT)测试:采用逐级递增负荷运动方式,使用RUN Med700跑台(Technogym,意大利)和CPX运动气体代谢分析仪(JAEGER,德国)。受试者测试前热身3min,正式试验采用预设的Bruce方案,即每3min增加速度或坡度直至力竭,通气数据由气体代谢分析仪自动采集。
判定VO2max标准:①继续运动后摄氧量的差小于5%或150mL/min或2mL/kg.min;②心率超过180次/min,呼吸商超过1.10;③受试者体力达到力竭,不能保持规定速度;④继续运动时摄氧量出现下降[13]。
判定VT标准:将肺通气量(VE)、二氧化碳呼出量、心率、吸氧量等指标急剧增加的拐点定义为VT。
(3)RE测试:参照席翼方法[13],设备同前。受试者空腹,适应跑台5min,设定坡度始终为0,正式实验采用递增负荷运动方式,到3min时将跑速加到11.5km/h,以该速度持续跑6min,取最后2min摄氧量的平均值即为RE值。每次实验前校对气体代谢分析仪,保持受试者各项测试条件一致。获取RE(绝对值,单位:L/min)和 R-RE(VO2/Wt,单位:mL/kg/min)。
一些种类的微藻可以直接产生乙醇。在黑暗异养条件下,部分微藻可以生存并合成碳水化合物。如果建立黑暗缺氧条件,积累的淀粉的氧化反应就会变得不完全,根据微藻种类不同,生成氢气、二氧化碳、乙醇、乳酸、甲酸、乙酸等产品[38]。也可以利用基因工程等手段定向选育具有乙醇生产能力的微藻。
1.4 尿液UCF-miRNA表达谱检测和实时荧光定量PCR法验证
(1)分别于17周有氧耐力训练前清晨空腹安静状态下、PB5000测试结束后1小时内、恢复24小时和48小时后安静状态接取中段尿100ml,离心,取上清,冻干浓缩备用[18]。
(2)总RNA提取和MicroRNA芯片测定UCF-miRNA表达谱。采用miRCURY RNA isolation kit(EX-300112 Exiqon)提取small RNA(<1000nt);miRCURYTM Array Labelling kit试剂盒进行miRNA标记操作,严格按照说明书进行操作。miRNA芯片的杂交、清洗按照miRCURYTMArray microarray kit(Exiqon)试剂盒说明书进行操作,采用 Molecular Devices公司的Genepix 4000B双通道激光图像扫描仪以635 nm单波长进行扫描。Genepix Pro 6.0软件分析数据。数据预处理后根据各张芯片的global mean进行片间校正。使用SAM(significance analysis of microarrays,SAM.version2.1)筛选差异表达miRNA。筛选条件为:FDR(false discoveryrate)<5%,Fold change>2。
(3)实时荧光定量PCR(qRT-PCR)验证差异表达UCF-miRNA相对表达量。尿液miRNA使用mirVanaTMPairs miRNA isolation kit(Ambion)试剂盒抽提。以10ng总RNA为模板合成cDNA,使用MMLV反转录酶试剂盒(Thermo),根据mirBASE提供的miRNA种子区序列,设计茎环反转录引物(北京六合华大基因公司合成),反应条件:65℃加热5min,置冰中2min。然后放入 PCR仪16℃热激30min,42℃ 60min,85℃,5min 停止反转录。Bio-Rad iCycler PCR System进行qRT-PCR检测(PCR相关试剂均购自天根生化科技有限公司)。反应体系按照SYBR Premix Ex TaqTMⅡ(Perfect Real Time)说明书进行配置。使用Bio-Rad iCycler PCR System进行 Real Time PCR反应,程序:95℃ 5min;95℃ 10s,60℃ 30s,40cycle;以上循环结束后进行65℃-95℃的融解曲线分析。每个样品平行三次,溶解曲线为单一峰。按照2-ΔΔCt计算样品的相对表达丰度。
1.5 UCF-miRNA差异表达谱的获得及其与有氧耐力指标的相关性分析
(1)计算训练前后R-RE变化率η(R-RE),采用K-means法进行聚类分析,根据聚类分析结果将学员分为对有氧训练的①高敏感表型组(High Response genotype Group,以下简称HR组)和②普通敏感表型组(Common Response genotype Group,以下简称CR组)。比较两组UCF-miRNA表达水平,提取HR组UCF-miRNA差异表达谱。以相对于基线表达值比率升高或降低超过2倍为显著性差异表达,比较 HR组和CR组UCF-miRNAs表达谱,提取HR组的UCF-miRNAs特征性表达谱。
(2)UCF-miRNA特征性表达谱/R-RE关联模型的miRNA入选标准:(1)microRNA芯片检测结果与qRT-PCR验证结果一致;(2)UCF-miRNA的基线表达值在HR组和CR组之间无显著性差异(±10%以内);(3)文献显示该miRNA与运动性适应相关的生理过程存在关联。
(3)计算HR组差异表达的UCF-miRNA在基线值-0h阶段(以下简称为阶段Ⅰ)和0h-24h阶段(以下简称为阶段Ⅱ)的表达量变化率η(UCF-miRNA):
η(UCF-miRNA)=[(阶段终点表达量——阶段起点表达量)/阶段起点表达量]×100%
(4)计算η(UCF-miRNA)与各有氧耐力指标变化率之间的Pearson相关系数,分析其相关性。
1.6 数据处理与分析
采用SPSS 17.0软件对结果进行处理。各项指标数据均采用“±s”表示。有氧耐力训练前后各项指标的变化率η(Χ)=[(训练值-基础值)/基础值]×100%。均数间比较采用两独立样本的t检验分析,P<0.05表示两组间的差异有统计学意义。聚类分析采用k-means法。两组数据之间的相关性分析采用Bivariate过程,计算Pearson相关系数并用r表示。
2 研究结果
2.1 参训学员各项指标在17周有氧训练前后的变化
参训者在17周有氧训练前后PB5000、VT和RE水平均有显著提高(P<0.05),具体数据见表2、表3。
表2 有氧训练前后参训者PB5000和有氧耐力指标比较±s)
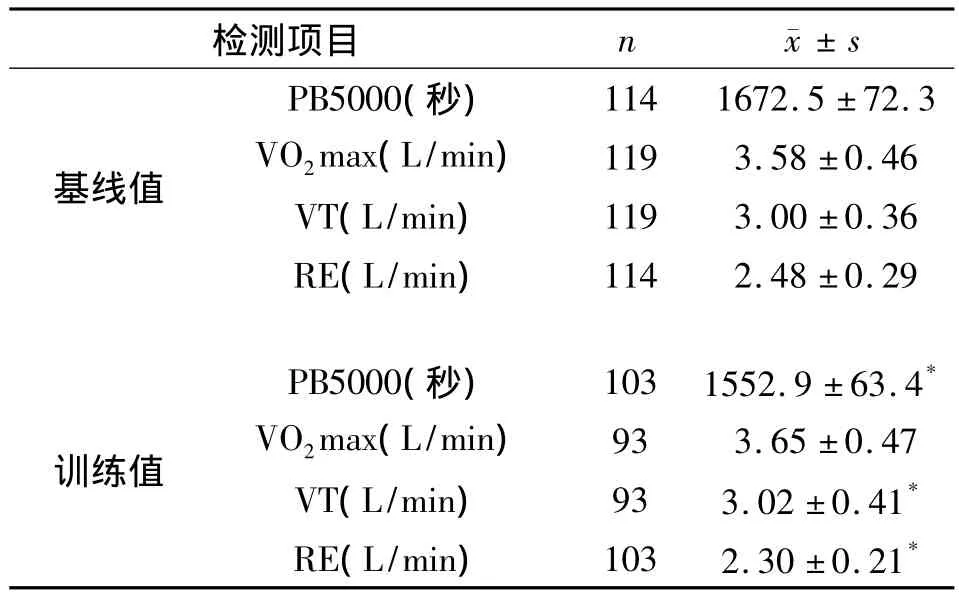
表2 有氧训练前后参训者PB5000和有氧耐力指标比较±s)
注:*P<0.05表示有显著性差异,下同。
检测项目n±s基线值PB5000(秒) 114 1672.5±72.3 VO2 max(L/min) 119 3.58±0.46 VT(L/min) 119 3.00±0.36 RE(L/min) 114 2.48±0.29训练值PB5000(秒) 103 1552.9±63.4*VO2max(L/min) 93 3.65±0.47 VT(L/min) 93 3.02±0.41*RE(L/min) 103 2.30±0.21*
表3 有氧训练对参训者R-VO2 max、R-VT和R-RE的影响(±s)

表3 有氧训练对参训者R-VO2 max、R-VT和R-RE的影响(±s)
检测项目 训练前 训练后 变化率PB5000 1672.5±72.3 1552.9±63.4* -0.70±0.05 R-VO2 max(mL/kg/min) 55.22±5.88 57.58±7.16* 0.029±0.033 R-VT(mL/kg/min) 45.98±4.40 46.90±6.44 0.009±0.052 R-RE(mL/kg/min) 38.41±3.52 35.98±4.05*-0.051±0.078
2.2 训练前后PB5000变化率与有氧耐力指标变化率的相关性分析
参训者训练前后PB5000变化率与RE和RRE变化率相关性最强,相关系数分别为0.721(RE)和0.763(R-RE);与 VT(r=-0.630)、RVT(r=-0.685)和R-VO2max(r=0.670)的变化率具有较强的相关性;与VO2max(r=-0.264)变化率呈弱相关。
2.3 对R-RE变化率进行聚类分析结果
k-mean法对参训学员17周训练后R-RE相对于基线值变化百分率进行聚类分析,将实验对象分为HR组和CR组,聚类结果见图1。
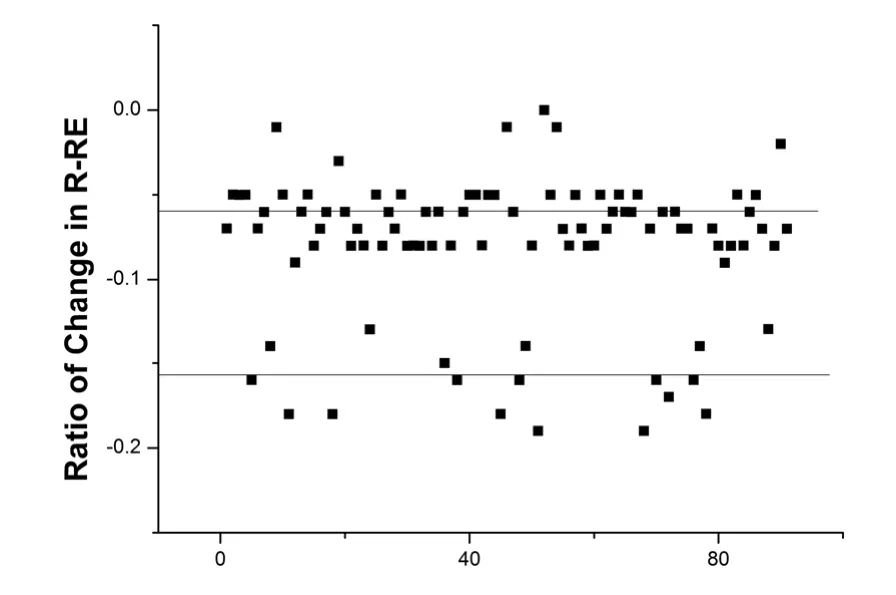
图1 k-mean法对参训学员17周训练后R-RE相对于基线值变化百分率聚类分析散点图。聚类中心分别为15.9%(HR group)和6.7%(CR group)。
比较两组间R-RE变化率,HR组显著高于CR组(P<0.05),具体结果见表7。
表4 聚类分析后各组参训学员R-RE相对于基线值变化百分率比较(±s)

表4 聚类分析后各组参训学员R-RE相对于基线值变化百分率比较(±s)
组别 n Class center R-RE HR group 20 -0.159 -0.159±0.03*CR group 71 -0.067 -0.067±0.02
2.4 HR组UCF-miRNA特征性表达谱
与CR组表达水平相比较,HR组差异表达12条UCF-miRNA。查询文献确认已知功能与运动性适应有关,经qRT-PCR确认表达模式与基因芯片结果一致。其中8条以上调为主要特征,4条以平稳或下调为主要特征[15-25]。UCF-miRNAs表达模式特征分别见图2(上调)和图3(下调或保持平稳)。
2.5 差异表达的UCF-miRNA与训练前后PB5000变化率及R-RE变化率的相关性分析
分别检测各个差异表达的UCF-miRNA在阶段Ⅰ和阶段Ⅱ相对表达量变化率与训练前后PB5000变化率和R-RE变化率之间的Pearson相关系数,结果表明:(1)阶段ⅠmiR181、miR9和miR34a的变化率与训练前后PB5000变化率相关系数分别是 rmiR181=0.799、rmiR9=0.645、rmiR34a=0.677(P<0.05);与训练前后R-RE变化率与PB5000变化率相关系数分别是 rmiR181=0.795、rmiR9=0.700、rmiR34a=0.751(P <0.05);(2)阶段 ⅡmiR378、miR 126、miR 222、miR 146a、miR 21、miR 696和miR 192的变化率与训练前后R-RE变化率相关系数分别是rmiR378=0.682、rmiR126=0.723、rmiR222=0.563、rmiR1146a=0.577、rmiR21=0.522、rmiR696=0.623、rmiR192=0.842(P <0.05)。
3 分析与讨论
本研究探讨了运动性适应过程中UCF-miRNA表达谱与有氧运动能力的可训练性之间的关联,发现:具有显著高于普通水平的R-RE变化率表型的个体在单次训练过程中有12个UCF-miRNA显示出明显特征性的表达模式,文献表明,该组中miRNA参与调控运动性适应的基本生理过程。本研究结果显示:该组UCF-miRNA表达量变化率与17周有氧训练前后PB5000和R-RE等有氧耐力指标的变化率具有显著的相关性。表明HR组UCF-miRNA特征性表达谱与有氧运动能力的可训练性水平具有显著的关联性,可用来建立关联模型对其加以鉴别和评估。本研究结果提示:运动能力的可训练性水平与miRNA调控基因选择性表达的模式有关,可以通过特征性UCF-miRNA表达谱对其加以分辨和评估。可以利用特征性的UCF-miRNA表达谱在有氧能力发展敏感期准确预测其发展潜力,将具有相关运动天赋的运动员早期选拔出来加以系统培养。
运动训练性敏感性与机体对训练应激的适应能力密切相关,表观遗传学基因调控机制在机体响应环境压力的适应性重塑过程中起到关键性作用,其调控模式决定了基因表达的选择性和表达效率,进而影响机体对环境因素的适应能力[26]。miRNAs编辑作用是表观遗传学基因调控的重要途径,可能是导致运动能力及其可训练性个体差异主要原因[27]。临床研究已经证实:尿液内 UCF-miRNA与机体生理病理状态密切关联,可以作为疾病诊断、病因分析和预后判断的生物标记。R-RE反映了机体的氧利用效率,是评估有氧耐力的重要指标[13],本研究以R-RE变化率为有氧能力的可训练性的标志将实验对象分为HR组和CR组,提取HR组UCF-miRNA特征性表达谱进行比较分析。结果显示:HR组差异表达的12条UCF-miRNA均与运动性适应过程中的基因表达调控有关,其调控作用覆盖了蛋白质合成、细胞能量代谢、线粒体生物合成、低氧适应等适应反应通路,显著改变心肌骨骼肌组织对机械应力的响应及重塑、线粒体有氧代谢能力及抗自由基损伤、糖脂代谢酶活性、运动性疲劳耐受和恢复等生物过程的基因表达时序性和表达水平,其作用特点与较高的有氧耐力训练敏感性存在关联。HR组UCF-miRNAs特征表达谱显示的运动性适应有关的代谢调控特征符合我们对优秀运动员的训练适应特点的经验性了解,说明UCF-miRNA表达特征与运动性适应之间存在本质上的一致性。
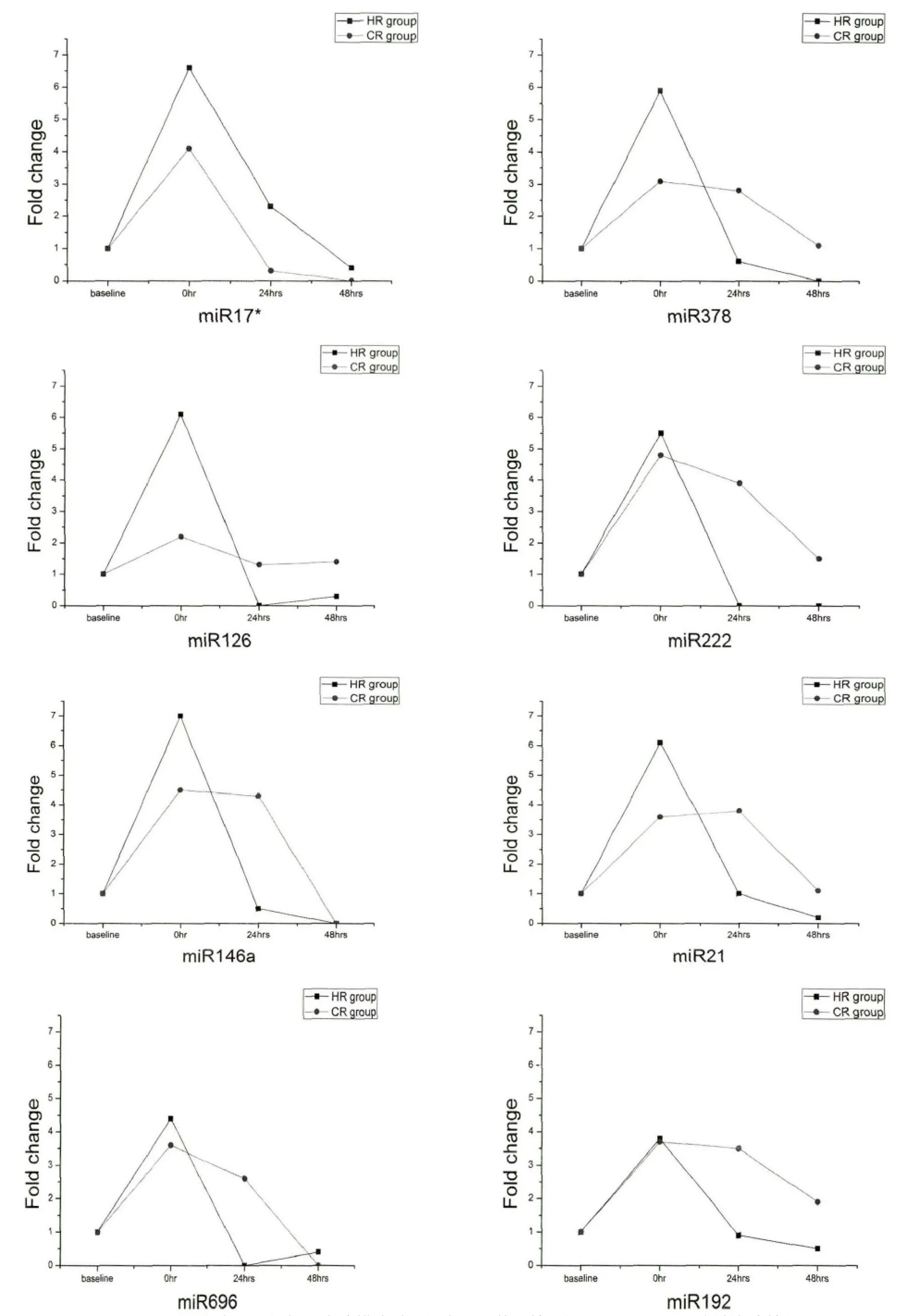
图2 HR组与CR组相比表达模式以上调为主要差异特征的UCF-miRNAs连续表达特征
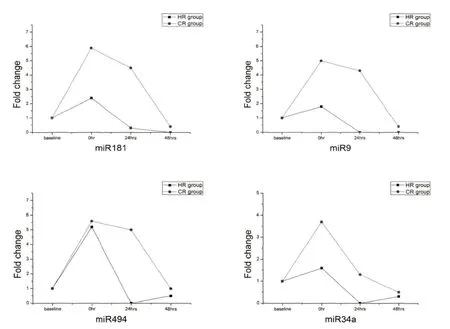
图3 HR组与CR组相比表达模式以平稳或下调为主要差异特征的UCF-miRNAs连续表达特征
本研究分析单次运动训练过程中三个特征阶段的UCF-miRNA表达量变化率与有氧运动能力各项指标之间的相关性。结果显示:在阶段Ⅰ,miR133、miR9、miR34a表达量变化率与17周有氧训练前后PB5000和R-RE的提高率具有显著的相关性;在阶段Ⅱ,miR378、miR126、miR222、miR146a、miR21、miR696、miR192表达量变化率与17周有氧训练前后R-RE的提高率具有显著的相关性。Boggish等[18]的研究表明 miR146a表达水平与VO2max水平具有正相关关系,本研究的结果与之相近。
关于UCF-miRNA的表达行为机理及其与组织miRNA和循环miRNA的确切关系目前尚不清楚,目前无法对结果进行深入的功能性解读,本研究结果目前只能作为有氧能力可训练性的生物标记。对于本研究发现的UCF-miRNA表达谱反映的运动性适应调控机理的深入分析有待于进一步研究加以阐明。
本研究以尿液UCF-miRNA表达谱作为生物标记鉴别与分析有氧运动能力的可训练性的表观遗传学调控模式。并且建立了UCF-miRNA与有氧能力的训练敏感性的关联模型和一种简便易行的无创检测方案,可以用于早期评估有氧运动能力的发展潜力和训练适应状态。
4 研究结论
本研究发现典型有氧训练周期的单次训练过程中UCF-miRNAs表达谱与有氧运动能力的训练敏感性之间存在密切的联系并初步建立了关联模型,同时也提出了一种采用表观遗传学指标进行运动员选材和指导训练的无创检验方法,以UCF-miRNAs表达特征作为生物标记评估运动能力的发展潜力和训练适应状态。
[1]马力宏.人类体质与运动能力的遗传学基础[J].天津体育学院学报,2001,16(4):1-5.
[2]于红.表观遗传学:生物细胞非编码RNA调控的研究进展[J].遗传,2009(11):1077-1086.
[3]刘海平,胡杨,汪洪波.低氧诱导因子-1a基因C958G单核苷酸多态性与有氧运动能力关联性研究[J].体育科学,2008,28(7):61-65.
[4]郑永霞,焦炳华.miRNA的生物形成及调控基因表达机制[J].生命的化学,2010(6):821-826.
[5]Williams AH,Liu N,van Rooij E et al.MicroRNAControl of Muscle Development and Disease[J].Curr Opin Cell Biol 2009,21(4):461-469.
[6]Zhang C.MicroRNAs inVascular Biology and Vascular Disease[J].J Cardiovasc Transl Res,2010,3(3):235-240.
[7]Dang CV.Re-Thinking the Warburg Effect with Mycmicroma Naging Glutamine Metabolism[J].Cancer Res 2010,70(5):859-862.
[8]Bartel,D.P.MicroRNAs:Genomics,Biogenesis,Mechanism,and Function[J].Cell,2004,116(2):281-97.
[9]Kosaka N,Iguchi H,Ochiya T.CirculatingMicroRNA in Body Fluid:A New Potential Biomarker for Cancer Diagnosis and Prognosis[J].Cancer Sci,2010,101(10):2087-2092.
[10]李锦,白雪源.血和尿中miRNA作为疾病诊断的特异性标志物[J].医学综述,2010,16(19):2881-2883.
[11]李义志,骆军保,汤长发.HSP1A1基因+190G>C多态与有氧运动能力表型指标的关联性研究[J].天津体育学院学报,2013(2):180-184.
[12]聂晶,胡扬,何子红,等.VEGF及其受体基因多态性与有氧运动能力表型的关联研究[J].体育科学,2010,30(8):77-85.
[13]王国军,席翼,武雅琼.我国普通男性青年跑节省化(RE)测试方法的建立[J].中国体育科技,2013(1):34-40.
[14]王鹏宇.1500m耐力训练中实施心率和血乳酸监测的研究[J]. 吉林体育学院学报,2013,29(3):61-64.
[15]Xu Y,Fang F,Zhang J,et al.MiR-17 Suppresses Tumorigenicity of Prostate Cancer by Inhibiting Mitochondrial Antioxidant Enzymes[J].PLoSOne 2010,5(12):e14356.
[16]Rottiers V,N r AM.MicroRNAs inMetabolism and Metabolic Disorders[J].Nat Rev Mol Cell Biol,2012,13(4):239-250.
[17]Fernandes T,Hashimoto NY,Magalhaes FC,et al.AerobicExercise Training-Induced Left Ventricular Hypertrophy Involves Regulatory Micrornas,Decreased Angiotensin-Converting Enzyme-Angiotensin II,and Synergistic Regulation of Angiotensin-Converting Enzyme 2-Angiotensin[J].Hypertension,2011,58(2):182-9.
[18]Baggish AL,Hale A,Weiner RB,et al.DynamicRegulation of Circulating Microrna During Acute Exhaustive Exercise and Sustained Aerobic Exercise Training[J].J Physiol,2011,589(16):3983-94.
[19]Weber M,Baker M,Moore J,et al.MiR-21 isInduced in Endothelial Cells by Shear Stress and Modulates Apoptosis and eNOS Activity[J].Biochemical and Biophysical Research Communications,2010,393(4):643-648.
[20]Aoi W,Naito Y,Mizushima K,et al.TheMicroRNA MiR-696 Regulates PGC-1αin Mouse Skeletal Muscle in Response to Physical Activity[J].Am J Physiol Endocrinol Metab,2010,298(4):E799-806.
[21]Putta S,Lanting L,Sun G,et al.Natarajan InhibitingMicroRNA-192 Ameliorates Renal Fibrosis In Diabetic Nephropathy [J].J Am Soc Nephrol,2012,23(3):458-469.
[22]Godfrey R,Theologou T,Dellegrottaglie S,et al.TheEffect of High-Intensity Aerobic Interval Training on Postinfarction Left Ventricular Remodelling[J].BMJ Case Rep,2013,[Epub ahead of print]
[23]Leung AKL,Sharp PA.MicroRNAFunctions in Stress Responses[J].Molecular Cell,2010,40(2):205-215.
[24]Alexander MS,Shi X,Voelker KA,et al.Foxj3Transcriptionally Activates Mef2c and Regulates Adult Skeletal Muscle Fiber Type Identity[J].Dev Biol,2010,337(2):396-404,.
[25]Mangala LS,Zhang Y,He Z,et al.Effects ofSimulated Microgravity on Expression Profile of MicroRNA in Human Lymphoblastoid Cells[J].JBiol Chem,2011,286(37):32483-32490.
[26]冀国华,吕柯,曲丽娜,李莹辉.应激诱导的MicroRNAs调控作用研究进展[J].航天医学与医学工程,2012(6):449-453.
[27]Bye A,R sj H,Aspenes ST,et al.CirculatingMicroRNAs and Aerobic Fitness——the HUNT-Study[J].PLoS One,2013,8(2):e57496.
