亚麻MAPK基因克隆及盐碱胁迫下的表达分析
2015-11-11
亚麻MAPK基因克隆及盐碱胁迫下的表达分析
于莹1,陈宏宇2,程莉莉1,赵东升1,袁红梅1,吴广文1,关凤芝1*
(1.黑龙江省农业科学院经济作物研究所,哈尔滨150086;2.东北农业大学生命科学学院,哈尔滨150030)
摘要:为研究MAPK基因与亚麻应答盐碱胁迫的关系,利用生物信息学方法从亚麻基因组中预测得到20个亚麻MAPK基因,命名为LuMPK1~LuMPK20,这20个MAPK基因都含有T[D/E]Y保守结构域,除LuMPK5和LuMPK20以外,其他基因都是两两一组。利用PCR技术克隆得到5个亚麻MAPK基因(LuMPK3、LuMPK8、LuMPK11、LuMPK14、LuMPK16),这5个基因序列与预测序列完全一致。利用数字基因表达谱数据,分析亚麻盐碱胁迫下这5个基因的表达情况。结果表明,这5个基因的表达均受到盐碱胁迫影响。LuMPK3、LuMPK11、LuMPK14、LuMPK16在中性盐胁迫下上调表达,LuMPK3、LuMPK8、LuMPK11、LuMPK14在碱性盐胁迫下上调表达,LuMPK16在碱性盐胁迫下下调表达。研究为亚麻MAPK基因功能,尤其是耐盐碱功能研究奠定理论基础。
关键词:亚麻;MAPK;基因克隆;盐碱胁迫;表达分析
网络出版时间2015-3-13 15:22:00
[URL]http://www.cnki.net/kcms/detail/23.1391.S.20150313.1522.009.html
于莹,陈宏宇,程莉莉,等.亚麻MAPK基因克隆及盐碱胁迫下的表达分析[J].东北农业大学学报, 2015, 46(3): 21-28.
亚麻是一种世界性纤维作物和油料作物,在我国经济作物生产中具有重要地位[1]。近年来,耕地面积萎缩,为解决麻类作物与粮食作物争地矛盾,麻类作物走出粮田,拓展种植区域,向非耕地发展是麻类作物可持续发展的必由之路。亚麻种植向盐碱地发展对保证国家粮食安全和亚麻纤维供应具有重要意义。我国东北地区是世界三大盐碱土集中分布区之一[2],土壤中的致害盐类除以NaCl为主的中性盐,还有以Na2CO3和NaHCO3为主的碱性盐[3]。但关于植物碱性盐胁迫的相关研究报道较少。
促有丝分裂原活化蛋白激酶(Mitogen-activat⁃ed protein kinases, MAPKs)是植物外界刺激信号传导物质,能将外界各种刺激信号,包括高盐胁迫、渗透胁迫、低温胁迫等传导到细胞内,引起相应基因表达[4]。MAPKs与其他信号分子组成MAPK级联途经,通过依次磷酸化将逆境信号传递下去,即MAPKs→MAPKKs→MAPKKKs[5]。
目前,科学家已从拟南芥[6]、水稻[7]、玉米[8]、小麦[9]、大麦[10]、燕麦[11]、烟草[12]等植物中分离得到MAPK基因。研究表明,MAPK基因与植物耐盐性有密切关系[13-15]。但是,亚麻MAPK基因还没有相关报道。
本研究从亚麻基因组中预测得到20个亚麻MAPK基因,命名为LuMPK1~LuMPK20,分析这些基因结构、蛋白结构、亚细胞定位及进化关系等信息。利用PCR技术克隆得到5个亚麻MAPK基因,利用数字基因表达谱(DGE)数据,分析盐碱胁迫下这5个基因的表达情况,可为亚麻MAPK基因功能的研究奠定理论基础。
1 材料与方法
1.1材料
植物材料种植及胁迫处理:亚麻(Linum usita⁃tissimum L.)种子由黑龙江省农业科学院经济作物研究所提供。将亚麻种子播种于装有蛭石花盆中,待生长3周后(约10 cm),移入含有25 mmol·L-1Na2CO3胁迫溶液中处理18 h后取样,提取总RNA,并反转录成cDNA,留作基因克隆的模板。总RNA提取采用原平皓植物RNA快速提取试剂盒(HF109-02),反转录采用东洋纺SYBR Green Re⁃altime PCR Master Mix试剂盒(QPK-201T)。
1.2方法
1.2.1亚麻MAPK基因的预测
从phytozome网站(http://www.phytozome.net/)下载亚麻蛋白质数据进行本地化。利用已经报道的其他植物MAPK蛋白序列进行多序列比对,构建MAPK训练集。利用隐马尔科夫模型(Hidden markov model, HMM)[16]和MAPK训练集搜索本地亚麻蛋白质数据库,获得可能的亚麻MAPK基因。利用PLANTSP程序(http://plantsp.genomics.purdue. edu/)检测亚麻MAPK基因保守结构域。
1.2.2亚麻MAPK基因结构分析、序列比对及进化树构建
亚麻MAPK基因结构分析在phytozome网站(http://www.phytozome.net/)上进行。在线预测亚麻MAPK蛋白分子质量和等电点(http://web.expasy.org/compute_pi/)。亚细胞定位分析采用在线程序CEL⁃LO v.2.5 server(http://cello.life.nctu.edu.tw/)[17]。利用DNAMAN软件对亚麻MAPK蛋白序列进行多序列比对。为研究亚麻MAPK基因的进化关系,利用MEGA v5软件将20个拟南芥MAPK基因与20个亚麻MAPK基因构建进化树[18]。1.2.3亚麻MAPK基因的克隆
根据预测亚麻MAPK基因序列,利用Primer 5.0软件设计引物,引物序列见表1。

表1 亚麻MAPK基因的引物序列Table 1 Primer sequences of flax MAPK genes
PCR反应程序:94℃5 min;94℃30 s,58℃30 s,72℃2 min(30个循环);72℃10 min。将PCR扩增产物进行测序,利用DNAMAN 6.0软件对PCR产物测序结果进行同源性比对分析。PCR扩增采用东洋纺KOD Plus Neo试剂盒(Lot: 266600),PCR反应体系按照试剂盒说明书进行。引物合成及测序工作由江苏金唯智公司完成。
1.2.4亚麻MARK基因在盐碱胁迫下的表达分析
盐碱胁迫下亚麻数字基因表达谱(DGE)数据[19]被用来研究亚麻MAPK基因在盐碱胁迫下的表达模式。DGE测序的亚麻样品盐碱胁迫处理条件为25 mmol·L-1Na2CO3(pH 11.6)(Alkaline salt stress, AS2)、50 mmol·L-1NaCl(Neutral salt stress, NSS)和6 mmol·L-1NaOH(pH 11.6)(Alkaline stress, AS),蒸馏水作对照。
2 结果与分析
2.1亚麻MAPK基因的预测
利用隐马尔科夫模型(HMM)预测亚麻MAPK基因,结果得到24个搜索结果高于600分,被初步认为是可能的亚麻MAPK基因(见图1)。采用在线软件PLANTSP检测亚麻MAPK保守结构域,结果有4个基因无MAP激酶结构域(Lus10021945、Lus10034601、Lus10038472、Lus10041234),说明可能的亚麻MAPK基因有20个。按照HMM预测的分值,从高到低,将亚麻MAPK基因命名为LuMPK1~LuMPK20。
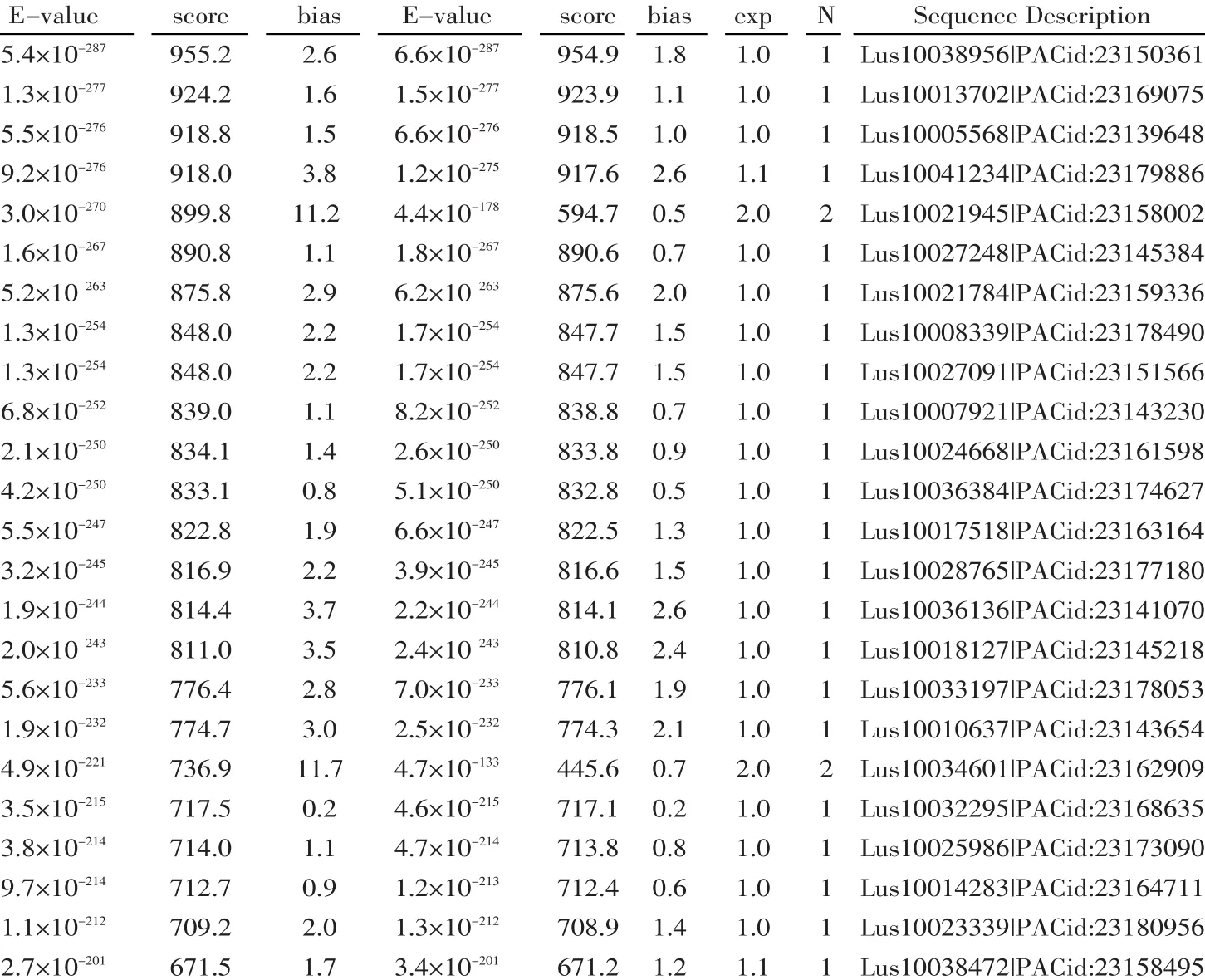
图1 亚麻MAPK基因的隐马尔科夫模型(HMM)预测结果Fig. 1 HMM predicted results of MAPK gene from flax
2.2亚麻MAPK基因结构分析、序列比对及进化树构建
对20个亚麻MAPK基因进行结构分析(见表2)。结果表明,所有基因均含有内含子,但是数目为2~11个不等。基因长度为1 431~14 267 bp,有10倍的差距,但是CDS长度为1 104~1 851 bp,相差不大,说明主要是内含子长度差别较大。蛋白长度为367~616 aa,其中有5对基因的长度相同(LuMPK6/LuMPK7、LuMPK8/LuMPK20、LuMPK11/LuMPK12、LuMPK13/LuMPK14、LuMPK15/LuMPK 16)。蛋白分子质量为42.2~69.7 ku,等电点为5.46~ 9.21。其中有4个基因的等电点大于7(LuMPK2、LuMPK3、LuMPK18、LuMPK19),说明其属于碱性蛋白。亚麻基因组测序数据没有染色体定位信息,所以按照scaffold信息定位基因。亚细胞定位预测结果表明,5个基因定位在细胞质、细胞核、线粒体上,9个基因定位在细胞核中,6个基因定位在细胞质中。
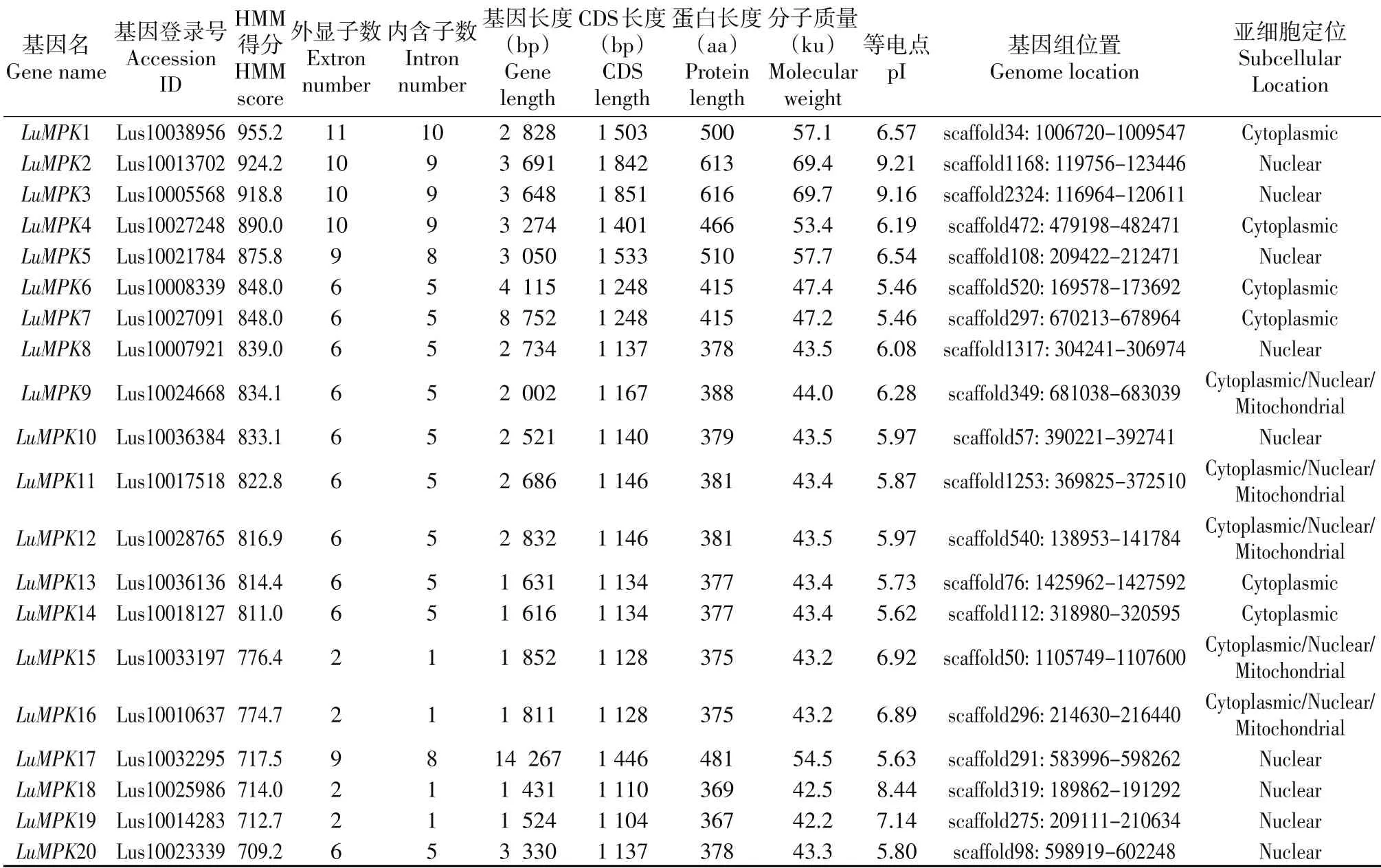
表2 亚麻MAPK基因结构分析Table 2 List of MAPK gene structure in flax
对20个亚麻MAPK基因进行序列比对分析(见图2)。结果表明,所有亚麻MAPK基因均含有植物MAPK基因特有的T[D/E]Y保守结构域,其中有5个基因含有TDY结构域,15个基因含有TEY结构域。将20个亚麻MAPK基因与20个拟南芥MAPK基因构建进化树(见图3)。
图3结果表明,20个亚麻MAPK基因被分为4个组(A~D),其中5个在A组,6个在B组,4个在C组,5个在D组。除LuMPK5和LuMPK20以外,其他基因均两两一组。

图2 亚麻MAPK蛋白序列比对Fig. 2 Alignment of MAPK proteins in flax

图3 亚麻MAPK蛋白与拟南芥MAPK蛋白进化关系分析Fig. 3 Phylogenetic relationships of MAPK families between flax and Arabidopsis
2.3亚麻MAPK基因的克隆
利用基因特异引物和PCR方法,从盐碱胁迫后亚麻全植株中提取总RNA并反转录(见图4A)。扩增得到目的基因产物,进行琼脂糖凝胶电泳检测(见图4B~F)。
结果表明,分别克隆得到1个约2 000 bp和4个约1 200 bp的目的条带。将PCR产物切胶回收,并测序。测序结果比对证实PCR扩增产物为Lus10005568(LuMPK3)、Lus10007921(LuMPK8)、Lus10017518(LuMPK11)、Lus10018127(LuMPK14)和Lus10010637(LuMPK16)的完整序列。
2.4亚麻MAPK基因在盐碱胁迫下的表达分析
利用DGE测序数据分析亚麻LuMPK3、LuMPK8、LuMPK11、LuMPK14、LuMPK16基因在盐碱胁迫下的表达模式(见图5)。
结果表明,LuMPK3、LuMPK8、LuMPK11、LuMPK14、LuMPK16这5个基因的表达均受到盐碱胁迫影响。LuMPK3、LuMPK11、LuMPK14、LuMPK16在中性盐胁迫下上调表达,LuMPK3、LuMPK8、LuMPK11、LuMPK14在碱性盐胁迫下上调表达,LuMPK16在碱性盐胁迫下下调表达。说明这5个基因可能与亚麻应答盐碱胁迫密切相关。
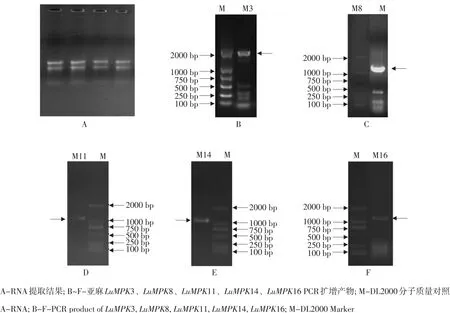
图4 亚麻MAPK基因的克隆Fig. 4 Cloning of MAPKgene in flax

图5 亚麻LuMPK3、LuMPK8、LuMPK11、LuMPK14、LuMPK16基因在盐碱胁迫下的表达模式Fig. 5 Expression pattern of LuMPK3, LuMPK8, LuMPK11, LuMPK14, LuMPK16 under saline and alkaline stress
3 讨论与结论
促有丝分裂原活化蛋白激酶是一类丝氨酸/苏氨酸蛋白激酶,广泛存在于植物、动物和真菌中。植物MAPK是重要信号传导物质,在植物应答非生物逆境过程中发挥重要作用。目前,许多植物MAPK基因均已被分离出来,其中拟南芥有20 个[6],水稻有17个[7],玉米有19个[8],杨树21个[20],烟草17个[12],番茄16个[21],苹果26个[22]。本研究从亚麻基因组中预测得到20个MAPK基因,与拟南芥中数目一致。亚麻MAPK基因中有5个含有TDY结构域,15个含有TEY结构域;水稻中有11个含有TDY结构域,5个含有TEY结构域;拟南芥中有8个含有TDY结构域,12个含有TEY结构域。说明不同物种间MAPK基因数目和结构域分类不同,这些基因可能参与植物不同的代谢活动。
通过构建进化树,发现亚麻MAPK基因大多数均是两两一组,每一组基因序列均非常相似,这些基因在进化上可能是复制而来。很难利用荧光定量PCR方法设计引物区分不同基因表达模式。所以,采用DGE测序数据分析亚麻MAPK基因的表达模式。利用PCR方法只克隆得到5个亚麻MAPK基因,说明其他MAPK基因在样品中没有表达或表达量太低,也可能是假基因或者基因组测序拼接不准确及PCR技术本身原因。
近年来,许多研究发现在逆境胁迫下植物MAPK基因对盐胁迫会做出积极应答[23-24]。本文通过对5个亚麻MAPK基因在盐碱胁迫下的表达分析,发现这5个基因的表达均受盐碱胁迫影响。A组和B组MAPK被认为与植物应答环境胁迫相关[25],参与拟南芥应答环境胁迫的AtMPK4就属于B组[26],本研究中LuMPK14属于A组,LuMPK8和LuMPK11属于B组,C组MAPK研究较少,但是本研究中LuMPK16属于C组,其表达也受盐碱胁迫影响,有报道D组MAPK与非生物胁迫有关[27-28],本研究中LuMPK3属于D组。因此,推测A~D组MAPK在亚麻响应盐胁迫过程中发挥重要作用。这些基因具体功能及与亚麻应答胁迫分子机制关系有待进一步研究。
为研究MAPK基因与亚麻应答盐碱胁迫关系,本研究利用生物信息学方法从亚麻基因组数据库预测得到20个亚麻MAPK基因,命名为LuMPK1~LuMPK20,分析这20个MAPK基因的基因结构、蛋白结构、亚细胞定位及进化关系等信息。利用PCR技术克隆得到5个亚麻MAPK基因(LuMPK3、LuMPK8、LuMPK11、LuMPK14、LuMPK16)全长。利用DGE数据,分析盐碱胁迫下这5个基因表达情况。结果表明,5个基因的表达均受到盐碱胁迫影响。
[参考文献]
[1]杨学,李柱刚.亚麻学[M].哈尔滨:黑龙江科学技术出版社, 2009: 3-5.
[2]王春裕,武志杰,石元亮,等.中国东北地区的盐渍土资源[J].土壤通报, 2004, 35(5): 643-647.
[3]杨书华,张春宵,朴明鑫,等. 69份玉米自交系的苗期耐盐碱性分析[J].种子, 2011, 130(3): 1-6.
[4]Rodriguez M C, Petersen M, Mundy J. Mitogen-activated protein kinase signaling in plants[J]. Annu Rev Plant Biol, 2010, 61: 621-649.
[5]Nakagami H, Pitzschke A, Hirt H. Emerging MAP kinase path⁃ways in plant stress signalling[J]. Trends in Plant Sci, 2005(10): 1360-1385.
[6]Mizoguchi T, Hayashida N, Yamaguchi-Shinozaki K, et al. AT⁃MPKs: A gene family of plant MAP kinases in Arabidopsis thali⁃ana[J]. FEBS Lett, 1993, 336: 440-444.
[7]Lieberherr D, Thao N P, Nakashima A, et al. A sphingolipid elici⁃tor-inducible mitogen-activated protein kinase is regulated by the small GTPase OsRac1 and heterotrimeric G-protein in rice[J]. Plant Physiol, 2005, 138: 1644-1652.
[8]Lalle M, Visconti S, Marra M, et al. ZmMPK6, a novel maize MAP kinase that interacts with 14-3-3 proteins[J]. Plant Mol. Biol, 2005, 59: 713-722.
[9]Takezawa D. Elicitor- and A23187-induced expression of WCK-1, a gene encoding mitogen-activated protein kinase in wheat[J]. Plant Mol Biol, 1999, 40: 921-933.
[10]Knetsch M, Wang M, Snaar-Jagalska B E, et al. Abscisic acid in⁃duces mitogen-activated protein kinase activation in barley aleu⁃ron protoplasts[J]. Plant Cell, 1996(8): 1061-1067.
[11]Huttly A K, Phillips A L. Gibberellin-regulated expression in oat aleurone cells of two kinases that show homology to MAP kinase and a ribosomal protein kinase[J]. Plant Mol Biol, 1995, 27: 1043-1052.
[12]Wilson C, Anglmayer R, Vicente O, et al. Molecular cloning, func⁃tional expression in Escherichia coli, and characterization of multi⁃ple mitogen-activated protein kinases from tobacco[J]. Eur J Bio⁃chem, 1995, 233: 249-257.
[13]Jonak C,Ökrész L, Bögre L, et al. Complexity, cross talk and inte⁃gration of plant MAP kinase signaling[J]. Curr Opin Plant Biol, 2002(5): 415-424.
[14]Singh R, Lee M O, Lee J E, et al. Rice mitogen-activated protein kinase interactome analysis using the yeast two-hybrid system[J]. Plant Physiol, 2012, 160: 477-487.
[15]陈宏宇,冯树丹,刘相国,等. NaCl和Na2CO3胁迫下的高粱MAPKs基因家族表达模式[J].吉林大学学报:理学版, 2012, 50 (1): 139-146.
[16]Eddy S R. Profile hidden Markov models[J]. Bioinformatics, 1998, 14: 755-763.
[17]Yu C S, Chen Y C, Lu C H. Prediction of protein subcellular local⁃ization[J]. Proteins 2006, 64: 643-651.
[18]Tamura K, Peterson D, Peterson N, et al. MEGA5: Molecular evo⁃lutionary genetics analysis using maximum likelihood, evolution⁃ary distance, and maximum parsimony methods[J]. Mol Biol Evol, 2011, 28: 2731-2739.
[19]Yu Y, Huang W, Chen H, et al. Identification of differentially ex⁃pressed genes in flax (Linum usitatissimum L.) under saline-alka⁃line stress by digital gene expression[J]. Gene, 2014, 549: 113-122.
[20]Nicole M C, Hamel L P, Morency M J, et al. MAP-ping genomic organization and organ-specific expression profiles of poplar MAP kinases and MAP kinase kinases[J]. BMC Genom, 2006(7): 223.
[21]Kong F, Wang J, Cheng L, et al. Genome-wide analysis of the mi⁃togen-activated protein kinase gene family in Solanum lycopersi⁃cum[J]. Gene, 2012, 499: 108-120.
[22]Zhang S, Xu R, Luo X, et al. Genome-wide identification and ex⁃pression analysis of MAPK and MAPKK gene family in Malus do⁃mestica[J]. Gene, 2013, 531: 377-387.
[23]Xu H, Sun X, Wang X, et al. Involvement of a cucumber MAPK gene (CsNMAPK) in positive regulation of ROS scavengence and osmotic adjustment under salt stress[J]. Sci Hortic-Amsterdam, 2011, 127: 488-493.
[24]Zhang J, Zou D, Li Y, et al. GhMPK17, a cotton mitogen-activat⁃ed protein kinase, is involved in plant response to high salinity and osmotic stresses and ABA signaling[J]. PloS One, 2014(9): e95642.
[25]MAPK Group. Mitogen-activated protein kinase cascades in plants: A new nomenclature[J]. Trends Plant Sc, 2002(7): 301-308.
[26]Kosetsu K, Matsunaga S, Nakagami H, et al. The MAP kinase MPK4 is required for cytokinesis in Arabidopsis thaliana[J]. Plant Cell, 2010, 22: 3778-3790.
[27]Jammes F, Song C, Shin D, et al. MAP kinases MPK9 and MPK12 are preferentially expressed in guard cells and positively regulate ROS-mediated ABA signaling[J]. Proc Natl Acad Sci, 2009, 106: 20520-20525.
[28]Shi J, Zhang L, An H, et al. GhMPK16, a novel stress-responsive group D MAPK gene from cotton, is involved in disease resistance and drought sensitivity[J]. BMC Mol Biol, 2011(12): 22.
Yu Ying, Chen Hongyu, Cheng Lili, et al. Cloning and expression analysis of MAPK gene under saline-alkaline stress in flax [J]. Journal of Northeast Agricultural University, 2015, 46(3): 21-28. (in Chinese with English abstract)
Cloning and expression analysis of MAPK gene under saline-alkaline
stress in flax/YU Ying1, CHEN Hongyu2, CHENG Lili1, ZHAO Dongsheng1, YUAN Hongmei1, WU Guangwen1, GUAN Fengzhi1(1.Institute of Industrial Crops, Heilongjiang Academy of Agricultural Sciences, Harbin 150086, China; 2. School of Life Sciences, Northeast Agricultural University, Harbin 150030, China)
Abstract:In order to investigate the relationship between flax MAPKgenes and saline-alkaline stress, 20 flax MAPK genes, named LuMPK1-LuMPK20, repectively, were predicted from flax genome by bioinformatics methods. These 20 MAPK genes all contained T[D/E]Y conserved domains and existed in pairs except LuMPK5 and LuMPK20. Five MAPK genes (LuMPK3, LuMPK8, LuMPK11, LuMPK14, LuMPK16) were cloned by PCR and their sequences were consistent with the predicted sequences. The expressions of these genes were analyzed by digital gene expression under saline and alkaline stresses. The results showed that the expressions of these five genes were subject to saline and alkaline stresses. LuMPK3, LuMPK11, LuMPK14, LuMPK16 were up-regulated under neutral salt stress, and LuMPK3, LuMPK8, LuMPK11, LuMPK14 were up-regulated under alkaline salt stress. LuMPK16 was down-regulated under alkaline salt stress. This study laid the theoretical foundation for the function of MAPK genes in flax, especially the saline-alkaline tolerance function.
Key words:flax;MAPK;gene cloning;saline-alkaline stress;expression analysis
*通讯作者:关凤芝,研究员,研究方向为亚麻遗传育种。E-mail: kj-gfz@163.com
作者简介:于莹(1981-),女,助理研究员,博士,研究方向为亚麻分子育种。E-mail: yuying_1981_0451@163. com
基金项目:哈尔滨市青年科技创新人才专项(2013RFQYJ011);黑龙江省农业科学院院级课题重点项目(2012ZD011);黑龙江省博士后资助经费项目(LBH-Z13182)
收稿日期:2014-12-19
文章编号:1005-9369(2015)03-0021-08
文献标志码:A
中图分类号:S563.2;Q785
