多元自动化基因组工程
2015-10-27李丹高海军
李丹 高海军
(北京理工大学生命学院,北京 100081)
多元自动化基因组工程
李丹 高海军
(北京理工大学生命学院,北京 100081)
基因组编辑技术在基因组工程研究中应用广泛,其中位点特异性核酸酶编辑技术和CRISPR/Cas系统在单基因编辑方面贡献卓越,但由于基因组的庞大,这些技术又有一定的局限性。多元自动化基因组工程(MAGE)是一种新型基因组编辑技术,可同时作用于多个基因,具有快速、高效的特点,已被用于大肠杆菌的基因敲除和基因替换。主要介绍了MAGE的原理、具体操作流程及技术进展,并结合MAGE技术的应用,讨论其发展趋势。
多元自动化基因组工程;基因组编辑技术;基因组工程;大肠杆菌
多种生物全基因组测序工作已经完成,相关测序结果已在基础科学研究中广泛应用,大量用于基因组工程(genome engineering)研究的新技术应运而生。基因组工程是指为了实现某一目标对生物体中特定的基因进行有针对性遗传修饰的技术[1],它开启了科学研究新的一页。基因组工程应用广泛,可被用于敲除或失活一个基因,修复不利突变[2,3],或增加生物种群的多样性,如农药抗性植物技术的发展[4,5],或作为科学研究的工具如通过观察基因失活导致的异常现象来阐述该基因的功能。
将一个DNA片段插入活细胞基因组的技术,如转座子失活技术,具有一定的随机性。DNA片段被随机放置在染色体上,可能会干扰其他基因功能的行使或灭活基因,甚至会造成比较严重的副作用,如触发细胞癌变。更重要的是,这些技术没有可重复性,不能保证同一序列插入不同细胞的同一位置[6,7]。近年来基因组工程研究最大的突破是能够特定地修饰生物染色体中某一区域的DNA序列,从而提高基因组修饰的精度,防止细胞毒性的产生,同时也增加了基因组操作的可重复性。近期发展起来的基因组编辑技术(genome editing)很好地诠释了这一点[8,9]。采用基因组编辑技术可以对几乎任意基因进行修饰、编辑。位点特异性核酸酶编辑的技术[10]以及最近刚兴起的由成簇的、规律间隔的短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)/Cas(CRISPR-associa-ted)系统介导的基因组靶向修饰技术[11]是其中较重要的两种。
1 基因组编辑技术(genome editing)
基因组编辑技术中用到的位点特异性核酸酶主要包括锌指核酸酶(zinc-finger nucleases, ZFN)[12-14]和转录激活因子样效应因子核酸酶(transcription activator-like effector nucleases,TALEN)[8,9,15-17],它们是由序列特异性DNA结合结构域(锌指蛋白或转录激活因子样效应因子)与非特异性DNA切割结构域(Fok I限制性核酸内切酶中的非特异性DNA切割结构域)组合而成的人工核酸酶。ZFN和TALEN都能在靶位点切割双链DNA形成DSBs(DNA double-strand break)[18],引发细胞内的非同源性末端连接修复,即非同源性末端接合(nonhomologous end joining,NHEJ)[19,20]或同源重组修复(homology-directed repair,HDR),对基因进行较精确的遗传修饰。目前已建立了ZFN文库和TALEN文库,可从中获取相关特异性DNA结合结构域,进行一系列基因改造[21-23]。
由CRISPR/Cas系统介导的基因组靶向修饰技术是近期兴起的一种基因组定向编辑技术[11,24]。CRISPR/Cas 系统广泛分布于细菌和古生菌基因组中,为细胞提供一种获得性免疫保护机制,即通过一套RNA介导的DNA切割机制使细菌免受外源DNA的侵扰[25-27]。其中II型CRISPR/Cas系统的研究最为深入,该系统识别外源侵入的短片段DNA分子(即间隔分子)并将其整合到CRISPR基因组内,转录并处理成短的CRISPR RNAs(crRNAs)。这些crRNAs与反式激活的crRNA,即trans-activating rRNA(tracrRNA)结合,介导DNA的序列特异性切割,最终在Cas蛋白作用下将外源的“致病”DNA分子沉默。
这些基因组编辑技术在实验和临床等方面应用广泛,极大地提高了人们对模式生物的处置和研究能力,也为治疗遗传性疾病、纠正遗传错误提供了一个平台[2-5,28-33]。
ZFN、TALEN和CRISPR/Cas系统推动整个基因组工程的发展。由于微生物基因组的规模十分庞大,单纯利用产生DSBs来激活同源重组修复机制(HDR)或非同源末端连接修复机制(NHEJ)的方法很难实现多元化,且多处切口同时存在时,会给细胞带来严重的损伤。此外,NHEJ修复机制在断裂处也较容易出现微小的基因片段的插入或缺失,导致移码突变,最终使基因失活。因此迫切需要一种高效的、多元的编辑技术[19,20]。
2 多元自动化基因组工程(MAGE)
哈佛医学院遗传系的Church研究小组在2009年8月的Nature杂志上发表了一项新技术——多元自动化基因组工程(multiplex automated genome engineering,MAGE)[34],并于2012年4月申请了美国国家专利[35]。
区别于一次仅改造一个基因的技术,多元自动化基因组工程(图1)可对细胞染色体上的多个基因或位点进行修饰,方式多样,可以是插入、错配或缺失,产生多种多样的基因突变型菌株[34,36]。
2.1 原理
2.1.1 λ噬菌体Red重组系统 MAGE基于λ噬菌体Red重组酶的同源重组系统,Red 重组系统由3种蛋白组成:Exo蛋白、Beta蛋白和Gam蛋白。其中Exo蛋白是一种核酸外切酶,结合在双链DNA末端,从5'端向3'端降解DNA,产生3'突出端;Beta蛋白结合在单链DNA上,介导互补单链DNA退火;Gam蛋白可与RecBCD 酶结合,抑制其对外源DNA的降解。在内源性λ重组系统中,控制exo、bet、gam三个基因的PL启动子受到CI857阻遏蛋白的抑制,CI857阻遏蛋白由cI基因表达产生,是一个温敏性阻遏子,它在30-34℃条件下,阻遏PL启动子的功能,使其不能形成λ red蛋白;而在42℃条件下,该阻遏子被抑制,λ red 蛋白正常表达[37-41]。
2.1.2 MAGE基本原理 基于Red重组原理,Church研究小组构建了一套科学、完整的MAGE循环系统,首先30℃下培养菌体,达到对数期后温度变换为42℃,该条件下,CI857阻遏蛋白被抑制,λ-Red蛋白即Exo蛋白、Beta蛋白和Gam蛋白正常表达,接着将菌体冷藏于4℃,防止上述生成的蛋白质被降解,同时将人工合成的多个简并寡核苷酸通过电击的方式转入细胞中,每个寡核苷酸都靶定了基因组上的一段特定序列,结合后通过同源重组的方式替换靶定序列。MAGE的的优势在于可重复进行上述循环,并能反复检测,使更多类的寡核苷酸转入细胞,以促进基因组的快速变异(图2)[34]。
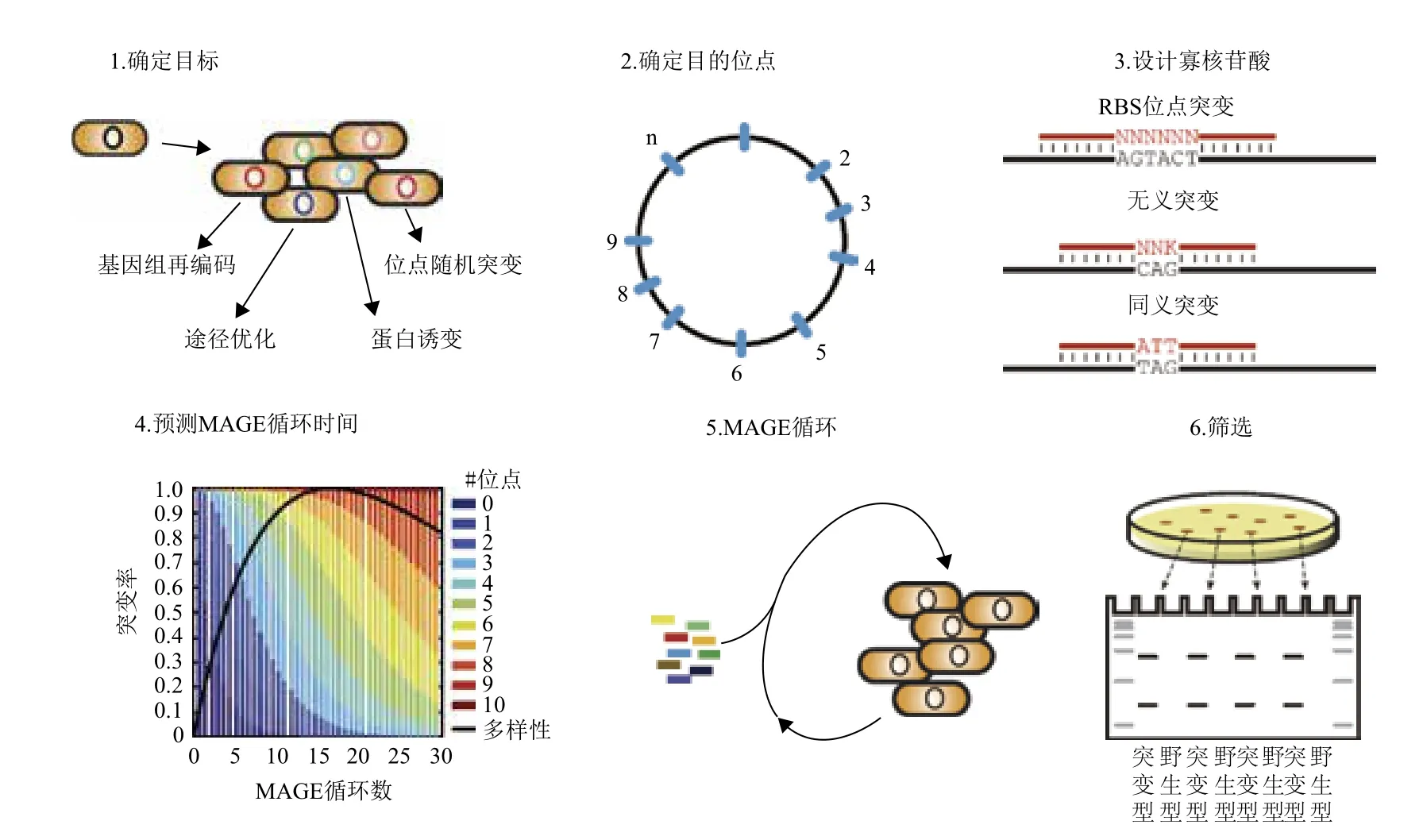
图1 多元自动化基因组工程(MAGE)[42]
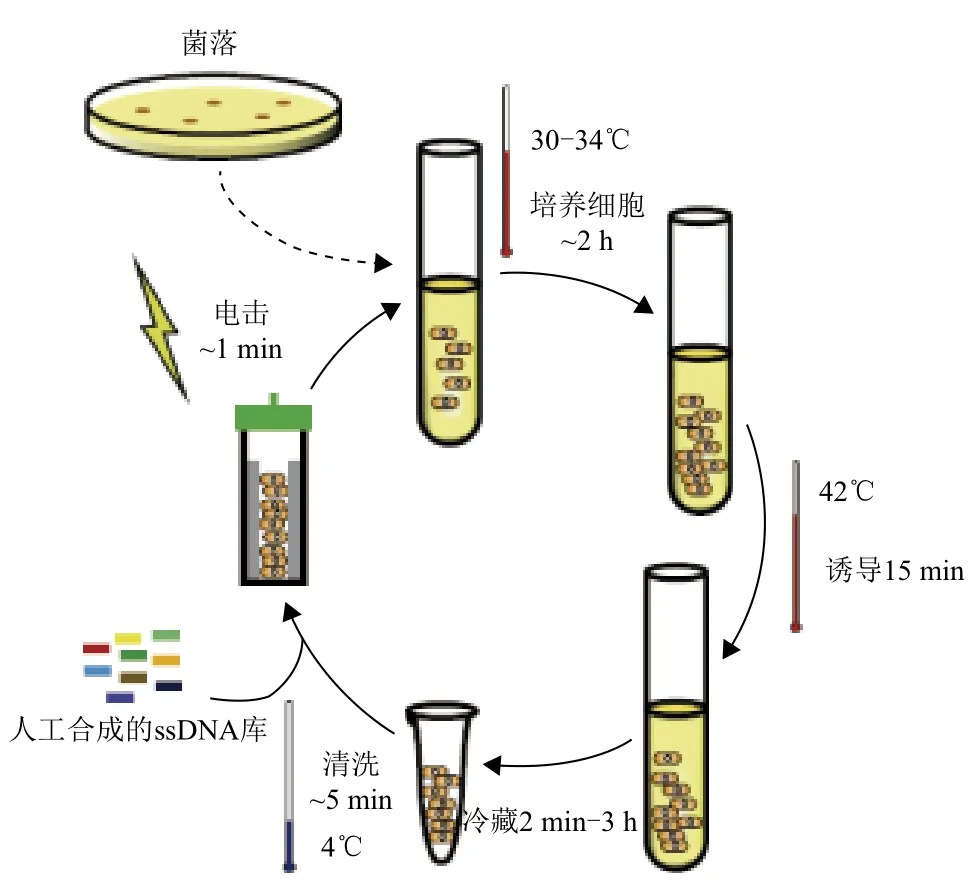
图2 MAGE循环详细过程[42]
2.2 MAGE自动化装置
由于上述过程是周期性、循环式的,操作起来比较繁琐、费时费力,Church研究小组设计了一个自动化装置[34],使得上述过程实施起来省时省力,更加快速、有效的产生各种组合的基因组。
图3为详细的MAGE自动化装置原理图,该装置由3部分组成,分别是可实时监控细胞浓度的人工温控气候培养箱(绿色)、使细胞在人工培养箱和培养液、缓冲液间交替更换的流控系统(蓝色)和实时生产感受态细胞以便与人工合成的DNA片段库电转结合的装置(黄色)。这3部分相辅相成,使得MAGE循环重复、有序进行。
根据不同的MAGE步骤(生长、热休克和冷却),培养基放置于不同温度条件下的人工培养箱里(30℃、42℃和4℃),此过程中需要注意的是接受了外来DNA片段的感受态细胞需要通过滤膜过滤并用wash buffer重悬方可进入下一循环[34]。
2.3 MAGE的优势
MAGE能将大量人工合成的具有各种突变(包括碱基错配、插入和缺失)的DNA库导入宿主细胞进行重组,可以快速高效地在全基因组尺度上对菌株的DNA序列进行设计和修饰,极大地加快了细胞优化的进程。它的优势主要体现在以下几方面:
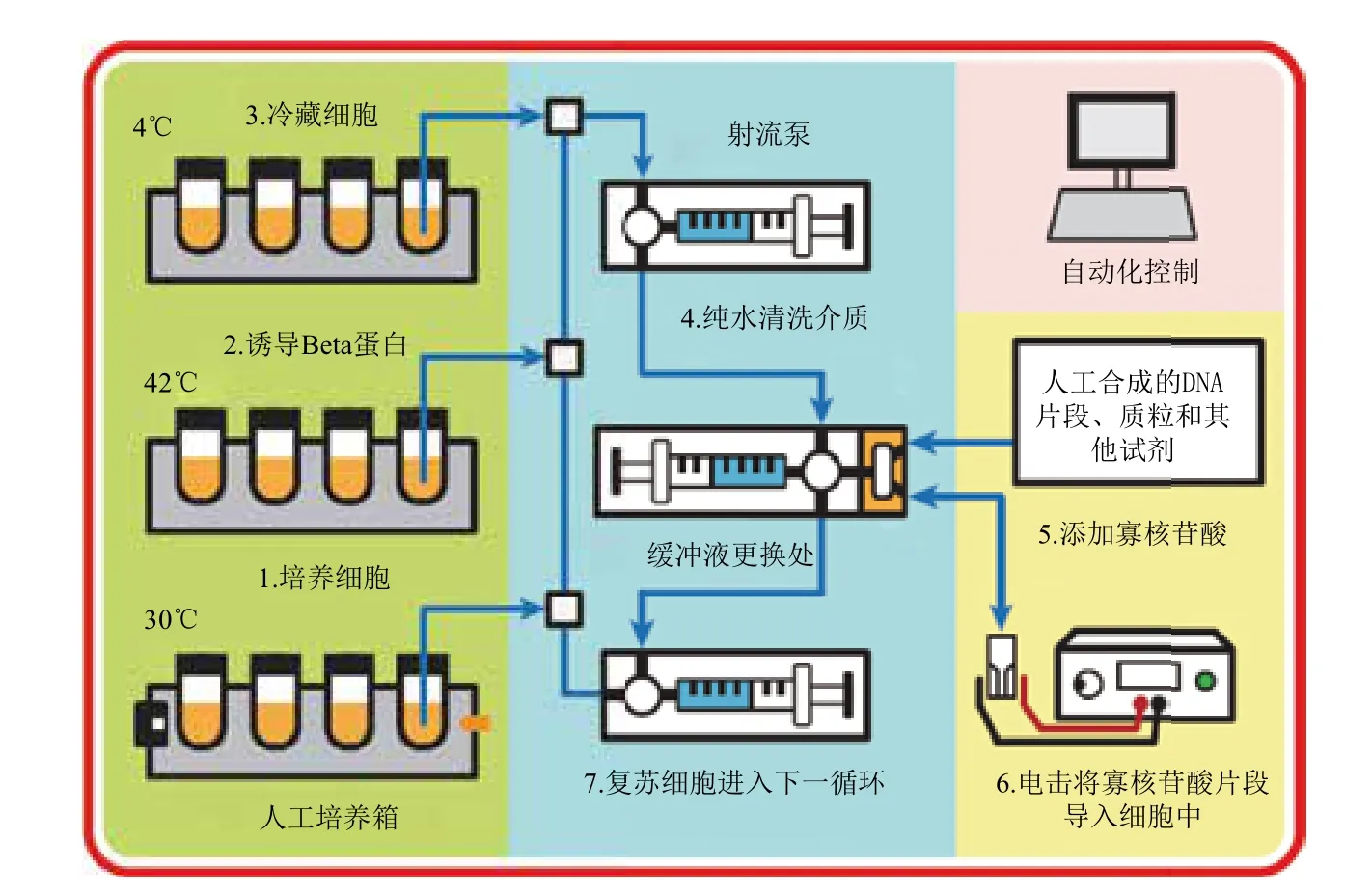
图3 MAGE自动化装置[34](颜色标识见电子版)
区别于以往的技术,MAGE将ssDNA导入细胞中,这样使得Red重组效率由导入dsDNA 片段时的0.2%提高到25%[43,44];MAGE可同时作用于基因组的多个位点。MAGE同时将大量靶向基因组不同部位的序列的ssDNA库转入细胞中,这样可产生各种组合的突变型,真正实现了基因组的可编辑、可组装[34];MAGE通过上述自动化装置,重复周期性的导入ssDNA,快速、高效地得到各种突变型,“一天之内可生成数十亿个变化”[1];导入的ssDNA片段进行磷硫酰化硫修饰以防止细胞本身核酸外切酶的降解,有研究表明,对外来DNA片段进行硫代磷酸化修饰,重组效率增加了2-3倍[44-46];导入的ssDNA片段作用于复制叉的后随链上,研究表明此法比之前作用于前导链时重组效率提高了近30倍[43,47]。
2.4 MAGE的应用及改进
现在已有成功应用MAGE的例子,如大肠杆菌DXP合成途径改造。随着其应用的推广,也有一些学者对该技术的缺陷和不足进行改进,并成功应用到大肠杆菌色氨酸合成等途径中。
2.4.1 经典的大肠杆菌DXP合成途径改造 番茄红素合成途径涉及20个内源性基因,包括dxs、 dxr、ispD、ispE、isp、GispH、idi、ispA、appY、rpoS、crl、elbA、elbB、yjiD、purH、rnlA、yggT、ycgZ、ymgA和ariR[48,49]。Church研究小组针对这20个基因进行番茄红素表达的优化,将分别靶定每个基因的ssDNA库导入细胞中,经过35个MAGE循环,产生了近150亿个基因变异体,经过合适的筛选方法得到高产番茄红素突变株。另外,分支途径上的4个基因(ytjC、fdhF、aceE和gdhA)[50]因引入两个无义突变而失活,进一步提高了番茄红素的产量[34]。由于自动化装置的存在,每个循环只需2-2.5 h,此工作仅在3 d内完成,使得番茄红素的产量提高了近5倍。由此可见,与之前的单基因操作相比,MAGE可以同时优化多个基因,其高效率也得到了证实。
2.4.2 分层次结合组装基因组工程(CAGE) 该技术[51]克服了MAGE仅作用于有限的十几、二十个位点的缺陷,通过精确的操作,将E. coli 全基因组中314个TAG终止密码子替换成同义的TAA密码子。研究人员将314个TAG密码子分成32个小组,每个小组包含10个TAG-TAA的改变,再分别进行小规模MAGE重组,然后利用细菌天然结合能力,获得更大规模的换成TAA密码子的菌株,直到获得不依赖TAG的全新突变菌株。该技术使得基因组工程从核苷酸水平上升到了兆碱基水平(the megabase scale)。该技术也可应用于模式微生物操作中,用来生产各种药品或化学制品。
2.4.3 共选择的多元自动化基因组工程(Coselection MAGE) 共选择的多元自动化基因组工程(CoSMAGE)也是改进MAGE的一种方法,MAGE同时作用于染色体多个位点,可以得到大量的突变株,但筛选过程过于复杂繁琐,从而导致大多数的突变型无法得到鉴定而丧失意义,CoS-MAGE由于co-marks的存在,能更好、更有效的进行突变株的筛选。T7启动子作为一个co-marks,将其插入与大肠杆菌色氨酸合成途径(此途径产生靛蓝)相关的12个基因的上游,经过几轮MAGE后,筛选靛蓝产量提高的菌株,然后再进行MAGE,如此几轮下来产生了80种不同T7启动子组合,经过筛选,得到了一株靛蓝产量提高62%的突变株。这说明MAGE可以通过插入co-marks构建一个突变基因组合库来快速有效地筛选到理想菌株[52,53]。
2.4.4 可定位的多重染色体重组技术(TRMR) 即使如上述添加co-marks来提高筛选效率,仍不能满足理论存在的突变型筛选工作。上述大肠杆菌色氨酸生物合成过程中,12个基因被工程改造,理论上产生了212种突变型,但事实上研究者们只鉴定出来80种。因此,在大量突变株存在的条件下,一个高通量的筛选方法是很有必要的。2010年,科罗拉多大学的Warner等[54]发明了一种可定位的多重染色体重组技术(trackable multiplex recombineering,TRMR),利用分子条码[55]、微阵列及MAGE技术,在一天时间内修饰了E. coli内95%的基因;在一周内,完成影响E. coli生长的近4 000个基因的遗传定位。具体操作是通过合成带有分子条码的,编码相关突变的插入序列,电击转入宿主细胞,然后利用分子条码技术追踪到相关突变,量化突变率,进行遗传性状定位。这样大大提高了突变株筛选效率,使得MAGE技术在应用中更进一步。
3 结语
综上所述,多元自动化基因组工程(MAGE)技术是一种大幅度修改和进化细胞基因组的技术,它可将大量人工合成的具有各种突变(包括碱基错配、插入和缺失)的单链DNA库导入宿主细胞进行重组,快速高效地得到各种突变株。尽管现在已有应用MAGE及改进版的MAGE的成功案例,但MAGE有一定的局限性,表现在以下几点:(1)除2013年有文献提到一种类似方法应用于谷氨酸棒状杆菌中[56],MAGE仅用于大肠杆菌这一模式菌株中,可能与其作用机制(如导入的片段作用于后随链)有关,其广泛应用还有待进一步的探索。(2)经过几轮MAGE,可以产生大量的基因突变型,上述应用已提到一些筛选改进方法,但仍未能对大多数突变型进行筛选,造成多数突变型的丢失。(3)MAGE突变位点的选择以及突变序列的设计对理论储备和经验提出了更高的要求。(4)MAGE可作用于多个位点,其同源重组效率与单一位点重组相比明显下降,如何提高这一效率还需更多的研究探索[42]。
[1] Carr PA, Church GM. Genome engineering[J]. Nature Biotechnology, 2009, 27(12):1151-1162.
[2] Cho SW, Kim S, Kim JM, et al. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease[J]. Nature Biotechnology, 2013, 31(3):230-232.
[3] Mali P, Yang L, Esvelt KM, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339(6121):823-826.
[4] Li T, Liu B, Spalding MH, et al. High-efficiency TALEN-based gene editing produces disease-resistant rice[J]. Nature Biotechnology,2012, 30(5):390-392.
[5] Shukla VK, Doyon Y, Miller JC, et al. Precise genome modification in the crop species Zea mays using zinc-finger nucleases[J]. Nature, 2009, 459(7245):437-441.
[6] Sasaki E, Suemizu H, Shimada A, et al. Generation of transgenic non-human primates with germline transmission[J]. Nature, 2009,459(7246):523-527.
[7] Yang SH, Cheng PH, Banta H, et al. Towards a transgenic model of Huntington’s disease in a non-human primate[J]. Nature, 2008,453(7197):921-924.
[8] Miller JC, Tan S, Qiao G, et al. A TALE nuclease architecture for efficient genome editing[J]. Nature Biotechnology, 2011, 29(2):143-148.
[9] Mussolino C, Morbitzer R, Lutge F, et al. A novel TALE nuclease scaffold enables high genome editing activity in combination with low toxicity[J]. Nucleic Acids Research, 2011, 39(21):9283-9293.
[10] Gaj T, Gersbach CA, Barbas CF 3rd. ZFN, TALEN, and CRISPR/ Cas-based methods for genome engineering[J]. Trends in Biotechnology, 2013, 31(7):397-405.
[11] Mussolino C, Cathomen T. RNA guides genome engineering[J]. Nature Biotechnology, 2013, 31(3):208-209.
[12] Beerli RR, Barbas CF 3rd. Engineering polydactyl zinc-finger transcription factors[J]. Nature Biotechnology, 2002, 20(2):135-141.
[13] Liu Q, Segal DJ, Ghiara JB, et al. Design of polydactyl zinc-finger proteins for unique addressing within complex genomes[J]. Proc Natl Acad Sci USA, 1997, 94(11):5525-5530.
[14] Maeder ML, Thibodeau-Beganny S, Osiak A, et al. Rapid “opensource” engineering of customized zinc-finger nucleases for highly efficient gene modification[J]. Molecular Cell, 2008, 31(2):294-301.
[15] Moscou MJ, Bogdanove AJ. A simple cipher governs DNA recognition by TAL effectors[J]. Science, 2009, 326(5959):1501.
[16] Deng D, Yan C, Pan X, et al. Structural basis for sequence-specific recognition of DNA by TAL effectors[J]. Science, 2012, 335(6069):720-723.
[17] Mercer AC, Gaj T, Fuller RP, et al. Chimeric TALE recombinases with programmable DNA sequence specificity[J]. Nucleic Acids Research, 2012, 40(21):11163-11172.
[18] Wyman C, Kanaar R. DNA double-strand break repair:all’s well that ends well[J]. Annual Review of Genetics, 2006, 40:363-383.
[19] Maresca M, Lin VG, Guo N, et al. Obligate ligation-gated recombination(ObLiGaRe):custom-designed nuclease-mediated targeted integration through nonhomologous end joining[J]. Genome Research, 2013, 23(3):539-546.
[20] Cristea S, Freyvert Y, Santiago Y, et al. In vivo cleavage of transgene donors promotes nuclease-mediated targeted integration[J]. Biotechnology and Bioengineering, 2013, 110(3):871-880.
[21] Moehle EA, Rock JM, Lee YL, et al. Targeted gene addition into a specified location in the human genome using designed zinc finger nucleases[J]. Proc Natl Acad Sci USA, 2007, 104(9):3055-3060.
[22] Orlando SJ, Santiago Y, DeKelver RC, et al. Zinc-finger nucleasedriven targeted integration into mammalian genomes using donors with limited chromosomal homology[J]. Nucleic Acids Research,2010, 38(15):e152.
[23] Chen F, Pruett-Miller SM, Huang Y, et al. High-frequency genome editing using ssDNA oligonucleotides with zinc-finger nucleases[J]. Nature Methods, 2011, 8(9):753-755.
[24] Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096):816-821.
[25] Wiedenheft B, Sternberg SH, Doudna JA. RNA-guided genetic silencing systems in bacteria and archaea[J]. Nature, 2012, 482(7385):331-338.
[26] Terns MP, Terns RM. CRISPR-based adaptive immune systems[J]. Curr Opin Microbiol, 2011, 14(3):321-327.
[27] Bhaya D, Davison M, Barrangou R. CRISPR-Cas systems in bacteria and archaea:versatile small RNAs for adaptive defense and regulation[J]. Annu Rev Genet, 2011, 45:273-297.
[28] Hwang WY, Fu Y, Reyon D, et al. Efficient genome editing in zebrafish using a CRISPR-Cas system[J]. Nature Biotechnology,2013, 31(3):227-229.
[29] Townsend JA, Wright DA, Winfrey RJ, et al. High-frequency modification of plant genes using engineered zinc-finger nucleases[J]. Nature, 2009, 459(7245):442-445.
[30] Wood AJ, Lo TW, Zeitler B, et al. Targeted genome editing across species using ZFNs and TALENs[J]. Science, 2011, 333(6040):307.
[31] Tesson L, Usal C, Menoret S, et al. Knockout rats generated by embryo microinjection of TALENs[J]. Nature Biotechnology,2011, 29(8):695-696.
[32] Meng X, Noyes MB, Zhu LJ, et al. Targeted gene inactivation in zebrafish using engineered zinc-finger nucleases[J]. Nature Biotechnology, 2008, 26(6):695-701.
[33] Sander JD, Cade L, Khayter C, et al. Targeted gene disruption in somatic zebrafish cells using engineered TALENs[J]. Nature Biotechnology, 2011, 29(8):697-698.
[34] Wang HH, Isaacs FJ, Carr PA, et al. Programming cells by multiplex genome engineering and accelerated evolution[J].Nature, 2009, 460(7257):894-898.
[35] Church GM, Isaacs FJ, Wang HH. Multiplex automated genome engineering:United States, 8569041 B2[P]. 2013-10-23.
[36] Jeong J, Cho N, Jung D, et al. Genome-scale genetic engineering in Escherichia coli[J]. Biotechnology Advances, 2013, 31(6):804-810.
[37] Murphy KC. Lambda Gam protein inhibits the helicase and chistimulated recombination activities of Escherichia coli RecBCD enzyme[J]. J Bacteriol, 1991, 173(18):5808-5821.
[38] Takahashi N, Kobayashi I. Evidence for the double-strand break repair model of bacteriophage lambda recombination[J]. Proc Natl Acad Sci USA, 1990, 87(7):2790-2794.
[39] Muniyappa K, Shaner SL, Tsang SS, et al. Mechanism of the concerted action of recA protein and helix-destabilizing proteins in homologous recombination[J]. Proc Nat Acad Sci USA, 1984, 81(9):2757-2761.
[40] Copeland NG, Jenkins NA, Court DL. Recombineering:a powerful new tool for mouse functional genomics[J]. Nature Reviews Genetics, 2001, 2(10):769-779.
[41]Muniyappa K, Radding CM. The homologous recombination system of phage lambda. Pairing activities of beta protein[J]. J Biol Chem, 1986, 261(16):7472-7478.
[42]Gallagher RR, Li Z, Lewis AO, et al. Rapid editing and evolution of bacterial genomes using libraries of synthetic DNA[J]. Nature Protocols, 2014, 9(10):2301-2316.
[43] Mosberg JA, Lajoie MJ, Church GM. Lambda red recombineering in Escherichia coli occurs through a fully single-stranded intermediate[J]. Genetics, 2010, 186(3):791-799.
[44] Liang J, Wang Z, He X, et al. DNA modification by sulfur:analysis of the sequence recognition specificity surrounding the modification sites[J]. Nucleic Acids Research, 2007, 35(9):2944-2954.
[45] Wang L, Chen S, Xu T, et al. Phosphorothioation of DNA in bacteria by dnd genes[J]. Nature Chemical Biology, 2007, 3(11):709-710.
[46] Eckstein F, Gish G. Phosphorothioates in molecular biology[J]. Trends in Biochemical Sciences, 1989, 14(3):97-100.
[47] Yu D, Sawitzke JA, Ellis H, et al. Recombineering with overlapping single-stranded DNA oligonucleotides:testing a recombination intermediate[J]. Proc Natl Acad Sci USA, 2003, 100(12):7207-7212.
[48] Jin YS, Stephanopoulos G. Multi-dimensional gene target search for improving lycopene biosynthesis in Escherichia coli[J]. Metabolic Engineering, 2007, 9(4):337-347.
[49] Kang MJ, Lee YM, Yoon SH, et al. Identification of genes affecting lycopene accumulation in Escherichia coli using a shot-gun method[J]. Biotechnology and Bioengineering, 2005, 91(5):636-642.
[50] Alper H, Jin YS, Moxley JF, et al. Identifying gene targets for the metabolic engineering of lycopene biosynthesis in Escherichia coli[J]. Metabolic Engineering, 2005, 7(3):155-164.
[51] Isaacs FJ, Carr PA, Wang HH, et al. Precise manipulation of chromosomes in vivo enables genome-wide codon replacement[J]. Science, 2011, 333(6040):348-353.
[52] Wang HH, Kim H, Cong L, et al. Genome-scale promoter engineering by coselection MAGE[J]. Nature Methods, 2012, 9(6):591-593.
[53] Carr PA, Wang HH, Sterling B, et al. Enhanced multiplex genome engineering through co-operative oligonucleotide co-selection[J]. Nucleic Acids Research, 2012, 40(17):e132.
[54] Warner JR, Reeder PJ, Karimpour-Fard A, et al. Rapid profiling of a microbial genome using mixtures of barcoded oligonucleotides[J]. Nature Biotechnology, 2010, 28(8):856-862.
[55] Shoemaker DD, Lashkari DA, Morris D, et al. Quantitative phenotypic analysis of yeast deletion mutants using a highly parallel molecular bar-coding strategy[J]. Nature Genetics, 1996, 14(4):450-456.
[56] Binder S, Siedler S, Marienhagen J, et al. Recombineering in Corynebacterium glutamicum combined with optical nanosensors:a general strategy for fast producer strain generation[J]. Nucleic Acids Research, 2013, 41(12):6360-6369.
(责任编辑 狄艳红)
Multiplex Automated Genome Engineering
Li Dan Gao Haijun
(School of Life Sciences,Beijing Institute of Technology,Beijing 100081)
Genome editing is widely used in genome engineering research and site-specific nuclease technologies and CRISPR/Cas system focus on single gene editing. Owing to the huge size of genome, there are some limitations on the applications of these technologies. Multiplex Automated Genome Engineering(MAGE)is a new, fast and efficient genome editing technology, which can operate multiple genes simultaneously, and be used in knockout and replacement of Escherichia coli genes. This review illustrates the recent advances in the theory,operation protocol and technological innovation of MAGE, its application and development trend were also discussed.
multiplex automated genome engineering;genome editing;genome engineering;Escherichia coli
10.13560/j.cnki.biotech.bull.1985.2015.06.009
2014-09-22
国家重大科学仪器设备开发专项项目(2012YQ0401400802)
李丹,女,硕士研究生,研究方向:微生物学;E-mail:ld_bit@126.com
高海军,男,博士,副教授,研究方向:微生物学;E-mail:hj_gao@aliyun.com
