3种鳢属鱼类ITS序列及系统进化分析
2015-10-21朱树人孟庆磊朱永安等
朱树人 孟庆磊 朱永安等
摘要[目的]阐明乌鳢、斑鳢、月鳢核基因序列(ITS)的遗传变异情况,并用ITS序列研究鳢属系统进化关系。[方法]利用PCR扩增3种鳢的ITS,经克隆、拼接得到ITS全长后进行分析。[结果]乌鳢、斑鳢和月鳢ITS全序列长度为902、 927、902或903 bp。3种鳢ITS的G+C含量较高,约占72%。3种鳢之间核苷酸差异明显大于种内,明显的差异性ITS片段可以鉴别3种鳢。采用NJ和ML方法建立的进化树都表明乌鳢和斑鳢的遗传距离最近,而乌鳢和月鳢遗传距离最远。[结论]该研究为鳢属的分类鉴定、系统进化关系以及种间杂交提供基础资料。
关键词乌鳢;斑鳢;月鳢;ITS序列;亲缘关系
中图分类号S961.6文献标识码A文章编号0517-6611(2015)31-047-03
Analysis on ITS Sequences and Phylogenetic Relationship of Three Kinds of Channa
ZHU Shuren1,2,3, MENG Qinglei1, ZHU Yongan1 et al
(1.Shandong Provincial Key Laboratory Freshwater Genetics and Breeding, Shandong Freshwater Fisheries Research Institute, Jinan,Shandong 250017; 2.Key Laboratory of Freshwater Aquatic Genetic Resources Gertificated by Ministry of Agriculture, Shanghai Ocean University, Shanghai 201306; 3.Department of Biology, East China Normal University, Shanghai 200241)
Abstract [Objective] The aim was to examine sequence variation in the internal transcribed spacer (ITS) isolates from Channa argus, C. maculata and C. asiatica, and to reconstruct their phylogenetic relationship using ITS sequences. [Method]The ITS sequences were amplified from each sample and the amplicons were cloned and spliced the full length of ITS sequences.[Result] The full length of ITS sequences of C. argus, C. maculata and C. asiatica were 902, 927, 902 or 903 bp. The content of G+C (about 72%) was higher than A+T. For three species of snakehead, interspecific differences were significantly greater than the intraspecific differences. The obvious difference of ITS fragments could identify three species of snakehead. Based on NJ and ML method, phylogenetic analysis showed that C. argus and C. maculata had the lowest genetic distance, while C. argus and C. asiatica had the highest genetic distance. [Conclusion]The results of this study could be useful for classification, phylogenetic relationship and interspecific hybrids of Channa.
Key words Channa argus; C. maculata ; C. asiatica; ITS sequences; Phylogenetic relationship
真核生物的核糖體都有3种rRNA,即18S rRNA、5.8S rRNA 和28S rRNA。这3种rRNA 基因都是以串联重复形式存在,编码这些rRNA的基因都按一定顺序排列在DNA上。ITS包括ITS1和ITS2,分别位于18S rRNA与5.8S rRNA,5.8S rRNA 和28S rRNA之间。由于ITS1和ITS2进化速度较快,两者广泛应用于鱼类物种的鉴定,特别用于分类较相似的鱼类之间[1-4]。可以根据ITS两边rRNA 具有很高的保守性,设计引物扩增ITS序列片段。
笔者对我国较为常见的乌鳢、斑鳢、月鳢种间的分子遗传距离和进化关系进行研究,旨在促进对其种内遗传多样性的认识,为分析鳢属种间杂交提供借鉴。
1材料与方法
1.1材料
3种鳢属鱼类样本于2013年5~10月份采集。乌鳢样本采集于山东微山湖,斑鳢和月鳢都采自广东。每种鳢采集5尾,样本均采自水上作业的渔民或附近的水产品市场,剪取少量胸鳍,保存于无水乙醇中,贴好标签放置于4 ℃冰箱备用。
1.2DNA提取
将胸鳍组织从乙醇中取出,充分漂洗晾干并剪碎,采用标准的酚-氯仿法提取基因组DNA。取2 μl DNA用1.5%琼脂糖凝胶电泳检测,并稀释到合适浓度用于PCR扩增。
1.3PCR扩增及测序
PCR扩增引物参照通用引物,均由上海生工生物工程有限公司合成,上游引物NSF1787F(5′CCGTA GGTGA ACCTG CGG3′)和下游引物FishLSU5Rev(5′TTAAA TTCAG CGGGT TGTCT3′)[5]。
PCR反应体系为20 μl,包含10×PCR buffer 2 μl,10 mmol/L dNTP 0.5 μl,25 mmol/L Mg2+ 2.4 μl,10 umol/L 引物NSF1787F 、FishLSU5′Rev各 1 μl,Taq(5 U/μl) 0.1 μl,DNA模板 2 μl,其他用 ddH2O 补充。PCR扩增程序采用降落PCR,94 ℃预变性3 min,然后是35个循环,每个循环包括94 ℃变性40 s,退火40 s(初始温度为64 ℃,每次循环降0.5 ℃,降至61 ℃),72 ℃延伸1.5 min,最后72 ℃再延伸10 min。
PCR产物经电泳检测纯化、回收、克隆后由上海迈浦生物科技有限公司进行测序。
1.4数据分析
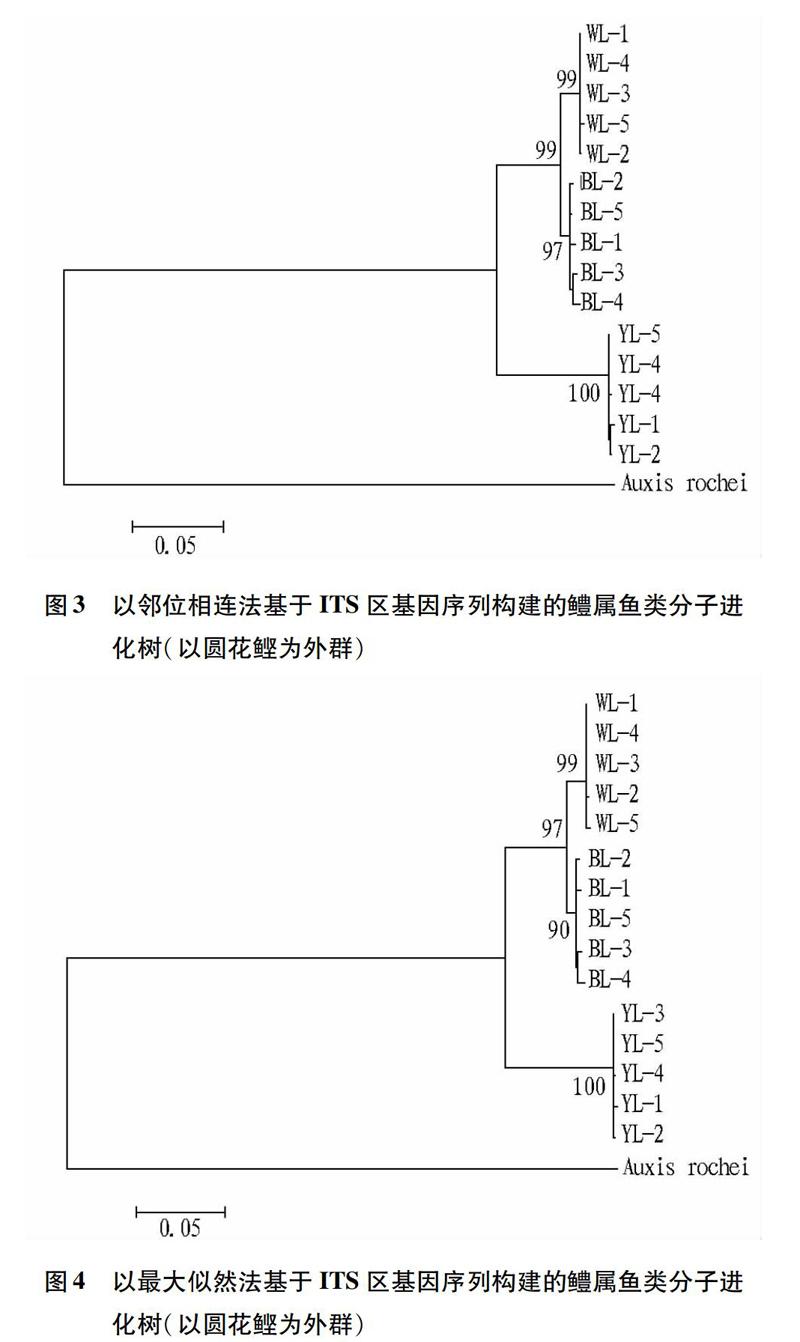
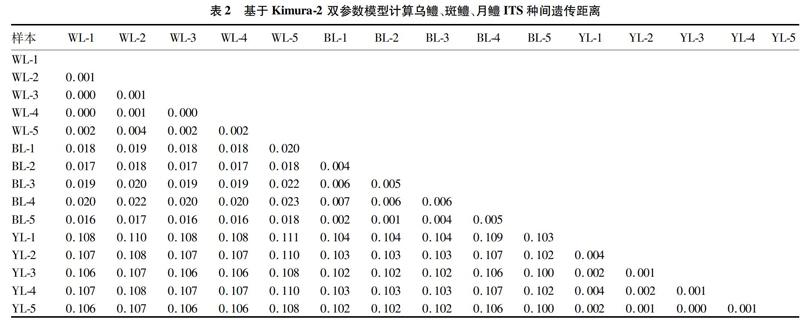
利用Clustal X(1.83)[6]对序列进行对位排列,并结合人工校正。通过MEGA5.0软件[7-8]分析序列的碱基组成、变异位点,同时采用Kimura双参数法计算种间的遗传距离。在遗传距离分析的基础上,以鲈形目鱼类圆花鲣(Auxis rochei,Genbank AB193743)为外群,采用Kimura 2parameter模型,Bootstrap估算重复次数1 000次,采用NJ法(Neighborjoining)和ML法(Maximum Likehood)构建系统进化树。
2结果与分析
2.1阳性克隆鉴定结果
ITS基因序列阳性克隆提取质粒后进行PCR鉴定(图1),得到约1 200 bp的条带,与基因组PCR结果完全一致,表明克隆已经成功。
2.2序列测定结果
采用双向测序,经过拼接后得到了完整的核糖体ITS序列(包括部分18S rRNA、ITS1、5.8S rRNA、ITS2和部分28 S rRNA序列)。经过NCBI上的Blast程序在GenBank基因数据库中进行同源比对,发现测得的ITS序列与许多鱼类的核糖体保守区域具有很高的同源性,其中与鲈形目鱼类的同源性最高,证明测得序列的正确性(图2)。乌鳢和斑鳢测得的ITS序列片段大小在1 200~1 300。把克隆测得ITS序列向Genbank进行了提交。样品ITS序列登录号如下:WL1,KJ451597;WL2,KJ451598;WL3,KJ451599;WL4,KJ451600;WL5,KJ451601;BL1,KJ451602;BL2,KJ451603;BL3,KJ451604;BL4,KJ451605;BL5KJ451606;YL1,KJ451607;YL2,KJ451608;YL3,KJ451609;YL4,KJ451610;YL5,KJ451611。
2.4系统发育关系分析
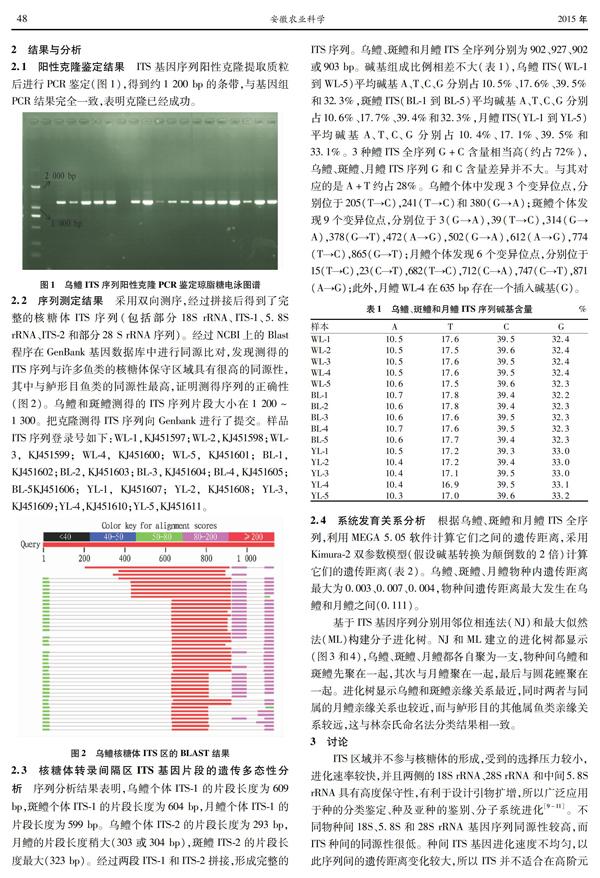
根据乌鳢、斑鳢和月鳢ITS全序列,利用MEGA 5.05软件计算它们之间的遗传距离,采用Kimura2双参数模型(假设碱基转换为颠倒数的2倍)计算它们的遗传距离(表2)。乌鳢、斑鳢、月鳢物种内遗传距离最大为0.003、0.007、0.004,物种间遗传距离最大发生在乌鳢和月鳢之间(0.111)。
基于ITS基因序列分别用邻位相連法(NJ)和最大似然法(ML)构建分子进化树。NJ和ML建立的进化树都显示(图3和4),乌鳢、斑鳢、月鳢都各自聚为一支,物种间乌鳢和斑鳢先聚在一起,其次与月鳢聚在一起,最后与圆花鲣聚在一起。进化树显示乌鳢和斑鳢亲缘关系最近,同时两者与同属的月鳢亲缘关系也较近,而与鲈形目的其他属鱼类亲缘关系较远,这与林奈氏命名法分类结果相一致。
3讨论
ITS区域并不参与核糖体的形成,受到的选择压力较小,进化速率较快,并且两侧的18S rRNA、28S rRNA 和中间5.8S rRNA具有高度保守性,有利于设计引物扩增,所以广泛应用于种的分类鉴定、种及亚种的鉴别、分子系统进化[9-11]。不同物种间18S、5.8S和28S rRNA 基因序列同源性较高,而ITS种间的同源性很低。种间ITS基因进化速度不均匀,以此序列间的遗传距离变化较大,所以ITS并不适合在高阶元物种间进行分子系统发育研究,适合在属以下分类中进行比较研究。
该研究中,用ITS分析乌鳢和斑鳢物种内具有较大的相似度,同属鳢科鱼类的两者在ITS有明显的差异性片段(特别是在ITS2中斑鳢明显多出30 bp大小的片段,即TACCC CCACA CTCTG GGGCC CGGGG CGGGT)。因此ITS不适合用于研究物种内个体之间的差异性,这段明显的差异性片段可以用于准确判定形态学上较难辨别的乌鳢和斑鳢[12],而两者与月鳢无论在形态上还是ITS序列上差异较大(如月鳢在800 bp左右多出CTTCG CCGGG GCTA),很容易用于ITS序列鉴别。在果蝇ITS研究中表明高GC含量是一个比较原始的特征[13],这种特征也存在于鱼类[14]。该研究中,3种鳢G+C含量占72%左右,明显高于A+T,表明鳢属鱼类比较原始,其起源和分化可能较早,故鳢属鱼类种间明显的序列长度多态性是在漫长的进化中积累形成的,同样序列变异较小的种类可能分化较晚,进化时间相对较短。
该研究采用NJ和ML方法建立了分子进化树,2种方法都表明乌鳢和斑鳢的遗传距离较近(约0.020),亲缘关系最近,而与同属的月鳢遗传距离相对较远,这为种间杂交提供了基础资料。鳢科鱼类与同目的圆花鲣遗传距离较大,这与ITS序列不大适合在高阶元物种间分子系统发育研究相符合。
綜上所述,鳢属ITS序列较快的核苷酸进化速率,适合用于该属内种间物种鉴别和系统进化学研究。目前,发现的鳢属鱼类并不完整,GenBank数据库中对鳢属鱼类ITS序列数据还较少,今后有必要对更多的鳢属鱼类进行测定,结合形态学特征及其他分子标记,这将有助于科研工作者对鳢属新物种的发现和分类进化系统有更进一步的认识。
43卷31期朱树人等3种鳢属鱼类ITS序列及系统进化分析
参考文献
[1] PLEYTE K A,DUNCAN S D,PHILLIPS R B.Evolutionary relationships of the salmonid fish genus Salvelinus inferred from DNA sequences of the first internal transcribed spacer(ITS1)of ribosomal DNA[J].Molecular phylogenetics and evolution,1992,1(3):223-230.
[2] MATEJUSOV I,GELNAR M,MCBEATH A J,et al.Molecular markers for gyrodactylids(Gyrodactylidae:Monogenea)from five fish families(Teleostei)[J].International journal for parasitology,2001,31(7):738-745.
[3] CHOW S,NAKAGAWA T,SUZUKI N,et al.Phylogenetic relationships among Thunnus species inferred from rDNA ITS1 sequence[J].Journal of fish biology,2006,68:24-35.
[4] SAJDAK S L,PHILLIPS R B.Phylogenetic relationships among Coregonus species inferred from the DNA sequence of the first internal transcribed spacer(ITS1)of ribosomal DNA[J].Canadian journal of fisheries and aquatic sciences,1997,54(7):1494-1503.
[5] SIYA L,MELISSA A W,DAVID M W,et al.Identification of larval sea basses(Centropristis spp.)using ribosomal DNA-specific molecular assays [J].Fishery bulletin,2008,106(2):183-193.
[6] THOMPSON J D,GIBSON T J,PLEWNIAK F,et al.The CLUSTAL_X windowsinterface:Flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Research,1997,5:4876-4882.
[7] KIMURA M.A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences [J].Journal of Molecular Evolution,1980,16:111-120.
[8] TAMURA K,PETERSON D,PETERSON N,et al.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods [J].Molecular Biology and Evolution,2011,28:2731-2739.
[9] 王庆,傅洪拓.核rDNA ITS区序列在水产动物研究中的应用 [J].安徽农学学报,2007,13(11):99-101.
[10] 徐田军,刘楚吾,刘丽,等.基因间隔序列(ITS)在水产动物种质鉴定和遗传多样性分析中的应用 [J].湛江海洋大学学报,2006,26(1):84-88.
[11] 杨成龙,陈章蛾,吴小平,等.基因组ITS序列分析鉴定红曲霉菌株 [J].核农学报,2015,29(2):252-259.
[12] ZHU S R,MA K Y,XING Z J,et al.The complete mitochondrial genome of Channa argus,C.maculata and hybrid snakehead fish [C.maculata(♀)× C.argus (♂)][J].Mitochondrial DNA,2013,24:217-218.
[13] RODR?GUEZ-TRELLES F,TARRO R,AYALA F J.Evidence for a high ancestral GC content in Drosophila[J].Molecular biology and evolution,2000,17(11):1710-1717.
[14] BOOTON G C,KAUFMAN L,CHANDLER M,et al.Evolution of the ribosomal RNA internal transcribed spacer one(ITS-1)in cichlid fishes of the Lake Victoria region[J].Molecular phylogenetics and evolution,1999,11(2):273-282.
