茶树DNA甲基转移酶基因CsDRM2的克隆及表达分析
2015-08-24周艳华曹红利岳川王璐郝心愿王新超杨亚军中国农业科学院茶叶研究所农业部茶树生物学与资源利用重点实验室国家茶树改良中心浙江杭州310008
周艳华,曹红利,岳川,王璐,郝心愿,王新超,杨亚军(中国农业科学院茶叶研究所/农业部茶树生物学与资源利用重点实验室/国家茶树改良中心,浙江 杭州 310008)
茶树DNA甲基转移酶基因CsDRM2的克隆及表达分析
周艳华,曹红利,岳川,王璐,郝心愿,王新超*,杨亚军*
(中国农业科学院茶叶研究所/农业部茶树生物学与资源利用重点实验室/国家茶树改良中心,浙江 杭州 310008)
DNA甲基化作为表观遗传修饰的主要途径之一,能够通过对基因组DNA的修饰参与植物对外界环境胁迫的响应,DNA甲基化需要DNA甲基转移酶的参与。实验室前期研究表明,茶树在冷驯化过程中发生了甲基化反应。通过RACE克隆,获得了茶树中DNA甲基转移酶CsDRM2基因的cDNA全长序列(NCBI登录号KR057963)。CsDRM2序列全长2328bp,含1815bp的开放阅读框,编码604个氨基酸,预测蛋白质分子量为67.39 kD,理论等电点(PI)为4.85。生物信息学分析结果显示,茶树CsDRM2蛋白与芝麻和烟草的亲缘关系最近,CsDRM2的氨基酸序列与其他植物的DRM2相似性均高于65%。CsDRM2基因表达分析的结果发现,随着冷驯化的进行,CsDRM2基因的表达量呈现出增加的趋势,参与茶树冷驯化过程的甲基化响应。
茶树;DNA甲基转移酶基因CsDRM2;克隆;表达分析;冷驯化
表观遗传主要是研究没有DNA序列变化的、可遗传的基因表达的改变[1]。许多研究结果表明,表观遗传在植物生长发育和逆境胁迫响应过程中起着极其重要的作用,其中DNA甲基化是表观遗传学的重要修饰形式之一[2]。DNA甲基化是在DNA甲基转移酶的催化下完成的,甲基转移酶将 SAM的甲基基团转移到胞嘧啶的 5-位碳原子上从而完成甲基化过程[3]。DNA甲基转移酶能够通过改变其表达水平调节植物DNA甲基化或去甲基化的进行[4]。本实验室前期的研究发现,茶树冷驯化过程中发生了DNA的甲基化和去甲基化过程[5],这个过程必然涉及到DNA甲基转移酶的催化过程。在植物中已经发现了4种DNA甲基转移酶,其中结构域重排甲基转移酶DRM能够催化DNA的从头甲基化[6,7],其作用位点为CG、CNG和CHH[8]。DRM家族主要包括DRM1和DRM2,两者具有较高的同源性。但DRM1的表达水平低于DRM2[6]。本实验室在前期冷驯化转录组测序所获得的表达序列标签(EST)序列中,获得了一条与DRM2基因5'端高度同源EST序列[9]。本实验根据这条EST序列为基础,采用SMART-RACE和RT-PCR技术,获得了该基因的全长cDNA序列,并采用Real-time PCR技术分析了 CsDRM2基因在冷驯化不同时期的表达情况,为研究该基因在茶树冷驯化过程中的作用奠定基础。
1 材料与方法
1.1 试验材料
研究材料为种植于中国农业科学院茶叶研究所国家茶树改良中心的特早生国家级茶树品种‘龙井43’。自2013年11月2日开始,随着外界温度的变化,采摘成熟、完整、无病虫害的茶树叶片,分成两份,一份进行电导率的测定[10],一份液氮速冻后于-80℃冰箱中保存备用。根据电导率测定情况,选取11月2日(驯化前)、12月19日(驯化后)和2014年2月20日(脱驯化)3个时期的样品作为研究对象。
分子生物学试剂:RNAprep pure植物总RNA提取试剂盒(北京天根生化科技有限公司);SMART RACE cDNA Amplification Kit、Advantage®Polymerase Mix(美国Clontech);PCR2.1®vector、大肠杆菌DH5α感受态细胞、Marker、Taq酶、SYBR Premix Ex TaqTM(Perfect Real Time)试剂盒、PrimeScriptTMRT reagent kit with gDNA Eraser (Perfect Real Time)试剂盒(TaKaRa公司);逆转录酶SuperScriptⅢ(美国Invitrogen);琼脂糖凝胶回收技术盒(美国Axygen);其他试剂均为国产分析纯。引物合成及测序由上海华津生物技术有限公司完成。
1.2 R NA提取、cDNA合成及荧光定量 PCR (qRT-PCR)
按照离心柱型植物总RNA快速提取试剂盒说明书的要求提取不同冷驯化阶段茶树叶片的RNA。对RNA进行浓度、完整性等质量检验后,调整浓度至1μg·μL-1,-80℃保存备用。参照SMART RACE cDNA synthesis kit操作步骤,各取1 μg茶树叶片总RNA为模板,合成3′ cDNA链,用于RACE-PCR;用5 μg茶树总RNA为模板,先用DNase Ⅰ去除RNA中的DNA污染,然后根据SuperScript Ⅲ试剂盒的说明合成 cDNA用于 qRT-PCR和反转录PCR(RT-PCR)。
1.3 茶树CsDRM2基因的克隆
在实验室前期获得的茶树冷驯化转录组基础上,经过生物信息学分析筛选出与DNA甲基转移酶DRM2基因5′ 端高度同源的EST系列,根据获得的EST序列信息用primer5.0软件设计3′ RACE引物。以提取的茶树叶片总 RNA为材料,通过SMART-RACE-PCR技术扩增该基因的3′ 端序列。RACE特异引物见表1。PCR扩增程序:94℃ 30sec,68℃ 30sec,72℃ 3 min,30个循环。PCR产物经1%的凝胶电泳检测,切胶回收,连接转化,蓝白斑筛选,菌落PCR验证阳性克隆后送上海华津生物科技有限公司测序。测序结果拼接后得到完整开放阅读框(ORF)的全长序列,在ORF两端设计引物(表1)进行PCR扩增并测序验证,从而得到CsDRM2基因的cDNA全长序列。
1.4 生物信息学分析
生物信息学分析参照曹红利等[11]的方法进行。
1.5 CsDRM2基因的表达分析
以PTB基因作为内参基因,参照SYBR Premix Ex TaqⅡTM(Perfect Real Time)的操作说明书对CsDRM2基因的表达进行荧光定量PCR分析,PCR反应体系为:SYBR Premix Ex Taq 25 μL、上/下游引物(10 μmol·L-1)各1 μL、ROX Dye II 1 μL、cDNA 2 μL,加水至终体积50 μL。反应在ABI PRISM 7500实时定量PCR仪上进行,程序为95℃ 15 sec;95℃5 sec,60℃ 34 sec,40个循环。反应结束后分析荧光值变化曲线和融解曲线,采用2-ΔΔCT法分析结果[12],每个样品3次重复。引物序列见表1。
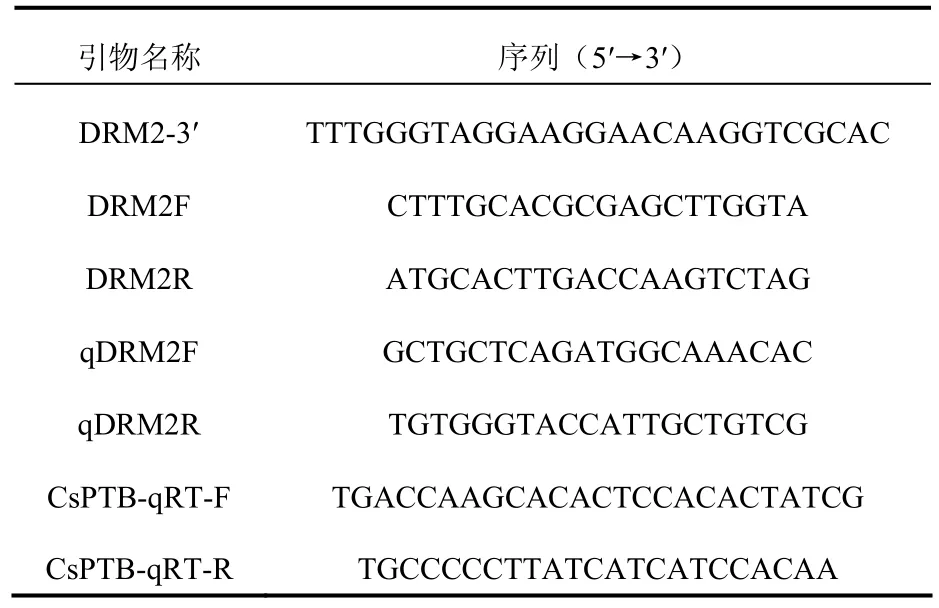
表1 试验引物及其序列Table 1 Sequence of primers applied

图1 3'-RACE(A)和RT-PCR(B)电泳结果(M表示2000bp Marker)Fig. 1 Electrophoresis of 3'RACE (A) and RT-PCR (B)(M is 2000bp Marker)
2 结果与分析
2.1 CsDRM2 cDNA全长序列的克隆
根据本实验室前期茶树冷驯化样品转录组数据库中筛选出的一条与DRM2基因5'端高度同源的EST序列,设计3'RACE特异引物,以提取的冷驯化阶段的RNA为材料,运用SMARTer RACE技术获得DRM2基因的3'端序列(图1)。测序后用DNAstar软件将原始EST序列与3'端测序获得的序列进行拼接,获得具有开放阅读框(ORF)的序列。再在ORF两端设计特异引物,进行RT-PCR(图2),测序验证,获得完整的序列。经BLAST搜索比对,证明该序列编码完整的结构域重排甲基转移酶,命名为CsDRM2(NCBI登录号:KR057963)。
2.2 茶树CsDRM2基因的生物信息学分析
茶树CsDRM2基因cDNA全长2328bp,其中开放阅读框(ORF)长1815bp,编码604个氨基酸,蛋白质分子量为67.39 kD,理论等电点(PI)为4.85。SingalP进行信号肽预测显示,该氨基酸序列无信号肽位点,属于非分泌性蛋白质;TNHMM预测显示该蛋白无跨膜螺旋结构;WoLF PSORT亚细胞定位预测显示其在细胞核中的可能性最高,因此DRM2基因要定位在细胞核内,在细胞核中催化DNA发生甲基化。
将CsDRM2编码的氨基酸序列与GenBank中已经登录的葡萄(Vitis vinifera, XP_010660894.1)、梅花(Prunus m ume, XP_008230579.1)、芝麻(Sesamum in dicum, XP_011097426.1)、黄瓜( Cucumis sativus, XP_004164820.1)、 烟 草(Nicotiana t abacum, BAC67060.1)、苹果(Malus domestica, XP_008379234.1)、白梨(Pyrus x bretschneideri, XP_009334102.1)、大豆(Glycine max,XP_003524549.1)等11种植物的DRM2氨基酸序列进行比对,结果显示,茶树与上述物种的DRM2氨基酸序列相似性较高,相似性高于65%(图2)。DRM2包含N-末端的UBA结构域和C-末端的甲基转移酶结构域,并且C末端的氨基酸序列高度保守。

图2 茶树与其他植物的DRM2氨基酸序列比对Fig. 2 Multiple alignment on amino acid sequences of tea and various plant

图3 茶树与其他植物的DRM2氨基酸序列的进化树分析Fig. 3 Phy logenetic tree analysis on DRM2 protein of tea plants

图4 茶树CsDRM2的三级结构预测Fig. 4 The tertiary structure homology-modeling of CsDRM2

图5 CsDRM2在不同冷驯化阶段的表达水平Fig. 5 RT-PCR expression of CsDRM2 at various stages of cold acclimatization
用MEGA5.0软件构建茶树与其他18个物种的DRM2氨基酸序列进化树发现(图3),植物中DRM2可以分为两种类型,即A型和B型,其中葡萄、苹果等的属于A型,B型的成员较少,茶树CsDRM2属于B型,芝麻、烟草和茄属植物中的DRM2也属于B型。在进化树上,茶树CsDRM2蛋白与芝麻的关系最近(图3)。
SOPMA 预测的CsDRM2的蛋白质二级结构显示,其与已报道的拟南芥、葡萄的DRM2的二级结构有较高的相似性。主要是以α螺旋、延伸链、β转角以及无规则卷曲为主,它们所占的百分比为36.42%、13.08%、9.44%、41.06%。三级结构分析结果显示(图4),茶树CsDRM2在高级结构上与其他的DRM2具有较高的相似性,与数据库中人类DNA甲基转移酶的三级结构(PDB:4onj.1)相似性达84%,说明DRM2在生物体中的结构较保守,反映了DRM2介导的DNA甲基化广泛存在于生物中。
2.3 CsDRM2在不同冷驯化阶段的表达分析
以PTB基因作为内参基因,用荧光定量PCR技术分析了CsDRM2在3个不同冷驯化阶段的茶树叶片的基因表达情况,结果表明(图5),在冷驯化不同阶段之间,CsDRM2基因的表达量具有明显差异,与对照(11.2)相比,随着冷驯化的进行,CsDRM2的表达量呈现上升趋势,在脱驯化时期表达量达到最大,与前期本实验室甲基化研究结果的趋势类似[5],说明CsDRM2的表达与茶树冷驯化之间有一定关系,这种表达差异影响到茶树叶片的甲基化水平和状况。
3 讨论
对模式植物拟南芥的研究发现,拟南芥中至少存在3条甲基化途径,且每条途径与一种特定的甲基转移酶关联[13]。甲基转移酶 1(MET1)是哺乳动物中的DNA甲基转移酶1(DNMT1)的同系物,能够维持CG双核苷酸位点的甲基化;染色质甲基化酶3(CMT3)是植物特有的甲基转移酶,能够使CHG(H=A,T 或 C)位点优先发生甲基化;结构域重新甲基化酶2(DRM2)是哺乳动物DNMT3的同系物,能够独自完成DNA从头甲基化过程。3种甲基转移酶的共同作用完成了DNA甲基化的维持以及从头甲基化过程。Cao等[14]通过研究拟南芥RNA介导的DNA甲基化过程中甲基转移酶DRM基因的作用,最后证明DRM能够诱导CG、CNG以及非对称位点序列建立最初的RNA介导的DNA甲基化。本研究克隆获得了茶树CsDRM2基因的全长。对其进行生物信息学分析,发现茶树DRM2蛋白与芝麻和烟草的亲缘关系最近,CsDRM2的氨基酸序列与其他植物的DRM2相似性均高于65%。不同冷驯化阶段的茶树叶片荧光定量PCR结果表明,随着冷驯化的进行,DRM2基因的表达量逐渐增加,在冷驯化时期的表达量高于冷驯化之前,在脱驯化时期表达量达到最大。CsDRM2基因在不同冷驯化阶段茶树叶片中表达量的变化趋势与我们前期进行的MSAP和HPLC检测到的甲基化水平变化趋势相一致[5],说明茶树能够通过改变CsDRM2基因的表达水平来调节基因组DNA甲基化的变化,从而参与到对低温胁迫的响应中去。当然,茶树中是否还存在其他类型的DNA甲基化转移酶基因、这些基因是否也参与到茶树DNA甲基化的过程,仍需要进行后续的深入研究。
[1] Bender J. DNA methylation and epigenetics [J]. Annu Rev Plant Biol,2004,(55):41-68.
[2] Vanyushin B F and Ashapkin V V. DNA methylation in higher plants: past, present and future [J]. Biochim Biophys Acta,2011,1809(8):360-368.
[3] Chan S W, Henderson I R, Jacobsen S E. Gardening the genome: DNA methylation in Arabidopsis thaliana [J]. Nat Rev Genet,2005,6(5):351-360.
[4] 赵云雷,叶威武,王俊娟,等.DNA甲基化与植物抗逆性研究进展[J].西北植物学报,2009,29(7):1479-1489.
[5] 周艳华,曹红利,岳川,等.冷驯化不同阶段茶树 DNA甲基化模式变化分析[J].作物学报,2015,41:印刷中.
[6] Cao X and Jacobsen S E. Role of the arabidopsis DRM methyltransferases in de novo DNA methylation and gene silencing [J]. Curr Biol, 2002,12(13):1138-1144.
[7] Chan S W, Zilberman D, Xie Z, et al. RNA silencing genes control de novo DNA methylation [J]. Science,2004,303(5662):1336.
[8] Wada Y, Ohya H, Yamaguchi Y, et al. Preferential de novo methylation of cytosine residues in non-CpG sequences by a domains rearranged DNA methyltransferase from tobacco plants [J]. J Biol Chem, 2003,278(43):42386-42393.
[9] Wang X-C, Zhao Q-Y, Ma C-L, et al. Global transcriptome profiles of Camellia sinensis during cold acclimation [J]. BMC Genomics,2013,14(1):415.
[10] 杨亚军,郑雷英,王新超.冷驯化和 ABA对茶树抗寒力及其体内脯氨酸含量的影响[J].茶叶科学,2004,24(3):177-182.
[11] 曹红利,岳川,郝心愿,等.茶树胆碱单加氧酶CsCMO的克隆及甜菜碱合成关键基因的表达分析[J].中国农业科学,2013,46(15):3087-3096.
[12] Livaka K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the2-ΔΔCTmethod [J]. Methods,2001,25(4):402-408.
[13] Deleris A, Greenberg M V, Ausin I, et al. Involvement of a Jumonji-C domain-containing histone demethylase in DRM2-mediated maintenance of DNA methylation [J]. EMBO Rep, 2010,11(12):950-955.
[14] Cao X, Aufsatz W, Zilberman D, et al. Role of the DRM and CMT3 methyltransferases in RNA-directed DNA methylation [J]. Curr Biol,2003,13(24):2212-2217.
Cloning and Expression Analysis on DNA Methyltransferase Gene,CsDRM2, of Camellia sinensis
ZHOU Yan-Hua,CAO Hong-Li,YUE Chuan,WANG Lu,HAO Xin-Yuan,WANG Xin-Chao*,YANG Ya-Jun*
(Tea Research Institute of the Chinese Academy of Agricultural Sciences / Key Laboratory of Tea Biology and Resources Utilization,Ministry of Agriculture / National Center for Tea Improvement,Hangzhou,Zhejiang 310008,China)
As one of the major pathways of epigenetic regulation, DNA methylation can modify genomic DNA and take part in responding to the environmental stresses of the plants. The process requires the involvement of DNA methyltransferase. It was found,in our previous study, to occur during the cold acclimation. In this study, a full-length cDNA sequence of CsDRM2 gene was obtained using the RACE technique. Subsequently, it was submitted to NCBI and assigned an accession number of KR057963. The cDNA length of CsDRM2 was 2,328 bp containing a 1,815 bp open reading frame and the encoding 604 amino acid residues. The predicted molecular weight was 67.39 kD and theoretical isoelectric point pH 4.85. The comparison on their sequences showed a similarity greater than 65% between CsDRM2 and other reported DRMs. The phylogenetic tree analysis on CsDRM2 indicated its close genetic relationships with Sesamum indicum and Nicotiana tabacum. The qRT-PCR results on CsDRM2 revealed differential expressions of the gene at different stages of the cold acclimation. It suggested that CsDRM2 participated in the DNA methylation of the tea plant in response to cold stress.
tea plant; DNA methyltransferase gene CsDRM2; cloning; gene expression; cold acclimation
S571.1
A
2015-01-07 初稿;2015-03-09 修改稿
国家自然科学基金(31170650);国家茶叶产业技术体系(CARS-23);浙江省农业新品种选育重大专项(2012C2905-3)。
周艳华(1990-),女,硕士研究生,主要从事茶树育种与抗性机制研究。
E-mail: wangxinchao@caas.cn;yjyang@tricaas.com
