连接酶-ELISA反应检测循环DNA基因突变研究
2015-08-24崔海忠肖娜张永平陈大贵唐一通
崔海忠,肖娜,张永平,陈大贵,唐一通△
新技术交流
连接酶-ELISA反应检测循环DNA基因突变研究
崔海忠1,肖娜2,张永平1,陈大贵1,唐一通2△
目的 研究一种简便、灵敏的单核苷酸多态性(SNP)分型方法,使其能够在简单实验条件下进行常规的临床样本检测。方法 设计针对突变位点的检测探针,通过检测探针的连接、通用扩增、标记和ELISA反应,根据检测位点对应反应管显色值判定突变位点的基因型。以表皮生长因子受体(EGFR)基因外显子21和18上的3个SNP突变位点L858R、L861Q和G719C为检测对象,对62例肺癌血浆循环DNA样本进行检测,并与直接测序结果进行比较。结果 通过对3个突变位点的检测,2种方法均在L858R位点检出杂合子突变。直接测序法仅能够明确检出2例杂合子突变,另外1例样本因在突变位点出现不明显的套峰而无法明确判定突变类型。而新方法能够明确检出6例杂合子突变。结论 建立了一种基于连接酶-ELISA的简便、灵敏的SNP突变检测方法,适合于在简单实验条件下对不均一样本进行常规突变检测。
多态性,单核苷酸;突变;基因型;DNA连接酶类;酶联免疫吸附测定
单核苷酸多态性(single nucleotide polymorphism,SNP)被认为是疾病易感性和药物反应的决定因素之一,开展SNP研究对遗传学、医学、药物开发与合理用药等的发展都具有重要意义。目前,SNP突变检测方法主要有限制性片段长度多态性聚合酶链反应(PCR-RFLP)[1]、DNA测序、PCR-单链构象多态性(PCR-SSCP)[2]、实时荧光定量PCR(Real-Time PCR)[3]、TaqMan PCR[4]、焦磷酸测序(Pyrosequencing)[5-6]、质 谱 技 术(Mass Spectrometry)[7]、SNPstream技术[8]、分子灯塔技术[9]、液相芯片技术[10]等。传统的DNA测序检测灵敏度较低[11],因此在对肿瘤组织、穿刺活检组织或体液样本等不均一样本进行检测时有较大局限性。虽然质谱、Pyrosequencing等技术检测灵敏度较高,但其对实验条件、仪器设备、试剂等具有很高的要求,在简单实验条件下的常规临床检测中难以广泛应用。笔者通过对构建质粒模板的检测,建立了一种新的SNP检测方法,其灵敏度可达5%[12]。本文进一步通过对非小细胞肺癌循环DNA的检测,研究一种基于连接酶-ELISA的简便、灵敏的SNP突变检测方法,使其适合于在简单实验条件下对不均一样本进行常规临床检测。
1 材料与方法
1.1 实验材料 寡核苷酸探针:表皮生长因子受体(EGFR)基因外显子21和18上的3个突变位点L858R、L861Q、G719C的检测探针序列,见表1。对检测探针进行通用引物扩增的Tag1序列和Tag2的互补序列(CTag2)分别为Tag1:5′biotin-GGGTTCGTGGTAGAGCGTCGGAGT-3′;CTag2:5′digoxin-CCAGACGACACCGAGATAGCAGCC-3′。Tag1和CTag2序列5′端分别为生物素(biotin)和地高辛(digoxin)分子修饰。所有核酸序列均由上海生工生物有限公司合成。
样本来源:收集湖北文理学院附属医院及枣阳临床学院2013年6月—2014年8月确诊的非小细胞肺癌患者血浆样本62例(36例腺癌,26例鳞癌)。其中男37例,女25例。
实验试剂:低分子质量DNA Marker-A购自生工生物工程(上海)有限公司;2×PCR Mastermix购自天根生化科技北京有限公司;链亲和素磁珠、磁性分离器购自西安金磁纳米生物技术有限公司;Taq DNA ligase购自New England BioLabs;QIAamp DNA Blood Mini Kit(QIAGEN)购自上海叶舟生物科技有限公司。其余试剂均为分析纯。
1.2 实验方法
1.2.1 EGFR基因扩增 用QIAamp DNA Blood Mini Kit抽提血浆样本循环DNA。对外显子18和21进行PCR扩增,扩增引物为:21-forward:5′-GAGCTTCTTCCCATGATGATCT-3′;21-reverse:5′-GAAAATGCTGGCTGACCTAAAG-3′;18-forward:5′-GAGGTGACCCTTGTCTCTGTGT-3′;18-reverse:5′-CCCAAACACTCAGTGAAACAAA-3′。扩增体系为:3 μL循环DNA样本,0.5 μmol/L扩增引物,10 μL 2×Taq PCR Mastermix,去离子水补足20 μL。扩增条件为:95℃4 min,95℃30 s,60℃30 s,72℃40 s,30个循环;72℃4 min。测定各样本扩增产物序列。
1.2.2 突变检测 (1)连接反应。针对某个SNP位点,用上游的2条特异性检测探针和下游的1条公用探针进行检测。L858R、L861Q、G719C 3个突变位点的检测探针同前期建立方法[12]。每组3个检测管(标记为WT、MT和对照CT管)用来检测1个SNP位点。WT和MT管分别加入2 μL 10×连接反应缓冲液,2 μL扩增产物和1 U Taq ligase,在MT管中加入0.1 pmol突变型特异性检测探针和公用检测探针,WT管中加入0.1 pmol野生型特异性检测探针和公用检测探针,去离子水补足20 μL。对照管CT中不加入模板序列,其他成分与检测管相同。反应条件为:94℃1 min,57℃2 min;5个循环;95℃5 min。(2)通用引物扩增。取2×PCR Mastermix 10 μL,各管连接反应产物2 μL,2 μmol/L通用扩增引物Tag1和CTag2,去离子水补足20 μL。95℃30 s;95℃25 s,65℃45 s,72℃20 s;20个循环,72℃1 min进行扩增反应。(3)ELISA检测。将扩增产物移入96孔板对应孔中,链亲和素磁性微粒进行纯化,将磁性微粒保存于20 μL Tris-HCl缓冲液(10 mmol/L,pH 7.5)中。各孔中加入50 μL HRP标记抗地高辛抗体溶液(20%胎牛血清1∶1 000稀释),恒温摇床中室温,70 r/min,30 min。磁性分离,200 μL清洗缓冲液(1× PBST)清洗3次,加入TMB底物缓冲液100 μL,室温显色5 min,2 mol/L H2SO4终止反应,设定背景波长595 nm,检测波长450 nm,酶标仪(ELX 800 UV,BIO-TEK INSTRUMENTS,INC)测定各孔光密度(OD450)值。根据各检测位点WT管、MT管和CT管对应各孔显色值(ODWT、ODMT和ODCT)判定所检测SNP位点基因型(ODCT<0.05时,以ODCT=0.05计算)。以ODWT/ODCT和ODMT/ODCT比值作为判断标准:若ODWT/ODCT>5,同时ODMT/ODCT>5,则此检测位点为杂合子突变;若只有ODWT/ODCT>5,则此检测位点为野生纯合子;若只有ODMT/ ODCT>5,则此检测位点为突变纯合子。
1.2.3 检测流程 野生纯合子SNP位点的检测流程,见图1。通过连接反应,WT管中野生型检测探针和公用探针能完成连接,而MT管中突变型检测探针和公用探针不能连接,经通用引物Tag1和CTag2的通用扩增和标记,通过ELISA进行分型检测,检测结果可通过观察WT管和MT管对应显色孔显色值来判定。结果判定方法为,若只在WT管对应显色孔有显色值,则检测位点为野生型;若WT、MT管对应显色孔均有显色值,则为杂合突变型;若只在MT管对应显色孔有显色值,则为纯合突变型。
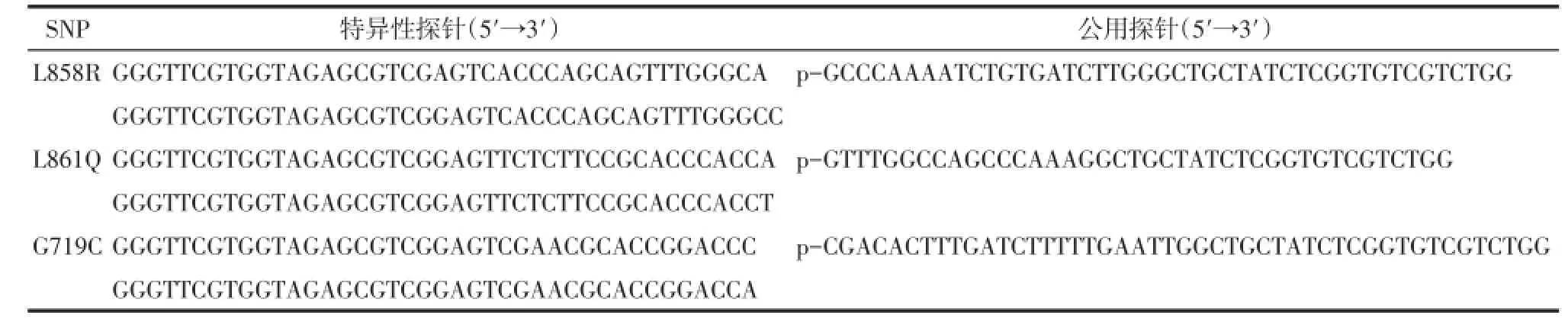
Tab.1 The sequences of oligonucleotide probes表1 寡核苷酸检测探针序列
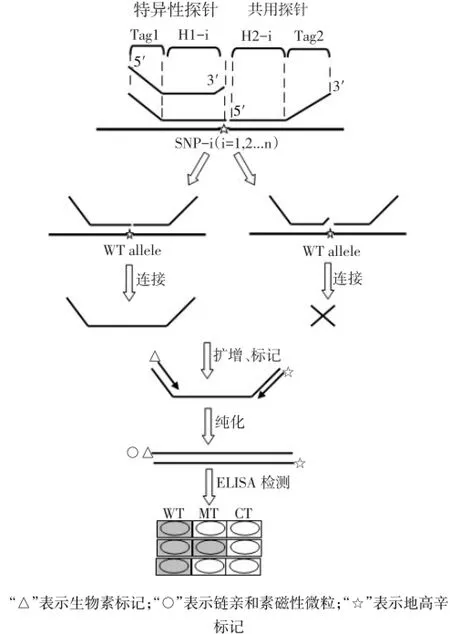
Fig.1 Schematic representation of ligation-ELISA assay图1 连接酶-ELISA分型方法检测流程图
2 结果
对62例样本L858R、L861Q和G719C 3个突变位点进行了检测,并与直接测序结果进行了比较。2种方法未检测到L861Q和G719C位点的突变,但均检测到L858R位点有杂合子突变。直接测序仅可以明确检测出8#、15#2例样本是L858R位点杂合子突变,而23#样本出现类似杂峰的突变碱基对应峰,无法得出明确的基因型判断;6#、11#、14#样本因为突变碱基对应峰无法从杂峰中分离,更加不能检出突变基因型,见图2。连接酶-ELISA方法共检测出6#、8#、11#、14#、15#、23#共6例样本是L858R杂合子突变。且6个突变型样本对应WT管在450 nm处的ODWT在1.58和1.82之间,MT管在450 nm 处ODMT均高于0.96,ODWT/ODCT和ODMT/ODCT均大于10,见表2。野生型样本中(37#、42#、50#、58#样本),各WT管在450 nm处ODWT大于1.5,MT管的450 nm处ODMT均低于0.16,ODWT/ODCT大于10,而ODMT/ODCT均小于5。另外,ODMT(杂合)/ODMT(野生)均高于5。且ODWT/ODCT及ODWT/ODMT也均高于10。因此,新建立方法有着简便、灵敏的特点,适合于从不均一的样本中进行突变等位基因的检测。6例突变样本(6#、8#、11#、14#、15#、23#)和4例野生型样本(37#、42#、50#、58#)对应各检测管的显色值见表2。
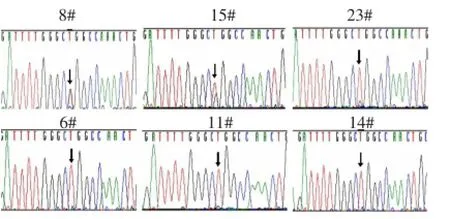
Fig.2 Results of direct sequencing for L858R in EGFR of circulating DNA samples图2 循环DNA样本EGFR基因L858R位点测序图
Tab.2 Genotyping of the circulating DNA samples for L858R in EGFR with ELISA表2 循环DNA样本EGFR基因L858R位点ELISA基因型分型检测 (n=62,±s)

Tab.2 Genotyping of the circulating DNA samples for L858R in EGFR with ELISA表2 循环DNA样本EGFR基因L858R位点ELISA基因型分型检测 (n=62,±s)
检测管6#样8 本#中各检测11 管#对应显色14 值#15# WT1.65±0.071.78±0.131.63±0.121.82±0.161.63±0.08 MT1.22±0.071.81±0.150.96±0.051.27±0.081.60±0.06 CT0.04±0.030.08±0.020.06±0.020.07±0.020.07±0.01检测管23#样37 本#中各检测42 管#对应显色50 值#58# WT1.58±0.091.52±0.101.82±0.151.71±0.071.88±0.14 MT1.41±0.020.12±0.040.14±0.060.15±0.030.16±0.02 CT0.06±0.030.06±0.030.07±0.020.05±0.040.08±0.02
3 讨论
3.1 SNP检测技术及应用现状 近年来SNP的检测方法被广泛研究。但SNP检测技术在检测成本、灵敏度和常规性等方面仍需改进。如DNA直接测序的灵敏度仅有20%[11],在对不均一样本进行突变检测时,无法将突变等位基因对应峰线从背景峰中明确分离,从而造成假阴性结果。虽然焦磷酸测序、实时定量PCR等方法具有较高灵敏度,报道称可达到5%[13]和1%[14],但这些方法需依赖昂贵仪器或专业人员的技术支持,且操作复杂,不能在常规实验条件下进行突变检测,限制了其在基层医疗和研究机构的应用。
3.2 建立方法灵敏度及应用性分析 本研究方法具有较高检测灵敏度,通过在检测探针上设计通用扩增引物序列Tag1和Tag2,完成连接反应后,进行连接探针的二次扩增,以大大增加检测产物的数量,从而进一步在ELISA水平进行快速检测,其检测灵敏度可达5%[12],因此适合于从不均一样本中对突变等位基因进行检测。通过对62例样本的检测,结果表明,直接测序结果仅能够明确检出2例,而其他3例因无法有效区分背景峰,不能明确判定。而本方法能够根据ELISA结果从中明确判定出6例杂合子样本,具有较高的检测灵敏度。此外,与其他检测方法如焦磷酸测序、荧光定量PCR、质谱等方法相比,本方法操作简单,不需借助昂贵的实验仪器设备,只需普通PCR仪、酶标仪等简单实验条件即能完成。同时,所需检测探针长度均小于50 bp,合成成本低,也没有对荧光探针等昂贵试剂及高质量检测样本的要求,具有较低的检测成本和易操作性,因此更加适合于在普通简单实验条件下进行检测。
3.3 建立方法特异性分析 为保证本方法具有较高的检测特异性,在设计检测探针时,除依靠特异性检测探针3′端碱基与突变碱基的配对识别能力外,还在其3′端上游第3个碱基处引入1个错配碱基以增强探针的识别能力。同时,特异性检测探针和公用探针的H1和H2序列的Tm值与通用扩增引物Tag1和Tag2序列的Tm值相差7~8℃,以减少连接反应和后续的扩增反应之间的影响。
总之,本研究通过对非小细胞肺癌循环DNA样本的检测,建立了一种基于连接酶-ELISA的简便、灵敏的SNP突变检测方法。该方法操作简便,有较高的检测特异性和灵敏度,适合于在一般实验条件下对不均一样本进行常规突变检测。但因ELISA方法的固有局限性,本方法不能对未知SNP位点进行突变检测,且在检测通量上也有限制,不适合于进行高通量检测。
[1]Qiao W,Wang T,Zhang L,et al.Association study of single nucleotide polymorphisms in XRCC1 gene with the risk of gastric cancer in chinese population[J].Int J Biol Sci,2013,9(7):753-758.doi: 10.7150/ijbs.6783.eCollection 2013.
[2]Serrano ML,Yunis JJ.Identification of three new mutations in the RB1 gene in patients with sporadic retinoblastoma in Colombia[J].Biomedica, 2013, 33(1):53-61.doi: 10.1590/S0120-41572013000100007.
[3]Hung CC,Chiou MH,Huang BH,et al.Impact of genetic polymorphisms in ABCB1,CYP2B6,OPRM1,ANKK1 and DRD2 genes on methadone therapy in Han Chinese patients[J].Pharmacogenomics,2011,12(11):1525-1533.doi:10.2217/pgs.11.96.
[4]Farivar TN,Nezam MK,Johari P.Genotyping of hepatitis C virus isolated from hepatitis patients in Southeast of Iran by Taqman Realtime PCR[J].J Pak Med Assoc,2011,61(6):586-588.
[5]Kang SH,Pyo JY,Yang SW,et al.Detection of BRAF V600E mutation with thyroid tissue using pyrosequencing:comparison with PNA-clamping and real-time PCR[J].Am J Clin Pathol,2013,139 (6):759-764.doi:10.1309/AJCPN3ULH6YWBHPH.
[6]Liu TF,Zhang XL,Cai WZ,et al.Studies on the correlation of PON1 gene rs854572 single nucleotide polymorphism to clopidogrel resistance[J].Med J Chin PLA,2012,37(10):982-985.[刘滕飞,张效林,蔡文芝,等.PON1基因rs854572单核苷酸多态性与氯吡格雷抵抗的相关性研究[J].解放军医学杂志,2012,37(10): 982-985].
[7]Mauger F,Gelfand DH,Gupta A,et al.High-specificity singletube multiplex genotyping using Ribo-PAP PCR,tag primers,alkali cleavage of RNA/DNA chimeras and MALDI-TOF MS[J].Hum Mutat,2013,34(1):266-273.doi:10.1002/humu.22227.
[8]Fei LJ,Ji LD,Zhang LN,et al.Application of SNPstream for medical genetics research[J].Chin J Med Genet,2012,29(1):9-12.[费丽娟,季林丹,张莉娜,等.SNPstream基因分型技术在医学遗传学研究中的应用[J].中华医学遗传学杂志,2012,29(1):9-12].doi:10.3760/cma.j.issn.1003-9406.2012.01.003.
[9]Liu XQ.Association study of the C2 and C3 and age-related macular degeneration in Chinese population by SnaPshot methods[J].Int J Lab Med,2013,34(3):285-287.[刘小琦.采用SNaPshot方法对中国老年黄斑变性与C2和C3基因单核苷酸多态性进行相关性研究[J].国际检验医学杂志,2013,34(3):285-287].doi: 10.3969/j.issn.1673-4130.2013.03.012.
[10]Xiao F,Zhang WM,Zhang WY.Relations of epithelial-mesenchymal transition and expression of IGF-1R to the efficacy of gefitinib as second-line therapy for late-stage non-small cell lung cancer[J].Med J Chin PLA,2011,36(6):563-568.[肖锋,张为民,张文颖.EMT和IGF-1R表达与吉非替尼二线治疗晚期非小细胞肺癌疗效的关系[J].解放军医学杂志,2011,36(6):563-568].
[11]Gillian E,Zhu GS,Alexandros M,et al.EGFR mutation testing in lung cancer:a review of available methods and their use for analysis of tumour tissue and cytology samples[J].J Clin Pathol,2013,66(2):79-89.doi:10.1136/jclinpath-2012-201194.
[12]Cui HZ,Xiao N,Zhang YP,et al.A new method for detection of single nucleotide polymorphisms with ligase-ELISA[J].J Biol,2014,31 (4):95-98.[崔海忠,肖娜,张永平,等.基于连接酶-ELISA反应的单核苷酸多态性分型新方法[J].生物学杂志,2014,31(4):95-98].doi:10.3969/j.issn.2095-1736.2014.04.095.
[13]Dufort S,Richard MJ,de Fraipont F.Pyrosequencing method to detect KRAS mutation in formalin-fixed and paraffin-embedded tumor tissues[J].Analytical Biochem,2009,391(2):166-168.doi: 10.1016/j.ab.2009.05.027.
[14]Lang AH,Heinz D,Simone GR,et al.Optimized allele-specific real-time PCR assays for the detection of common mutations in KRAS and BRAF[J].J Mol Diagn,2011,13(1):23-28.doi:10.1016/ j.jmoldx.2010.11.007.
(2014-09-15收稿 2014-12-10修回)
(本文编辑 李鹏)
Study for gene mutation detection of circulating DNA with ligase-ELISA reaction
CUI Haizhong1,XIAO Na2,ZHANG Yongping1,CHEN Dagui1,TANG Yitong2△
1 Zaoyang Clinical College,Medical College of Hubei University of Arts and Science,Zaoyang 441200,China;2 Key Laboratory of Molecular Medicine,Medical College of Hubei University of Arts and Science
△Corrsponding Author E-mail:yitongtang@126.com
Objective To establish a single nucleotide polymorphisms genotyping(SNP)method for a convenient,accurate,and routine analysis of clinical samples.Methods Based on the design of oligonucleotide probe,the assay was performed through three steps:the conjunction of the detection probe,universal amplification,labeling and ELISA reaction.The genotype of each SNP was revealed by reading signals of each set of reaction tubes.This assay was applied to detect sixtytwo plasma samples of lung cancer for circulating DNA for three SNPs of EGFR,c.2573T>G(L858R),EGFR,c.2582T>A (L861Q),EGFR,c.2155 G>T(G719C).Results were compared with those obtained by direct sequencing.Results The heterozygote mutation was identified for L858R by both methods,although no mutation was detected for L861Q and G719C.Six samples were identified as heterozygotes with the new method,and only two samples were unambiguously identified as heterozygotes by the direct sequencing.Two additional samples could not be identified as heterozygotes because the peak of mutant allele was very low compared with that of wild allele.Conclusion The developed method enabled accurate identification of SNP in a convenient manner,and which is adapted to routine analysis from heterogeneous samples unambiguously.
polymorphism,single nucleotide;mutation;genotype;DNA ligases;enzyme-linked immunosorbent assay
R394-33
A DOI:10.11958/j.issn.0253-9896.2015.05.023
湖北省卫生计生科研基金(WJ2015MB266);襄阳市科技局项目(襄科业[2012]43号);湖北省教育厅项目(Q20132604);湖北省教育厅高校青年教师深入企业计划项目(XD2014245)
1湖北文理学院医学院枣阳临床学院(邮编441200);2湖北文理学院医学院分子医学重点实验室
崔海忠(1972),男,主治医师,硕士,主要从事肿瘤学方面研究
△E-mail:yitongtang@126.com
