犬细小病毒山东分离株VP 2基因的克隆与遗传变异分析
2015-08-08于永乐张传美杨海燕杨瑞梅张洪亮
于永乐,张传美,张 倩,杨海燕,杨瑞梅,张洪亮,单 虎
(1.青岛农业大学动物科技学院,山东 青岛 266109;2.山东省黄岛出入境检验检疫局,山东 青岛 266555)
犬细小病毒(CPV)是引起幼犬出血性胃肠炎和心肌炎的主要病原体之一。CPV-2最早发现于1978年,1978-2000年间,CPV-2先后发生了3次重要的基因变异,产生了CPV-2a、CPV-2b和CPV-2c 3种主要的抗原变异体[1]。到目前为止,新型变异株New CPV-2a、New CPV-2b、CPV-2c(a)和CPV-2c(b)已逐步取代了原始毒株在多个国家广泛流行。在我国,1982年最早报道了此病的发生,此后的30多年伴随着该病毒的不断进化和变异。尽管目前为预防该病,疫苗接种工作已在国内普及,但由于毒株不断变异等原因,该病毒仍然广泛流行,给我国养犬业带来极大的损失。
CPV是单股、负链、线性DNA病毒,基因组全长5.2kb,包含两个大开放阅读框ORF1(编码结构蛋白VP1和VP2)和ORF2(编码非结构蛋白NS1和NS2),其中VP2蛋白是主要的衣壳蛋白和病毒保护性抗原蛋白,同时具有自我装配形成病毒样颗粒(VLPS)的能力。此外,研究表明,VP2基因某些区域的点突变会对病毒抗原性的差异造成影响[2]。本研究对山东地区分离的12株不同的CPV进行VP2基因扩增,并与国内外流行毒株进行序列比较及遗传变异分析,构建系统进化树,确定毒株类型,以便及时了解CPV流行毒株的差异及变异情况,为加强CPV的防治工作和制备有效的疫苗提供理论支持和参考。
1 材料与方法
1.1 材料 12株不同的CPV(命名为SDCP1-12),由青岛农业大学山东省预防兽医学重点实验室分离鉴定并保存。质粒小量提取试剂盒为美国Omega公司产品;DNAiso Regent、DNA-Marker DL-2000、Taq DNA聚合酶、pMD 18-T Vector,均购自宝生物工程(大连)有限公司;DNA胶回收试剂盒,购自上海生工生物工程技术服务有限公司。
1.2 方法
1.2.1 引物合成与VP2基因的扩增 根据GenBank上发表的CPV基因序列(登录号:KF803643),设计1对扩增VP2全基因的引物,由北京赛百盛基因技术有限公司合成。扩增片段长度为1755 bp。上游:5′-ATGAGTGATGGAGCAGTTC-3′,下游:5′-TTAA TATAATTTTCTAGGTGC-3′,按照DNAiso Regent说明书提取病毒DNA,以提取12株CPV的DNA为模板进行PCR扩增,反应结束后,PCR产物于1%琼脂糖凝胶进行电泳鉴定。
1.2.2 PCR产物的回收、测序与序列分析 对PCR产物用试剂盒进行胶回收和纯化,将目的片段与pMD18-T载体连接,并转化DH5α感受态细胞,挑菌提取质粒进行PCR鉴定,并将阳性质粒进行序列测定。用DNAStar软件将12株VP2基因的序列与CPV参考株序列以及国内外流行毒株序列进行比较分析。用MEGA 5.0构建核苷酸系统进化树。
2 结果
2.1 VP2基因扩增、测序及同源性分析 PCR扩增12株CPV,电泳结果出现与预期1755 bp大小一致的目的条带。经过序列测定,获得12株CPV的VP2核苷酸序列。12株分离株(SDCP1-12)之间的核苷酸同源率为98.9%~100.0%,氨基酸同源率为98.8%~100.0%;与7株国外参考株CPV-b、CPV-15、V120、CPV-39、Taichung、V139、V203、56/00相比较,核苷酸同源率为98.8%~99.4%,氨基酸同源率为97.9%~100.0%;与疫苗株Vac1的核苷酸同源率为98.6%~99.0%,氨基酸同源率为97.4%~98.5%。表明各分离株与参考毒株同源率较高,差异小;但与疫苗株同源性相对较低,差异大。
2.2 VP2基因中非同义置换的碱基和氨基酸 将分离株核苷酸序列和氨基酸序列与参考毒株对比,与原始的CPV 2a/2b亚型毒株CPV-15/CPV-39相比,第297位氨基酸发生了Ser→Ala突变,第426位氨基酸发生了Asn→Asp突变,据此判定本次分离株SDCP1-4、SDCP6、SDCP9-11为New CPV-2a亚型,SDCP5、SDCP7、SDCP8、SDCP12为New CPV-2b亚型。所有分离株在87位(Met→Leu)、101位(Ile→Thr)、300位(Ala→Gly)、305位(Asp→Tyr)的突变相一致,这4处氨基酸的替换被认为是区分CPV-2及其变异体的重要依据[3]。此外,SDCP1第321位氨基酸N→D,497位氨基酸Q→L;SDCP5第167位氨基酸N→K,399位氨基酸T→I,549位氨基酸Q→L;SDCP10第551位氨基酸M→V;SDCP11第136位氨基酸L→M,182位氨基酸T→S;SDCP12第380位氨基酸F→C,这9处氨基酸的替换从未被报道,是本次分离株所特有的氨基酸突变。
2.3 系统进化树分析 对12个分离株与Gen⁃Bank发表的61个国内外CPV分离株进行比对,构建系统进化树(图1)。从图中可以看出,除了SD⁃CP4和SDCP6外,其余各分离株聚集形成一个大的分支,与我国台湾地区、泰国、韩国CPV株关系较近处于一个分支上,而与意大利、美国和越南株进化关系较远。与国内毒株相比,8个New CPV-2a亚型毒株与吉林、黑龙江、甘肃、北京等地毒株亲缘关系较近;4个New CPV-2b亚型毒株与广东、广西等地毒株亲缘关系较近,这与王净等[4]研究一致,认为我国长江以北地区主要以CPV-2a流行为主,而广东、云南、四川等南方地区则以CPV-2b流行为主。值得讨论的是除了CPV07-04外,其余所有的山东株与本次分离株在拓扑分类上距离较远。总体看来,12个分离株并没有形成独立的分支。
3 讨论
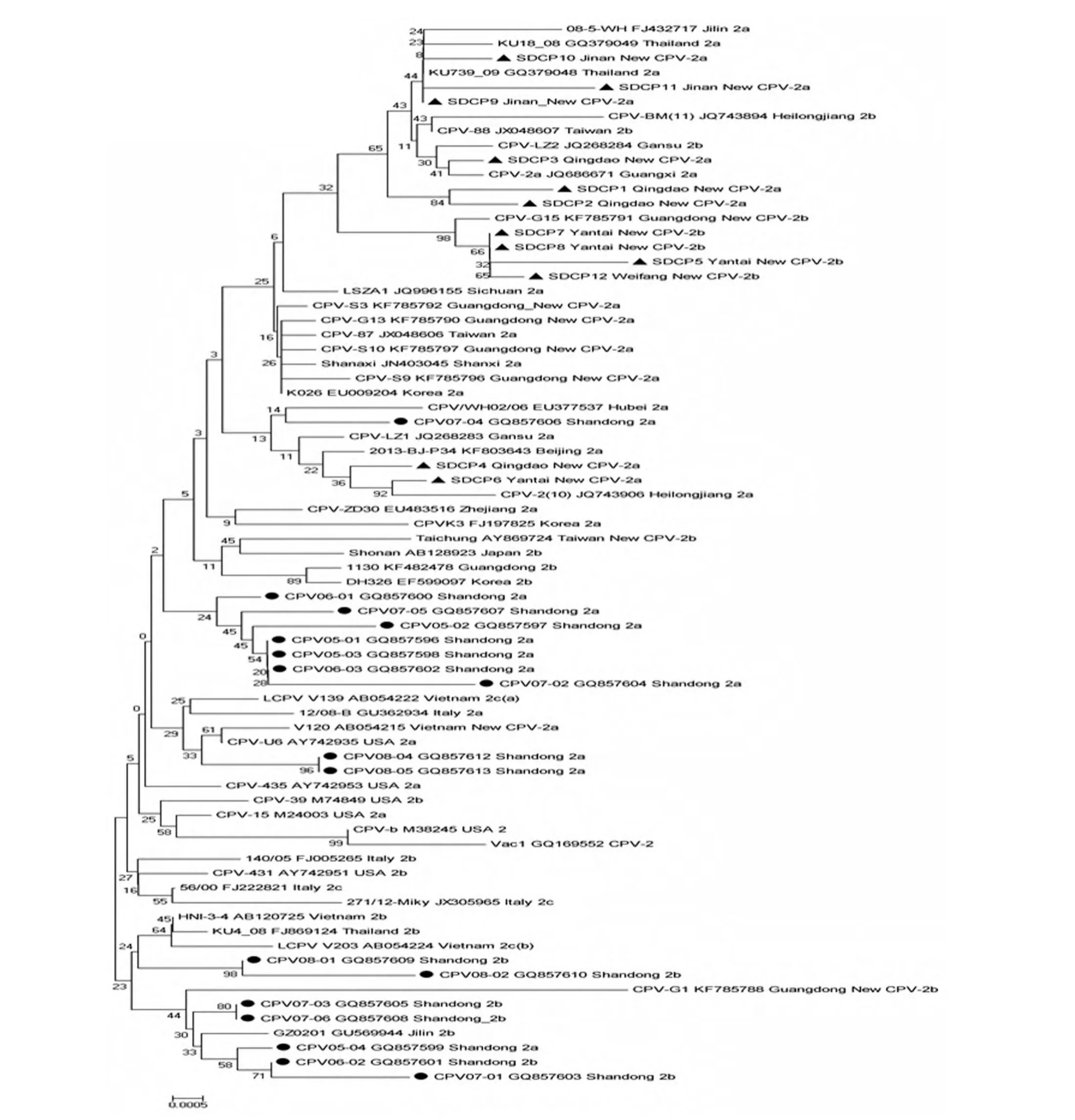
图1 73株C PV VP 2基因核苷酸系统进化树
目前,CPV-2及其抗原变异株在世界范围内广泛流行,New CPV-2a和New CPV-2b已基本替代了CPV-2a和2b在亚洲主要国家(中国、泰国和韩国)[5]流行,而CPV-2c在欧洲(意大利、德国、西班牙、法国)[6]、北美洲(美国)[7]和南美洲(巴西、阿根廷)[8]的流行比例正逐渐扩大。随着CPV感染率和致病性持续升高,广泛的分子流行病学调查极为重要。
CPV-2基因变异主要为集中在VP2基因的非同义置换,通过VP2基因定向漂移产生新抗原变异株[9]。本研究12个分离株VP2氨基酸序列共产生18处非同义突变位点,其中第182位氨基酸T→S替换与2012年四川分离株YAZA4相一致[10];第267位氨基酸F→Y替换并没有展示在犬细小病毒的衣壳表面,此位点氨基酸改变可能不会影响病毒的抗原性[11];第321位N→D和324位Y→I替换在空间位置上恰好临近323位,先前研究表明,93位(Lys→Asn)和323位(Asp→Asn)突变,使CPV获得结合犬细胞表面TfR的能力[12],因此,321和324位氨基酸的突变可能引起CPV宿主范围的改变;第380位和399位氨基酸位于350-400位B细胞抗原表位区内,其亲水性和抗原指数较高,是蛋白潜在跨膜区[13],此处氨基酸的突变可能会影响病毒的抗原性;第440位Y→I替换在多个国家和地区已有报道,该位点处于三重纤突的顶端,是强阳性选择位点[14],此处氨基酸的改变可能促使新型变异株的出现;第136位氨基酸L→M,167位氨基酸N→K,497位氨基酸Q→L,549位氨基酸Q→L,551位氨基酸M→V,这5处突变均未见报道,这种变化是否会引起表型或生物学改变,有待进一步验证和研究。
进化分析显示,本次分离株与2005-2008年山东分离株进化关系较远,与2009年之后的分离到的各亚型毒株(泰国,中国台湾地区,吉林,黑龙江,甘肃,广东,广西等地)亲缘关系较近,我们认为造成两者差异的原因有两个:一是CPV高的突变速率和持续的选择压力迫使新分离毒株产生了不同的遗传变异;二是山东沿海和国内外贸易及人员来往密切,有将国外毒株带到国内的可能。然而,具体的进化机制和演变过程还需要进一步研究。本研究对2013年分离到的12株CPV流行毒株进行了VP2基因克隆与遗传变异分析,8株为New CPV-2a亚型,4株为New CPV-2b亚型,与国内外参考毒株核苷酸和氨基酸同源率在98%以上,且分离株VP2基因及其推导的氨基酸的变异主要以点突变为主,关键氨基酸位点未见明显改变。
[1]Hoelzer K,Parrish C R.The emergence of parvoviruses of carni⁃vores[J].Vet Res,2010,41(6):39-51.
[2]Muz D,Oguzoglu T C,Timurkan M O,et al.Characterization of the partial VP2 gene region of canine parvoviruses in domestic cats from Turkey[J].Virus Genes,2012,44:301-308.
[3]Hong C,Decaro N,Desario C,et a l.Occurrence of canine parvo⁃virus type 2c in the United States[J].J Vet Diagn Invest,2007,19(5):535-539.
[4]王净,李英俊,李刚,等.犬细小病毒北京分离株VP2基因克隆及变异分析[J].中国兽医学报,2011,31(5):649-653.
[5]Phromno S,Sirinarumitr K,Sirinarumitr T.Sequence analysis of VP2 gene of canine parvovirus isolates in Thailand[J].Virus Genes,2010,41(1):23-29.
[6]Decaro N,Buonavoglia C.Canine parvovirus-a review of epidemi⁃ological and diagnostic aspects,with emphasis on type 2c[J].Vet Microbiol,2012,155:1-12.
[7]Kapil S,Cooper E,Lamm C,et al.Canine parvovirus types 2c and 2b circulating in North American dogs in 2006 and 2007[J].J Clin Microbiol,2007,45:4044-4047.
[8]Gallo Calderon M,Wilda M,Boado L,et al.Study of canine par⁃vovirus evolution:comparative analysis of full-length VP2 gene sequences from Argentina and international field strains[J].Virus Genes,2012,44:32-39.
[9] Hoelzer K,Shackelton L A,Parrish C R,et al.Phylogenetic anal⁃ysis reveals the emergence,evolution and dispersal of carnivore parvoviruses[J].J Gen Virol,2008,89(9):2280-2289.
[10]梁璐琪,张恒,魏胜男,等.藏獒源犬细小病毒的分离鉴定[J].中国兽医科学,2012,42(02):144-149.
[11]Agbandje M,McKenna R,Rossmann M G,et al.Structure deter⁃mination of feline panleukopenia virus empty particles[J].Pro⁃teins,1993,16:155-171.
[12]Hueffer K,Parker J S L,Weichert W S,et al.The natural host range shift and subsequent evolution of canine parvovirus resulted from virus-specific binding to the canine transferring receptor[J].J Virol,2003,77:1718-1726.
[13]郭伟,商晓桂,单同领,等.犬细小病毒上海分离株全基因的克隆及进化树分析[J].中国动物传染病学报,2009,17(3):27-32.
[14]Pereira C A,Leal E S,Durigon E L.Selective regimen shift and demographic growth increase associated with the emergence of high-fitness variants ofcanine parvovirus[J].Infect Genet Evol,2007,7(3):399-409.
